Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Botanical Sciences
versión On-line ISSN 2007-4476versión impresa ISSN 2007-4298
Bot. sci vol.91 no.3 México sep. 2013
Genética
Variación genética en poblaciones mexicanas de Swietenia macrophylla King, una especie tropical en expansión geográfica reciente
Genetic variation in Mexican populations of Swietenia macrophylla King, a tropical species in recent geographic expansion
José Enrique Trujillo-Sierra1, Patricia Delgado-Valerio2, Ivón Ramírez-Morillo1, Virginia Rebolledo-Camacho3 y Nidia Pérez-Nasser4
1 Centro de Investigación Científica de Yucatán AC. Mérida, Yucatán, México.
2 Facultad de Agrobiología Presidente Juárez, Universidad Michoacana de San Nicolás de Hidalgo. Uruapan, Michoacán, México.
3 lnstituto de Investigaciones Forestales, Universidad Veracruzana. Xalapa, Veracruz, México.
4 Centro de Investigación en Ecosistemas, Universidad Nacional Autónoma de México. Morelia, Michoacán, México.
2 Autor para la correspondencia: dvalerio@umich.mx.
Enviado: 4 de septiembre de 2012
Aceptado: 12 de diciembre de 2012
Resumen
Swietenia macrophylla King, es una especie tropical que históricamente ha sido explotada por su alta calidad maderable y actualmente está catalogada como amenazada de extinción. En este estudio se usaron cuatro microsatélites nucleares polimórficos para estimar los niveles de variación y estructura genética de seis poblaciones fragmentadas de S. macrophylla, distribuidas en la Península de Yucatán y sur de Veracruz, México. Se encontró un promedio de variación genética moderado (HE = 0.600) y un nivel de endogamia alto y significativo (FIS = 0.309). El tamaño efectivo (NE) fue heterogéneo entre las poblaciones con un promedio de 19.4. La estructura genética fue baja pero significativa (FST = 0.095), y la tasa de migración alta (M > 3.83). El dendrograma y el análisis de inferencia Bayesiana de la estructuración genética y espacial definieron tres grupos altamente diferenciados, pero no siguen un patrón de aislamiento por distancia (P = 0.486). Los resultados aportan evidencias de que S. macrophylla es susceptible a la pérdida de variación genética por el efecto de la fragmentación del hábitat y apoyan la hipótesis de expansión reciente de las poblaciones por el efecto fundador de algunos genotipos. Finalmente, se sugieren dos probables rutas de dispersión de las poblaciones; la primera a través de la zona del Caribe y la segunda por la vertiente del Golfo de México.
Palabras clave: caoba, expansión, fragmentación, México, variación genética.
Abstract
Swietenia macrophylla King is a tropical species that historically had been exploited as a timber resource of high quality and currently listed as threatened of extinction. The present study evaluated genetic variation and structure of six fragmented populations of S. macrophylla from the Yucatan Peninsula and southern Veracruz, Mexico, using four reproducible and polymorphic nuclear microsatellites. A moderate value of genetic variation was found (HE = 0.600), and a high and significant coefficient of inbreeding (FIS = 0.309; P = 0.0001). The effective population size (NE) was heterogeneous among populations with an average of 19.4. The genetic structure was moderate but significant (FST = 0.095) and the migration rate high (M > 3.83). Two and three highly differentiated groups were defined with the dendrogram and the Bayesian inference analyzes of spatial and genetic structure; however, the isolation by distance analysis between pairs of populations was not significant (P = 0.486). The results provide us evidences of S. macrophylla is susceptible to lost genetic variation to habitat fragmented, and support a recent expansion hypothesis based on the founder effect of some genotypes. Finally two probable dispersal routes are suggested; the first through the Caribbean zone and the second in the Gulf of Mexico coastal.
Key words: expansion, genetic variation, habitat fragmentations, mahogany, Mexico.
Los bosques tropicales del mundo han sufrido una severa reducción de sus poblaciones como resultado de su sobreexplotación. La fragmentación y deterioro del hábitat han ocasionado que las poblaciones originales sean cada vez más pequeñas y aisladas, sometidas a problemas crecientes de viabilidad genética y demográfica (Hedrick, 2001; Frankham et al., 2002). El conocimiento del efecto de la fragmentación del hábitat de los bosques tropicales sobre la dinámica evolutiva y ecológica de las especies es crucial para su adecuado manejo y conservación (Nason y Hamrick, 1997).
Teóricamente, se espera que las poblaciones con hábitat fragmentados presenten bajos niveles de variación genética y una diferenciación alta entre las poblaciones remanentes, en comparación con las poblaciones de distribución continua, debido fundamentalmente a la reducción del flujo genético entre ellas, a la pérdida de alelos por el efecto de la deriva genética y al incremento de los niveles de endogamia (Slatkin, 1987; Ellstrand y Elam, 1993; Frankham et al., 2002). Se impide el potencial adaptativo de las especies con una reducción importante en su adecuación (p. ej., menor producción de semilla, frutos, viabilidad y germinación; Hall et al., 1996; Nason y Hamrick, 1997; Lowe et al., 2005). A esta predicción también se le conoce como hipótesis no adaptativa, la cual se ha corroborado en la mayoría de las especies de árboles tropicales estudiados (Carapa guianensis, Hall et al., 1994; Pithecellobium elegans, Hall et al., 1996; Samanea saman, Ward et al., 2005; Swietenia humilis, White et al., 1999; Swietenia macrophylla, Novick et al., 2003; Lowe et al., 2003, 2005; Breed et al., 2012; Symphonia globulifera, Aldrich et al., 1998; Terminalia amazonia, Pither et al., 2003). En contraparte, existen investigaciones que sugieren que no todas las especies se ajustan a la hipótesis no adaptativa cuando la variación genética resulta de adaptaciones de los árboles a nichos específicos, definidos por componentes bióticos y abióticos (sistemas reproductivos, mecanismos de dispersión de polen y semillas, suelo, luz, etc; Bawa, 1990). Este comportamiento adaptativo se ha observado en poblaciones fragmentadas con bajos niveles de endogamia y tamaños efectivos grandes (Astrocaryum mexicanum, Eguiarte et al., 1992; Caryocar brasiliense, Collevatti et al., 2001; Psytrochia faxlucens, Pérez-Nasser et al., 1993; Swietenia macrophylla, Céspedes et al., 2003). Muchas de las especies de árboles que siguen este patrón, son bastante resilientes a los efectos adversos de la fragmentación del hábitat, debido fundamentalmente a su longevidad, elevada variación genética intrapoblacional y a los altos niveles de flujo genético, los cuales contrarrestan los efectos de la deriva genética y de la endogamia (Hamrick, 2004).
Aunque con estos estudios se pueden apreciar dos tendencias en la historia evolutiva de árboles tropicales, aún son insuficientes para poder delinear patrones generales del efecto que la fragmentación del hábitat tiene sobre la diversidad genética. Una especie interesante que puede aportar evidencias sobre estos aspectos es Swietenia macrophylla King (caoba). Esta especie es representativa de los bosques tropicales; sin embargo, su acelerada sobreexplotación ha fragmentado y disminuido drásticamente su hábitat natural, por lo que está catalogada como una especie amenazada de extinción (CITES, 2002). Se distribuye en México, América Central y en la cuenca del Amazonas (Perú, Bolivia y Brasil; Navarro, 1999). Para México, su área de distribución actual se reduce a pequeñas poblacionales fragmentadas en los estados de Puebla, sur de Tamaulipas, Veracruz y en la Península de Yucatán (Quintana Roo, Campeche y Yucatán; Peninngton y Sarukhán, 1968; Navarro 1999).
Swietenia macrophylla junto con S. humilis (Zucc) y S. mahagoni (L. Jac), conforman el género Swietenia, y a pesar de que es un grupo de especies muy reducido, no existen estudios filogenéticos, por lo que el origen y evolución de las tres especies no es clara y muchos procesos biológicos dentro y entre estos taxa están pobremente entendidos. Ecológicamente, las poblaciones de S. macrophylla tienen una gran capacidad para adaptarse a condiciones ambientales distintas, desde condiciones húmedas hasta condiciones de extrema sequía, por lo que la especie sigue diferentes patrones de variación ecológica y geográfica (Whitmore, 1983; Gillies et al., 1999). Por ejemplo, existe correlación entre la distribución geográfica de las poblaciones y su capacidad regenerativa, de supervivencia y crecimiento de plántulas (Newton et al., 1999; Ward y Lugo, 2003; Navarro y Hernández, 2004). otro aspecto importante de S. macrophylla tiene que ver con la forma de dispersión de sus semillas, las cuales son muy pesadas, y a pesar de ser aladas tienden a caer muy cerca del árbol madre (32 a 36 m), con una distancia máxima de 80 m (Gullison et al., 1996). La caoba es monoica de entrecruzamiento obligado y requiere para su polinización de insectos como abejas, palomillas y trips (Gillies et al., 1999). Sin embargo, la reducción y fragmentación de las poblaciones podrían estar alterando esta asociación mutualista (con las especies polinizadoras), y con ello tener un efecto importante en la pérdida de la variación genética y aumento de los niveles de endogamia.
Actualmente existen varios estudios que han estimado los niveles de variación genética intra e interpoblacional de Swieteneia macrophylla, basados en diferentes marcadores moleculares como RAPDs (Gillies et al., 1999), microsatélites nucleares, SSRn (Céspedes et al., 2003; Novick et al., 2003; Lowe et al., 2003; Lemes et al., 2002, 2003; Degen et al., 2012) y microsatélites de cloroplasto, SSRcp (Lemes et al., 2010). Los estudios realizados con SSRn y RAPDs indican que las poblaciones de Mesoamérica contienen niveles de variación y estructura genética significativamente menores a los de las poblaciones del Amazonas (SSRn, HE = 0.657; HE = 0.781 respectivamente). En contraste, los resultados obtenidos por Lemes et al. (2010) con el uso de SSRcp muestran mayores niveles de variación genética para las poblaciones de Mesoamérica que en la zona del Amazonas (H = 0.639 y H = 0.176, respectivamente). No obstante, a estas diferencias, los estudios apuntan a que la baja variación genética de las poblaciones de caoba de Mesoamérica podrían deberse a eventos de expansión geográfica muy recientes (efecto fundador), donde las poblaciones más aisladas y fragmentadas están conformadas por tamaños efectivos pequeños, así como a cambios drásticos de la vegetación que se presentaron en esta región durante fases glaciales del Pleistoceno (Novick et al., 2003; Lemes et al., 2010). Cabe mencionar que estos estudios sólo han incluido cuatro poblaciones de México, y algunos de ellos con tamaños de muestra pequeños (1 a 4 individuos/población) por lo que los resultados no son muy contundentes (Lemes et al., 2010; Degen et al., 2012).
En este estudio se analizaron los niveles de variación y estructura genética de seis poblaciones de Swietenia macrophylla asociadas a hábitats fragmentados en la Península de Yucatán y sur de Veracruz, con el uso de marcadores moleculares microsatélites nucleares (SSRn). Dado que S. macrophylla es una especie de entrecruzamiento obligado, pero que está representada en México por poblaciones fragmentadas, se espera que contenga valores bajos de diversidad genética, una estructura genética marcada, tamaños efectivos pequeños y valores altos de endogamia, que sigue una historia evolutiva acorde con la hipótesis no adaptativa (poblaciones genéticamente más vulnerables). Los objetivos específicos fueron: (1) estimar los niveles de variación genética, el índice de endogamia y el tamaño efectivo de las poblaciones mexicanas de S. macrophylla y (2) determinar la estructura geográfica de la variación genética y el flujo genético de las poblaciones. Adicionalmente, con los resultados se analizó la hipótesis de expansión reciente de la caoba en América Central.
Materiales y métodos
Colecta de material vegetativo. Se colectó un total de 81 muestras de hojas y raíces de 11 a 16 árboles por población en seis localidades reportadas por Pennington y Sarukán (1968) y Navarro (1999). Tres poblaciones distribuidas en el estado de Quintana Roo, dos en el estado de Campeche y una en el sur de Veracruz (Cuadro 1, Figura 1). Las muestras de hojas se colectaron en las poblaciones representadas por árboles con ramas bajas y las raíces en las poblaciones en donde los árboles eran mayores de 40 m de altura y sin ramificaciones accesibles. El material se colocó en bolsas de plástico y se almacenó en un ultracongelador a -80 °C.

Obtención de datos moleculares. El ADN total fue obtenido con el método Mini-prep CTAB 2X (Vázquez-Lobo, 1996). Para seleccionar loci polimórficos se realizó un análisis preliminar con 24 individuos usando nueve microsatélites, de los que sólo cuatro fueron reproducibles y polimórficos, los cuales fueron seleccionados y usados en este estudio; sm01 (Lemes et al., 2002), mac83, mac49 and mac52 (White y Powell, 1997). Las reacciones de amplificación y condiciones de PCR se hicieron de acuerdo con el método propuesto por Céspedes et al. (2003). Para la separación de los fragmentos se usaron geles de poliacrilamida al 6% (7 M Urea) los cuales se corrieron a 2,500 V durante 2:30 h. La visualización de los fragmentos se hizo por medio de la técnica de tinción con nitrato de plata (Echt et al., 1996), y el tamaño de los fragmentos se determinó utilizando como referencia un marcador de diez pares de bases (pb).
Análisis de datos. Los niveles de variación genética intrapoblacional fueron estimados de acuerdo con Nei (1987), mediante los siguientes parámetros; número de alelos por locus (A), promedio de alelos efectivos (Ae), heterocigosis observada (HO) y esperada (HE). Estas estimaciones se realizaron con el programa Arlequín 3.1 (Excoffier et al., 2005). El tamaño efectivo (NE) de las poblaciones fue estimado con el método de desequilibrio de ligamiento (LD), el cual se desarrolló bajo el supuesto de que en una población aislada y panmíctica, la asociación entre los alelos de diferentes loci podría no ser azarosa, donde para loci neutrales y no ligados el LD podría deberse exclusivamente a la deriva genética (Hill, 1981; Waples, 1991). Para la estimación del NE se usó el programa NeEstimator ver 1.3 (Ovenden et al., 2007).
La estructura genética entre poblaciones, entre grupos de poblaciones y entre poblaciones dentro de grupos, se estimó por medio de un análisis jerárquico de varianza molecular (AMOVA) de acuerdo al modelo de mutación de alelos infinitos (IAM; Weir, 1996) y de mutaciones de un paso (SMM; Kimura y Ohta, 1978). Es interesante analizar ambos modelos, ya que los datos de microsatélites podrían ajustarse más a alguno de ellos. La FST es un estimador más preciso para poblaciones pequeñas y de divergencia reciente, mientras que el estimador RST es más exacto para poblaciones grandes y con tiempos de divergencia mayores o más antiguos (Balloux y Lugon-Moulin, 2002). La significancia estadística fue obtenida con 1,000 permutaciones no paramétricas y calculadas con el programa Arlequín 3.1 (Excoffier et al., 2005). Para determinar la estructuración geográfica y genética de las poblaciones se usó una aproximación probabilística con el algoritmo implementado en el programa BAPS de inferencia Bayesiana (Corander et al., 2008). Las estimaciones se hicieron asumiendo grupos (K) de 1 a 10, con diez réplicas para cada valor de K. Los análisis se hicieron con una partición inicial de 100 hasta obtener el valor de probabilidad marginal más grande (10,000 iteraciones).
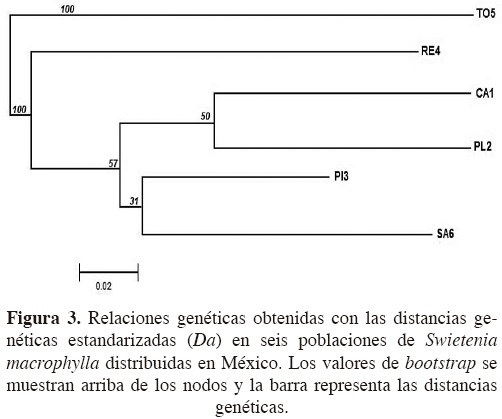
Para analizar las relaciones genéticas de las poblaciones se construyó un dendrograma de Neighborjoining, con base en las distancias genéticas estandarizadas (Da) de Nei et al. (1983). La robustez de la topología fue evaluada con el algoritmo de remuestreo bootstrap con 1000 réplicas (Takezaki y Nei, 1996) con el uso del programa Poptree2 (Takezaki et al., 2010). El flujo genético se estimó con el parámetro M, que es la tasa de migración (M = Nm) entre pares de poblaciones (Slatkin, 1995). Finalmente, se realizó un análisis de aislamiento por distancia entre pares de poblaciones y su distribución geográfica mediante una prueba de Mantel con 1,000 permutaciones. Para las estimaciones se usó el programa Arlequín 3.1 (Excoffier et al., 2005).
Resultados
Variación genética. Se observaron de 16 a 21 alelos por población con un total de 29 alelos (Cuadros 2, 3). El locus sm01 fue el más polimórfico con un total de diez alelos, y el locus mac52 fue el menos polimórfico con cinco alelos. La mayoría de los alelos se presentaron en más de una población con frecuencias superiores a 0.033 (Cuadro 2). El promedio de alelos por locus (Na) fue de 4.4, con un intervalo de 4.0 (PI3) a 5.2 (TO5), el cual fue mayor al estimado de alelos efectivos (Ae), con un promedio de 2.57 y un intervalo de 2.07 (PI3) a 3.20 (TO5).
La heterocigosis esperada (HE) varió entre 0.520 (PI3) a 0.688 (TO5) con un promedio de 0.600. La heterocigosis observada (HO) varió de 0.209 (PL2) a 0.603 (RE4) con un promedio de 0.420 (Cuadro 3). El promedio de heterocigosis esperada (HE) fue generalmente más alto que el promedio de heterocigosis observada (Ho), diferencia que fue estadísticamente significativa para tres loci (mac52, FIS = 0.621, P = 0.0001; mac83, FIS = 0.307, P = 0.003 y smo1 FIS = 0.328, P = 0.0001). Por tanto, el promedio del índice de fijación fue significativamente distinto al esperado en el equilibrio entre la tasa de mutación y la deriva genética (FIS = 0.309; P = 0.0001; Cuadro 3). Las poblaciones CA1, PL2 y TO5 presentaron los valores más altos y significativos de endogamia (P = 0.0001). El tamaño efectivo (NE) fue heterogéneo entre las poblaciones y varió de 2.2 en la población de PI3 a 82.1 en TO5, con un promedio de 20.1 individuos reproductivos.
Diferenciación genética. El análisis jerárquico de la varian-za molecular (AMOVA) obtenido con el modelo de mutación IAM presentó un valor más alto y significativo de varianza entre las poblaciones (Φst = 0.095; P = 0.0001), que el modelo de mutaciones de un paso SMM (Φst = 0.051; P = 0.068). De igual manera, la varianza entre individuos dentro de poblaciones y dentro de individuos fue mayor y significativa con el modelo IAM (Φct = 0.303, P = 0.0001; ΦSC = 0.610, P = 0.0001), que con el modelo SMM (Φct = 0.051, P = 0.265; ΦSC = 0.896, P = 0.105). Los resultados obtenidos con el programa BAPS, para determinar si existía una estructura geográfica (espacial) y genética de las poblaciones, generó K = 3 grupos con los valores de probabilidad más altos (lnP (D) = - 934.70; P = 0.999). El primero y segundo grupo incluyen a las poblaciones distribuidas en el estado de Campeche (RE4 y To5), y en el tercer grupo al resto de las poblaciones estudiadas (Figura 2).

Relaciones genéticas y aislamiento por distancia. El dendrograms obtenido con base en el algoritmo de Neighborjoining formó tres grupos; el primero y más basal incluye a la población de To5, seguido del segundo grupo que incluye a la población de RE4, ambas distribuidas en el estado de Campeche (Golfo de México). El tercer grupo se divide en dos subgrupos; el primero incluye a la población de Pi3 distribuida en el sur de Quintana Roo y SA6 distribuida en el sur de Veracruz, y el segundo subgrupo a las poblaciones PL2 y CA1 distribuidas en la parte norte y sur de Quintana Roo (Figura 3). La tasa de migración (M) fue alta y variable entre pares de poblaciones, presentándose el valor más bajo entre las poblaciones de PI3 y RE4 (M = 3.83), hasta valores infinitos entre las poblaciones de CA1 y PI3. No se presentó un patrón de aislamiento por distancia (r = 0.1950, P = 0.486), lo cual sugiere que la variación genética de las poblaciones no se asocia de manera importante con su distribución geográfica.
Discusión
Variación genética. En este estudio se encontró que el nivel promedio de variación genética de las poblaciones de Swie-tenia macrophylla distribuidas en la Península de Yucatán y sur de Veracruz, se encuentra dentro del intervalo obtenido con SSRn en especies de árboles tropicales (0.230-0.889, Lowe et al., 2005). Sin embargo, es inferior al obtenido en poblaciones de caoba distribuidas en la región del Amazonas (HE = 0.840, Lemes et al., 2002; HE = 0.781, Lemes et al., 2003) y en Mesoamérica (HE = 0.657, Novick et al., 2003), pero superior al valor obtenido para diferentes poblaciones de Costa Rica (HE = 0.470-0.540, Céspedes et al., 2003; HE = 0.447, Lowe et al., 2003). Estos resultados sugieren que las poblaciones más alejadas del área de mayor distribución de la especie (Amazonas) tienden a disminuir sus niveles de variación genética, como es el caso de las poblaciones distribuidas en México (región más norteña de distribución de la especie) y en Costa Rica.
Para el caso particular de las poblaciones que presentaron los mayores niveles de variación genética (To5, HE = 0.688; RE4, HE = 0.665), se puede considerar que la variación se ha mantenido por que las poblaciones todavía se encuentran dentro de fragmentos de selvas que no han sido intensamente perturbados (T05 con 30 ind./ha, ~ 17 ha; RE4 con 10 ind./ha, ~ 15 ha), mientras que las poblaciones menos diversas genéticamente (PI3, HE = 0.520; PL2, HE = 0.568) podrían estar asociadas al manejo forestal intensivo. La población de Pi3 se ubica entre las regiones Silvituc-Calakmul y Río Hondo, Quintana Roo; las cuales son regiones establecidas para la conservación de la biodiversidad (SEMARNAT, 2006). Sin embargo, en esta población se observó un escaso número de individuos (6 ind./ha, ~ 8 ha). Esto se debe probablemente a que limita con la zona de mayor actividad forestal (Felipe Carrillo Puerto y Pioneros del Río), donde se realiza el 78% del manejo forestal del estado de Quintana Roo (SEMARNAT, 2006). La población de PL2 por su parte, se encuentra en un área perturbada y se distribuye en la zona más norteña y aislada de la especie en el estado de Quintana Roo, y está también conformada por escasos individuos (8 ind./ha, ~ 18 ha). Estas características del hábitat de las poblaciones PI3 y PL2 se asocian a la pérdida de variación genética observada, la cual se ve afectada por procesos endogamia y de deriva genética.
El nivel promedio de endogamia obtenido fue alto (FIS = 0.309) y superior al reportado por Novick et al. (2003), para las poblaciones estudiadas en Mesoamérica (FIS = 0.149). Las poblaciones de mayor FIS fueron CA1 y PL2 distribuidas en Quintana Roo, zona de mayor deforestación, y TO5 distribuida en el sur de Campeche. Las dos primeras poblaciones contienen valores de A, Na, Ae y HE cercanos al promedio obtenido y la población TO5 valores mayores al promedio. Sin embargo, para las tres poblaciones la HO fue baja por lo que se desvía del valor esperado en el equilibrio Hardy-Weinberg y genera un alto índice de endogamia. Las poblaciones PI3 y SA6 presentan los menores valores de HE, el menor número de A, Na y Ae, lo que refleja el efecto de la deriva génica sobre la variabilidad genética (perdida de variantes alélicas). Este comportamiento indica que Swietenia macrophylla presenta una historia evolutiva acorde con la predicción no adaptativa, la cual ha sido corroborada en muchas especies de árboles tropicales. Por ejemplo, White et al. (1999) en un estudio con poblaciones de S. humilis encuentran que cuando el tamaño de los fragmentos decrece, disminuye la diversidad genética (HE = 0.548) e incrementa el índice de endogamia (FIS = 0.223). Hall et al. (1994) mencionan que la disminución de la diversidad genética también se puede deber a una interacción de factores. En su estudio con isoenzimas en poblaciones de Carapa guianensis de Centroamérica, encuentran bajos niveles de HE (0.120), con una relación significativa entre la disrupción de su sistema reproductivo y la subestructuración de sus poblaciones. Swietenia macrophylla es monoica y de entrecruzamiento obligado, y aunque sus semillas y polen son dispersados fundamentalmente por el viento, se ha reportado la existencia de dispersores de polen como abejas, palomillas y trips (Gillies et al., 1999; Lowe et al., 2003), no obstante, se desconoce todavía su efecto sobre la polinización (Pennington 2002, Cornelius et al., 2004). Los pocos estudios sobre dispersión de semillas en S. macrophylla han documentado que la mayoría caen alrededor de 36 m del árbol madre y en áreas abiertas se esparcen entre 60 a 80 m (Gullison et al., 1996; Rodríguez, 1994). Por lo que el mayor efecto de la pérdida de diversidad genética podría atribuirse en primer término a la fragmentación del hábitat. Sin embargo, se requiere de estudios más finos con los que se pueda evaluar el impacto potencial de la fragmentación del hábitat sobre la diversidad genética de poblaciones particulares y con distintas generaciones (p. ej., comparación de sitios pre y post fragmentados, así como análisis de progenie).
Estructura poblacional. El análisis de varianza molecular (AMOVA), utilizando ambos modelos de mutación, indica que la mayor diversidad genética se encuentra dentro de las poblaciones de Swietenia macrophylla y que no presentan una estructura genética marcada (FST = 0.0868; RST = 0.051). Muchas especies de árboles tropicales que se han estudiado con isoenzimas o SSRn muestran bajos valores de diferenciación genética (Hamrick et al., 1992; Eguiarte et al., 1992), pero existe un intervalo muy amplio que va desde 0.005 en Symphonia globulifera (Aldrich et al., 1998) hasta 0.290 en Caryocar brasiliense (Collevatti et al., 2001). Lowe et al. (2005) recopilaron datos de isoenzimas y SSRn para nueve especies de árboles tropicales y reportan un valor promedio de FST de 0.106, el cual es relativamente bajo y similar al que se obtuvo para S. macrophylla en este estudio. Los estimados de FST en diversas poblaciones de S. macrophylla con el uso de SSRn también han sido similares; por ejemplo, Cespedes et al. (2003) reportan para poblaciones fragmentadas de Costa Rica un valor de 0.063, Lemes et al. (2003) para la región del Amazonas reportan un valor de 0.100 y Novick et al. (2003) para poblaciones en Mesoamérica una FST de 0.109. Los dos últimos resultados difieren de los obtenidos por Lemes et al. (2010) para las mismas poblaciones, pero con el uso de SSRcp (FST = 0.360 en poblaciones de Mesoamérica y FST = 0.900 en poblaciones del Amazonas), lo que atribuyen al comportamiento diferente del genoma del cloroplasto y nuclear. Los procesos de deriva genética son más acentuados en el cloroplasto debido a dos aspectos; el primero es que el tamaño efectivo del cloroplasto es más pequeño que en el genoma nuclear y su tasa de evolución es mayor (Schaal et al., 1991). El segundo aspecto es el número de loci que se analizan en cada región del genoma, en el cloroplasto se generan resultados de un simple locus (haplotipo), mientras que en el genoma nuclear se analizan varios loci de manera independiente (Lemes et al., 2010). No obstante, en términos generales, los valores de FST son relativamente menores en Mesoamérica que en la región del Amazonas con ambos tipos de marcadores (incluyendo los resultados de FST obtenidos en este estudio). En estos términos se puede considerar que la diferenciación genética no se ve afectada por la fragmentación del hábitat, debido probablemente ha que ha pasado un tiempo muy corto desde los eventos de fragmentación para que reflejen su efecto.
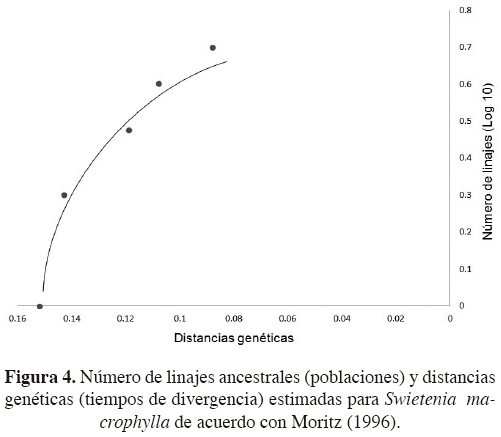
De acuerdo con Lemes et al. (2010), se consideran tres hipótesis que explican este patrón: eventos de dispersión a gran distancia, los efectos de las glaciaciones y las actividades antropogénicas históricas y actuales. Para la primera hipótesis acerca de eventos de dispersión a gran distancia, la presencia de los mismos haplotipos en las poblaciones de América Central analizadas por Lemes et al. (2010) y de la mayoría de los alelos encontrados en este estudio, implican un papel de la dispersión a gran distancia donde las montañas no han sido barreras efectivas, y posiblemente los huracanes que son muy frecuentes en esta zona, pudieron actuar como dispersores, y dejar una distribución relativamente uniforme de alelos. La asociación entre la distribución geográfica y la genética de las poblaciones estudiadas también concuerdan con esta hipótesis, ya que tanto el análisis de BAPS como el dendrograma obtenido, separan a las poblaciones más aisladas o ubicadas en la periferia del área de distribución de la especie (RE4 y TO5) de las poblaciones distribuidas más al sur (excepto SA6), en una zona más cercana con América Central, en donde se encuentra la mayoría de las poblaciones representativas de la especie. Por tanto, se podría sugerir que la dispersión de Swietenia macrophylla se inició de la zona sur hacia el noreste de México. La segunda hipótesis se basa en el efecto que las glaciaciones del Pleistoceno tuvieron en el clima regional, dejando áreas relativamente pequeñas para el establecimiento de la especie (Lemes et al., 2010). La reconstrucción de la vegetación en esta zona, entre los 20,000 y 10,500 B.P., muestra áreas restringidas con vegetación forestal de zonas húmedas y áreas muy amplias de vegetación de bosque espinoso, matorral y sabana (Piperno y Pearsall, 1998). Estos factores pudieron causar la extinción local de poblaciones y una drástica reducción de su tamaño efectivo, por lo que los signos de diversificación y estructuración pudieron ser modificados en ese periodo. Lemes et al. (2010) consideran que los niveles de variación y diferenciación genética que obtuvieron con SSRcp en S. macrophylla, y los obtenidos con SSRn por Novick et al. (2003), reflejan la expansión geográfica reciente de algunos haplotipos ancestrales a partir de los cuales se originaron haplotipos (alelos) derivados con un tamaño efectivo menor en las poblaciones más aisladas o de origen más reciente. En este estudio, la obtención del mejor ajuste del estimado de la estructura genética (FST) con el modelo IAM (poblaciones pequeñas y de divergencia reciente; Balloux y Lugon-Moulin, 2002), el alto flujo genético obtenido y los bajos niveles de heterocigosis, corroboran la hipótesis de la existencia de eventos de expansión geográfica recientes de las poblaciones de S. macrophylla distribuidas en México. Para el caso del tamaño efectivo (NE), no existen estudios con los cuales se pueda hacer su comparación. Sin embargo, el NE fue diferente en la mayoría de las poblaciones estudiadas, donde algunas de ellas contienen valores inferiores al promedio obtenido, como PI3 (NE = 2), RE (NE = 6.0) y SA6 (NE = 5.9), así como valores dos veces más altos (TO5, NE = 82.1), lo cual indica que en efecto, las poblaciones más aisladas geográficamente presentan NE más pequeños. Para apoyar más esta hipótesis se hizo un análisis sencillo en torno a la historia demográfica de las poblaciones (Moritz, 1996). Este análisis se basa en el supuesto de que las poblaciones que se encuentran en expansión demográfica muestran una relación filogenética (análisis de Neighborjoining) en forma de estrella, la cual resulta de una relación parabólica entre las distancias genéticas (como un estimado del tiempo de divergencia) y el logaritmo del número de poblaciones. Por el contrario, para poblaciones estables demográficamente se muestra una fuerte estructura filogenética y una relación exponencial. Los resultados son interesantes y sugieren que las poblaciones de S. macrophylla han tendido a incrementar su tamaño, lo cual se ve más acentuado en un tiempo reciente (Figura 4). La tercera hipótesis se sustenta en el efecto que las actividades antropogénicas han tenido en esta región desde hace más de 2,800 B.P., en donde la civilización Maya influyó severamente en la fragmentación y pérdida de áreas boscosas (Hodell et al., 2000). Estas actividades dejaron escasos árboles que, con el colapso de la sociedad Maya (entre 800 y 900 A.D.), pudieron actuar como dispersores, con la consecuente regeneración de las zonas forestales (Grogan et al., 2003; Nevle y Bird, 2008). Esto pudo haber acelerado la expansión de los linajes remanentes de S. macrophylla en México, en donde se observa que la mayoría de las poblaciones estudiadas no se encuentran en equilibrio Hardy-Weinberg, aspecto que podría indicar un origen reciente de las poblaciones por el efecto fundador de pocos genotipos que podrían estar generando los altos niveles de endogamia que actualmente observamos. En particular, la población de TO5 presentó los valores más altos de heterocigosis, pero también se desvía del equilibrio Hardy-Weinberg, presenta los valores más altos de endogamia y es la población más diferente genéticamente, basal en el dendrograma y con el NE más grande, por lo que podría considerarse como una población remanente, a partir de la cual se ha dado la dispersión o expansión de otras poblaciones cercanas. De acuerdo con lo anterior, se propone un escenario evolutivo en donde la dinámica de la dispersión se ha presentado en forma de parches a través de dos posibles zonas, vertiente del Golfo de México (poblaciones de Campeche) y zona del Caribe (poblaciones de Quintana Roo). Esto último también se sustenta con el dendrograma, en el cual dos de las poblaciones ubicadas en la zona del Golfo de México (TO5 y RE4) son más cercanas genéticamente que con el resto de las poblaciones distribuidas en el Caribe. En el caso de la población del sur de Veracruz (Golfo de México, SAY6), que forma parte del segundo grupo del dendrograma, podría considerarse como una población a partir de la cual se han originado otras poblaciones registradas para la zona norte-centro de México (sur de Tamaulipas, norte de Veracruz y Puebla; Peninngton y Sarukhán, 1968).
En conclusión, los resultados demuestran que las poblaciones de Swietenia macrophylla distribuidas en México contienen bajos niveles de variación y estructura genética. Estos resultados, junto con los valores altos de endogamia y los tamaños efectivos relativamente pequeños que se obtuvieron, son compatibles con la hipótesis no adaptativa. Se argumenta que los principales procesos que han moldeado la variación y distribución genética de las poblaciones están relacionados con cambios climáticos que se presentaron durante las glaciaciones del Pleistoceno, como con las actividades históricas y actuales de deforestación. Estas actividades han disminuido el tamaño de las poblaciones, lo que genera claros de vegetación y fragmentos de poblaciones aisladas que actualmente podrían encontrarse en proceso de expansión. Es recomendable realizar estudios complementarios que incluyan otras poblaciones distribuidas en el centro y norte de México y usar marcadores haploides (cloroplasto o mitocondria), para corroborar el patrón de dispersión sugerido en este trabajo.
Agradecimientos
Los autores agradecen a F. Chi, A. Navarro y M. Bautista por su ayuda en la colecta del material biológico y a G. Castillo por su asistencia en el trabajo de laboratorio. Al laboratorio de Biología Molecular del Centro de investigación Científica de Yucatán AC., y a dos revisores anónimos por sus invaluables comentarios. Este trabajo fue financiado por una beca de Maestría CONACYT-211087 de E. Trujillo y por el proyecto SEP-CONACYT-44373 de P. Delgado.
Literatura citada
Aldrich P.R., Hamrick J.L., Chavarriaga P y Kochert G .1998. Microsatellite analysis of demographic genetic structure in fragmented populations of the tropical tree Symphonia globulifera. Molecular Ecology 7:933-944 [ Links ]
Balloux F. y Lugon-Moulin N. 2002. The estimation of population differentiation with microsatellite markers. Molecular Ecology 11:155-165. [ Links ]
Bawa K.S. 1990. Plant pollinator interactions in tropical rain forests. Annals Review of Ecology Systems 21:399-422. [ Links ]
Breed M.F., Gardner M.G., Ottewell K.M., Navarro C.M. y Lowe A.J. 2012. Shifts in reproductive assurance strategies and inbreeding costs associated with habitat fragmentation in Central American mahogany. Ecology Letters 15:444-452. [ Links ]
Céspedes M., Gutierrez M.V., Holbrook N.M. y Rocha O.J. 2003. Restoration of genetic diversity in the dry forest tree Swietenia macrophylla (Meliaceae) after pasture abandonment in Costa Rica. Molecular Ecology 12:3201-3212. [ Links ]
CITES. 2002. Convention on International Trade in Endangered Species of Wild Fauna and Flora, Appendix II. <www.cites.org/resources/species.html> (consultado julio 2012). [ Links ]
Collevatti R.G., Grattapaglia D. y Hay J.D. 2001. High resolution microsatellite based analysis of the mating system allows the detection of significant biparental inbreeding in Caryocar brasiliense, an endangered tropical tree species. Heredity 86:60-67. [ Links ]
Corander J., Marttinen. P., Sirén J. y Tang J. 2008. Enhanced Bayesian modelling in BAPS software for learning genetic structures of populations. BMC Bioinformatics 9:539. [ Links ]
Cornelius J.P., Wightman K.E., Grogan J.E. y Ward S.E. 2004. Swietenia (American mahogany). En: Burley J., Evans J. y Youngquist J.A. Eds. Encyclopaedia of Forest Science, pp. 1720-1726, Academic Press, San Diego. [ Links ]
Degen B., Ward S.E. Lemes M.R., Navarro C., Cavers S. y Seb-benn A.M. 2012. Verifying the geographic origin of mahogany (Swietenia macrophylla King) with DNA-finerprints. Forensic Science International: Genetics 7:55-62. [ Links ]
Echt C.S., May-Marquardt P., Hseih M. y Zahorchak R. 1996. Characterization of microsatellite markers in eastern white pine. Genome 39:1102-1108. [ Links ]
Eguiarte L.E., Perez-Nasser N. y Piñero D. 1992. Genetic structure, outcrossing rate and heterosis in Astrocaryum mexicanum (tropical palm): implications for evolution and conservation. Heredity 69:217-228. [ Links ]
Ellstrand N.C. y Elam D.R. 1993. Population genetic consequences of small population size: implications for plant conservation. Annual Review of Ecology and Systematics 24:217-242. [ Links ]
Excoffier L., Laval G. y Schneider S. 2005. Arlequin (version 3.0): An integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online 1:47-50. [ Links ]
Frankham R., Ballou J.D. y Bricoe D.A. 2002. Introduction to Conservation Genetics. Cambridge University Press, Cambridge. [ Links ]
Gillies A.C.M., Navarro C., Lowe A.J., Newton A.C., Hernández M., Wilson J. y Cornelius J.P. 1999. Genetic diversity in mesoamerican populations of mahogany (Swietenia macrophy-lla), assessed using RAPDs. Heredity 83:722-732. [ Links ]
Grogan J., Ashton, M.S. y Galvão J. 2003. Big-leaf mahogany (Swietenia macrophylla) seedling survival and growth across a topographic gradient in southeast Pará, Brazil. Forest Ecology and Management 186:311-326. [ Links ]
Gullison R.E., Panfil S.N., Strouse J.J. y Hubbell S.P. 1996. Ecology and management of mahogany (Swietenia macrophylla King) in the Chimanes Forest, Beni, Bolivia. Botanical Journal of the Linnean Society 122:9-34. [ Links ]
Hall P., Orrell L.C. y Bawa K.S. 1994. Genetic diversity and mating system in a tropical tree, Carapa guianensis (Meliaceae). American Journal of Botany 81:1104-1111. [ Links ]
Hall P, Walker S. y Bawa K. 1996. Effect of forest fragmentation on genetic diversity and mating system in a tropical tree, Pithecellobium elegans. Conservation Biology 10:757-768. [ Links ]
Hamrick J.L., Godt M.J.W. y Sherman-Broyles S.L. 1992. Factors influencing levels of genetic diversity in woody plants species. New Forests 6: 95-124. [ Links ]
Hamrick J.L. 2004. Response of forest trees to global environmental changes. Forest Ecology and Management 197:323-335 [ Links ]
Hedrick P.W. 2001. Conservation genetics: where are we now? Trends in Ecology and Evolution 16: 629-636. [ Links ]
Hill W.G. 1981. Estimation of effective population size from data on linkage disequilibrium. Genetic Research 38:209-216. [ Links ]
Hodell D.A., Brenner M. y Curtis J.H. 2000. Climate change in the northern American tropics and subtropics since the last ice age: implications for environment an culture. En: Lentz D.L. Ed. Imperfect Balance: Landscape Transformations in the Precolumbian Americas, pp. 13-38, Columbia university Press, Nueva York. [ Links ]
Kimura M. y ohta T. 1978. Stepwise mutation model and distribution of allelic frequencies in finite populations. Proceedings of the National Academy of Sciences of the USA 75:2868-2872. [ Links ]
Lemes M.R., Brondani R.P.V. y Grattapaglia D. 2002. Multiplexed systems of microsatellite markers for genetic analysis of mahogany, Swietenia macrophylla King (Meliaceae), a threatened Neotropical timber species. Journal of Heredity 93:287-291. [ Links ]
Lemes M.R., Gribel R., Proctor J. y Grattapaglia D. 2003. Population genetic structure of mahogany (Swietenia macrophylla King, Meliaceae) across the Brazilian Amazon, based on variation at microsatellite loci: implications for conservation. Molecular Ecology 12:2875-2883. [ Links ]
Lemes M.R., Dick C.W., Navarro C., Lowe A.J., Cavers S. y Gribel R. 2010. Chloroplast DNA microsatellites reveal contrasting phylogeographic structure in Mahony (Swietenia macrophylla King, Meliaceae) from Amazonia and Central America. Tropical Plant Biology 3:40-49. [ Links ]
Lowe A.J., Jourde B., Breyne P., Colpaert N., Navarro C., Wilson J. y Cavers S. 2003. Fine-scale genetic structure and gene flow within Costa Rican populations of mahogany (Swietenia macrophylla). Heredity 90:268-275. [ Links ]
Lowe A.J., Boshier D., Ward M., Bacles C.F.E. y Navarro C. 2005. Genetic resource impacts of habitat loss and degradation; reconciling empirical evidence and predicted theory for neotropical trees. Heredity 95:255-273. [ Links ]
Moritz C. 1996. uses of molecular phylogenies for conservation. En: Harvey P.H., Brown A.J.L., Smith J.M. y Nee S.Eds. New Uses of New Phylogenies, 203-216, oxford university Press, oxford. [ Links ]
Nason J.D. y Hamrick J.L. 1997. Reproductive and genetic consequences of forest fragmentation: two case studies of neotropical canopy trees. Journal of Heredity 88:264-276. [ Links ]
Navarro C. 1999. Diagnóstico de la caoba (Swietenia macrophylla King) en Mesoamérica. Silvicultura-Genética. Centro Científico Tropical. PROARCA/CAPAS. [ Links ]
Navarro C. y Hernández G. 2004. Progeny analysis and population differentiation of Mesoamerican mahogany (Swietenia macrophylla). Agronomía Costarricense 28:37-51. [ Links ]
Nei M. 1987. Molecular Evolutionary Genetics. Columbia university Press, Nueva York. [ Links ]
Nei M., Tajima F. y Tateno Y. 1983. Accuraty of estimated phylogenetic trees from molecular data. II. Gene frequency data. Journal of Molecular Evolution 19:153-170. [ Links ]
Nevle R.J. y Bird D.K. 2008. Effects of syn-pandemic fire reduction and reforestation in the tropical Americas on atmospheric CO2 during European conquest. Palaeogeography, Palaeoclimatology, Palaeoecology 264:25-38. [ Links ]
Newton A.C., Watt A.D., Lopez F., Cornelius J.P, Mesén J.F. y Corea E.A. 1999. Genetic variation in host susceptibility to attack by the mahogany shoot borer, Hypsipyla grandella (Zeller). Agricultural and Forest Entomology 1 :11-18. [ Links ]
Novick R.R., Dick C.W., Lemes M.R., Navarro C., Caccone A. y Bermingham E. 2003. Genetic structure of Mesoamerican populations of big-leaf mahogany (Swietenia macrophylla) inferred from microsatellite analysis. Molecular Ecology 12:2885-2893. [ Links ]
Ovenden J., Peel D., Street R., Courtney A.J., Hoyle S.D., Peel S.L. y Podlich H. 2007. The genetic effective and adult census size of an Australian population of tiger prawns (Penaeus esculentus). Molecular Ecology 16:127-138. [ Links ]
Pennington T.D. y Sarukhán J. 1968. Manual para la Identificación de Campo de los Principales Arboles Tropicales de México. INIF-SARH-FAO. México, D.F. [ Links ]
Pennington T.D. 2002. Mahogany carving a future. Biologist 49:204-208. [ Links ]
Pérez-Nasser N., Eguiarte L.E. y Piñero D. 1993. Mating system and genetic structure of the distylous tropical tree Psychotria faxlucens (Rubiaceae). American Journal of Botany 80:45-52. [ Links ]
Piperno D.R. y Pearsall D.M. 1998. The Origins of Agriculture in the Lowland Neotropics. Academic Press, San Diego. [ Links ]
Pither R., Shore J.S. y Kellman M. 2003. Genetic diversity of the tropical tree Terminalia amazonia (Combretaceae) in naturally fragmented populations. Heredity 91:307-313. [ Links ]
Rodríguez S., Chavelas PB.J. y García Cuevas X. 1994. Dispersión de semillas y establecimiento de caoba después de un tratamiento mecánico del sitio. En: Snook L. y Barrera de Jorgenson A. Eds. Madera, Chicle, Caza y Milpa. Contribuciones al Manejo Integral de la Selvas de Quintana Roo, México pp. 81-90. PROAFT, INIFAP, USAID, WWF-US. Mérida. [ Links ]
Schaal B.A., O'Keane S.L. Jr. y Rogstad S.H. 1991. DNA variation in plant populations. Trends in Ecology and Evolution 6:329-333. [ Links ]
SEMARNAT. Secretaria de Medio Ambiente y Recursos Naturales. 2006. Atlas de experiencias comunitarias en manejo sostenible de los recursos naturales de Quintana Roo. SEMARNAT. México. [ Links ]
Slatkin M. 1987. Gene flow and the geographic structure of natural populations. Science 236:787-792. [ Links ]
Slatkin M. 1995. A measure of population subdivision based on microsatellite allele frequencies. Genetics 139:457-462. [ Links ]
Takezaki N. y Nei M. 1996. Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA. Genetics 144:389-399. [ Links ]
Takezaki N., Nei M. y Tamura K. 2010. POPTREE2: Software for Constructing Population Trees from Allele Frequency Data and Computing Other Population Statistics with Windows Interface. Molecular Biology and Evolution 27:747-752. [ Links ]
Vázquez-Lobo Y.A. 1996. Evolución de hongos endófitos del género Pinus L: implementación de técnicas moleculares y resultados preliminares. Tesis de Licenciatura. Facultad de Ciencias. Universidad Nacional Autónoma de México, México, D.F. 66 pp. [ Links ]
Waples R.S. 1991. Genetics methods for estimating the effective size of cetacean populations. En: Hoezel A.R. Ed. Genetic Ecology of Whales and Dolphins, pp. 279-300, Reports of International Whaling Commission. Special Issue 13, International Whaling Commission, Cambridge. [ Links ]
Ward S.E. y Lugo A.E. 2003. Twenty mahogany provenances under different conditions in Puerto Rico. En: Lugo A.E., Figueroa C.J.C. y Alayón M. Eds. Big Leaf Mahogany: Genetics, Ecology and Management. Ecological Studies 159, pp. 29-93, Springer-Verlag, Nueva York. [ Links ]
Ward M., Dick C.W., Gribel R. y Lowe A.J. 2005. To self, or not to self... A review of outcrossing and pollen-mediated gene flow in neotropical trees. Heredity 95:246-254. [ Links ]
Weir B.S. 1996. Genetic Data Analysis II. Sinauer Associates, Sunderland. [ Links ]
White G. y Powell W. 1997. isolation and characterization of microsatellite loci in Swietenia humilis (Meliaceae): an endangered tropical hardwood species. Molecular Ecology 6:851-860. [ Links ]
White G.M., Boshier D.H. y Powell W. 1999. Genetic variation within a fragmented population of Swietenia humilis Zucc. Molecular Ecology 8:1899-1909. [ Links ]
Whitmore J.L. 1983. Swietenia macrophylla and S. humilis (caoba, mahony). En: Janzen D.H. Ed. Costa Rica Natural History, pp. 331-333, university of Chicago Press, Chicago. [ Links ]














