Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Chapingo serie ciencias forestales y del ambiente
On-line version ISSN 2007-4018Print version ISSN 2007-3828
Rev. Chapingo ser. cienc. for. ambient vol.23 n.3 Chapingo Sep./Dec. 2017
https://doi.org/10.5154/r.rchscfa.2017.03.019
Artículo científico
Caracterización molecular mediante iniciadores ISSR de Magnolia mexicana DC. de dos regiones de Zongolica, Veracruz, México
1Universidad Autónoma Chapingo, Departamento de Fitotecnia. km 38.5 Carretera México-Texcoco. C. P. 56230. Chapingo, Texcoco, Estado de México, México.
2Universidad Autónoma Chapingo, Departamento de Preparatoria Agrícola, Área de Química. km 38.5 Carretera México-Texcoco. C. P. 56230. Chapingo, Texcoco, Estado de México, México.
Introducción:
Magnolia mexicana DC. es una especie amenazada según la NOM-059-SEMARNAT-2010, situación atribuida a la fragmentación y destrucción del hábitat. No existen estudios sobre la diversidad genética de M. mexicana, a pesar de que es endémica de nuestro país.
Objetivo:
Evaluar la variabilidad genética en dos poblaciones de M. mexicana mediante marcadores moleculares tipo ISSR.
Materiales y métodos:
Las colectas provienen de Amatitla y Zapotla en Zongolica, Veracruz. El ADN se extrajo de las hojas jóvenes. Se probaron 55 iniciadores ISSR, se seleccionaron los 10 que produjeron mayor número de bandas con polimorfismo y se amplificaron por PCR.
Resultados y discusión:
Los iniciadores ISSR mostraron 86 % de polimorfismo. El análisis de agrupamiento, con el método de varianza mínima de Ward, fue capaz de separar las colectas por su procedencia geográfica. El análisis de varianza molecular demostró que la mayor variabilidad (90.88 %) se encuentra dentro de cada población. El índice de diversidad de Shannon-Weaver fue de 0.47 y 0.41 para Amatitla y Zapotla, respectivamente.
Conclusión:
Las poblaciones de M. mexicana no han sufrido cambios en su estructura genética; no hay evidencia, a nivel genético, de alteraciones ocasionadas por la reducción de poblaciones o fragmentación del hábitat.
Palabras clave: Especie amenazada; hábitat fragmentado; variabilidad genética; índice de Shannon-Weaver
Introduction:
Magnolia mexicana DC. is a threatened species according to NOM-059-SEMARNAT-2010, a situation attributed to the fragmentation and destruction of its habitat. There are no studies on the genetic diversity of M. mexicana, even though it is endemic to our country.
Objective:
To evaluate genetic variability in two M. mexicana populations using ISSR molecular markers.
Materials and methods:
The collections come from Amatitla and Zapotla in Zongolica, Veracruz. The DNA was extracted from the young leaves. A total of 55 ISSR primers were tested; the 10 that produced the highest number of polymorphic bands were selected and amplified by PCR.
Results and discussion:
The ISSR primers showed 86 % polymorphism. Cluster analysis, using Ward's minimum variance method, was able to separate the collections by their geographical origin. Analysis of molecular variance showed that the greatest variability (90.88 %) is found within each population. The Shannon-Weaver diversity index was 0.47 and 0.41 for Amatitla and Zapotla, respectively.
Conclusion:
The M. mexicana populations have not undergone changes in their genetic structure; there is no evidence at genetic level of alterations caused by population reduction or habitat fragmentation.
Keywords: Threatened species; fragmented habitat; genetic variability; Shannon-Weaver index
Introducción
Las especies de la familia Magnoliaceae son consideradas primitivas debido a que los sépalos y pétalos no están claramente diferenciados, además están dispuestos en espiral y no en anillo como en las demás angiospermas (Cicuzza, Newton, & Oldfield, 2007). Esta característica es de gran interés en los estudios de evolución, biogeográficos, taxonómicos y ecológicos (Endress & Doyle, 2009). Magnolia mexicana DC. es una especie endémica de México que se encuentra en selvas altas perennifolias y bosque mesófilo de montaña (Palacios, 2006). El hábitat de la especie se encuentra seriamente amenazado; aproximadamente, 50 % de la superficie original del bosque mesófilo ha sido reemplazado por otros tipos de cobertura (Comisión Nacional para el Conocimiento y Uso de la Biodiversidad [CONABIO], 2010). Esta situación se relaciona con el cambio climático y actividades antropogénicas como la construcción de caminos, pastoreo y depredación (CONABIO, 2010; Nora, Albaladejo, González-Martínez, Robledo-Arnuncio, & Aparicio, 2011). Por esta razón, M. mexicana fue incluida en la lista de la NOM-059-SEMARNAT-2010 en la categoría de especie amenazada (Secretaría de Medio Ambiente y Recursos Naturales [SEMARNAT], 2010). Actualmente podemos encontrar poblaciones viables en los estados de Chiapas, Oaxaca, Veracruz y Puebla, algunas son remanentes asociados a cultivos, principalmente en cafetales, donde se utilizan como sombra (Palacios, 2006).
En las especies vegetales amenazadas como M. mexicana, es importante conocer la estructura genética poblacional, los niveles de variación y la diferenciación genética, así como los efectos de los mecanismos microevolutivos. La selección natural, endogamia, deriva génica, mutación y flujo génico son influenciados por la dinámica demográfica poblacional, causada por las tasas de natalidad, mortalidad y migración (Hamrick, Godt, & Sherman-Broyles, 1992). A pesar de que M. mexicana es una especie endémica de nuestro país, a la fecha no hay estudios sobre la diversidad genética existente. Los niveles de variación genética entre organismos de una especie o entre diferentes niveles taxonómicos se pueden determinar a través de los métodos tradicionales de comparaciones morfológicas y las técnicas modernas de análisis del ADN (Mohammadi & Prasanna, 2003). Las Inter Secuencias Simples Repetidas o ISSR (Inter-Simple Sequence Repeat) son un tipo de marcador genético que permite medir los niveles de variación en las regiones intermicrosatélite dispersas en el genoma. Los ISSR son marcadores semiarbitrarios por lo que se amplifican a partir de un iniciador complementario a la región microsatélite (Cornejo, Serrato, Rendón, & Rocha, 2014). Estos marcadores se han utilizado en especies como sorgo y banano (Godwin, Aitken, & Smith, 1997), cafeto (Aga, Bekele, & Bryngelsson, 2005) y plantas catalogadas primitivas como las cícadas (Xiao, Ge, Gong, Hao, & Zheng, 2004). El objetivo de este estudio fue evaluar la variabilidad genética presente en dos poblaciones de M. mexicana mediante el uso de marcadores moleculares tipo ISSR. Las poblaciones pertenecen a las regiones de Amatitla y Zapotla del municipio de Zongolica, Veracruz, México.
Materiales y métodos
Material vegetal
Debido a las dificultades técnicas relacionadas con la colecta de hojas jóvenes de M. mexicana en árboles de altura promedio de 35 m y con la finalidad de obtener ADN de buena calidad, se optó por evaluar material vegetal proporcionado por la unidad de manejo ambiental de Yoloxóchitl, ubicada en el municipio de Zongolica, Veracruz, México. Las semillas de Amatitla (18° 39´ 34.77” N, 96° 59´ 43.38” O) y Zapotla (18° 38´ 46.72” N, 96° 58´ 20.61” O), localidades ubicadas a una distancia de 2 248 m entre sí, fueron colectadas y germinadas in situ. Posteriormente seis y siete ejemplares correspondientes a cada localidad se usaron para obtener las hojas jóvenes y realizar la extracción de ADN.
Extracción y purificación de ADN genómico
La extracción de ADN se hizo de acuerdo con el método reportado por Dellaporta, Wood, y Hicks (1983). Para tal objetivo se pesaron 0.3 g de material vegetal y se suspendieron en una solución (pH 8.0) de Tris-HCl (100 mM), EDTA-Na2 (50 mM), NaCl (500 mM), 2-Mercaptoetanol (10 mM) y SDS (1.3 %), como amortiguador de extracción. Posteriormente, la solución se centrifugó a 12 000 rpm durante 20 min. El sobrenadante se mezcló con isopropanol frío y el ADN se dejó precipitar por 60 min, luego se agregó solución STE (Tris-HCl [50 mM], EDTA-Na2 [10 mM], NaCl [100 mM], pH 8.0) y ARNasa (10 mg·mL-1) y se incubó a 37 °C por una hora. Después se agregó acetato de sodio (3 M) e isopropanol frío y se centrifugó a 8 000 rpm por 5 min para la precipitación del ADN y eliminación del sobrenadante. El ADN se lavó con etanol (70 %) y se dejó secar, después se disolvió en amortiguador TE (Tris-HCl [10 mM], EDTA-Na2 [1 mM], pH 8.0) y se almacenó en refrigeración hasta su uso. La cantidad y pureza del ADN se midió en un Nanodrop Thermo Scientific® (modelo LITE, EUA) y la calidad se determinó mediante electroforesis en agarosa teñida con bromuro de etidio. Las bandas se visualizaron bajo luz ultravioleta en un transiluminador DigiDoc-it Imaging System (UVP®, EUA) y las imágenes obtenidas se documentaron mediante una cámara Kodak ® EDAS290.
Obtención de patrones ISSR-PCR
Se probaron 55 iniciadores ISSR (Sigma®) y se seleccionaron los 10 que produjeron el mayor número de bandas nítidas y con polimorfismo (Cuadro 1). Los segmentos de ADN se amplificaron mediante PCR. La mezcla para PCR tuvo un volumen total de 25 μL, incluyendo los reactivos siguientes: 5.2 μL de H2O grado biología molecular, 10 μL de dNTPs (dGTP, dATP, dTTP, dCTP a una concentración 500 μM), 2.5 μL de amortiguador de PCR (10X), 1.5 μL de MgCl2 (50 mM), 3.0 μL de iniciador (10 ng·μL-1), 0.3 μL de Taq ADN polimerasa (5 U·µL-1) y 2.5 μL de ADN genómico de las muestras homogeneizado a una concentración de 10 ng·µL-1. La mezcla se sometió a un ciclo inicial de desnaturalización de 8 min a 93 °C; 40 ciclos de 1 min a 93 °C, 1 min a la temperatura de alineamiento recomendada para cada iniciador (Cuadro 1), 1 min a 72 °C; y un ciclo de extensión final de 6 min a 72 °C. La amplificación se realizó en un termociclador marca Techne® modelo TC-412 (EUA). La separación de los productos amplificados se hizo mediante electroforesis en gel de agarosa al 1 % con amortiguador TAE (Tris acetato [40 mM], pH 7.6; Na2EDTA [1 mM]), teñido con bromuro de etidio para visualizar las bandas en luz ultravioleta del transiluminador DigiDoc-it Imaging System (UVP®). Las imágenes obtenidas se documentaron con la cámara Kodak ® EDAS290.
Cuadro 1 Iniciadores ISSR utilizados en dos poblaciones de Magnolia mexicana en la región de Zongolica, Veracruz.
| Iniciador | Secuencia (5´-3´) | Ta (°C) | BT | BP | P (%) |
|---|---|---|---|---|---|
| ISSR2 | (CA)8AAGCT | 62 | 10 | 10 | 100 |
| ISSR3 | (GA)8CTC | 58 | 9 | 5 | 55.5 |
| ISSR4 | (AG)8CTG | 58 | 9 | 9 | 100 |
| LOL9 | (CAC)3GC | 38 | 10 | 9 | 90.0 |
| MESL3 | (TG)7G | 46 | 6 | 6 | 100 |
| PI02 | (CA)6AGG | 46 | 5 | 5 | 100 |
| PI04 | (CT)8AGC | 58 | 10 | 8 | 80.0 |
| UBC840 | (GA)8CTT | 56 | 5 | 4 | 80.0 |
| UBC842 | (GA)8CTG | 58 | 8 | 5 | 62.5 |
| 17898B | (CA)6GT | 42 | 9 | 9 | 100 |
| 81 | 70 | 86.4 |
Ta: Temperatura de alineamiento, BT: bandas totales, BP: bandas polimórficas, P: polimorfismo.
Análisis estadístico
El número de bandas, producto de la amplificación, se cuantificó asignándose el valor 1 a la presencia y 0 a la ausencia, de esta manera se construyó una matriz básica de datos (MBD) para realizar los análisis. Las similitudes genéticas se obtuvieron con el coeficiente de Jaccard con trasformación √1-S y el análisis de agrupamiento se realizó con el método de varianza mínima de Ward; adicionalmente se calculó el análisis de varianza molecular (AMOVA), para cuantificar la distribución de la variabilidad molecular dentro y entre poblaciones (Balzarini, Bruno, Peña, Teich, & Di Rienzo, 2010), y finalmente se calculó el índice de diversidad de Shannon-Weaver.
Para obtener una representación espacial de la distribución de las muestras de M. mexicana se hizo un análisis de coordenadas principales (ACoorP) junto con un árbol de mínimo recorrido (AMR), que es una representación geométrica de los resultados provenientes del análisis de agrupamiento. Este análisis se sugiere para mejorar la interpretación al permitir considerar las deformaciones ocurridas en la proyección de las coordenadas principales (Balzarini et al., 2010). Todos los análisis se hicieron con el programa Info-Gen® (Balzarini & Di Rienzo, 2004).
Resultados y discusión
Uno de los objetivos del análisis con marcadores moleculares es la clasificación de los individuos de manera que se puedan distinguir grupos homogéneos entre ellos. Para tal fin se utilizan métodos jerárquicos de análisis de conglomerados (agrupamientos o cluster en inglés) (Balzarini et al., 2010). La Figura 1 muestra el dendrograma obtenido para las muestras de M. mexicana, el cual reflejó mayoritariamente la procedencia geográfica de los materiales. En dicha figura, a una altura de corte de 0.83, se pueden distinguir claramente dos grupos: el grupo uno formado por cinco de los siete ejemplares provenientes de Zapotla, mientras que el grupo dos estuvo integrado por cinco de los seis ejemplares procedentes de Amatitla. Estos resultados muestran que la diferenciación genética entre poblaciones fue suficiente para cambiar la frecuencia de bandas ISSR permitiendo su clara separación. No obstante, los patrones de bandas no reflejan necesariamente el patrón de variación en genes que controlan caracteres de importancia agronómica o aptitud geográfica, por lo que los marcadores ISSR no siempre discriminan genotipos con base en área de adaptación. Las diferencias entre estos tres individuos pudieron originarse por flujo genético entre las dos poblaciones o bien debido a la presencia de factores evolutivos similares (Slavov et al., 2013).
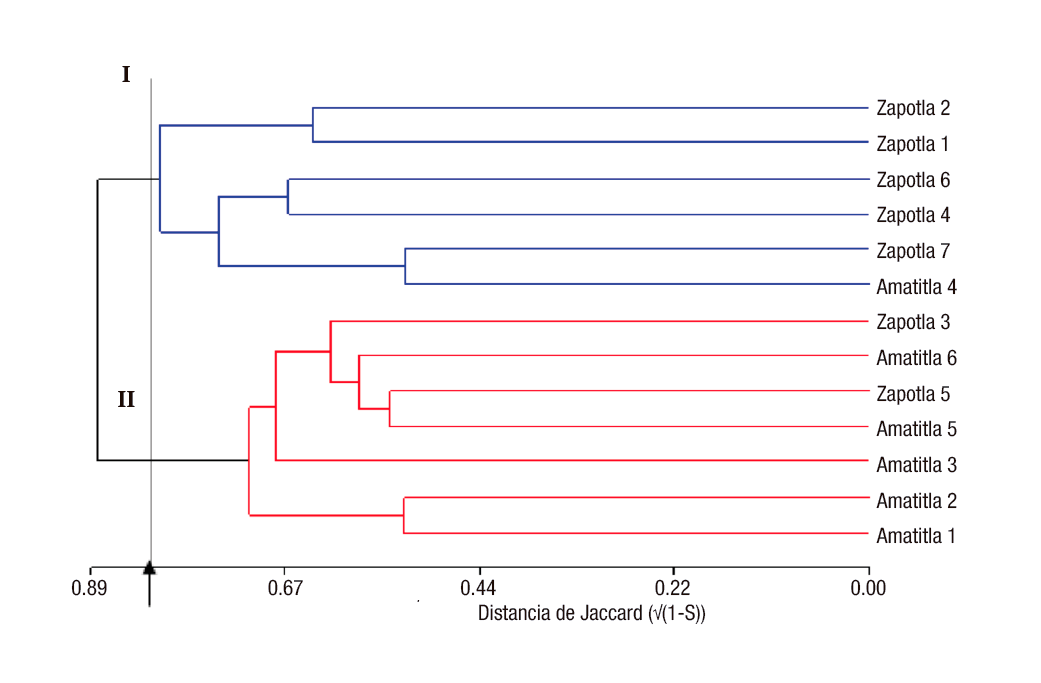
Figura 1 Dendrograma construido por el método de varianza mínima de Ward y las distancias de Jaccard entre muestras de Magnolia mexicana procedentes de Zongolica, Veracruz, México.
El análisis AcooP, en la Figura 2, muestra que todos los elementos provenientes de Amatitla pueden ser encerrados dentro de un círculo debido a su cercanía, mientras que fuera de este grupo se localizan los ejemplares de Zapotla. La misma tendencia se observa en el AMR (Figura 2) en el cual mediante líneas se conectan los elementos más cercanos entre sí, correspondientes a los árboles de Amatitla. Los análisis de agrupamientos ACooP y AMR reflejaron la distribución de los ejemplares según la zona de colecta Amatitla y Zapotla, mientras que en el dendrograma algunos ejemplares quedaron mal agrupados.
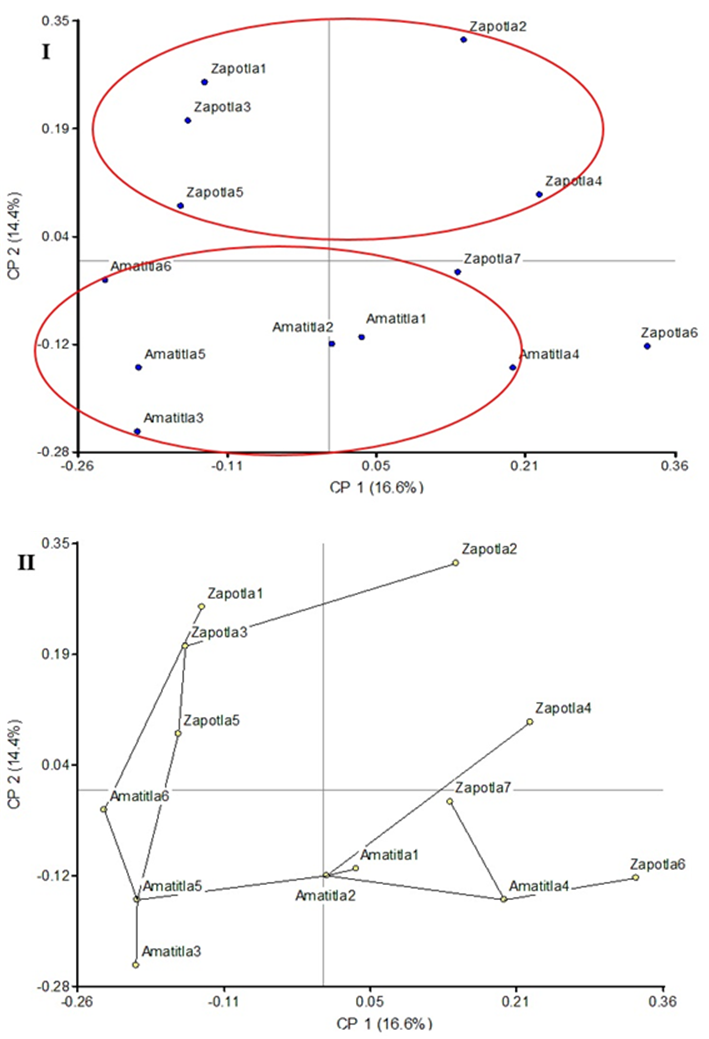
Figura 2 Análisis de Coordenadas Principales (I) y Árbol de Recorrido Mínimo (II) de 13 muestras de Magnolia mexicana procedentes de Zongolica, Veracruz, México.
Los marcadores ISSR son una técnica que puede detectar una alta variación polimórfica en la secuencia de ADN (Cornejo et al., 2014). Estos marcadores se han empleado con éxito para distinguir la variación, estructura y diferenciación genética de diferentes especies de árboles entre los que se encuentran Dendropanax arboreus (L.) Decne. & Planch. en la zona de los Tuxtlas, México (Figueroa-Esquivel, Puebla-Olivares, Eguiarte, & Núñez-Farfán, 2010); Theobroma speciosum Willd. ex Spreng. en Brasil (Giustina et al., 2014); Swietenia macrophylla King en Costa Rica (Céspedes, Gutiérrez, Holbrook, & Rocha, 2003); y S. humilis Zucc. en Centroamérica (White, Boshier, & Powell, 1999).
La fragmentación es un proceso de alteración de la calidad y extensión de un hábitat que conduce a una pérdida de biodiversidad a nivel intraespecífico, lo cual ocasiona deriva génica y endogamia y, con ello, la reducción de la variabilidad genética (Nora et al., 2011). En el presente estudio, el análisis AMOVA mostró que 90.88 % de la variación genética es atribuible a diferencias individuales dentro de las dos poblaciones muestreadas (Cuadro 2), por lo que el efecto de la fragmentación no es evidente a este nivel. En otros estudios, usando ISSR, se ha reportado alta variabilidad genética dentro de poblaciones de Magnolia wufengensis L. (90.9 %) (Chen, Chen, He, & Ma, 2014) y M. officinalis Rehder & E. H. (68.4 %) (Yu, Yang, Sun, & Liu, 2011), ambos trabajos realizados en China. Por tanto, los resultados del AMOVA en el presente estudio coinciden con la observación general de que las especies leñosas, perennes y exógenas mantienen la mayor parte de su variación dentro de las poblaciones (Hamrick et al., 1992).
Cuadro 2 Análisis de varianza molecular (AMOVA) de dos poblaciones de Magnolia mexicana procedentes de Zongolica, Veracruz.
| Fuente de variación | Suma de cuadrados | Grados de libertad | Cuadrados medios | P-valor | Componentes de varianza | Variación (%) |
|---|---|---|---|---|---|---|
| Entre poblaciones | 20.81 | 1 | 20.81 | 0.0300 | 1.27 | 9.12 |
| Dentro de población | 138.88 | 11 | 12.63 | 0.0175 | 12.63 | 90.88 |
| Total | 159.69 | 12 | 13.31 | 13.89 | 100.00 |
El cálculo del índice de diversidad de Shannon-Weaver permite dimensionar la diversidad genética, considerando un valor de cero cuando solo se presenta un alelo en la población; conforme aumenta el número de alelos, el valor también incrementa y con ello la diversidad genética (Pla, 2006). En el Cuadro 3 se presentan los índices de diversidad calculados para las poblaciones de M. mexicana muestreadas, los cuales fueron muy similares (0.47 y 0.41 para Amatitla y Zapotla, respectivamente). Balzarini et al. (2010) consideran que las muestras de dos poblaciones siempre tendrán un valor mayor que la media de sus diversidades determinada por separado, excepto si ambas poblaciones son idénticas en su composición. En este trabajo, el valor medio del índice de diversidad fue 0.44, mientras que la diversidad total fue 0.42, lo que indica que las poblaciones muestreadas presentan igual diversidad genética. Los valores del índice de Shannon-Weaver fueron similares a los estimados en otras especies del género Magnolia. Yu et al. (2011) reportaron un valor de diversidad de 0.41 en poblaciones de Magnolia officinalis (Rehder & E. H. Wilson) muestreadas en China, mientras que Newton et al. (2008) determinaron un índice de 0.56 en colectas de Magnolia sharpii Miranda y 0.50 en Magnolia schiedeana Schltdl., en México.
Cuadro 3 Índice de diversidad de Shannon-Weaver en dos poblaciones de Magnolia mexicana procedentes de Zongolica, Veracruz.
| Grupo | n | I | LI | LS | IBoot | EEBoot |
|---|---|---|---|---|---|---|
| Amatitla | 6 | 0.47 | 0.39 | 0.41 | 0.40 | 0.03 |
| Zapotla | 7 | 0.41 | 0.40 | 0.41 | 0.40 | 0.03 |
| Total | 2 | 0.42 | 0.41 | 0.42 | 0.42 | 0.03 |
n: número de muestras, I: índice de diversidad de Shannon-Weaver, LI: límite inferior, LS: límite superior, Iboot: 500 ciclos de remuestreo (bootstrap) con intervalo de confianza de 95 %, EEBoot: error estándar del remuestreo.
El grado de diferenciación genética aumenta, generalmente, cuando la distancia entre poblaciones incrementa (Pusadee, Jamjod, Chiang, Rerkasem, & Schaal, 2009). No obstante, Yu et al. (2011) consideran que otros factores han contribuido a maximizar la variabilidad genética de M. officinalis, como los relacionados con sus características ornamentales, la intromisión humana debida al desplazamiento por pobladores y las aves atraídas por la alta visibilidad de las semillas rojas de la especie. En M. mexicana se ha encontrado que los escarabajos del género Cyclocephala actúan como agentes polinizadores y dispersadores en la zona de Zongolica (Mora-Aguilar & Delgado, 2012). Algunos estudios demuestran que las tasas de polen inmigrante pueden ser elevadas en poblaciones fragmentadas, en especial si la polinización es entomófila, estimándose distancias de 21 m en Centaurea corymbosa Pourr. (Hardy et al., 2004) hasta 88.6 km en higuera Ficus sycomorus L. (Ahmed, Compton, Butlin, & Gilmartin, 2009).
Por otro lado, cabe destacar que las selvas altas perennifolias y los bosques mesófilos de montaña de zonas de Veracruz, como Zongolica y los Tuxtlas, tienen relictos de áreas conservadas (Sandoval et al., 2007). Los resultados obtenidos en el presente estudio sugieren que la reducción del hábitat y disminución de las poblaciones de M. mexicana aún no han reducido la variabilidad genética de las poblaciones naturales de manera importante. Sin embargo, es necesario realizar estudios en otras poblaciones de M. mexicana considerando un mayor número de localidades, para tener una estimación más precisa de los cambios en la estructura y variación genética de la especie en el estado de Veracruz, en particular, y de México, en general.
Conclusiones
Los resultados del presente estudio sugieren que las poblaciones de Magnolia mexicana no han sufrido cambios que modifiquen su estructura genética, por lo que no hay evidencia, a nivel genético, de alteraciones ocasionadas por la reducción de poblaciones o fragmentación del hábitat. Los niveles de diversidad genética de M. mexicana en las poblaciones estudiadas son semejantes a los reportados en otras especies de la familia Magnoliaceae, así como en otros árboles en bosques mesófilos de montaña. Los marcadores moleculares tipo ISSR demostraron su utilidad para evaluar la variabilidad genética presente en especies arbóreas como M. mexicana.
Agradecimientos
Se agradece el apoyo técnico invaluable de la M. C. María Elisa Alvarado Cano y Q. F. B. Ricardo Gaspar Hernández del Laboratorio de Mejoramiento Genético Asistido del Departamento de Fitotecnia de la Universidad Autónoma Chapingo, y el apoyo incondicional del Ing. Antonio Vázquez Lemus de la UMA-Yoloxochitl en Zongolica, Veracruz.
REFERENCIAS
Aga, E., Bekele, E., & Bryngelsson, T. (2005). Inter-simple sequence repeat (ISSR) variation in coffee forest trees (Coffea arabica L.) populations from Ethiopia. Genetica, 124, 213-221. doi: 10.1007/s10709-005-1484-6 [ Links ]
Ahmed, S., Compton, S. G., Butlin, R. K., & Gilmartin, P. M. (2009). Wind-borne insects mediate directional pollen transfer between desert fig trees. 160 kilometers apart. Proceedings of the National Academy of Sciences, 106(48), 20342-20347. doi: 10.1073/pnas.0902213106 [ Links ]
Balzarini, M., & Di Rienzo, J. (2004). Info-Gen: Software para análisis estadístico de datos genéticos. Argentina: Facultad de Ciencia Agropecuarias, Universidad Nacional de Córdoba. [ Links ]
Balzarini, M., Bruno, C., Peña, A., Teich, I., & Di Rienzo, J. (2010). Estadística en Biotecnología. Aplicaciones en Info-Gen. Córdoba, Argentina: Encuentro Grupo Editor. [ Links ]
Céspedes, M., Gutiérrez, M. V., Holbrook, N. M., & Rocha, J. (2003). Restoration of genetic diversity in the dry forest tree Swietenia macrophylla (Meliaceae) after pasture abandonment in Costa Rica. Molecular Ecology, 12(12), 3201-3212. [ Links ]
Chen, L., Chen, F., He, S., & Ma, L. (2014). High genetic diversity and small genetic variation among populations of Magnolia wufengensis (Magnoliaceae), revealed by ISSR and SRAP markers. Electronic Journal of Biotechnology. Retrieved from http://www.redalyc.org/articulo.oa?id=173332565003 [ Links ]
Cicuzza, D., Newton, A., & Oldfield, S. (2007). Red list of the Magnoliaceae. Cambridge, UK: Fauna & Flora International. Retrieved from https://www.bgci.org/plant-conservation/magnolia_red_list/ [ Links ]
Comisión Nacional para el Conocimiento y Uso de la Biodiversidad (CONABIO). (2010). El bosque mesófilo de montaña en México: amenazas y oportunidades para su conservación y manejo sostenible. México: Autor. [ Links ]
Cornejo, R. A., Serrato, D. A., Rendón, A., & Rocha, M. M. G. (2014). Herramientas moleculares aplicadas en ecología: aspectos teóricos y prácticos. México: INECC/SEMARNAT. Retrieved from http://www.publicaciones.inecc.gob.mx/?id_pub=710 [ Links ]
Dellaporta, S. L., Wood, J., & Hicks, J. B. (1983). A plant DNA minipreparation: Version II. Plant Molecular Biology Reporter, 1(4), 19-21. doi: 10.1007/BF02712670 [ Links ]
Endress, P. K., & Doyle, J. A. (2009). Reconstructing the ancestral angiosperm flower and its initial specializations. American Journal of Botany, 96(1), 22-66. doi: 10.3732/ajb.0800047 [ Links ]
Figueroa-Esquivel, E. M., Puebla-Olivares, F., Eguiarte, L. E., & Núñez-Farfán, J. (2010). Genetic structure of a bird-dispersed tropical tree (Dendropanax arboreus) in a fragmented landscape in Mexico. Revista Mexicana de Biodiversidad, 81(3), 789-800 Retrieved from http://www.redalyc.org/articulo.oa?id=42518439019 [ Links ]
Giustina, L. D., Luz, L. N., Vieira, F. S., Rossi, F. S., Soares-Lopes, C. R. A., Pereira, T. N. S., & Rossi, A. A. B. (2014). Population structure and genetic diversity in natural populations of Theobroma speciosum Willd. Ex Spreng (Malvaceae). Genetics and Molecular Research: GMR, 13(2), 3510-3519. doi: 10.4238/2014.February.14.5 [ Links ]
Godwin, I. D., Aitken, E. A. B., & Smith, L. W. (1997). Application of inter simple sequence repeat (ISSR) markers to plant genetics. Electrophoresis, 18(9), 1524-1528. doi: 10.1002/elps.1150180906 [ Links ]
Hamrick, J. L., Godt, M. J. W., & Sherman-Broyles, S. L. (1992). Factors influencing levels of genetic diversity in woody plant species. New Forests, 6(1-4), 95-124. doi: 10.1007/BF00120641 [ Links ]
Hardy, O. J., González-Martínez, S. C., Colas, B., Fréville, H., Mignot, A., & Olivieri, I. (2004). Fine-scale genetic structure and gene dispersal in Centaurea corymbosa (Asteraceae). II. Correlated paternity within and among sibships. Genetics, 168(3), 1601-1614. doi: 10.1534/genetics.104.027714 [ Links ]
Mohammadi, S. A., & Prasanna, B. M. (2003). Analysis of genetic diversity in crop plants-salient statistical tools and considerations. Crop Science, 43, 235-248. Retrieved from https://www.researchgate.net/publication/240787100_Analysis_of_Genetic_Diversity_in_Crop_Plants-Salient_Statistical_Tools_and_Considerations [ Links ]
Mora-Aguilar, E. F., & Delgado, L. (2012). A new species of Cyclocephala Dejean (Coleoptera: Scarabaeidae: Dynastinae: Cyclocephalini) from the cloud forests of Southeastern Mexico and description of the female of Cyclocephala berti Delgado. The Coleopterists Bulletin, 66(2), 139-142. doi: 10.1649/072.066.0209 [ Links ]
Newton, A., Gow, J., Robertson, A., Williams, L. A., Ramírez, M. N., González, E. M., & Ennos, R. (2008). Genetic variation in two rare endemic Mexican trees, Magnolia sharpii and Magnolia schiedeana. Silvae Genetica, 57(6), 348-356. Retrieved from https://www.thuenen.de/media/institute/fg/PDF/Silvae_Genetica/2008/Vol._57_Heft_6/57_6_348.pdf [ Links ]
Nora, S., Albaladejo, R. G., González-Martínez, S. C., Robledo-Arnuncio, J. J., & Aparicio, A. (2011). Movimiento de genes (polen y semillas) en poblaciones fragmentadas de plantas. Revista Ecosistemas, 20(2-3), 35-45. Retrieved from https://www.revistaecosistemas.net/index.php/ecosistemas/article/view/21/15 [ Links ]
Palacios, E. (2006). Cuarenta y ocho especies de la flora de Chiapas incluidas en el PROY-NOM-059-ECOL-2000. Retrieved from http://www.conabio.gob.mx/institucion/proyectos/resultados/W008_Fichas%20de%20especies.pdf [ Links ]
Pla, L. (2006). Biodiversidad: Inferencia basada en el índice de Shannon y la riqueza. Interciencia, 31(8), 583-590. Retrieved from http://www.redalyc.org/articulo.oa?id=33911906 [ Links ]
Pusadee, T., Jamjod, S., Chiang, Y. -C., Rerkasem, B., & Schaal, B. A. (2009). Genetic structure and isolation by distance in a landrace of Thai rice. Proceedings of the National Academy of Sciences, 106(33), 13880-13885. doi: 10.1073/pnas.0906720106 [ Links ]
Sandoval, M. J. B., Ramírez, S. A. F., Sheseña, H. I. M., Sormani, C., Ruiz de la Merced, F., Jarvio, A. D., & Farid, M. E. (2007). Evaluación del estado de conservación de los ecosistemas forestales de la región denominada Uxpanapa. Veracruz, México: Dirección General de Desarrollo Forestal, Gobierno del Estado de Veracruz - Pronatura A. C. [ Links ]
Secretaría del Medio Ambiente y Recursos Naturales (SEMARNAT). (2010). Norma Oficial Mexicana NOM-059-SEMARNAT-2010. Protección ambiental- Especies nativas de México de flora y fauna silvestres-Categorías de riesgo y especificaciones para su inclusión, exclusión o cambio-Lista de especies en riesgo. Diario Oficial de la Nación (Segunda sección). Ciudad de México, México. Retrieved from http://www.profepa.gob.mx/innovaportal/file/435/1/NOM_059_SEMARNAT_2010.pdf [ Links ]
Slavov, G., Robson, P., Jensen, E., Hodgson, E., Farrar, K., Allison, G., …Donnison, I. (2013). Contrasting geographic patterns of genetic variation for molecular markers vs. phenotypic traits in the energy grassMiscanthus sinensis. GCB Bioenergy, 5, 562-571. doi: 10.1111/gcbb.12025 [ Links ]
White, G. M., Boshier, D. H., & Powell, W. (1999). Genetic variation within a fragmented population of Swietenia humilis Zucc. Molecular Ecology, 8(11), 1899-1909. doi: 10.1046/j.1365-294x.1999.00790.x [ Links ]
Xiao, L. Q., Ge, X. J., Gong, X., Hao, G., & Zheng, S. X. (2004). ISSR variation in the endemic and endangered plant Cycas guizhouensis (Cycadaceae). Annals of Botany, 94(1),133-138. doi: 10.1093/aob/mch119 [ Links ]
Yu, H. H., Yang, Z. L., Sun, B., & Liu, R. (2011). Genetic diversity and relationship of endangered plant Magnolia officinalis (Magnoliaceae) assessed with ISSR polymorphisms. Biochemical Systematics and Ecology, 39(2), 71-78. doi: 10.1016/j.bse.2010.12.003 [ Links ]
Recibido: 01 de Marzo de 2017; Aprobado: 31 de Julio de 2017











 text in
text in 


