Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Chapingo serie ciencias forestales y del ambiente
versión On-line ISSN 2007-4018versión impresa ISSN 2007-3828
Rev. Chapingo ser. cienc. for. ambient vol.18 no.1 Chapingo ene./abr. 2012
https://doi.org/10.5154/r.rchscfa,2011.03.024
Optimización de un protocolo del aislamiento del ADN y de un sistema de amplificación ISSR-PCR para Ceratozamiamexicana Brongn. (Zamiaceae).
Optimization of a protocol for DNA isolation and ISSR-PCR amplification system for Ceratozamia mexicana Brongn. (Zamiaceae).
Nadia Guadalupe Sánchez-Coello1; Mauricio Luna-Rodríguez2; Mario Vázquez-Torres3; Lázaro Rafael Sánchez-Velásquez1; Nancy Santana-Buzzy4; Pablo Octavio-Aguilar1; Lourdes Georgina Iglesias-Andreu¶1.
1 Instituto de Biotecnología y Ecología Aplicada, Universidad Veracruzana. Av. de las Culturas Veracruzanas Núm.101 Col. E. Zapata C. P. 91090 Xalapa, Veracruz, México. Correo-e: xliglesias@gmail.com. (¶Autor para correspondencia).
2 Laboratorio de Alta Tecnología de Xalapa, Universidad Veracruzana. Calle Médicos Núm. 5, Col. Unidad del Bosque C. P. 91010. Xalapa, Veracruz, México.
3 Instituto de Investigaciones Biológicas, Universidad Veracruzana. Av. Luis Castelazo Ayala s/n, Col. Industrial Ánimas, C.P. 91190, Apartado Postal 294. Xalapa, Veracruz, México.
4 Centro de Investigación Científica de Yucatán. Calle 43 Núm. 130 Col. Chuburná de Hidalgo. C. P. 97200, Mérida, Yucatán, México.
Recibido: 16 de marzo, 2011
Aceptado: 1 de noviembre, 2011
RESUMEN
La mayoría de las cícadas contienen altas concentraciones de aceites esenciales, flavonoides, polifenoles y polisacáridos que interfieren en la extracción de ADN, causando productos de amplificación errados o inhibiendo la PCR. La optimización del aislamiento del ADN y el empleo de iniciadores de secuencias intergénicas repetidas simples (ISSRs) se investigaron en Ceratozamia mexicana Brongn., una cícada mexicana en peligro de extinción. El ADN obtenido de tejido foliar fresco, con un amortiguador modificado de cetil trimetil amonio, nos permitió obtener un ADN de buena calidad, sin pigmentos coloridos o contaminantes. La modificación al protocolo de extracción de ADN, basado en CTAB, fue un prelavado por 1 h, del tejido foliar, con una solución de 0.7 M de NaCl, para facilitar la lisis celular. El ADN extraído exitosamente se amplificó por PCR, usando seis iniciadores arbitrarios ISSR. Se observaron productos de amplificación reproducibles en todas las reacciones de PCR. Nuestros resultados muestran que la implementación mejora significativamente la calidad del ADN obtenido, usando una concentración baja de iniciadores (25 pM). Se detectaron 23 bandas fuertes, nueve de las cuales fueron polimórficas. Los resultados indican que el protocolo de optimización del aislamiento del ADN y en el sistema de PCR es viable para futuros trabajos en esta especie. Este trabajo es el primer protocolo de extracción de ADN y de ISSR reportado para esta especie ornamental en peligro de extinción.
Palabras clave: Ceratozamiamexicana, Cícadas, extracción de ADN, especies en peligro, ISSR.
ABSTRACT
Most of the cycads contain high concentrations of essential oils, flavonoids, polyphenols, and polysaccharides that interfere with DNA extraction, causing erroneous or no PCR products. The optimization of DNA isolation, employing inter-simple sequence repeats (ISSRs) primers were investigated in Ceratozamia mexicana Brongn., an endangered Mexican cycad. The DNA obtained from fresh-leaf tissues with a modified cetyltrimethylammonium bromide buffer protocol gave a good quality of DNA with no colored pigments and contaminants. The main modification to the CTAB-based DNA extraction protocol was the one hour leaf tissue soaking pre-treatment with a 0.7 M NaCl solution, to facilitate the cell lysis. The DNA extracted was successfully amplified by PCR using six arbitrary ISSR primers. Reproducible amplifiable products were observed in all PCR reactions. Our results show a significant improvement in the DNA quality obtained using low primer concentration (25 pM). 23 strong bands were detected, 9 of which were polymorphic. The results indicated that the optimized protocol for DNA isolation and PCR system is suitable for further work in this specie. This work is the first DNA extraction and ISSR protocols reported for this ornamental and endangered species.
Keywords: Ceratozamiamexicana, Cycads, DNA extraction, endangered species, ISSR.
INTRODUCCIÓN
Ceratozamia mexicana Brongn. (Zamiaceae) es una planta dioica (Norstog y Nicholls, 1997). Es una cícada endémica de México distribuida en la zona central del país, particularmente en Coacoatzintla, Veracruz (Vovides, 1983). Esta cícada, al igual que el resto del grupo, tiene un alto valor comercial como planta ornamental y constituye un importante recurso genético mexicano por su larga vida (Vovides y Iglesias, 1994). Debido a la pérdida y fragmentación de su hábitat, su distribución limitada y los atributos de su historia de vida (cruzamiento obligado, larga vida y dioicidad), esta especie se encuentra en la categoría de vulnerable y está protegida por el gobierno mexicano bajo la NOM-059 (SEMARNAT, 2010).
Con el fin de contribuir a la conservación de este importante recurso genético, es necesario desarrollar protocolos moleculares que permitan determinar los niveles de variación genética dentro de las poblaciones en diferentes condiciones ambientales (Avise, 1994, González-Astorga et al., 2006), entre sexos (Reamon- Büettner y Jung, 2000; Flachowsky et al., 2001; Prakash y Staden, 2006) y entre categorías de tamaño o edad (Octavio- Aguilar et al., 2009).
En el estado actual del conocimiento, contar con una técnica efectiva para el proceso de extracción de ADN es un paso crítico en todos los estudios de genética molecular (Boiteux et al., 1999). Contar con protocolos rápidos, fiables y de bajo costo para la extracción de ADN resulta siempre deseable. El ADN extraído de tejidos proveniente de la especie Ceratozamiamexicana, suele estar degradado o contaminado por aceites esenciales, polifenoles y proteínas. En particular, la cantidad de polisacáridos como componente de las hojas en las cícadas es alta (Yagi etal., 2002) y éstos dificultan la extracción y purificación del ADN (Aljanabi et al., 1999). En general, es difícil extraer y purificar ADN de buena calidad en cícadas debido a la presencia además de los polisacáridos, de grandes cantidades de metabolitos secundarios, proteínas así como taninos, alcaloides y polifenoles. Estos compuestos interfieren al precipitar junto con el ADN, por lo tanto degradan su calidad y reducen su rendimiento (Katterman and Shattuck, 1983).
Las técnicas basadas en PCR, como los Inter- microsatélites (Inter Simple Sequences Repeats-ISSR), desarrollado por Zietkiewicz et al. (1994), han demostrado ser muy útiles en los estudios de variación genética en cícadas (Jianguang et al., 2005), así como para determinar el sexo de las plantas (Gangopadhyay et al., 2007). La técnica ISSR es una modificación de la técnica de microsatélites (SSR) que utiliza un solo iniciador, consistente en repeticiones de di o trinucleótidos abundantes en el genoma. Los iniciadores ISSRs son ligeramente mayores (16-20 pb) por lo que pueden anclarse mejor al ADN y mejorar la fiabilidad y reproducibilidad del sistema (Reddy et al., 2002). Además, los ISSRs son marcadores moleculares significativamente más baratos que otros como los AFLPs (Polimorfismo de longitud de fragmentos amplificados). Con el fin de realizar un manejo y conservación eficiente de C.mexicana, es necesario realizar extensas investigaciones sobre la estandarización de las técnicas basadas en el uso de marcadores moleculares del ADN.
El objetivo del presente estudio es desarrollar un método eficiente, sencillo y económico de extracción de ADN, a partir del tejido foliar de C. mexicana Brongn. (Zamiaceae) para obtener un protocolo óptimo de extracción para futuras aplicaciones en esta especie ornamental y en peligro de extinción.
MATERIALES Y MÉTODOS
Material de estudio
Como material de estudio se utilizaron quince plantas masculinas y femeninas (el sexo se identificó a través de la observación del cono) de C. mexicana Brongn., de la población ubicada en Coacoatzintla, Ver., México. Se colectaron muestras de hoja fresca en los meses de febrero a abril de 2009. Las muestras fueron almacenadas en bolsas de plástico, colocadas dentro de una hielera de unicel y trasladadas al laboratorio, donde se mantuvieron a una temperatura de 4 °C, por pocos días hasta ser utilizadas en la extracción del ADN.
Aislamiento y detección de ADN
Los tejidos foliares colectados se lavaron con una solución de etanol (70 % v/v). Posteriormente 0.060 g (aproximadamente 1 cm2) de tejido foliar fue sumergido durante 1 hora en una solución de 0.70 M de NaCl (Cloruro de Sodio) con el fin de facilitar la lisis del tejido. Las muestras de hojas sin tratamiento de inmersión fueron utilizadas como control. La extracción de ADN se realizó por duplicado, siguiendo el método de Stewart y Vía (1993), basado en el uso de bromuro de cetiltrimetilamonio (CTAB) y una purificación adicional.
Las cuantificaciones de ADN fueron realizadas en un espectrofotómetro UV (Perkin Elmer) a 260 y 280 nm. La pureza se determinó mediante el cálculo de la relación de absorbancias (A260/A280) para todas las muestras. El ADN resuspendido se diluyó a una concentración de 50 ng·μL-1 de agua destilada doblemente esterilizada. Las muestras, debidamente etiquetadas, se conservaron hasta su uso a una temperatura de -20 °C. La integridad del ADN de los individuos masculinos y femeninos en estudio, fue analizada mediante electroforesis horizontal (CONSORT), en geles al 0.8 % de agarosa. Se usó una solución amortiguadora TBE 0.5 x y se aplicó una corriente constante de 130 V durante 60 min. Los geles se tiñeron con bromuro de etidio (1 mg·mL-1) y las bandas se visualizaron en un transiluminador UV (CONSORT TFX-20M).
Optimización del sistema ISSR-PCR
La amplificación del ADN purificado se basó en el sistema ISSR-PCR. El ADN de quince individuos (masculino y femenino), previamente mezclados en "grupos", según el sexo, fue analizado mediante el método de análisis masal segregante (Michelmore et al., 1991). Cada una de las amplificaciones se llevó a cabo empleando 25 μL de una mezcla de reacción que contenía (1x amortiguador PCR, pH 8.3), 3.0 mM MgCl2, 0.4 mM dNTPs, 1.25U de Taq ADN polimerasa (INVITROGEN®) y 50 ng de ADN de los grupos de individuos masculinos y femeninos previamente formados. Se utilizó un conjunto de 6 iniciadores ISSRs (Cuadro 1), provenientes de la Universidad Columbia Británica, Canadá. Para la amplificación y estandarización de las condiciones de PCR. Los ISSRs fueron seleccionados en base al polimorfismo mostrado en otras especies vegetales (Pollegioni et al., 2006).
Para optimizar las condiciones de reacción se estudiaron dos variables: concentración de iniciadores y temperaturas de anillamiento, como se describe en el Cuadro 2.

Las reacciones de PCR se realizaron en un termociclador de ADN (marca eppendorf ®.AG PTC-200) bajo las siguientes condiciones térmicas: paso inicial de desnaturalización de 94 °C durante 7min, seguido por 40 ciclos de 30s a 94 °C, 60 s, a diferentes temperaturas de anillamiento, por cada iniciador (52-55 °C), 90 s a 72 °C durante 10 min. En todos los casos se incluyeron controles negativos. 7 μL de productos de PCR fueron sometidos a electroforesis en geles de agarosa al 2 % (w / v) en una solución amortiguadora TBE 0.5X a 100 V, durante 1 hora y finalmente fueron teñidos con bromuro de etidio (0.5 μgmL-1). Se utilizaron dos marcadores de peso molecular de 50 y 200pb (marca Axygen CA 94587).
Después de la electroforesis, los geles se visualizaron bajo un transiluminador UV y fueron registrados utilizando el Sistema de Documentación de Gel (marca CONSORT SP-TF12).
Se determinó el número y el peso molecular de cada banda ISSRs obtenida; sin embargo, para el análisis, sólo las bandas reproducibles y bien definidas fueron consideradas en este estudio.
RESULTADOS Y DISCUSIÓN
Extracción y detección de ADN
Para la extracción de ADN se siguió el método propuesto por Stewart y Vía (1993), basado en uso del CTAB. Se obtuvieron 0,060 g de ADN por gramo de tejido de hoja fresca. Sin embargo, el análisis de la absorbancia (A260/A280) del ADN que dio un valor de 1.4 evidenció la presencia de altos niveles de contaminación por proteínas, polisacáridos y fenoles. La muestra de ADN obtenida resultó muy viscosa denotando la presencia de polisacáridos muy similar a lo reportado por Yagi et al. (2002). Tras la electroforesis, los patrones de bandas obtenidos (Figura 1a), mostraron que el ADN estaba un poco degradado. No se visualizaron, mediante electroforesis, la presencia de bandas bien definidas. En la Figura 1 se muestra el resultado de la electroforesis del ADN genómico extraído en 5 individuos masculinos y femeninos, respectivamente, de C. mexicana.

Se evaluó la eficacia en términos de cantidad y calidad del ADN el protocolo de extracción de ADN de Stewart y Vía (1993) para C. mexicana y se perfeccionó. El protocolo modificado, incluyó un pre-tratamiento del ADN con una solución de 0.70 M of NaCl. Esto permitió obtener un ADN de alta pureza (libre de aceites esenciales, polifenoles, flavonoides y polisacáridos) a partir de muestras frescas de hoja. La pureza de las muestras de ADN fue confirmada por el valor de absorbancias (A260/A280) de 1.8 o más obtenido. Valores entre 1.8 - 2.0 de absorbancia denota generalmente que el ADN es de alta calidad. Las bandas, después de la electroforesis, fueron más nítidas (Figura 1b).
Sistema ISSR-PCR
Las dos condiciones evaluadas en las reacciones ISSRs fueron: la concentración de los iniciadores y la temperatura de anillamiento, ya que ambas tienen un efecto en los resultados de la amplificación y en los patrones y reproducibilidad de las bandas obtenidas. En el Cuadro 3 se muestran las condiciones optimizadas para detectar marcadores ISSR-PCR en C. mexicana.
El ADN extraído resultó adecuado para efectuar las amplificaciones por PCR. Se observó la presencia de bandas de alta intensidad al emplear distintas concentraciones del iniciador UBC 834 (Figura 2). Los resultados preliminares del análisis de los iniciadores mostraron que la concentración de 25μl de oligonucleótidos (UBC), podría ser suficiente para obtener bandas claras, distintivas y reproducibles, que pudiesen ser seleccionadas para estudios posteriores (Figura 2 y 3).
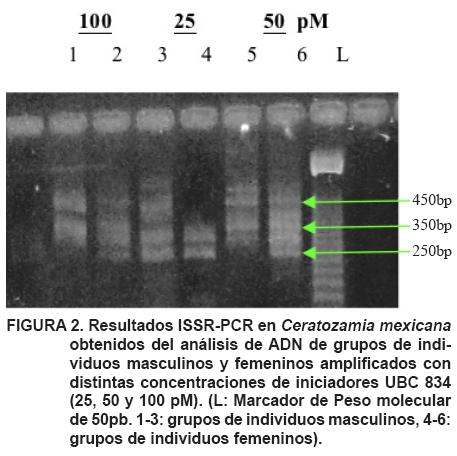

Un total de 23 bandas fueron amplificadas con los 6 iniciadores en estudio, de las cuales 9 fueron polimórficas (39,13 % de polimorfismo). Por cada iniciador se amplificaron en promedio 4.16 ± 2.13 bandas. Las bandas ISSRs detectadas en C. mexicana revelaron la presencia de fragmentos desde 200pb (UBC: 841 y 891) hasta 600 pb (UBC: 841 y 856). Los iniciadores UBC 841, 856 y 891 fueron los más polimórficos, mientras que los iniciadores UBC 890, 834 y 888 fueron monomórficos (Cuadro 1). No obstante no se puede generalizar que todos los individuos en los grupos examinados sean monomórficos para esos iniciadores.
Abunda en la literatura, protocolos de extracción de ADN en plantas que se han optimizado para obtener ADN de adecuada calidad (Dellaporta et al., 1983; Doyle y Doyle, 1990; Suman et al., 1999; Shah et al., 2000; Warude etal., 2003; Sarwat et al., 2006; Deshmukh et al., 2007) pero en la práctica estos procedimientos han sido empíricos, debido a la variabilidad en la composición de los tejidos vegetales. Las ventajas y desventajas de los diferentes métodos de extracción y purificación que se han utilizado en plantas han sido previamente discutidos (Aljanabi etal., 1999; Sharma et al., 2000). Aunque en cícadas se han evaluado distintos procedimientos de extracción de ADN, se encontró que el método basado en el uso del CTAB resulta el más efectivo en Cycas circinalis L. Miq. y en C.micholitzii Dyer (Gangopadhyay et al., 2007)
El proceso de extracción de ADN implica romper o digerir las paredes celulares para liberar los constituyentes celulares. A esto le sigue la lisis de las membranas celulares para liberar el ADN para su posterior amplificación. Los problemas encontrados en la extracción y purificación de ADN de alto peso molecular en ciertas especies vegetales conlleva la presencia de diferentes contaminantes. Entre ellos, los polisacáridos se encuentran entre los contaminantes más difíciles de separar del ADN (Murray y Thompson, 1980). Los polisacáridos pueden interferir con la actividad de varias enzimas tales como las polimerasas, ligasas y enzimas endonucleasas de restricción. La eliminación de los inhibidores de la polimerasa, tales como los polisacáridos, favorecen la amplificación por PCR del ADN (Lodhi et al., 1994).
El método de Stewart y Vía (1993) ha sido utilizado con éxito en la extracción del ADN de varias especies vegetales, como Pinus patula Schiede ex Schlechtendal et Chamisso (Luna-Rodríguez et al., 2005). Este procedimiento utiliza una mayor concentración (2 % p/v) de Polivinilpirrolidona (PVP) de elevado peso molecular es decir, (40000 a 2 % p/v). Este es un compuesto comúnmente utilizado por suprimir la oxidación polifenólica (Porebski et al., 1997) en protocolos de extracción de ADN basado en el uso del CTAB (Doyle y Doyle, 1990). La presencia de compuestos polifenólicos que se producen en muchas especies vegetales son también uno de los principales componentes bioactivos que se encuentran presentes en muchas de las especies de cícadas (Yagi et al., 2002; Peng-Quiao et al., 2008). En el presente estudio, se añadió suficiente PVP y ácido ascórbico en el momento de la homogenización del tejido. La adición de PVP, conjuntamente con el CTAB, puede contribuir a eliminar la presencia de compuestos fenólicos, mediante la formación de un complejo y hasta cierto punto, también pudo ayudar en la eliminación de otras impurezas. El tratamiento que repetidamente se realizó con una solución de cloroformo y alcohol isoamílico, garantizó asimismo la eliminación de la clorofila, y otros pigmentos colorantes, proteínas, etc. La degradación del ADN se evitó, en cierta medida, mediante la realización del procedimiento de extracción a 4 °C. Por lo tanto, el presente protocolo describe un método confiable, rápido, sencillo y consistente de extracción de ADN para la detección de marcadores moleculares (ISSR-PCR) en Ceratozamia mexicana.
La adición de concentraciones de NaCl a 0.7 M, junto con CTAB es un método conocido para eliminar polisacáridos (Murray y Thompson, 1980; Fang et al., 1992). Los rangos de concentración mencionados en la literatura varía entre 0.5 M (Clark, 1997) y 6 M (Aljanabi et al., 1999) y depende de la especie de planta bajo investigación. La mayoría de los polisacáridos se eliminan de manera eficaz una sola precipitación de sales en 0.5-2.5 M NaCI. El uso del procedimiento de Stewart y de la Via (1993) con 0,7 M de NaCl fue muy útil para obtener la mejor calidad y cantidad de ADN en hojas de C. mexicana. Los resultados del presente estudio mostraron métodos modificados de CTAB combinados con la adición de 0,7 M de NaCl para mejorar la lisis extrayendo un ADN de mayor calidad. Por lo tanto, se concluye que el protocolo actual describe un método de aislamiento de ADN fiable, rápido, sencillo y consistente para Ceratozamia mexicana. La simplicidad del procedimiento hace que sea muy práctico para la extracción de ADN del tejido de la hoja, especialmente para especies como las cícadas, en el que polisacáridos y polifenoles son un problema.
Después de obtener un ADN genómico total con una adecuada pureza y rendimiento, se llevó a cabo un sistema óptimo de PCR basado en cada cebador ISSR. En resumen, el ADN aislado por el método CTAB modificado generó productos de amplificaciones fuertes y fiables mostrando su compatibilidad para los 6 cebadores estudiados. El presente protocolo optimizado para la técnica ISSR puede servir como una herramienta eficaz para futuros estudios moleculares en genética de poblaciones. El presente estudio podría ayudar a establecer un programa eficaz de conservación para esta especie en peligro de extinción.
CONCLUSIONES
El sistema de extracción de ADN modificado del protocolo CTAB, muestra una buena calidad y cantidad de ADN, adecuado para realizar nuevos estudios en genética de poblaciones, evolución de las cícadas y sistemática, como muestran los resultados de la amplificación ISSR.
El NaCl facilita la lisis celular y optimiza la extracción de ADN por la liberación de los materiales nucleares y la precipitación de los polisacáridos, lo que mejora la calidad del extracto y por lo tanto, de los productos de amplificación.
Por último, este trabajo forma parte de un proyecto más amplio sobre la biología y la ecología poblacional de cícadas mexicanas, por lo que la importancia de este protocolo se encuentra en los estudios futuros hacia la conservación y el manejo de esta especie con importancia ornamental y ecológica.
AGRADECIMIENTOS
Este trabajo contó con apoyo por parte del Consejo Nacional de Ciencia y Tecnología (Proyectos Núm: 83156 y 152073). Los autores desean agradecer al Consejo Nacional de Ciencia y Tecnología por la ayuda otorgada y al Consejo Veracruzano de Ciencia y Tecnología, por el apoyo en el trabajo expuesto en el Congreso Forestal Mundial. Se agradece a Maricela Durán Sánchez por el apoyo en los análisis moleculares y a José Manuel Cabrera Hernández por sus comentarios y sugerencias.
LITERATURA CITADA
Aljanabi, S., Forget, L. & Dookun, A. (1999). An improved rapid protocol for the isolation of polysaccharide and polyphenol-free sugarcane DNA. Plant Molecular BiologicalReport, 17, 1-8. [ Links ]
Avise, J. C. (1994). Molecular Markers, Natural History, and Evolution. New York. Chapman & Hall. [ Links ]
Boiteux, L. S., Fonseca, M. & Simon, P. W. (1999). Effects of Plant Tissue and DNA Purification Method Randomly Amplified Polimorphic DNA-based Genetic Fingerprinting Analysis in Carrot. American Society of HorticultureScience, 124, 32-38. [ Links ]
Clark, M.S. (1997). Plant Molecular Biology, a Laboratory Manual. Berlin. Springer-Verlag. [ Links ]
Dellaporta, S. L., Wood, J. & Hocks, J. B. (1983). A plant DNA minipreparation: version II. Plant Molecular BiologyReport, 1, 19-21. [ Links ]
Deshmukh, A. S., Treeback, J. T., Long, Y. C., Viollet, B., Wojtaszewski, J., F. & Zierat, H. J. R. (2007). Role of adenosine 5'-monophosphate-activated protein kinase subunits in skeletal muscle mammalian target of rapamycin signaling. Molecular Endocrinology, 22(5), 1105-1112. [ Links ]
Doyle, J. J. & Doyle J. L. (1990). A rapid DNA isolation procedure from small quantities of fresh leaf tissues. Phytochemical Bulletin, 19, 11-15. [ Links ]
Fang, G., Hammar, S. & Rebecca, R. (1992). A quick and inexpensive method for removing polysaccharides from plant genomic DNA. Biotechniques, 13, 52-56. [ Links ]
Flachowsky, H., Schumann, E., Weber, W. E. & Peil, A. (2001). Application of AFLP for the detection of sex-specific markers in hemp. Plant Breeding, 120, 305-309. [ Links ]
González-Astorga, J., Vovides, A. P., Octavio-Aguilar, P., Aguirre-Fey, D., Nicolalde-Morejon, F. & Iglesias, C. (2006). Genetic diversity and structure of the cycad Zamialoddigesii Miq. (Zamiaceae): implications for evolution and conservation. Botanical Journal of the LinneanSociety, 152, 533-544. [ Links ]
Gangopadhyay, G., Roy, R. S., Ghose, K., Poddar, R., Bandyopadhyay, T., Basu, D. &, Mukherjee, K. K. (2007). Sex detection of Carica papaya and Cycas circinalis in pre-flowering stage by ISSR and RAPD. CurrentScience, 92(4), 524-526. [ Links ]
Jianguang, X., Shuguang, J. & Nian, L. (2005). Genetic variation in the endemic plant Cycas debaoensis on the basis of ISSR analysis. Australian Journal of Botany, 53, 141-145. [ Links ]
Katterman F. & Shattuck, V. L. (1983). An effective method of DNA isolation from the mature leaves of Gossypium species that contain large amounts of phenolic terpenoids and tannins. Preparative Biochemical, 13, 347-359. [ Links ]
Lodhi, M. A., Ye, G. N., Weeden, N. F. & Reisch, B. I. (1994). A simple and efficient method for DNA lysis from grapevine cultivars and Vitis species. Plant MolecularBiological Report, 12, 6-13. [ Links ]
Luna-Rodríguez, M., López-Upton, J. & Iglesias-Andreu L. G. (2005). Variabilidad morfométrica y molecular (RAPD) en una plantación de Pinus patula en Veracruz, México. Agrociencia, 39, 231-235. [ Links ]
Michelmore, R. W., Paran, I. & Kesseli, R. V. (1991). Identification of markers linked to disease resistance genes by bulked segregant analysis: A rapid method to detect markers in specific genome regions by using segregating populations. Proceedings of the National Academic ofScience, 88, 9828-9832. [ Links ]
Murray, M. G. & Thompson, W. F. (1980). Rapid isolation of high molecular weight DNA. Nucleic Acids Research, 8, 4321-4325. [ Links ]
Norstog, K. J. & Nicholls, T. J. (1997). The Biology of the Cycads. Ithaca, USA. Cornell University Press. [ Links ]
Octavio-Aguilar, P., González-Astorga,J. & Vovides, A. P. (2009). Genetic diversity through life history of Dioon edule Lindley (Zamiaceae, Cycadales). Plant Biology, 11, 525-536. [ Links ]
Prakash, S. & Staden, J. (2006). Sex identification in Encephalartosnatalensis (Dyer and Verdoorn) using RAPD markers. Euphytica, 152(2),197-200. [ Links ]
Peng-Quiao, M. O., Yu-Yuan, H., Xiao-Qing, Z., Guang-Lin, L., Zheng-Wen, L. & Bao-Xuan, N. (2008). Establishment of Cycas micholitzii ISSR-PCR optimal conditions with orthogonal optimization method. Bulletin of BotanicalResearch, 28(3), 304-309. [ Links ]
Pollegioni, P., Major, A., Bartoli, S., Ducci, F., Proietti, R., Malvolti, M. E. & Anazato, D., (2006). Application of microsatellite and dominant molecular markers for the discrimination of species and interspecific hybrids in genus Juglans. Acta Horticulture, 705, 191-197. [ Links ]
Porebski, S., Bailey, L. G. & Baum, B. R. (1997). Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components. PlantMolecular Biological Report, 15, 8-15. [ Links ]
Reddy, G S. N., Prakash, J. S. S., Matsumoto, G. I., Stackebrandt, E. & Shivaji, S. (2002). Arthrobacter roseus sp. nov., a psychrophilic bacterium isolated from an Antarctic cyanobacterial mat sample. International Journal ofSystematic Evolution and Microbiology, 52, 1017-1021. [ Links ]
Reamon-Büettner, S. M. & Jung, C. (2000). AFLP-derived STS markers for the identification of sex in Asparagusofficinalis L. Theoretical and Applied Genetics, 100, 432-438. [ Links ]
Sarwat, M., Negi, M. S., Lakshmikumaran, M. & Tyagi, A. K. (2006). A standardized protocol for genomic DNA isolation from Terminalia arjuna for genetic diversity analysis. Electronic Journal of Biotechnology, 9, 86-91. [ Links ]
SEMARNAT (2010). Protección Ambiental de Especies Nativasde México de Flora y Fauna Silvestre. Categorías deRiesgo y Especificaciones para su Inclusión, Exclusión o Cambio. Lista de Especies en Riesgo. México. Secretaria del Medio Ambiente y Recursos Naturales. [ Links ]
Shah, M. M., Yen, Y., Gill, K. S. & Baenzinger, P. S. (2000). Comparisons of RFLP and PCR-based markers to detect polymorphism between wheat cultivars. Euphytica, 114, 135-142. [ Links ]
Sharma, K. K., Lavanya, M. & Anjaiah, V. (2000). A Method for isolation and purification of peanut genomic DNA suitable for analytical applications. Plant MolecularBiology Reporter, 18, 393a-393h. [ Links ]
Stewart, C. N. & Via, L. E. (1993). A rapid CTAB DNA isolation technique useful for RAPD fingerprinting and other PCR applications. BioTechniques, 14, 748-751. [ Links ]
Suman, P. S. K., Ajit, K. S., Darokar, M. P. & Kumar, S. (1999). Rapid isolation of DNA from dry and fresh samples of plants producing large amounts of secondary metabolites and essential oils. Plant Molecular Biology Report, 17, 1-7. [ Links ]
Vovides, A. P. (1983). Zamiaceae. In: V. Sosa, ed., Flora de Veracruz, Fasc. 26, 29. Xalapa, México. Instituto Nacionalde Recursos Bióticos. [ Links ]
Vovides, A. P. & Iglesias, C. G. (1994). An intregrated conservation strategy for the cycad Dioon edule Lindl. Biodiversityand Conservation, 3, 137-141. [ Links ]
Warude, D., Chavan, P., Joshi, K. & Patwardhan, B. (2003). DNA isolation from fresh, dry plant samples with highly acidic tissue extracts. Plant Molecular Biological Report, 21, 467. [ Links ]
Yagi, F., Iwaya, T., Haraguchi, T. & Goldstein, I. J. (2002). The lectin from leaves of Japanese cycad, Cycas revoluta Thunb. (Gymnosperm) is a member of the jacalin-related family. European Journal Biochemistry, 269, 4335-4341. [ Links ]
Zietkiewicz, E., Rafalaski, A. & Labuda, D. (1994). Genome fingerprinting by simple sequence repeat (SSR) anchored polymerase chain reaction amplification. Genomics, 20, 176-183. [ Links ]














