Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista bio ciencias
versión On-line ISSN 2007-3380
Revista bio ciencias vol.7 Tepic 2020 Epub 18-Nov-2020
https://doi.org/10.15741/revbio.07.e762
Artículos originales
Diferentes niveles de expresión de ALDH1A1 y DYRK1B en pacientes con glioblastoma multiforme y su tiempo de sobrevida
1Centro Oncológico Estatal ISSEMyM, Unidad de Investigación Básica y Aplicada en Salud, Av. Solidaridad Las Torres No. 101. Toluca, Estado de México 50180, México.
2Centro Oncológico Estatal ISSEMyM, Departamento de Patología. Av. Solidaridad Las Torres No. 101. Toluca, Estado de México 50180, México.
El glioblastoma multiforme es la forma más maligna de astrocitoma del sistema nervioso central. La mediana de supervivencia global con glioblastoma es de 12 a 16 meses después de la cirugía de resección, la quimioterapia y el tratamiento adyuvante; sin embargo, aún no está claro qué factores moleculares están involucrados en la determinación de la supervivencia global. Informes recientes muestran la isoforma A1A de aldehído deshidrogenasa (ALDH1A1), una proteína que participa en los procesos metabólicos celulares, como un biomarcador potencial en glioblastoma. En este estudio, identificamos y evaluamos la presencia de ALDH1A1 en biopsias de glioblastoma de pacientes mexicanos mediante inmunohistoquímica, reacción en cadena de la polimerasa por retrotranscripción y análisis de espectroscopía de masas. Por otro lado, también analizamos mediante pruebas inmunohistoquímicas y western blot la presencia de DYRK1B, una quinasa implicada en la regulación de la progresión tumoral y en la regulación del ciclo celular.
El propósito de este estudio fue investigar si la expresión histológica, así como el patrón de expresión de ambas proteínas (ALDH1A1 y DIRK1B) tuvieron una correlación relevante con la supervivencia general de los pacientes diagnosticados con glioblastoma. Encontramos diferentes patrones de expresión y observamos que tanto ALDH1A1 como DYRK1B tienen patrones de expresión muy similares entre sí y ambas mostraron una importante correlación con la supervivencia general de los pacientes. Estos hallazgos nos permiten considerar que los niveles de expresión de ALDH1A1 y DYRK1B pueden ser posibles factores pronósticos en el diagnóstico o posibles biomarcadores para el tiempo de supervivencia en pacientes con glioblastoma.
Palabras clave: Glioblastoma; ALDH1A1; DYRK1B; tiempo de supervivencia; inmunohistoquímica; espectrometría de masas
Glioblastoma multiforme is the most malignant form of central nervous system astrocytoma. The overall survival median in patients with glioblastoma is 12 to 16 months after undergoing resection surgery, chemo radiotherapy and adjuvant treatment; However, it is still unclear which molecular factors are involved in the determination of overall survival. Recent reports pointed out a specific isoform of aldehyde dehydrogenase (ALDH1A1), a protein participating in cellular metabolic processes, as a potential biomarker in glioblastoma. In this study, the expression of ALDH1A1 in biopsies of glioblastoma from Mexican patients was identified and evaluated using immunohistochemical dyes, reverse transcription polymerase chain reaction (RT-PCR) and mass spectroscopy analyses. On the other hand, the presence of DYRK1B, a kinase involved in the regulation of tumor progression and in the regulation of the cell cycle was also analyzed using immunohistochemical detection and western blot. The purpose of this study was to investigate whether the histological expression, as well as the expression pattern of both proteins (ALDH1A1 and DIRK1B) presented a significant correlation with the overall survival in patients diagnosed with glioblastoma. Different expression patterns were found and both ALDH1A1 and DYRK1B showed similar expression patterns and an important correlation with the overall survival of patients. These findings allowed to consider the expression levels of ALDH1A1 and DYRK1B as possible prognostic factors in the diagnosis or possible biomarkers for survival time in patients with glioblastoma.
Key words: Glioblastoma; ALDH1A1; DYRK1B; survival time; immunohistochemistry; mass spectrometry
Introducción
El glioblastoma multiforme (GBM) es el tumor más maligno de los astrocitomas, y es clasificado por la Organización Mundial de la Salud como grado IV (Natsume A., 2008; Van Den Bent et al., 2017). También tiene el peor pronóstico de las neoplasias malignas del sistema nervioso central (Louis et al., 2001; Louis et al., 2016; Ostrom et al., 2017; Park et al., 2017), y la mediana de la supervivencia para el Glioblastoma reportada es solo de 12 a 15 meses (Wen & Kesari, 2008;(Stupp & Roila, 2009). Sin embargo, no está claro, qué mecanismos moleculares podrían estar relacionados con la tasa de supervivencia de los pacientes. Se ha demostrado que las proteínas involucradas en los principales procesos metabólicos celulares, pueden ser alteradas en enfermedades como el cáncer, especialmente para aquellos procesos involucrados en la promoción de la proliferación y la supervivencia. La familia de enzimas aldehído deshidrogenasa (ALDH) está compuesta por más de 12 isoformas con sub-isoformas de enzimas citosólicas y mitocondriales (Jelski et al., 2014; Tomita et al., 2016; Visus et al., 2007), todas con pesos moleculares de aproximadamente 55 kDa. Su función principal es la catálisis de la oxidación de aldehídos. Se ha demostrado que esta familia está involucrada en diferentes procesos metabólicos (Alexandra et al., 2012), incluida la oxidación a través del procesamiento metabólico de los ácidos carboxílicos (Jelski et al., 2014).
ALDH1A1 es una isoforma citosólica, que se encuentra abundantemente en el hígado y participa en la vía de señalización del metabolismo del alcohol, junto con alcohol deshidrogenasa. Otra función importante de ALDH1A1 es la conversión de retinol a ácido retinoico (Tomita et al., 2016), y estudios recientes sugieren que esto podría influir en los procesos de diferenciación y actuar como biomarcadores de diagnóstico en cáncer de pulmón y glioblastoma (Alexandra et al., 2012; Arnold et al., 2015; Cheng et al., 2016; Kalantari et al., 2016; Rasper et al., 2010; Wang, Wang, & Chen, 2018; Zhang et al., 2011). Otros investigadores han propuesto que la expresión de ALDH1A1 contribuye a la progresión del glioma, donde la sobreexpresión fue encontrada en gliomas de alto grado en comparación con el resto de los grados de gliomas (Xu et al., 2015). Mientras que en otro estudio se reportó que ALDH1A1 es un mal pronostico, de acuerdo con la expresión de ALDH1A1 en GBM (Schäfer et al., 2012). También se ha reportado que la DYRK1B (dual specificity tyrosine phosphorylation regulated kinase 1B) (Ashford et al., 2014; Chen et al., 2017; Gao et al., 2009; Jin et al., 2007) participa en la regulación del ciclo celular (Gao et al., 2009) y su expresión génica puede verse alterada en células tumorales (Pérez-Sánchez et al., 2018), por ejemplo, siendo sobreexpresada en algunos tipos de cáncer humano, tales como cáncer de pulmón de células no pequeñas, adenocarcinoma ductal pancreático, carcinoma de colon, cáncer cervical, cáncer de ovario (Chen et al., 2017; Deng et al., 2006) y glioblastoma (Zhou et al., 2017). El propósito de este estudio fue investigar si la expresión proteica de ALDH1A1 y DYRK1B tiene alguna correlación con el diagnóstico de GBM o con los tiempos de supervivencia con respecto a sus patrones de expresión.
Material y Métodos
Ensayos de viabilidad celular
La línea celular U87 MG derivada de cerebro humano (glioblastoma/astrocitoma) fue cultivada en DMEM-HG (Dulbecco’s Modified Eagle Medium High- Glucose, Gibco by Life Technologies), suplementada con 10 % de suero fetal bovino (Gibco by Life Technologies), 1 % L-glutamina (Sigma-Aldrich Cat # G7513), 100 U/mL penicilina y 100 mg/mL de estreptomicina (Gibco by Life Technologies), y se mantuvo a 37 °C en una atmósfera de 95 % de aire y 5 % de CO2. La viabilidad celular se determinó mediante la técnica de exclusión de azul Tripano (Strober, 2015) (Invitrogen Cat # 15250061, USA) 48 h después de la exposición con ALDH1A1 en diferentes concentraciones (Invitrogen Cat # 11388H07E25, USA), los resultados fueron analizados por ANOVA de una vía, seguida de la prueba de comparación múltiple de Tukey.
Biopsias humanas
Este estudio fue aprobado por el Comité de Ética en Investigación del Centro Oncológico Estatal (COE) ISSEMyM, Estado de México bajo el protocolo número: COE/UEI/PG/14/2017. No se requirieron consentimientos informados, debido a la naturaleza retrospectiva del estudio. Los datos sobre la historia y evolución de los pacientes se obtuvieron de los registros médicos del hospital y las muestras se obtuvieron de bloques de parafina. Diez muestras de pacientes con glioblastoma y una muestra de cada grado de glioma; GI, GII y GIII fueron utilizadas. Todos los diagnósticos fueron confirmados por imágenes y por el departamento de patología del COE. Todos los pacientes con glioblastoma considerados en este estudio fueron monitoreados desde el comienzo de su tratamiento (cirugía y radioterapia) y hasta su fallecimiento.
Inmunohistoquímica
Las secciones de tejido de cerebro sano, hígado sano y carcinoma ductal mamario infiltrante fueron amablemente donados por el departamento de patología del COE y se utilizaron como tejidos control. Los portaobjetos se bloquearon con 1 % de peróxido (Sigma) durante 5 minutos a temperatura ambiente, se lavaron con solución salina tamponada con fosfato (PBS) y se incubaron durante toda la noche a 4 ºC en presencia de anticuerpos para ALDH1A1 (D9J7R) XP (Cell Signaling, Danver, MA, USA, Cat. # 36671S 1: 400) o DYRK1B (Sigma Aldrich, Cat # HPA028786, 1: 400). Posteriormente, las muestras se lavaron con PBS y se incubaron a temperatura ambiente durante una hora con el anticuerpo secundario, usando un kit Brown/Rabbit ImmunoDetector DAB HRP Brown (Cat. # BSB 0003) de acuerdo con las instrucciones del fabricante. Las secciones se contra tiñeron con hematoxilina Harris (Sigma-Aldrich, HHS32-1L) y se montaron usando medios de montaje DPX. Los portaobjetos se examinaron con un microscopio óptico (Leica DM500). Las imágenes se obtuvieron con el software de imágenes de microscopios Leica Microsystems, con una amplificación de 40X. Diez campos independientes fueron contados y analizados por medio de un ANOVA de una vía seguido de la prueba de comparación múltiple de Newman-Keuls, y los gráficos se realizaron utilizando el software GraphPad Prisma.
Espectrometría de masas
Para detectar la presencia de ALDH1A1 mediante ionización por electrospray, se utilizó el análisis de espectrometría de masas por infusión directa (ESI-MS/MS) (Arnold et al., 2015; Kirwan et al., 2014). Las muestras de péptidos se analizaron usando un sistema de espectrometría de masas QTRAP 4500 (Sciex, San Francisco, CA) con una NanoSource. Las muestras se desparafinaron e incubaron en un buffer de extracción utilizando el kit Qproteome FFPE Tissue Kit (QIAGEN, USA Cat. # 37623) de acuerdo con las instrucciones del fabricante. Las proteínas totales se aislaron y se procesaron para la generación de péptidos. Brevemente, el extracto total de proteínas se redujo/alquiló y digirió con tripsina (Promega, Cat. # V5111). Los péptidos resultantes fueron extraídos, desalados con un sistema ZipipC-18 (Millipore, Cat. # ZTC18S096) y resuspendidos en 20 % acetonitrilo y 0.1 % ácido fórmico (Aebersold & Mann, 2003; Megger et al., 2017) La proteína ALDH1A1 humana recombinante (Invitrogen Cat. # 11388H07E25) se usó como control y recibió el mismo tratamiento de preparación.
La distribución de las moléculas en Q1 se analizó a una velocidad de exploración de 100 a 1000 m/z, Da, con 4500 Volts/150 °C, luego los iones para el análisis en MS3 fueron seleccionados usando un de-clustering potential de 120 y una energía de colisión de 20 (Arnold et al., 2015). La búsqueda en la base de datos para la identificación de proteínas se realizó utilizando el buscador de iones “Mascot MS/MS ion search” (Matrix Science: disponible en http://www.matrixscience.com) con la base de datos AA Swiss-Prot y NCBInr para Homo sapiens (humano). Se utilizaron tolerancias de escisión de ± 1.2 Da para el ion precursor y ± 0.6 Da para las masas de los fragmentos de iones. Se consideraron los valores de masa monoisotópica y los valores no restringidos de masa. Adicionalmente, las secuencias de péptidos coincidentes se analizaron utilizando el software MS-BLAST software (Washington University, Saint Louis, MO; disponible en http://genetics.bwh.harvard.edu/msblast/) contra las bases de datos Swiss-Prot and NCBInr para confirmar la coincidencia de los péptidos.
Imagen
Se tomaron imágenes representativas de 3 pacientes con glioblastoma multiforme de su registro clínico del hospital.
Transcripción inversa y reacción en cadena de la polimerasa (RT-PCR)
La presencia de ALDH1A1 se evaluó adicionalmente utilizando RT-PCR. Para la extracción de RNA, las muestras de biopsia se desparafinaron (Bonin & Stanta, 2013) y se lavaron dos veces en PBS frío, pH 6.8. Después se extrajo el RNA total utilizando el reactivo TRIzol (Invitrogen, Cat # 15596026. USA) siguiendo las instrucciones del fabricante. La concentración de RNA fue medida por densidad óptica utilizando la absorbancia de 260 nm en un espectrómetro de luz Multiskan GO (Thermo Scientific). La calidad del RNA fue verificada por electroforesis en geles de agarosa con ayuda de un sistema de imagen BioDoc-UVP, así como por la relación 260/280 nm. La RT-PCR se realizó utilizando 5 μg de RNA el cual fue tratado con DNAsa (New England BioLabs, Cat # M0303S) y utilizando la transcriptasa reversa MMLV (Invitrogen, Cat # 28025013, USA), siguiendo las instrucciones del fabricante. Para amplificar ALDH1A1, se usaron los siguientes cebadores: forward 5’CCG TGG CGT ACT ATG GAT GC-3´ y reverse 5´-CGC AAT GTT TTG ATG CAG CCT-3’, para β-Actina, los cebadores utilizados fueron: forward 5’-TGG CACC ACA CCT TCT ACA-3’ y reverse 5’-TCA CGC ACG ATT TCC C-3’.
Ensayos de Western blot
La extracción de proteína total se realizó usando un kit de buffer de lisis para tejido Qproteome FFPE Tissue Kit (QIAGEN, USA Cat. # 37623) siguiendo las instrucciones del fabricante. Muestras con 50 μg de contenido de proteína total fueron separadas en geles de SDS-PAGE al 12 % y transferidas en membranas de difluoruro de polivinilideno (Bio-Rad, Hercules, CA, USA). Las membranas fueron bloqueadas por 1 h a temperatura ambiente con leche descremada al 5 %, buffer Tris-salino con 0.1 % Tween-20, y luego se incubaron a 4 °C toda la noche con el anticuerpo primario contra ALDH1A1 (D9J7R) XP (Cell Signaling, Danver, MA, USA, Cat. # 36671S 1:1000) o DYRK1B humano (Sigma Aldrich, Cat # HPA028786, 1:1000). Las trasferencias se lavaron e incubaron con el anticuerpo secundario HRP-Cabra/anti-conejo (Invitrogen Life Technologies, Cat. #656120, 1:5000). Como control positivo, las membranas fueron desnudadas y se volvieron a probar con el anticuerpo monoclonal anti-β-actina (Sigma-Aldrich, Cat. # A3854, 1:1000) y luego con un anti-ratón de cabra (Invitrogen, Cat.#62-6520, 1:5000). Los anticuerpos secundarios se acoplaron con peroxidasa (Life Technologies, Rockford, IL, USA) y las proteínas se revelaron con sustrato de quimioluminiscencia mejorada Western Lightning® Plus-ECL, (Perki-nElmer, Inc., Waltham, MA, USA). Las imágenes de las películas se adquirieron digitalmente con un sistema de imagen BioDoc-It (UVP).
Resultados y Discusión
Identificación de ALDH1A1 en tejido de glio-blastoma
En primer lugar, se revisó la historia clínica de los pacientes (Tabla 1), así como las imágenes tomográficas (Figura 1 a-c) de los glioblastomas. Se caracterizaron las biopsias de tejido, y la presencia de ALDH1A1 se evaluó adicionalmente usando RT-PCR (Figura 1 d) y espectrometría de masas (Figura 2).
Tabla 1 Características clínicas del paciente con GBM.
| Gioma grade |
Age (years) |
Sex | Medical records | Tumo location | RT cycles |
CT | Time survival (months) |
|||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Male | Female | DM | HT | Ht | SMOH | ALD | Fro | Par | Occ | Right | Left | |||||
| GBM | 29 | + | + | + | + | 30 | Tmz,Bvz | 47 | ||||||||
| GBM | 42 | + | + | + | + | + | + | 30 | Tmz | 8 | ||||||
| GBM | 47 | + | + | + | + | + | + | + | 25 | Tmz | 6 | |||||
| GBM | 59 | + | + | + | + | + | + | + | + | 34 | Tmz | 15 | ||||
| GBM | 60 | + | + | + | + | 0 | neither | 1 | ||||||||
| GBM | 60 | + | + | + | + | + | + | 0 | neither | 1 | ||||||
| GBM | 61 | + | + | + | + | + | + | + | 30 | Tmz,Cbp | 3 | |||||
| GBM | 62 | + | + | + | + | + | 22 | Tmz | 2 | |||||||
| GBM | 65 | + | + | + | + | + | + | 32 | Tmz | 4 | ||||||
| GBM | 72 | + | + | + | + | + | + | + | 29 | Tmz | 3 | |||||
DM= Diabetes mellitus; HT= Hipertiroidismo; Ht= Hipertensión; SMOH= Fumador; ALD= Alcohólico. Fro=Frontal; Par= Parietal; Occ= Occipital. Tmz= Temozolamida; Bvz= Bevacizumab; Cbp= Carboplatino.

Figura 1 Glioblastoma (GBM). Imágenes representativas de resonancia magnética de tres pacientes diferentes con GBM. Las imágenes muestran tumores únicos con necrosis central y refuerzo periférico asociado con el medio de contraste de Gadolinio (GD) asociado con un extenso edema de anillo periférico; a) Tumor en corte axial. b) Tumor en corte coronal y en c) Tumor en corte parasagital. d) Análisis de RT-PCR específico para aldh1a1 y β-actina en las muestras del paciente con GBM, como control negativo no se agregó DNA en el ensayo.

Figura 2 ALDH1A1 detectado en tejido de glioblastoma. a) Un espectro de masas representativo del ion precursor 796.3 m/z de ALDH1A1 en MS3 usando un método de péptidos basado en ESI-MS/MS. El ion precursor corresponde al péptido ANNTFYSAGVFTK, esta secuencia fue analizada por Blastnucleotide y correspondió a la cadena alfa de la aldehído deshidrogenasa (https://blast.ncbi.nlm.nih.gov). b) Parámetros espectrométricos de masas utilizados para la detección de ALDH1A1.
Efecto de ALDH1A1 en el crecimiento celular de células de glioblastoma U87-MG
Para evaluar el efecto de ALDH1A1 sobre la viabilidad celular, células de glioblastoma humano U87-MG fueron tratadas con diferentes concentraciones de proteína ALDH1A1 recombinante purificada (Figura 3). Los cultivos se evaluaron a las 48 horas después del tratamiento, se cuantificó el número de células viables usando el ensayo de exclusión de azul Tripano. El vehículo sin proteína se utilizó como control. Se observó que ALDH1A1 inducía un aumento en el número de células viables debido al aumento en la proliferación, de una manera dependiente de la concentración. Se cree que el aumento en la viabilidad celular está relacionado con los procesos metabólicos celulares, ya que otros autores también lo han informado (Tomita et al., 2016; Yue et al., 2015) con respecto a la participación de ALDH1A1 en el proceso de crecimiento celular y proliferación.
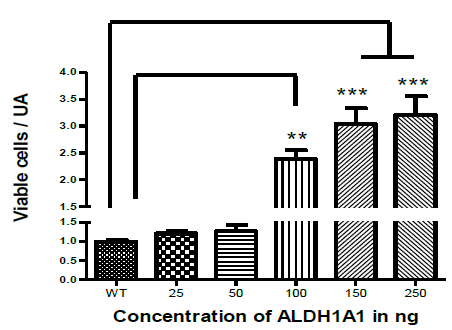
Figura 3 ALDH1A1 aumenta la viabilidad celular en células U87-MG de glioblastoma. ALDH1A1 indujo un aumento en el número de células viables de una manera dependiente de la concentración. Efecto de ALDH1A1 en la viabilidad celular en células U87-MG. La gráfica muestra unidades arbitrarias de la células U87-MG viables, determinada por exclusión del azul Tripano a las 48 horas después de tratarse con diferentes concentraciones de ALDH1A1 purificada recombinante. Los controles fueron tratados con el vehículo sin proteína. Las concentraciones de 25 y 50 ng/μL no mostraron diferencia significativa en comparación con el grupo control. El control es diferente de los grupos; 100 ng/µL, 150 ng/µL y 250 ng/µL. La concentración de 250 ng/µL es diferente en comparación con el grupo control, pero no lo es para la concentración de 150 ng/µL o 100 ng/µL. ANOVA de una vía seguido de la prueba de comparación múltiple de Tukey. **p<0.001, ***p<0.0001. Las barras son medias + S.D., n=3.
Tejidos de glioblastoma presentaron inmunoreactividad a ALDH1A1 y DYRK1B
La expresión de ALDH1A1 y DYRK1B se evaluó en los tejidos de glioblastoma mediante tinción inmunohistoquímica. Se observaron diferentes patrones de expresión y se clasificaron según su intensidad en las células tumorales (Figura 3, 4). Se establecieron tres categorías: focal, débil disperso y negativo en neoplasia (pero positiva en endotelio). DYRK1B también mostró inmunorreactividad en tejido de glioblastoma, y su patrón de expresión era muy similar al de ALDH1A1, se observaron varianzas iguales entre ALDH1A1 y DYRK1B, (p=0.2881), por lo que se utilizó la misma clasificación. Con las observaciones anteriores, la expresión de ALDH1A1 y DYRK1B fue evaluada en gliomas de bajo grado (Figura 4) por inmunohistoquímica. Interesantemente, en el grado I fue negativo; en el grado II mostró un focal débil, mientras que en el grado III se observó una expresión débil disperso. Se cree que estas diferencias en los patrones de expresión en el glioblastoma (Figura 5) pueden tener alguna relación con el progreso de la enfermedad, y es importante continuar investigando. La expresión de ALDH1A1 y DYRK1B también se observó en las muestras de tumores mediante la técnica de Western blot (Figura 6 a-b), en el grupo focal, la señal fue más intensa con respecto a la débil disperso y neoplasia negativa en comparación con la muestra de cerebro sano, mientras que para DYRK1B la señal fue más intensa en focal y débil disperso en comparación con la muestra de cerebro sano.

Figura 4 Expresión de ALDH1A1 y DYRK1B en gliomas. Imágenes representativas de ensayos inmunohistoquímicos para ALDH1A1 y DYRK1B en diferentes grados de gliomas. Para ALDH1A1; a) Cerebro sano, b) Hígado sano como control positivo, c) Glioma Grado I (GI), d) Glioma Grado II (GII), d) Glioma Grado III (GIII). Para DYRK1B; f) Cerebro sano., g) Carcinoma ductal invasivo de mama como control positivo, h) Glioma Grado I (GI), i) Glioma Grado II (GII), j) Glioma Grado III (GIII). Imágenes en magnificación 40X.

Figura 5 Patrones de expresión de ALDH1A1 y DYRK1B en Glioblastoma. Imágenes representativas de ensayos inmunohistoquímicos para ALDH1A1 y DYRK1B. Se usó tejido cerebral humano sano como control negativo para ALDH1A1 y DYRK1B, se utilizó tejido hepático sano como control positivo para ALDH1A1 y se utilizó tejido de carcinoma ductal invasivo como control positivo para DYRK1B. Se observaron tres patrones de expresión diferentes en los tejidos de pacientes con tejido de glioblastoma; focal, débil disperso y negativo en neoplasia (pero positivo en endotelio). Imágenes en magnificación 40X.

Figure 6 Expresión de ALDH1A1 y DYRK1B en tejidos de Glioblastoma y tiempo de supervivencia en pacientes con Glioblastoma. Una imagen representativa de Western blot. a-b) Muestra la expresión de ALDH1A1, DIRK1B y como control β-actina, determinado por Western blot. c-d) Porcentaje de células positivas para ALDH1A1 y DYRK1B con respecto a su patrón de expresión observado. ANOVA de una vía seguido de la prueba de comparación múltiple de Tukey. e) Gráfica del tiempo de supervivencia con respecto al patrón de expresión de ALDH1A y DIYRK1B. Kaplan-Meier, n=10.
Los patrones de expresión de DYRK1B y ALDH1A1 tienen una correlación significativa con la supervivencia general
El número de células positivas por campo para ALDH1A1 y DYRK1B se cuantificó, analizó y evaluó frente a la supervivencia general por paciente (Figura 6e). Para cada paciente, el tropismo entre ALDH1A1 y DYRK1B en las muestras fue equivalente. Debido a que los patrones de inmunoreactividad mostraron resultados interesantes, se analizó la correlación de la cantidad de células positivas vs el tiempo de supervivencia con un valor de -0.874, por lo que se cree que esta tendencia juega un papel importante en el desarrollo de la enfermedad. La mayor supervivencia global se observó en la clasificación de expresión de neoplasia negativa, mientras que la clasificación de expresión débil disperso tuvo la más baja.
Discusión
El glioblastoma multiforme es una enfermedad con un pronóstico particularmente pobre y una supervivencia general corta. Se ha demostrado que las proteínas involucradas en los principales procesos metabólicos de las células pueden alterarse en enfermedades como el cáncer, especialmente en aquellas involucradas en procesos que promueven la proliferación y la supervivencia. La presencia de DYRK1B en el glioblastoma, que se ha caracterizado en este tipo específico de cáncer (Vilenchik et al., 2019), y que han sido encontradas de manera estable en muestras clínicas, ha mostrado un patrón de expresión similar a ALDH1A1, y una correlación inversa con la supervivencia general de los pacientes en este estudio, aunque existe controversia con respecto a la presencia de ALDH1A1 y la supervivencia, como Adam et al. 2012 que describieron ALDH1A1 como un marcador de diferenciación astrocítica durante el desarrollo cerebral y un mejor marcador de pronóstico en pacientes que sufren glioblastoma primario. Con suerte, esto permitirá que surjan nuevas líneas de investigación sobre las vías de señalización en el glioblastoma, con la esperanza de dilucidar los mecanismos en los que convergen las vías para la señalización del ciclo celular y apoptosis. Hay otros informes, como Rasper et al. 2010 donde informan que se encontraron células ALDH1+ cerca de los vasos tumorales o alrededor de áreas necróticas, hipotetizando que las células madre tumorales de GBM se localizan predominantemente en la proximidad de los vasos tumorales que muestran una estrecha interacción con células endoteliales, también Tomita et al. 2016 indicaron que ALDH1A es un nuevo marcador de células madre y está presente en diferentes tipos de cáncer. Se han planteado muchas preguntas al respecto, y es necesario continuar investigando las consecuencias de la sobreexpresión de DYRK1B en el glioblastoma y las diferencias en los patrones histológicos de expresión. La relación entre la expresión de ALDH1A1 y el pronóstico clínico en diferentes grados de glioma (Alexandra et al., 2012; Liu et al., 2017; Xu et al., 2015) y entre pacientes con GBM es cada vez más evidente. En conjunto, la alta expresión de ALDH1A1 se propone como pronóstico pobre. Además, ALDH1A1 se considera un objetivo prometedor para mejorar el tratamiento de GBM de acuerdo con su papel previo como mediador de la resistencia a la temozolomida en pacientes con GBM (Schäfer et al., 2012), lo que requiere un mayor número de investigaciones de su comportamiento a nivel molecular. Por otro lado, aunque ALDH1A1 ha sido reportado en varios tipos de cánceres (Gong et al., 2014), incluido el glioblastoma, también es importante que investiguemos más el por qué ocurren estos diferentes patrones de expresión. Los análisis de correlación de los patrones de expresión de ALDH1A1 y DYRK1B vs la supervivencia general dejan muchas preguntas sin respuesta. Se cree que DYRK1B, así como ALDH1A1, tienen una participación importante en la progresión de la enfermedad y deben estudiarse más a fondo en GBM, dado su potencial como marcadores pronósticos (Gong et al., 2014; Rasper et al., 2010) o nuevos objetivos terapéuticos. Aunque ALDH1A1 y DYRK1B (Friedman, 2013; He et al., 2018; Huangfu & Anderson, 2005; Olmez et al., 2015) son proteínas completamente diferentes, es necesario realizar más estudios para determinar las vías en las que están involucrados, y sí hay alguna otra que compartan. Una perspectiva que tenemos para el futuro es cuantificar los niveles de expresión de ALDH1A1 con respecto a DYRK1B usando qPCR, y evaluar cómo esto se correlaciona con la activación de otras quinasas dependientes del crecimiento activas en el sistema neurológico, tal como Akt, Erk, o ciclina D1. Además, determinar los niveles de expresión de ALDH1A1 y DYRK1B en plasma en comparación con los niveles encontrados en el tejido tumoral ayudaría a evaluar si las interrupciones en la expresión de estos dos genes pueden detectarse en etapas más tempranas de la enfermedad a través de exámenes de sangre periférica. El hallazgo en este proyecto permitió observar la detección de DYRK1B en el glioblastoma, con diferentes patrones de expresión y un comportamiento de expresión que se parece mucho al observado en ALDH1A1, así como la tendencia que tienen estos patrones con respecto al tiempo de supervivencia.
Conclusión
El hallazgo más relevante en este proyecto fue la detección de DYRK1B en GBM, con diferentes patrones de expresión y un comportamiento de expresión que se parece mucho al observado para ALDH1A1. Estos hallazgos permiten considerar que los niveles de expresión de ALDH1A1 y DYRK1B pueden ser factores pronósticos en el diagnóstico o posibles biomarcadores para la supervivencia general en pacientes con glioblastoma.
Agradecimientos
Este trabajo fue apoyado por el Instituto de Seguridad Social del Estado de México y Municipios (ISSEMyM), el Consejo Mexiquense de Ciencia y Tecnología (COMECyT) y la Universidad Autónoma del Estado de México (UAEMex), beca número 848-2007. Queremos agradecer a Andrés Reyes y Verónica Fabela por la asistencia de laboratorio.
REFERENCIAS
Aebersold, R. & Mann, M. (2003). Mass spectrometry-based proteomics. Nature, 422(6928), 198-207. https://doi.org/10.1038/nature01511 [ Links ]
Alexandra Adam, S., Schnell, O., Pöschl, J., Eigenbrod, S., Kretzschmar, H. A., Tonn, J. C. and Schüller, U. (2012). ALDH1A1 is a marker of astrocytic differentiation during brain development and correlates with better survival in glioblastoma patients. Brain Pathology, 22(6), 788-797. https://doi.org/10.1111/j.1750-3639.2012.00592.x [ Links ]
Arnold, S. L., Kent, T., Hogarth, C. A., Schlatt, S., Prasad, B., Haenisch, M. and Isoherranen, N. (2015). Importance of ALDH1A enzymes in determining human testicular retinoic acid concentrations. Journal of Lipid Research, 56(2), 342-357. https://doi.org/10.1194/jlr.M054718 [ Links ]
Ashford, A. L., Oxley, D., Kettle, J., Hudson, K., Guichard, S., Cook, S. J. and Lochhead, P. A. (2014). A novel DYRK1B inhibitor AZ191 demonstrates that DYRK1B acts independently of GSK3β to phosphorylate cyclin D1 at Thr 286 , not Thr 288. Biochemical Journal, 457(1), 43-56. https://doi.org/10.1042/BJ20130461 [ Links ]
Bonin, S. & Stanta, G. (2013). Nucleic acid extraction methods from fixed and paraffin-embedded tissues in cancer diagnostics. Expert Review of Molecular Diagnostics, 13, 271-282. https://doi.org/10.1586/erm.13.14 [ Links ]
Chen, Y., Wang, S., He, Z., Sun, F., Huang, Y., Ni, Q. and Cheng, C. (2017). DYRK1B overexpression is associated with breast cancer growth and a poor prognosis. Human Pathology, 66, 48-58. https://doi.org/10.1016/j.humpath.2017.02.033 [ Links ]
Cheng, P., Wang, J., Waghmare, I., Sartini, S., Coviello, V., Zhang, Z. and Nakano, I. (2016). FOXD1-ALDH1A3 signaling is a determinant for the self-renewal and tumorigenicity of mesenchymal glioma stem cells. Cancer Research, 76(24), 7219-7230. https://doi.org/10.1158/0008-5472.CAN-15-2860 [ Links ]
Deng, X., Ewton, D. Z., Li, S., Naqvi, A., Mercer, S. E., Landas, S. and Friedman, E. (2006). The kinase Mirk/DYRK1B mediates cell survival in pancreatic ductal adenocarcinoma. Cancer Research, 66(8), 4149-4158. https://doi. org/10.1158/0008-5472.CAN-05-3089 [ Links ]
Friedman, E. (2013). MIRK/DYRK1B kinase in ovarian cancer. International Journal of Molecular Sciences, 14(3), 5560- 5575. https://doi.org/10.3390/ijms14035560 [ Links ]
Gao, J., Zheng, Z., Rawal, B., Schell, M. J., Bepler, G. and Haura, E. B. (2009). MIRK/DYRK1B, a novel therapeutic target, mediates cell survival in non-small cell lung cancer cells. Cancer Biology & Therapy, 8(17), 1671-1679. https:// doi.org/10.4161/cbt.8.17.9322 [ Links ]
Gong, Y., Wang, J., Huo, L., Wei, W., Ueno, N. T. and Woodward, W. A. (2014). Aldehyde dehydrogenase 1 expression in inflammatory breast cancer as measured by immunohistochemical staining. Clinical Breast Cancer, 14(3). https://doi.org/10.1016/j.clbc.2013.12.006 [ Links ]
He, M., Gu, J., Zhu, J., Wang, X., Wang, C., Duan, C. and Li, J. (2018). Up-regulation of DYRK1B promote astrocyte activation following lipopolysaccharide-induced neuroinflammation. Neuropeptides, 69(April), 76-83. https://doi. org/10.1016/j.npep.2018.04.008 [ Links ]
Huangfu, D. & Anderson, K. V. (2005). Signaling from Smo to Ci / Gli : conservation and divergence of Hedgehog pathways from Drosophila to vertebrates. 3-14. https://doi.org/10.1242/dev.02169 [ Links ]
Jelski, W., Laniewska-Dunaj, M., Orywal, K., Kochanowicz, J., Rutkowski, R. and Szmitkowski, M. (2014). The Activity of Alcohol Dehydrogenase (ADH) Isoenzymes and Aldehyde Dehydrogenase (ALDH) in the Sera of Patients with Brain Cancer. Neurochemical Research, 39(12), 2313-2318. https://doi.org/10.1007/s11064-014-1402-3 [ Links ]
Jin, K., Park, S., Ewton, D. Z. and Friedman, E. (2007). The survival kinase MIRK/DYRK1B is a downstream effector of oncogenic K-ras in pancreatic cancer. Cancer Research, 67(15), 7247-7255. https://doi.org/10.1158/0008-5472.CAN-06-4099 [ Links ]
Kalantari, E., Saadi, F. H., Asgari, M., Shariftabrizi, A., Roudi, R. and Madjd, Z. (2016). Increased Expression of ALDH1A1 in Prostate Cancer is Correlated With Tumor Aggressiveness: A Tissue Microarray Study of Iranian Patients. Applied Immunohistochemistry & Molecular Morphology: AIMM / Official Publication of the Society for Applied Immunohistochemistry, 00(00), 1-7. https://doi.org/10.1097/PAI.0000000000000343 [ Links ]
Kirwan, J. A., Weber, R. J. M., Broadhurst, D. I. and Viant, M. R. (2014). Direct infusion mass spectrometry metabolomics dataset: A benchmark for data processing and quality control. Scientific Data, 1, 1-13. https://doi.org/10.1038/sdata.2014.12 [ Links ]
Liu, X. Y., Xu, X. X., Yin, X. L., Zhang, X. X., Li, X. L. and Lu, X. H. (2017). Relationship between Glioblastoma Heterogeneity and Survival Time: An MR Imaging Texture Analysis. 1-7. https://doi.org/10.3174/ajnr.A5279 [ Links ]
Louis, D. N., Holland, E. C. and Cairncross, J. G. (2001). Glioma classification: A molecular reappraisal. The American Journal of Pathology, 159(3), 779-786. https://doi.org/10.1016/S0002-9440(10)61750-6 [ Links ]
Louis, D. N., Perry, A., Reifenberger, G., Deimling, A. Von, Figarella, D., Webster, B. and Ellison, D. W. (2016). The 2016 World Health Organization Classification of Tumors of the Central Nervous System: a summary. Acta Neuropathologica. https://doi.org/10.1007/s00401-016-1545-1 [ Links ]
Megger, D. A., Padden, J., Rosowski, K., Uszkoreit, J., Bracht, T., Eisenacher, M. and Sitek, B. (2017). One Sample, One Shot - Evaluation of sample preparation protocols for the mass spectrometric proteome analysis of human bile fluid without extensive fractionation. Journal of Proteomics, 154, 13-21. https://doi.org/10.1016/j.jprot.2016.11.021 [ Links ]
Natsume A. Y. J. (2008). Gene therapy for high-grade glioma: current approaches and future directions. Cell Adhes Migr, 2, 186e91. [ Links ]
Olmez, I., Shen, W., Mcdonald, H. and Ozpolat, B. (2015). Dedifferentiation of patient-derived glioblastoma multiforme cell lines results in a cancer stem cell-like state with mitogen-independent growth. Journal of Cellular and Molecular Medicine, 19(6), 1262-1272. https://doi.org/10.1111/jcmm.12479 [ Links ]
Ostrom, Q. T., Gittleman, H., Liao, P., Vecchione-Koval, T., Wolinsky, Y., Kruchko, C. and Barnholtz-Sloan, J. S. (2017). CBTRUS Statistical Report: Primary brain and other central nervous system tumors diagnosed in the United States in 2010-2014. Neuro-Oncology, 19(suppl_5), v1-v88. https://doi.org/10.1093/neuonc/nox158 [ Links ]
Park, M., Koo, S., Jong, L., Chang, H., Gu, S., Eui, K. and Ahn, S. (2017). Elderly patients with newly diagnosed glioblastoma: can preoperative imaging descriptors improve the predictive power of a survival model? Journal of Neuro-Oncology, pp 423-431. https://doi.org/10.1007/s11060-017-2544-3 [ Links ]
Pérez-Sánchez, G., Jiménez, A., Quezada-Ramírez, M. A., Estudillo, E., Ayala-Sarmiento, A. E., Mendoza-Hernández, G. and Segovia, J. (2018). Annexin A1, Annexin A2, and DYRK1B are upregulated during GAS1-induced cell cycle arrest. Journal of Cellular Physiology, 233(5), 4166-4182. https://doi.org/10.1002/jcp.26226 [ Links ]
Rasper, M., Schäfer, A., Piontek, G., Teufel, J., Brockhoff, G., Ringel, F. and Schlegel, J. (2010). Aldehyde dehydrogenase 1 positive glioblastoma cells show brain tumor stem cell capacity. Neuro-Oncology 12(10), 1024-1033. https:// doi.org/10.1093/neuonc/noq070 [ Links ]
Schäfer, A., Teufel, J., Ringel, F., Bettstetter, M., Hoepner, I., Rasper, M. and Schlegel, J. (2012). Aldehyde dehydrogenase 1A1-a new mediator of resistance to temozolomide in glioblastoma. Neuro-Oncology, 14(12), 1452-1464. https://doi.org/10.1093/neuonc/nos270 [ Links ]
Strober, W. (2015). Trypan Blue Exclusion Test of Cell Viability. (November), 3-5. https://doi.org/10.1002/0471142735.ima03bs111 [ Links ]
Stupp, R. & Roila, F. (2009). Malignant glioma: ESMO clinical recommendations for diagnosis, treatment and follow-up. Annals of Oncology, 20(SUPPL. 4), 126-128. https://academic.oup.com/annonc/article/20/suppl_4/iv126/148299 [ Links ]
Tomita, H., Tanaka, K., Tanaka, T. and Hara, A. (2016). Aldehyde dehydrogenase 1A1 in stem cells and cancer. Oncotarget, 7(10), 11018-11032. https://doi.org/10.18632/oncotarget.6920 [ Links ]
Van Den Bent, M. J., Weller, M., Wen, P. Y., Kros, J. M., Aldape, K. and Chang, S. (2017). A clinical perspective on the 2016 WHO brain tumor classification and routine molecular diagnostics. Neuro-Oncology, 19(5), 614-624. https://doi. org/10.1093/neuonc/now277 [ Links ]
Visus, C., Ito, D., Amoscato, A., Maciejewska-Franczak, M., Abdelsalem, A., Dhir, R. and DeLeo, A. B. (2007). Identification of human aldehyde dehydrogenase 1 family member a1 as a novel CD8+ T-cell-defined tumor antigen in squamous cell carcinoma of the head and neck. Cancer Research, 67(21), 10538-10545. https://doi.org/10.1158/0008-5472.CAN-07-1346 [ Links ]
Vilenchik, M., Kuznetsova, A., Frid, M., Duey, M., Damiani, A., De Leon, L. and Potapova, O. (2019). Implication of DYRK1B kinase in dormant glioblastoma cancers and utilization of DYRK1B inhibitors as a novel therapeutic strategy for glioblastoma. Journal of Clinical Oncology, 37(15_suppl):e14670-e14670. https://cancerres.aacrjournals.org/content/79/13_Supplement/3038# [ Links ]
Wang, Y., Wang, W. and Chen, Z. (2018). Expression of Aldehyde Dehydrogenase 1A1 (ALDH1A1) as a Prognostic Biomarker in Colorectal Cancer Using Immunohistochemistry. 2864-2872. https://doi.org/10.12659/MSM.910109 [ Links ]
Wen P. & Kesari S. (2008). Malignant gliomas in adults. The New England Journal of Medical, 359 (5), 492-507 DOI: 10.1056/NEJMra0708126 [ Links ]
Xu, S. L., Liu, S., Cui, W., Shi, Y., Liu, Q., Duan, J. J. and Bian, X. W. (2015). Aldehyde dehydrogenase 1A1 circumscribes high invasive glioma cells and predicts poor prognosis. American Journal of Cancer Research, 5(4), 1471-1483. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4473324/ [ Links ]
Yue, L., Huang, Z. M., Fong, S., Leong, S., Jakowatz, J. G., Charruyer-Reinwald, A. and Ghadially, R. (2015). Targeting ALDH1 to decrease tumorigenicity, growth and metastasis of human melanoma. Melanoma Research, 25(2), 138-148. https://doi.org/10.1097/CMR.0000000000000144 [ Links ]
Zhang, Q., Taguchi, A., Schliekelman, M., Wong, C.-H., Chin, A., Kuick, R. and Hanash, S. (2011). Comprehensive Proteomic Profiling of Aldehyde Dehydrogenases in Lung Adenocarcinoma Cell Lines. International Journal of Proteomics, 2011, 1-8. https://doi.org/10.1155/2011/145010 [ Links ]
Zhou, Q., Phoa, A. F., Abbassi, R. H., Hoque, M., Reekie, T. A., Font, J. S. and Kassiou, M. (2017). Structural Optimization and Pharmacological Evaluation of Inhibitors Targeting Dual-Specificity Tyrosine Phosphorylation-Regulated Kinases (DYRK) and CDC-like kinases (CLK) in Glioblastoma. Journal of Medicinal Chemistry, 60(5), 2052- 2070. https://doi.org/10.1021/acs.jmedchem.6b01840 [ Links ]
Como citar este artículo: Sánchez-Hernández, L., Saavedra Sotelo, R., Torres-Corona, D. D., Aguirre-Quezada, D. E., Gómez-García, E. (2020). Different levels of expression of ALDH1A1 and DYRK1B in patients with glioblastoma multiforme and their overall survival. Revista Bio Ciencias 7, e762. doi: https://doi.org/10.15741/revbio.07.e762
Recibido: 29 de Junio de 2019; Aprobado: 06 de Febrero de 2020











 texto en
texto en 


