Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista bio ciencias
versión On-line ISSN 2007-3380
Revista bio ciencias vol.6 spe Tepic 2019 Epub 14-Nov-2019
https://doi.org/10.15741/revbio.06.nesp.e540
Artículos de revisión
Los últimos 50 años de Salmonella en México: Fuentes de aislamiento y factores que influyen en su prevalencia y diversidad
1Centro de Investigación en Alimentación y Desarrollo A.C., Coordinación Regional Culiacán, Laboratorio Nacional para la Investigación en Inocuidad Alimentaria, Sinaloa, México.
2CONACYT-Centro de Investigación en Alimentación y Desarrollo A.C., Coordinación Regional Culiacán, Laboratorio Nacional para la Investigación en Inocuidad Alimentaria, Sinaloa, México.
3Centre for Environment, Fisheries and Aquaculture Science (CEFAS), Weymouth, United Kingdom.
Esta revisión describe el panorama de la investigación de Salmonella en términos de diversidad y factores que influyen su prevalencia en México; haciendo énfasis en ecología, cambio climático y estrategias que emplea para persistir en distintos ambientes. Para ello, se realizó una extensiva revisión científica de trabajos realizados en México entre enero de 1968 y marzo de 2018, donde se evidencia: la presencia al menos de 216 serotipos diferentes de Salmonella enterica circulando en México, siendo Enteritidis, Typhimurium, Anatum, Agona y Meleagridis los más prevalentes; los alimentos de origen animal son la principal fuente de aislamiento de Salmonella (42.76 %); y los estados ubicados en ecorregiones de climas cálidos-húmedos presentan los índices más elevados de salmonelosis no tifoidea en México. Las condiciones climáticas consistentes de las zonas tropicales favorecen la proliferación de Salmonella, y en México no es la excepción. Como parte de ello, la supervivencia y adaptación de Salmonella depende de tres estrategias fundamentales: 1) resistencia contra antibióticos de primera línea, 2) habilidad para utilizar fuentes alternas de carbono, y 3) capacidad de formar biopelículas. El uso de alguna estrategia o la combinación de ellas por las poblaciones de Salmonella circulantes podría aumentar el riesgo para la salud de la población. Por ello, esta revisión tiene como objetivo analizar de manera exhaustiva los elementos que le confieren una mayor capacidad de adaptación, favoreciendo las dinámicas de dispersión de Salmonella, y contribuyendo de esta manera al entendimiento de su naturaleza y a la epidemiología debida a este patógeno en México.
Palabras clave: Salmonella; cambio climático; México; biopelículas; resistencia antimicrobiana; diversidad genética.
This review describes the outlook of Salmonella research in terms of diversity and factors that influence its prevalence in Mexico; emphasizing in ecology, climate change and the strategies used to persist in different environments. To this end, an extensive scientific review of studies carried out in Mexico between January 1968 and March 2018 has evidenced the following: the presence of at least 216 different Salmonella enterica serotypes circulating in Mexico, being Enteritidis, Typhimurium, Anatum, Agona and Meleagridis the most prevalent; animal origin food are the main source of Salmonella isolation (42.76 %); and the states located in eco-regions of hot-humid climates have the highest rates of non-typhoid salmonellosis in Mexico. The climatic conditions of the tropics favor Salmonella proliferation, and Mexico is not the exception. As part of this, the survival and adaptation of Salmonella depends on three fundamental strategies: 1) resistance against firstline antibiotics, 2) ability to use alternate carbon sources, and 3) ability to form biofilms. The use of a strategy or their combination by the circulating populations of Salmonella could increase the risk to public health. Therefore, this review aimed to exhaustively analyze the elements that give Salmonella a greater adaptation capacity, favoring its dispersion dynamics; and thus contribute to the understanding of its nature and the epidemiology caused by this pathogen in Mexico.
Key words: Salmonella; climatic change; Mexico; biofilms; antimicrobial resistance; genetic diversity
Introducción
Salmonella es mundialmente reconocida como el principal agente etiológico de enfermedades transmitidas por alimentos y agua (WHO, 2018a). El centro para el control y prevención de enfermedades de los Estados Unidos de América y la autoridad Europea de Seguridad Alimentaria reportan anualmente más de 1,000,000 y 100,000 infecciones por Salmonella no tifoidea, respectivamente (CDC, 2018a; EFSA, 2015), mientras en México el número alcanza alrededor de 70,000 casos de este padecimiento cada año (DGE, 2017).
A pesar de que esta bacteria reside predominantemente en el tracto gastrointestinal de animales de sangre caliente y de sangre fría, su reconocida ubicuidad en ambientes naturales, donde despliega estrategias de supervivencia, le permiten prevalecer por periodos prolongados en suelos y sedimentos adaptándose a condiciones estresantes de temperatura, pH, desecación, estrés osmótico y nutrimental, así como depredación, facilitando su supervivencia, dispersión ambiental y alcance de nuevos hospederos (Donlan & Costerton, 2002; Tamagnini & Paraje, 2015).
Salmonella es persistente y muestra un comportamiento estacional, particularmente cuando concurren temperaturas y precipitaciones elevadas, como es el caso de las regiones tropicales y subtropicales donde las condiciones ambientales son ideales para el establecimiento de la bacteria (Simental & Martinez-Urtaza, 2008; Zhang et al., 2010; WHO, 2018b). Otro factor importante en la persistencia ambiental de Salmonella es su resistencia a antibióticos, lo que aumenta el riesgo de infecciones en humanos y animales debido a la aparición de clones multiresistentes, dificultando aún más el tratamiento terapéutico relacionado con enfermedades y brotes (Angulo et al., 2004; Zaidi et al., 2012). Además de esto, la capacidad de Salmonella para formar biopelículas en distintos escenarios facilita su adherencia para sobrevivir en superficies vivas o inertes, un paso crucial para infectar un nuevo hospedero (Donlan & Costerton, 2002; Ledeboer & Jones, 2005; Steenackers et al., 2012).
El objetivo de esta revisión fue describir el estado que guarda el conocimiento sobre la diversidad de Salmonella y los factores que contribuyen a su persistencia y prevalencia con la finalidad de tener un mejor entendimiento de su paso por el ambiente y tratar de discernir como esto puede impactar en este patógeno y por ende, en la epidemiología de la población de nuestro país. Dicha información será de utilidad para generar futuras líneas de investigación que ayudarán en el diseño de estrategias para la vigilancia y control de este microorganismo.
Resultados y Discusión
Epidemiología de Salmonella en México
El género Salmonella consta de dos especies: bongori y enterica, siendo ésta última dividida en seis subespecies: enterica, salamae, arizonae, diarizonae, houtenae e indica (Brenner et al., 2000). De éstas, destaca la subespecie enterica, la cual es representada por más de 2,600 serotipos que en conjunto, generan anualmente millones de infecciones, que varían desde una gastroenteritis aguda (salmonelosis no tifoidea, NTS) hasta fiebre tifoidea (serotipos Typhi y Paratyphi) en los seres humanos (CDC, 2013). Los principales síntomas de NTS son inflamación del tracto gastrointestinal que se acompaña con diarrea no sanguinolenta, vómito, náusea, dolor de cabeza, calambres abdominales y mialgia. En individuos sanos la NTS es una infección autolimitante, con un periodo de recuperación de 2 a 4 días; sin embargo, grupos susceptibles de la población, tales como niños, adultos mayores e inmunocomprometidos, son vulnerables a sufrir padecimientos graves que incluso pueden provocar la muerte (CDC, 2018b). De los 2600 serotipos Typhimurium y Enteritidis lideran los reportes de brotes diarreicos, por lo que se consideran los principales responsables de la NTS (CDC, 2018b).
Los sistemas de vigilancia epidemiológica son una herramienta eficaz para conocer con relativa certeza la incidencia de brotes diarreicos generados por el consumo de alimentos contaminados, así como el impacto en la morbilidad y mortalidad de la población. Desafortunadamente, estos sistemas solo funcionan de manera adecuada en países desarrollados, mientras que en los países en desarrollo su funcionabilidad es deficiente. Esta disfuncionalidad está asociada al desinterés de la población por acudir al médico en caso de un evento diarreico, probablemente debido al entendimiento general del comportamiento autolimitante que comúnmente exhiben dichas infecciones, y al compromiso económico que conlleva el proceso de consulta clínica y tratamiento, así como las deficiencias en los sistemas de salud pública que desmotivan al paciente a ejercer sus derechos como beneficiario del sistema.
Las autoridades de salud en México han hecho un gran esfuerzo para fortalecer el Sistema Nacional de Vigilancia Epidemiológica (SINAVE), que se encarga de recolectar la información de eventos epidemiológicos, incluyendo los casos de salmonelosis, los cuales son reportados semanalmente, cumpliendo con la Norma Oficial Mexicana NOM-017-SSA2-1994.
En este sentido, el SINAVE muestra en el periodo comprendido de 1984 a 2017 un aumento considerable de fiebre tifoidea pasando de 7,629 a 45,280 casos; y para fiebre paratifoidea/ otras salmonelosis de 31,943 a 104,471 casos, respectivamente (Figura 1 y 2). Estos datos estadísticos muestran a Sinaloa y Tamaulipas como los estados con mayor índice de fiebre tifoidea con 12.9 % y 10.25 %, respectivamente. Para el caso de fiebre paratifoidea y otras salmonelosis, Chiapas (12.99 %), Veracruz (9.37 %) y Tabasco (8.31 %) son los estados con mayor número de casos (INEGI, 2017) registrados en los meses de marzo a octubre (DGE, 2017). Esta información muestra un incremento de aproximadamente el doble de casos de salmonelosis en la población femenina en relación al género masculino (DGE, 2017). Estos hallazgos concuerdan con las dinámicas a nivel mundial y específicamente con lo citado por Reller et al., (2008) donde se reporta un aumento de salmonelosis en mujeres en los años comprendidos de 1968 a 2000. Las posibles causas de esta tendencia mundial y nacional se asocian a los siguientes factores: la población mexicana femenina es mayor a la población masculina, susceptibilidad biológica, mayor frecuencia de exposición con fuentes potenciales de contaminación.
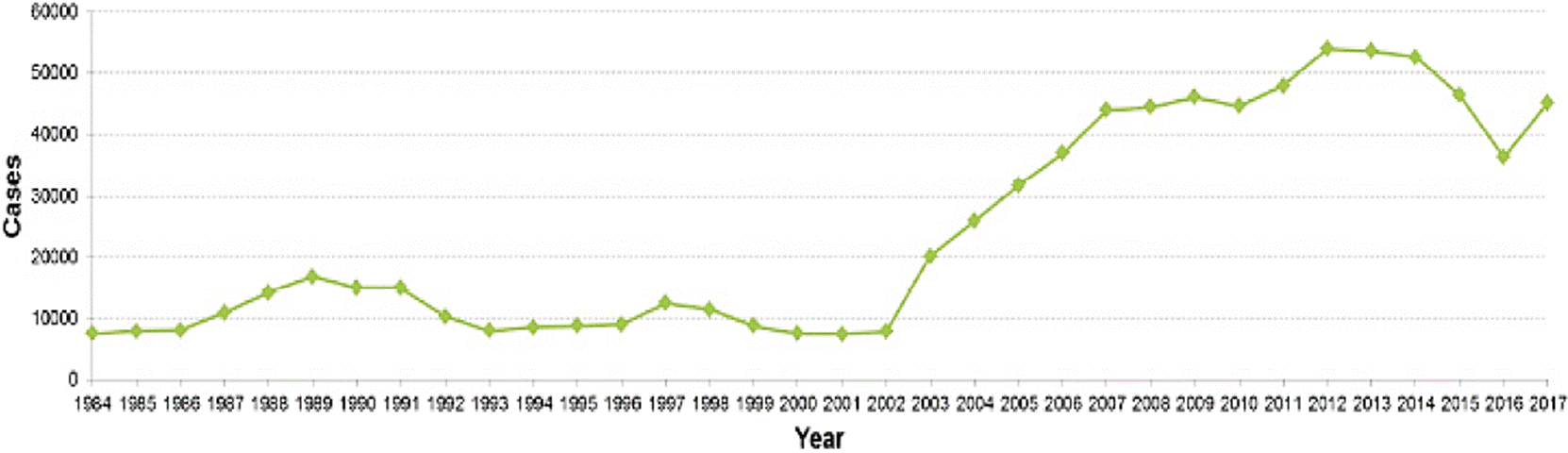
Figura 1 Prevalencia de fiebre tifoidea en México durante el período de 1984 a 2017 (Anuario de Morbilidad 1984-2017. DGE, 2017).

Figura 2 Prevalencia de fiebre paratifoidea y otras salmonelosis en México durante el período de 1984 a 2017. (Anuario de Morbilidad 1984 -2017. DGE, 2017).
En esta revisión de los años 1968 a 2018 se han detectado en México un amplio rango de serotipos de Salmonella aislados del ambiente, alimentos, humano y animales, destacando a Enteritidis, Typhimurium, Anatum, Agona, Meleagridis, Oranienburg, Derby, Infantis, Ohio y Havana como los serotipos más predominantes (Tabla 1); este patrón de predominancia de serotipos concuerda en gran medida con el estudio realizado por Gutiérrez-Cogco et al., (2000) donde presenta a Typhimurium, Enteritidis, Derby, Agona y Anatum como los principales serotipos de Salmonella que circulaban en el país en el período de 1972 a 1999, aislados de diversas fuentes (humanos, ambiente, alimentos).
Tabla 1 Diversidad de serotipos de Salmonella en México aislados de diferentes fuentes desde 1968 hasta 2018.
| Isolation origin | ||||||||
|---|---|---|---|---|---|---|---|---|
| Serotype | Animal | Human | Enviroment | Food of animal origin | Food of plant origin | Total | % | References* |
| Enteritidis | 7 | 190 | 1 | 53 | 6 | 257 | 6.61 | 27, 2, 46, 44, 11, 10, 35, 19, 42, 36, 22, 15 |
| Typhimurium | 17 | 63 | 38 | 100 | 29 | 247 | 6.52 | 27, 2, 46, 34, 44 |
| Anatum | 25 | 38 | 18 | 120 | 4 | 205 | 5.41 | 27, 24, 18, 2, 46, 34, 44, 1, 29, 41,
10, 35, 13, 40, 36 |
| Agona | 3 | 67 | 5 | 106 | 8 | 189 | 4.99 | 27, 46, 44, 27, 14,
29, 10, 35, 17, 40, 36, 15 |
| Meleagridis | 1 | 41 | 0 | 118 | 0 | 160 | 4.22 | 27, 46, 44 |
| Oranienburg | 32 | 43 | 50 | 2 | 0 | 127 | 3.35 | 38, 27, 2, 43, 46, 39, 19, 29, 14, 17 |
| Derby | 14 | 4 | 0 | 66 | 0 | 84 | 2.21 | 38, 24, 2, 44, 41, 13, 17, 32 |
| Infantis | 4 | 35 | 7 | 36 | 0 | 82 | 2.16 | 24, 18, 2, 34, 44, 28, 29, 41, 46, 13, 40 |
| Ohio | 0 | 67 | 0 | 2 | 0 | 69 | 1.82 | 34, 13 |
| Havana | 2 | 8 | 0 | 49 | 0 | 59 | 1.55 | 24, 44, 13 |
| Reading | 0 | 13 | 0 | 38 | 0 | 51 | 1.34 | 44, 46, 40 |
| Albany | 1 | 15 | 0 | 29 | 0 | 45 | 1.18 | 27, 44, 46, |
| Saintpaul | 11 | 0 | 27 | 6 | 0 | 44 | 1.16 | 27, 2, 28,14, 13, 17,18 |
| Arizonae | 24 | 0 | 0 | 1 | 12 | 37 | 0.97 | 30, 35, 36 |
| Adelaide | 0 | 13 | 0 | 23 | 0 | 36 | 0.95 | 44, 41, 40 |
| Give | 16 | 0 | 17 | 2 | 0 | 35 | 0.92 | 27, 2, 28, 14, 1, 29 |
| Wourthington | 0 | 0 | 0 | 30 | 0 | 30 | 0.79 | 46, 13 |
| Seftenberg | 15 | 0 | 1 | 11 | 0 | 27 | 0.71 | 2, 28, 13, 17, 32, 40 |
| Newport | 3 | 11 | 4 | 4 | 0 | 22 | 0.58 | 27, 2, 46, 34, 28, 44 |
| Minnesota | 9 | 0 | 8 | 3 | 0 | 20 | 0.52 | 27, 2, 28, 29, 13 |
| Typhi | 0 | 0 | 6 | 11 | 3 | 20 | 0.52 | 4, 32, 36, 21 |
| Muenchen | 4 | 9 | 0 | 5 | 0 | 18 | 0.47 | 27, 2, 46, 44 |
| Weltevreden | 9 | 0 | 7 | 0 | 1 | 17 | 0.44 | 39, 27, 28,14 |
| Choleraesuis | 3 | 0 | 0 | 2 | 12 | 17 | 0.44 | 2, 35, 40, 36 |
| London | 1 | 0 | 0 | 15 | 0 | 16 | 0.42 | 2, 32 |
| Hadar | 12 | 0 | 0 | 0 | 0 | 12 | 0.31 | 2 |
| Sandiego | 9 | 0 | 3 | 0 | 0 | 12 | 0.31 | 38, 24, 28,14 |
| Braenderup | 5 | 0 | 1 | 6 | 0 | 12 | 0.31 | 38, 28, 47 |
| Poona | 5 | 0 | 7 | 0 | 0 | 12 | 0.31 | 18, 2, 28, 18 |
| Stanleyville | 0 | 3 | 1 | 7 | 0 | 11 | 0.29 | 44, 39, 46 |
| Muenster | 1 | 0 | 5 | 4 | 0 | 10 | 0.26 | 24, 28, 41 |
| Brandenburg | 3 | 0 | 0 | 6 | 0 | 9 | 0.23 | 2, 46 |
| Gallinarum | 0 | 0 | 0 | 1 | 8 | 9 | 0.23 | 35, 36 |
| Bredeney | 1 | 0 | 0 | 7 | 0 | 8 | 0.21 | 24, 17, 40 |
| Heidelberg | 7 | 0 | 0 | 0 | 0 | 7 | 0.18 | 2 |
| Duesseldorf | 6 | 0 | 0 | 0 | 0 | 6 | 0.16 | 2 |
| Bovismorficans | 2 | 0 | 2 | 2 | 0 | 6 | 0.16 | 2, 28, 17 |
| Kentucky | 4 | 0 | 0 | 2 | 0 | 6 | 0.16 | 24, 2,10 |
| Azteca | 0 | 0 | 0 | 6 | 0 | 6 | 0.16 | 41 |
| Cerro | 0 | 0 | 0 | 6 | 0 | 6 | 0.16 | 46 |
| Lomita | 0 | 0 | 0 | 6 | 0 | 6 | 0.16 | 32 |
| Cannstatt | 0 | 0 | 0 | 6 | 0 | 6 | 0.16 | 46, 32 |
| Panama | 0 | 6 | 0 | 0 | 0 | 6 | 0.16 | 44 |
| Montevideo | 2 | 0 | 4 | 0 | 0 | 6 | 0.16 | 27, 2, 28 |
| Luciana | 3 | 0 | 2 | 0 | 0 | 5 | 0.13 | 27, 28 |
| Pomona | 0 | 0 | 5 | 0 | 0 | 5 | 0.13 | 28 |
| Houtenae | 0 | 0 | 0 | 1 | 4 | 5 | 0.13 | 35 |
| Vejle | 0 | 0 | 4 | 0 | 0 | 4 | 0.10 | 39 |
| Javiana | 1 | 0 | 2 | 1 | 0 | 4 | 0.10 | 27, 28, 32 |
| Edimburg | 0 | 0 | 0 | 1 | 3 | 4 | 0.10 | 35, 36 |
| Tennessee | 2 | 0 | 0 | 1 | 0 | 3 | 0.08 | 2, 32 |
| Soahanina | 2 | 0 | 1 | 0 | 0 | 3 | 0.08 | 27, 28 |
| Schwarzengrund | 0 | 0 | 0 | 3 | 0 | 3 | 0.08 | 13 |
| Acquatoria | 0 | 0 | 0 | 3 | 0 | 3 | 0.08 | 13 |
| Suberu | 0 | 0 | 3 | 0 | 0 | 3 | 0.08 | 39 |
| Urbana | 0 | 0 | 3 | 0 | 0 | 3 | 0.08 | 39 |
| Paratyphi | 0 | 0 | 3 | 0 | 0 | 3 | 0.08 | 4 |
| Degania | 2 | 0 | 0 | 0 | 0 | 2 | 0.05 | 30 |
| Binza | 0 | 0 | 0 | 2 | 0 | 2 | 0.05 | 13 |
| Tonev | 0 | 0 | 1 | 1 | 0 | 2 | 0.05 | 39 |
| Salamae | 0 | 0 | 0 | 0 | 2 | 2 | 0.05 | 36 |
| Sundsvall | 0 | 0 | 2 | 0 | 0 | 2 | 0.05 | 28 |
| Galiema | 0 | 0 | 2 | 0 | 0 | 2 | 0.05 | 39 |
| Othmarshem | 0 | 0 | 2 | 0 | 0 | 2 | 0.05 | 39 |
| Soerenga | 0 | 0 | 2 | 0 | 0 | 2 | 0.05 | 39 |
| Gaminara | 1 | 0 | 0 | 0 | 0 | 1 | 0.03 | 27 |
| Cayar | 1 | 0 | 0 | 0 | 0 | 1 | 0.03 | 27 |
| Rubislaw | 1 | 0 | 0 | 0 | 0 | 1 | 0.03 | 24 |
| Shamba | 1 | 0 | 0 | 0 | 0 | 1 | 0.03 | 30 |
| Selby | 1 | 0 | 0 | 0 | 0 | 1 | 0.03 | 30 |
| Bere | 1 | 0 | 0 | 0 | 0 | 1 | 0.03 | 30 |
| Bunnik | 1 | 0 | 0 | 0 | 0 | 1 | 0.03 | 30 |
| Drypool | 0 | 0 | 0 | 1 | 0 | 1 | 0.03 | 17 |
| Newlands | 0 | 0 | 0 | 1 | 0 | 1 | 0.03 | 17 |
| Winnipeg | 0 | 0 | 0 | 1 | 0 | 1 | 0.03 | 39 |
| Pullorum | 0 | 0 | 0 | 0 | 1 | 1 | 0.03 | 36 |
| Bongor | 0 | 0 | 0 | 0 | 1 | 1 | 0.03 | 36 |
| Kiambu | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 28 |
| Texas | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 28 |
| Amherstiana | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Augusten- burg | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Breda | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Bulovka | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Coeln | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Corvallis | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Djugu | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Nchanga | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Nitra | 0 | 0 | 1 | 0 | 0 | 1 | 0.03 | 39 |
| Stanley | 0 | 0 | 1 | 0 | 0 | 1 | 0.30 | 93 |
| Others | 612 | 187 | 43 | 711 | 76 | 1,629 | 41.27 | 12, 27, 24, 30, 38, 2, 46, 28, 18, 14, 39, 4, 24 , 37, 20, 35, 5, 39, 8, 10, 32, 42, 40, 20, 31, 36, 1, 22, 15, 9, 16, 3, 47, 25 |
*1.Acedo-Félix et al., 2009; 2. Alaniz-de la O et al., 1999; 3. Alcázar-Montañez et al., 2006; Becerra-Tapia & Botello, 1995; 5. Bello-Pérez et al., 1990; 6. Benavides-Plasencia et al., 2005; 7. Bessudo et al., 1973; 8. Camacho et al., 2010; 9. Cerna-Cortes et al., 2013; 10. Charles-Hernández et al., 2007; 11. Chávez-de la Peña et al., 2001; 12. Cueto-Medina et al., 2015; 13. Escartin et al., 1999; 14. Estrada-Acosta et al., 2014; 15. Estrada-García et al., 2004; 16. Félix-Fuentes et al., 2005; 17. Fernández-Escartín & Torres, 1996; 18. Figueroa et al., 2005; 19. Gallegos-Robles et al., 2008; 20. Gallegos-Robles et al., 2009; 21. García-Gómez et al., 2002; 22. Gómez-Aldapa et al., 2014; 23. González-Cortés et al., 1974; 24. Grabert, 1968; 25. Hernández et al., 2007; 26. Hernández-Domínguez et al., 2008; 27. Jiménez et al., 2011; 28. Jiménez et al., 2014; 29. López-Cuevas et al., 2009; 30. Martínez et al., 1999; 31. Morales-Hernández et al., 2009; 32. Nayarit-Ballesteros et al., 2016; 33. Olarte & Galindo, 1973; 34. Paniagua-Contreras et al., 2008; 35. Quiñonez-Ramírez et al., 2000; 36. Quiroz-Santiago et al., 2009; 37. Rubio et al., 2013; 38. Silva-Hidalgo et al., 2012; 39. Simental & Martínez-Urtaza, 2008; 40. Talavera et al., 2011; 41. Torres et al., 2011; 42. Varela-Guerrero et al., 2013; 43. Vázquez-Garcidueñas et al., 2014; 44. Zaidi et al., 2006; 45. Zaidi et al., 2007; 46. Zaidi et al., 2008; 47. Zaidi et al., 2012.
Este tipo de información expone una importante área de oportunidad para fortalecer los sistemas epidemiológicos en el estudio de Salmonella en México, ya que una de las principales limitantes al momento de realizar la vigilancia epidemiológica radica en las técnicas utilizadas para los monitoreos necesarios para este proceso; las metodologías empleadas suelen ser laboriosas, costosas, inespecíficas y toman un tiempo considerable para la obtención de resultados, y finalmente la búsqueda es restringida a solo dos serotipos (Typhi y Paratyphi). Es por ello, que en años recientes se promueve entre la comunidad científica la adopción de estrategias modernas que aborden las limitantes presentadas por los métodos convencionales y ofrezcan alternativas rápidas, confiables, sencillas o automatizadas y económicas. La secuenciación masiva de ácidos nucleicos es una herramienta que está revolucionando el análisis microbiológico en la actualidad, permitiendo obtener información relevante de individuos específicos en tiempo record mediante la detección, monitoreo, caracterización y análisis de microorganismos patógenos a través de genómica comparativa y funcional, también aplicada en la reconstrucción de las dinámicas de dispersión de dichos microorganismos y de su historia evolutiva.
Las herramientas genómicas innovadoras vendrán a revolucionar la vigilancia epidemiológica, ofreciendo herramientas útiles para el monitoreo, prevención y diseño de estrategias para el control de microorganismos patógenos responsables de brotes epidemiológicos. Lo que a su vez impactará fuertemente en la inocuidad alimentaria a lo largo de la cadena de la producción de alimentos y la salud pública.
Factores predisponentes para la prevalencia de Salmonella
Las condiciones ambientales extremas como olas de calor, altas precipitaciones pluviales y características intrínsecas del patógeno que incluyen la formación de biopelículas y la resistencia antimicrobiana en clones de Salmonella, generan una condición propicia para el establecimiento, persistencia y dispersión de la bacteria en nichos ambientales (Angulo et al., 2004; Ledeboer & Jones, 2005; Akil et al., 2014).
Cambio climático
El cambio climático se define como la variación del clima en la tierra durante un lapso extendido de tiempo, que se atribuye a causas naturales o a la actividad humana de manera directa o indirecta, afectando los parámetros de temperatura, precipitaciones y nubosidad. (WHO, 2018b). La atmósfera está compuesta por diversos gases, los cuales retienen el calor del sol alrededor de la tierra, haciéndola un planeta habitable. Entre estos gases se encuentran el dióxido de carbono, el óxido nitroso y el metano, que son llamados gases de efecto invernadero y sus niveles han ido en aumento debido a distintas actividades antropogénicas. Uno de estos gases, el CO2, es el que más atrapa calor en las capas inferiores de la atmósfera, esto ha generado un aumento de 0.85 °C en la temperatura media del planeta en el último siglo, permitiendo que se produzca el calentamiento global y concomitante al cambio climático (WHO, 2018b).
La salud humana puede verse seriamente afectada por el cambio climático en términos de su influencia en la interacción entre humanos y patógenos, ya que distintos fenómenos climáticos pueden promover la aparición de infecciones asociadas a microorganismos transmitidos por el aire, por vectores o por el agua, especialmente entre regiones y poblaciones susceptibles (Wu et al., 2016). Esto ha sido evidenciado por Akil et al., (2014) y Jiang et al., (2015) quienes reportan que de manera concomitante las altas temperaturas y la intensidad de lluvias son factores que tienen una relación directa con el aumento de brotes asociados a patógenos como Salmonella.
Esta dinámica climatológica ha tenido un impacto directo en algunas regiones del territorio mexicano, ya que los eventos climáticos como huracanes, son más frecuentes y más intensos, y generan graves inundaciones, especialmente en las zonas ubicadas cercanas al Océano Atlántico y la parte costera al Norte del país (CONAGUA, 2013). Ejemplo de ello son los elevados casos de fiebre tifoidea en estados como Sinaloa y Tamaulipas (DGE, 2017) y paratifoidea/ otras salmonelosis en Chiapas, Veracruz y Tabasco; estos estados tienen temperaturas y precipitación pluvial similar y son azotados por ciclones y huracanes (CONAGUA, 2013; INEGI, 2017), siendo los meses de marzo a octubre donde se presenta el mayor número de casos de salmonelosis (DGE, 2017).
Estos datos concuerdan con la distribución geográfica de fiebre tifoidea ubicándola en países en vías de desarrollo, los cuales convergen geo-espacialmente en el trópico de cáncer, y cuentan con un clima tropical (Lee et al., 2016). Sin embargo, aunque México se encuentre ubicado dentro de esta franja de clima tropical, su territorio es dividido por ecorregiones, que se distinguen por características medioambientales únicas de cada ecorregión que pueden ofrecer nichos específicos que pueden representar un reto a superar para cualquier microorganismo durante su fase ambiental.
La diversidad de microclimas y su efecto sobre la presencia de patógenos como Salmonella, son expuestos en trabajos contrastantes como los de Simental & Martínez-Urtaza (2008) y Jiménez et al., (2014) quienes evidencian la influencia que marcan la distribución geográfica y estacional; los primeros asociando a Salmonella a periodos de lluvia en sitios como Bahía de Todos Santos, Baja California, México; mientras los segundos, detectando a la bacteria de manera consistente independientemente de la estación del año en ríos de Sinaloa. La divergencia entre estos dos estudios puede ser debido a las diferencias en microclimas en estas regiones, debidos a las ecorregiones en los que se ubican los dos estados, identificando el California-mediterránea para el caso de Baja California y el de selvas cálidas-secas para Sinaloa (INEGI, 2017).
Para enfrentar el cambio climático es necesario el esfuerzo y coordinación conjunta del gobierno, sociedad civil y sector salud para formular estrategias y sensibilizar a la población para minimizar el calentamiento global y sus efectos secundarios, así como establecer oportunamente estrategias de prevención de enfermedades infecciosas. Esto principalmente como resultado del incremento de la población mundial, ya que tan solo México cuenta con un poco más de 112 millones de habitantes (INEGI, 2017), lo que lleva a la intensificación de los efectos adversos del cambio climático, ya que las actividades antropogénicas son una causa principal de este fenómeno.
Formación de biopelículas
Una vez que Salmonella es liberada al ambiente, enfrenta condiciones no hospedantes que inducen a la bacteria a activar mecanismos de adaptación (Winfield & Groisman, 2003; Martínez-Urtaza et al., 2004). Uno de los mecanismos de adaptación más utilizados por bacterias es la formación de biopelículas, que son asociaciones bacterianas rodeadas por una matriz polimérica adheridas a superficies vivas o inertes (Costerton et al., 1999), y pueden ser formadas en tres interfases: líquido-aire, sólido-aire y sólido-líquido (Constantin, 2009; Steenackers et al., 2012). Estas estructuras confieren protección contra factores ambientales estresantes u hostiles, además de representar una fuente constante y persistente de diseminación bacteriana (Donlan & Costerton, 2002; Jensen et al., 2010). Las biopelículas de Salmonella están constituidas por agua (70-97 %), ácidos nucleicos, exopolisacáridos como celulosa y ácido colánico, así como proteínas tipo fimbria curli y BapA (Steenackers et al., 2012). Varios estudios han demostrado que las bacterias, dependiendo de las condiciones ambientales y la superficie donde se adhieren, producen distintos exopolisacáridos y proteínas como componentes de la matriz de la biopelícula, siendo distintas las composiciones de las biopelículas formadas en tejidos animales, plantas o superficies inertes (vidrio, plástico, acero inoxidable) (Costerton et al., 1999; Boddicker et al., 2002; Steenackers et al., 2012). Pero indudablemente, la formación de biopelículas en cualquier espacio (ambiente, hospedero) en donde sea formada, mejora la capacidad de Salmonella para sobrevivir (Ledeboer & Jones, 2005).
En este sentido, en México Medrano-Félix et al. (2018a) demostraron que la capacidad de serotipos de Salmonella ambientales prevalentes en una región agrícola para desarrollar biopelículas en la interfase líquido-aire, depende de los niveles de pH y la salinidad del agua. Silva-Hidalgo et al., (2016) detectaron el gen bapA en serovares de Salmonella enterica subsp. enterica aislados de animales en cautiverio, resaltando la importancia de este gen en la conformación de biopelículas y como ello favorece la resistencia de esta bacteria en hábitats naturales. Otros estudios demostraron la capacidad de Salmonella Poona y Michigan para formar biopelículas en cortezas de melón cantaloupe (Annous et al., 2005) y Salmonella Typhimurium y Javiana en cladodios de nopal (Opuntia ficus-indica), agua y suelo (De los Santos et al., 2012) mostrando un aumento en la resistencia de este patógeno a los desinfectantes. El análisis en conjunto de estos trabajos exhibe el ciclo de vida de Salmonella envuelto por la protección de estas estructuras de sobrevivencia que le permiten permanecer viable ante la exposición en diferentes escenarios. Si bien estos son un esfuerzo para explicar el rol de las matrices exopoliméricas en la biología de las variantes de Salmonella que circulan en el país, aún falta ahondar y ampliar el estudio en este tema y conocer de qué manera los factores ambientales predominantes en México regulan e influyen en la formación/composición de las biopelículas, y si éstas fungen como un catalizador en el éxito de alcanzar a un nuevo hospedero.
Resistencia a antimicrobianos (AMR)
La RAM es la habilidad de los microorganismos para evitar que los antibióticos actúen contra ellos (WHO, 2016). Una de las principales causas que detonan la resistencia a los antimicrobianos, es su uso indiscriminado (Angulo et al., 2004). El éxito de bacterias con AMR es debido a su capacidad de mutar e intercambiar material genético entre diferentes especies bacterianas.
Un número importante de grupos de investigación han enfocado sus esfuerzos a caracterizar los diferentes niveles de resistencia que pueden presentar diversos serotipos de Salmonella que van desde uni-resistencia hasta resistencia múltiple a antibióticos como ampicilina, cloranfenicol, estreptomicina, sulfonamidas y tetraciclina (Bessudo et al., 1973; Olarte & Galindo, 1973; González-Cortes et al., 1974; Alaniz-de la O. et al., 1996; Miranda et al., 2009; Camacho et al., 2010; Talavera et al., 2011; Jiménez et al., 2011; Pérez-Montaño et al., 2012; Nayarit-Ballesteros et al., 2016; Aguilar-Montes de Oca et al., 2017), siendo todos estos fármacos de primera elección en el tratamiento de padecimientos ocasionados por Salmonella.
Lo anterior fue expuesto en un estudio de caso de un brote gastrointestinal por Salmonella Oranienburg ocurrido en una prisión mexicana, donde se observó una alta diversidad de patrones de resistencia (17 perfiles AMR), entre los que se encontraron predominantemente las resistencias a ampicilina, carbenicilina y cefalotina (Vázquez-Garcidueñas et al., 2014). La AMR no se limita a serotipos patógenos típicos del humano, los cuales se exponen comúnmente a antimicrobianos, sino que se observa también en cepas que predominan en ambientes no hospederos, tal como lo describen López-Cuevas et al., (2009) quienes detectaron una alta resistencia a tetraciclina en Salmonella Typhimurium (12/13) aislada de agua en el valle de Culiacán, Sinaloa; más tarde Lugo-Melchor et al., (2010), tras estudios más detallados de estas cepas empleando PCR y PFGE, identificó el gen tet(A) (que confiere resistencia a tetraciclina) en el transposón Tn1721 en el DNA genómico. El desarrollo de AMR en Salmonella durante su paso por el ambiente, puede ser mediado por cambios genéticos promovidos por la exposición de las poblaciones bacterianas a fármacos utilizados de manera intensiva e indiscriminada tanto en actividades agrícolas como pecuarias.
Debido al reto que el control bacteriano mediante el uso fármacos implica hoy en día, es necesario encontrar alternativas, que estén disponibles para toda la población y que garanticen una protección duradera, para disminuir el uso de antibióticos y sus efectos adversos, generando líneas alternas de investigación, como son los bacteriófagos (ampliamente utilizados como control biológico y fagoterapia) o el desarrollo de vacunas para otros serotipos diferentes a Salmonella Typhi.
Diversidad de Salmonella en ambientes no hospederos
La aparición de casos de salmonelosis no tifoidea en humanos se relaciona tradicionalmente con el consumo de productos de origen animal contaminados con Salmonella (WHO, 2018a); sin embargo, en los últimos años ha aumentado el número de brotes asociados con el consumo de productos frescos (CDC, 2018b). La excreción de animales portadores constituye la principal ruta de liberación de patógenos entéricos del hospedero hacia el ambiente. Estos desechos, pueden contaminar ríos, lagos y otros humedales al ser depositados directamente en los cuerpos de agua; otras vías de deposición son el arrastre resultado de precipitaciones y/o de actividades antropogénicas como agricultura y ganadería; o por su liberación a través de las desembocaduras de drenajes provenientes de ciudades u otros asentamientos humanos. Por ello, se considera al agua como un importante vehículo para la dispersión de Salmonella en el ambiente (Winfield & Groisman, 2003).
Conocer los mecanismos y entender las dinámicas de dispersión de Salmonella en el ambiente requiere de estudios minuciosos que reconstruyan la dispersión histórica del microorganismo por el tiempo y espacio, considerando tanto zonas geográficas como fuentes de aislamiento, estudiando los perfiles de diversidad de poblaciones de Salmonella y estableciendo el papel específico que juega cada uno de los factores durante el paso de Salmonella del ambiente hacia a un nuevo hospedero. Conforme se logre profundizar en estos procesos e interacciones biológicas, se mejorará el diseño e implementación de estrategias eficientes para la prevención, diagnóstico y control de Salmonella, con el objetivo de evitar brotes epidemiológicos y minimizar el impacto de Salmonella en la salud pública.
A continuación, se describe la diversidad de Salmonella encontrada por distintos autores en una variedad de matrices como agua, superficies y alimentos, así como sus posibles implicaciones en la diseminación de la bacteria.
Agua, suelo y superficies inertes
El agua de irrigación no tratada y los suelos incorporados con compostas son considerados las principales fuentes de contaminación que causan la pérdida de inocuidad alimentaria. El agua es un vehículo que puede magnificar la contaminación puntual y generar una gran dispersión de Salmonella (Martínez-Urtaza et al., 2004; Levantesi et al., 2012).
La presencia de Salmonella en agua de riego, forma parte de una serie de prácticas agrícolas y de manufactura deficientes, que puede aumentar la probabilidad de contaminar los alimentos en empaques agrícolas e incluso desde su producción en el campo. Estudios sobre la inocuidad de melón, chile, tomate y cilantro en los estados de Coahuila, Michoacán, Guerrero, Sinaloa y Sonora coinciden en que el agua de riego constituye un vehículo de contaminación por Salmonella (Gallegos-Robles et al., 2008; Figueroa et al., 2005; Hernández-Domínguez et al., 2008; López-Cuevas et al., 2009; Acedo-Félix et al., 2009; Morales-Hernández et al., 2009; Estrada-Acosta et al., 2014). La diversidad de serotipos encontrados en estos estudios puede atribuirse al hecho de que los cuerpos de agua están al aire libre, por lo que la incorporación de especies de Salmonella es multifactorial incluyendo aguas residuales (industriales o domésticas), heces de animales silvestres mamíferos, reptiles y aves), así como inundaciones y escorrentías promoviendo procesos de eutroficación benéficos para el microorganismo (Simental & Martínez-Urtaza, 2008; Jiménez et al., 2014).
En México, los serotipos más comunes de Salmonella aislados de agua, suelo de cultivo y superficies de empaques son Oranienburg, Typhimurium, Saintpaul, Anatum y Give, de los cuales las principales fuentes de aislamiento son agua de río, canales de riego y arroyos. El aislamiento de estos serotipos involucró el uso de diversas técnicas, tales como reacción en cadena de la polimerasa (PCR, por sus siglas en inglés), serotipificación (Figueroa et al., 2005; Simental & Martínez-Urtaza, 2008; Hernández-Domínguez et al., 2008; Morales-Hernández et al., 2009; López-Cuevas et al., 2009), polimorfismo de longitud de fragmentos de restricción (RFLP, por sus siglas en inglés) (Gallegos-Robles et al., 2008), así como análisis de electroforesis en gel de campo pulsado (PFGE, por sus siglas en inglés), que buscaban establecer la diversidad con base a las relaciones genéticas entre aislados de distintas fuentes, incorporando la georeferenciación para obtener patrones geográficos diferenciales (Estrada-Acosta et al., 2014; Jiménez et al., 2014). En estos dos últimos estudios, se vislumbró la diversidad intraserotipo que puede existir en un nicho ecológico, llegando a obtener hasta 22 perfiles diferentes de una población de 58 cepas comprendida de siete serotipos; mientras que algunos perfiles mostraban ser exclusivos de un punto en particular, algunos otros se encontraban distribuidos en diversos puntos de ese espacio.
Si bien, la técnica de PFGE fue un estándar utilizado ampliamente por la CDC, y contribuyó al conocimiento estableciendo la diversidad genética microbiana con base a su alcance, de acuerdo a los estándares actuales, esta herramienta no es suficiente para discriminar y establecer relaciones microbianas espacio-temporal confiables debido a su capacidad limitada para distinguir entre dos individuos estrechamente relacionados genéticamente. Esta es la razón por lo que se sugiere el uso de estrategias más modernas y eficientes como la secuenciación de genomas completos.
Entre las habilidades que Salmonella emplea para sobrevivir se encuentra la flexibilidad metabólica, ya que se ha demostrado que una vez que este patógeno se ha incorporado a los cuerpos de agua, no se ve limitado por la escasez y el tipo de fuentes de carbono disponibles. Medrano-Félix et al., (2018b) reportaron la facilidad de adaptación de serotipos de Salmonella de origen clínico al consumir una amplia variedad de fuentes de carbono. De igual manera, los serotipos ambientales mostraron una mayor actividad metabólica, pero con una diversidad limitada de fuentes de carbono; además observando un efecto superior en ensayos de adhesión, invasión y viabilidad celular en células epiteliales, demostrando con esto una mayor capacidad patogénica (Estrada-Acosta et al., 2017). Estos hallazgos sugieren que el estudio y vigilancia de Salmonella debería no ser limitada a únicamente los serotipos hospederoespecífico del humano.
Por otro lado, estudios de genómica comparativa entre aislados de Salmonella obtenidos a partir de muestras de agua superficial y de heces de animales domésticos asintomáticos arrojaron que la diversidad genética entre las distintas cepas aisladas son mínimas entre sí, lo que sugiere una relación estrecha entre los aislados locales de ambas fuentes, aportando evidencia que apoya la teoría de que los animales constituyen una fuente importante para la contaminación de los cuerpos de agua con Salmonella y que a su vez, ayudan en la transmisión de la bacteria a nuevos hospederos (Jiménez et al., 2011; 2014).
Alimentos de origen vegetal
El agua de riego contaminada con Salmonella ha sido asociada a brotes epidemiológicos, lo cual puede ser producto de procesos de desinfección ineficientes y prácticas higiénicas inadecuadas (en campo y empaque), derivando en la presencia de este microorganismo en alimentos de consumo fresco (Levantesi et al., 2012). En México, diversos estudios muestran la presencia de Salmonella en vegetales crudos y procesados, tales como lechuga y cilantro (García-Gómez et al., 2002), melón cantaloupe y chile (Morales-Hernández et al., 2009; Gallegos-Robles et al., 2008, 2009), tomate (Estrada-Acosta et al., 2014), germinado de frijol (Cerna-Cortes et al., 2013), perejil, brócoli, coliflor, espinaca (Quiroz-Santiago et al., 2009), ensalada de frutas (Félix-Fuentes et al., 2005), salsas para tacos (Estrada-García et al., 2004), tortillas de maíz (Gómez-Aldapa et al., 2013), jugo de betabel fresco (Gómez-Aldapa et al., 2014), entre otros. El melón, perejil y cilantro presentaron las concentraciones más altas de Salmonella, mientras que Typhimurium, Arizonae, Choleraesuius y Enteritidis fueron los serotipos más prevalentes.
La información mostrada en estos estudios concuerda con investigaciones de brotes ocurridos en Estados Unidos de los cuales el 11 % fueron causados por los serotipos de Salmonella Enteritidis y Typhimurium, siendo los vegetales de hojas frescas el alimento más comúnmente relacionado a los mismos (Painter et al., 2013; Herman et al., 2015); además de estos reportes es necesario señalar que México se ha visto involucrado en brotes por Salmonella en productos frescos tales como papaya, pepino, melón, mango, tomate (CDC, 2018b), por lo que es necesario enfocarse más en el estudio de este patógeno en productos frescos, ya que los datos reportados en México son escasos.
Alimentos de origen animal
Los animales domésticos son los mayores reservorios de Salmonella, cuya producción de carnes y derivados son los vehículos mayormente reportados en brotes de salmonelosis transmitidas por alimentos (WHO, 2018a). Desde la década de los 90s, en México se aisló Salmonella en un 42.76 % en alimentos de origen animal, principalmente de carne de cerdo, res y pollo (Charles-Hernández et al., 2007; Zaidi et al., 2006; Hernández et al., 2007; Zaidi et al., 2008; Camacho et al., 2010 ; Rubio et al., 2013; Varela-Guerrero et al., 2013; Nayarit-Ballesteros et al., 2016), seguido de chorizo, longaniza y cecina (Bello-Pérez et al., 1990; Escartin, 1999; Charles-Hernández et al., 2007; Torres et al., 2011). En el 2005, Mancera et al., 2005, aislaron un bajo porcentaje (0.25 %) de Salmonella Enteritidis a partir de la yema de huevo. Esto es relevante debido a que el principal producto alimenticio de la canasta básica mexicana es el huevo y su contaminación de origen podría significar un importante riesgo de salud pública. Otra fuente importante de Salmonella son los productos marinos, que comúnmente se consumen crudos. La diversidad de serotipos de Salmonella en ceviche de pescado (Fernández-Escartín & Torres-Vitela, 1996), almejas (Quiñonez-Ramírez et al., 2000) y moluscos (Simental & Martínez-Urtaza, 2008) vendidos en establecimientos fijos y ambulantes muestra una mayor incidencia de este patógeno durante las estaciones más cálidas.
Por todo lo antes expuesto, la contaminación de alimentos por Salmonella es de primordial importancia para la secretaría de salud en México, por lo que existen normas para el control de alimentos que puntualizan los requisitos mínimos de buenas prácticas de higiene que deben observarse en el proceso de alimentos y sus materias primas a fin de evitar su contaminación. Aún con estas medidas existe un alto número de serotipos de Salmonella en alimentos de origen animal, evidenciando el alto riesgo de consumo de estos productos bajo un proceso de cocción deficiente.
Diversidad de Salmonella en ambientes hospederos
En general Salmonella enterica puede encontrarse en una variedad de vertebrados, incluyendo seres humanos, mascotas, animales salvajes, terneros, aves de corral, cerdos, ovejas, aves silvestres y reptiles (CDC, 2018b). Esta bacteria se transporta de forma asintomática en el intestino o la vesícula biliar de muchos animales y excretada de forma intermitente a través de las heces (Gunn et al., 2014).
Animales
Durante más de medio siglo, Salmonella ha sido reportada en animales en México. En esta revisión, los animales ocupan el segundo lugar como fuente de aislamiento (23.42 %) de la bacteria (Tabla 1), de los cuales el ganado vacuno (10.14 %), aves de corral (5.63 %) y cerdo (3.38 %) presentaron la más alta prevalencia, seguidos por ovejas, perros, cucaracha americana e iguanas con el 2.02, 0.56, 0.33 y 0.11 %, respectivamente. Los principales serotipos encontrados fueron Oranienburg, Anatum, Arizonae y Typhimurium (Grabert, 1968; Figueroa et al., 2005; Jiménez et al., 2011; Cueto-Medina et al., 2015).
Por otro lado, estudios con animales en cautiverio han mostrado la presencia de Salmonella; Martínez et al., (1999) estudiaron las heces de diferentes especies de reptiles en un serpentario de la ciudad de Puebla, encontrando 7.77 % de aislados de Salmonella. Asimismo, Silva-Hidalgo et al., (2012) reportaron la presencia de esta bacteria en el 11.6 % de las muestras de heces de mamíferos, aves y reptiles cautivos, así como roedores e insectos que viven en estrecho contacto con las jaulas de los animales del zoológico en estudio; encontrando los serotipos Sandiego, Oranienburg, Weltevreden, Braenderup y Derby. El análisis intraserotipo mediante PFGE mostró cepas indistinguibles (nula diversidad genética), con alguna excepción aislada, lo cual sugiere la circulación de esta variante de Salmonella entre los habitantes animales en estos ambientes, que pueden diseminarse a través de agua, alimentos, fomites o vectores como aves acuáticas y roedores.
Humanos
Los seres humanos adquieren la infección por Salmonella después de la ingestión de alimentos o agua contaminados. El padecimiento lo causan principalmente serotipos hospedero-específico como los son Typhi y Paratyphi, sin embargo las infecciones en humanos no están restringidas solo a estos.
Desde que se documenta la vigilancia epidemiológica en México, la fiebre tifoidea ha ido en aumento mientras que la fiebre paratifoidea y otras salmonelosis se han mantenido en un rango constante de cerca de 100, 000 casos por año en la última década (DGE, 2017a) (Figura 1 y 2), resaltando que estos datos pudieran ser más elevados si se registraran todos los eventos de este padecimiento.
En el año de 1972 y principios de 1973 se presentó una epidemia de fiebre tifoidea (ocasionada por Salmonella Typhi) en la Ciudad de México, la cual se expandió rápidamente a áreas circunvecinas afectando a más de 10,000 personas (Bessudo et al., 1973; Olarte & Galindo, 1973; González-Cortes et al., 1974). Otros dos brotes importantes relacionados con el consumo común de alimentos contaminados fueron reportados; el primero ocurrió dentro de un hospital, involucrando a 155 personas infectadas por Salmonella Enteritidis (Chávez-de la Peña et al., 2001); el segundo tuvo lugar dentro de una prisión, donde 150 reclusos presentaron diarrea y otros síntomas de enfermedad entérica ocasionada por Salmonella Oranienburg (Vázquez-Garcidueñas et al., 2014). En ambos casos la fuente de infección no fue identificada. Si bien los alimentos de origen animal han sido descritos como la principal causa de brotes por Salmonella, la presencia de esta bacteria en individuos sanos y enfermos puede convertir al humano como portador, diseminador y por ende responsable de la transmisión de esta enfermedad (Zaidi et al., 2006; Hernández et al., 2007; Paniagua-Contreras et al., 2008); esto se ve reflejado ubicando al humano como la tercera fuente de aislamiento de Salmonella en México, con un 21.46 % (Tabla 1). Esta problemática acentúa la necesidad de implementar programas de vigilancia continuos y más amplios para el seguimiento de fuentes de contaminación, con el uso de técnicas más sensibles y más rápidas para la discriminación de patógenos.
Conclusiones
Salmonella es un patógeno ampliamente distribuido en México. La diversidad fenotípica indica la presencia de al menos 216 serotipos circulando en el país, siendo los serotipos Enteritidis, Typhimurium, Anatum, Agona y Meleagridis los más prevalentes. La capacidad infecciosa y de resistencia de los serotipos circulantes en México depende de la presión selectiva de las variaciones ambientales incluyendo la temperatura y la precipitación pluvial. De la misma manera esta presión selectiva es visible en el aumento exacerbado de serotipos de Salmonella con multiresistencia a los antibióticos, lo cual le provee a la bacteria con un incrementado factor de virulencia.
En esta revisión de los últimos 50 años de Salmonella en México fueron incluidos estudios de Salmonella que abordaron temas referentes a las interacciones de esta bacteria con el entorno en términos epidemiológicos, diversidad y resistencia, sin embargo, la información es limitada. Esto da pauta a fortalecer y realizar más investigaciones, profundizando en temas que permitan dilucidar con mayor precisión el papel que las variaciones climáticas, resistencia antimicrobiana y ambiental (biopelículas) juegan en la adaptación y aparición de cepas más virulentas de este patógeno, con base a la diversidad genética mediante el análisis de datos genómicos que puedan relacionar la dinámica de dispersión, estacionalidad y trazabilidad. Esta información coadyuvará a la aplicación de las acciones correspondientes por parte de las autoridades pertinentes para el debido control de este patógeno y permitir con ello la disminución de la morbilidad y mortalidad ocasionada por Salmonella dentro de la población mexicana.
Agradecimientos
Los autores agradecen a la QFB. Célida Isabel Martínez Rodríguez y LI. Jorge Luis Manjarrez Vázquez por su asistencia técnica.
REFERENCIAS
Acedo-Félix, E., Núñez-Hernández, Y., Pérez-Morales, R., Iñiguez-Palomares, C, M. and Castillón-Campaña, L. (2009). Caracterización polifásica de Salmonella spp. aislada de campos agrícolas de melón (Cucumis melo) y cilantro (Coriandrum sativum). Interciencia, 34(6): 419-423. http://www.scielo.org.ve/scielo.php?script=sci_arttext&pid=S0378-18442009000600010. [ Links ]
Aguilar-Montes de Oca, S., Talavera-Rojas, M., Soriano-Vargas, E., Barba-Leon, J., Vazquez-Navarrete, J. and Acosta-Dibarrat, J. et al. (2017). Phenotypic and genotypic profile of clinical and animal multidrug-resistant Salmonella enterica isolates from Mexico. Journal of Applied Microbiology, 124: 67-74. https://doi.org/10.1111/jam.13615 [ Links ]
Akil, L., Ahmad, H.A. and Reddy, R.S. (2014). Effects of Climate Change on Salmonella infections. Foodborne Pathogens Disease, 11: 974-980. https://doi.org/10.1089/fpd.2014.1802. [ Links ]
Alaniz-de la O, R., Ríos-Ibarra, M.L., Rosas-Barbosa, B.T. and Juan-Morales, A.L. (1996). Resistencia a antimicrobianos de cepas de Salmonella aisladas de fuentes animales. Veterinaria México, 28(3): 215-220. www.medigraphic.com/cgi-bin/resumen.cgi?IDARTICULO=16105 [ Links ]
Alcázar-Montañez, C.D., Rubio, L.M.S., Núñez, E.F. and Alonso, M.R.A. (2006). Detección de Salmonella spp y Listeria monocytogenes en quesos frescos y semimadurados que se expenden en vía pública en la ciudad de México. Veterinaria México, 37(4):417-429. https://www.redalyc.org/articulo.oa?id=42337402 [ Links ]
Angulo, F.J., Nargund, V.N. and Chiller, T.C. (2004). Evidence of an association between use of anti-microbial agents in food animals and antimicrobial resistance among bacteria isolated from humans and the human health consequences of such resistance. Journal of veterinary medicine. B, Infectious diseases and veterinary public health, 51: 374-379. https://doi.org/10.1111/j.1439-0450.2004.00789.x [ Links ]
Annous, B.A., Solomon, E.B., Cooke, P.H. and Burke. A (2005). Biofilm formation by Salmonella spp on cantaloupe melons. Journal of Food Safety, 25:276.287. https://doi.org/10.1111/j.1745-4565.2005.00024.x [ Links ]
Becerra-Tapia, N. & Botello, A.V. (1995). Bacterias coliformes totales, fecales y patógenas en el sistema lagunar ChantutoPanzacola, Chiapas, México. Hidrobiológica, 5: 87-94. http://www.redalyc.org/pdf/578/57850207.pdf [ Links ]
Bello-Pérez, L.A., Ortiz-Dillanes, D.M., Pérez-Memije, E. and Castro-Domínguez, V. (1990). Salmonella en carnes crudas: un estudio en localidades del estado de Guerrero. Salud Pública de México, 32(1): 74-79. http://www.redalyc.org/pdf/106/10632110.pdf [ Links ]
Benavides-Plasencia, L., Aldama-Ojeda, A.L. and Vázquez, H.J. (2005). Vigilancia de los niveles de uso de antibióticos y perfiles de resistencia bacteriana en hospitales de tercer nivel de la Ciudad de México. Salud Pública de México, 47(3): 219-226. http://www.medigraphic.com/cgi-bin/new/resumen.cgi?IDARTICULO=6615 [ Links ]
Bessudo, M.D., Olarte, J., Mendoza-Hernández, P., Galindo, E., Carrillo, J. and Gutiérrez-Trujillo, G. (1973). Aislamiento de S. typhi resistente a altas concentraciones de cloranfenicol. Boletin de la Oficina Sanitaria Panamericana, 1-6. http://iris.paho.org/xmlui/bitstream/handle/123456789/10871/v74n1p1.pdf?sequence=1 [ Links ]
Boddicker, J.D., Ledeboer, N.A., Jagnow, J., Jones, B.D. and Clegg, S. (2002). Differential binding to and biofilm formation on, HEp-2 cells by Salmonella enterica Serovar Typhimurium is dependent upon allelic variation in the fimH gene of the fim gene cluster. Molecular Microbiology, 45(5): 1255-1265. https://doi.org/10.1046/j.13652958.2002.03121.x [ Links ]
Brenner, F.W., Villar, R.G., Angulo, F.J., Tauxe, R. and Swaminathan, B. (2000). Salmonella Nomenclature. Journal of Clinical Microbiology, 38(7): 2465-2467. https://jcm.asm.org/content/jcm/38/7/2465.full.pdf [ Links ]
Camacho, R.O., Acedo, R.L.E., Moreno, I.G.M., Sánchez, M.R.I., Castillón, C.L.G. and Navarro, N.M. (2010). Detección de Salmonella resistente a los antibióticos en vísceras de pollo. Biotecnia, XII: 3-11. https://es.scribd.com/document/286356725/Salmonella# [ Links ]
CDC (Center for Disease Control and Prevention) (2013). Surveillance for foodborne disease outbreaks. United States (2009-2010) Morbidity and Mortality Weekly Report (MMWR), 62: 41-47. https://www.ncbi.nlm.nih.gov/pubmed/23344696 [ Links ]
CDC (Center for Disease Control and Prevention) (2018a). Salmonellosis-nontyphoidal. https://wwwnc.cdc.gov/travel/yellowbook/2018/infectious-diseases-related-to-travel/salmonellosis-nontyphoidal [ Links ]
CDC (Center for Disease Control and Prevention) (2018b). Salmonella Home Page. https://www.cdc.gov/salmonella/index.html [ Links ]
Cerna-Cortes, J.F., Gómez-Aldapa, C., Rangel-Vargas, E., Ramírez-Cruz, E. and Castro-Rosas, J. (2013). Presence of indicator bacteria, Salmonella and diarrheagenic Escherichia coli pathotypes on mung bean sprouts from public markets in Pachuca, Mexico. Food Control, 31: 280-283. https://doi.org/10.1016/j.foodcont.2012.10.010 [ Links ]
Charles-Hernández, G.L., Medina-Solis, C.E. and Hernández-Romano, J. (2007). Prevalencia de Salmonella sp en alimentos en el estado de Tamaulipas durante el año 2005. Revista de Investigación Clínica, 59(6): 437-443. http://www.medigraphic.com/pdfs/revinvcli/nn-2007/nn076e.pdf [ Links ]
Chávez-de la Peña, M.E., Higuera-Iglesias, A.L., Huertas-Jiménez, M.A., Báez-Martínez, R., Morales-de León, J. and Arteaga-Cabello, F. (2001). Brote por Salmonella Enteritidis en trabajadores de un hospital. Salud Pública de México, 43: 211-216. http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S0036-36342001000300006 [ Links ]
CONAGUA (Comisión Nacional del Agua, Sistema Meteorológico Nacional). (2013) Reporte del Clima en México. http://smn1conagua.gob.mx/climatologia/analisis/reporte/Anual2013.pdf [ Links ]
Constantin, O.E. (2009). Bacterial biofilm formation at air liquid interfaces. Innovative Romanian Food Biotechnology, 5: 18-22. http://www.bioaliment.ugal.ro/revista/5/Paper%2053.pdf [ Links ]
Costerton, J.W., Stewart, P.S. and Greenberg, E.P. (1999). Bacterial biofilms: A common cause of persistent infections. Science, 284: 1318-1322. http://doi.org/10.1126/science.284.5418.1318 [ Links ]
Cueto-Medina, S.M., Castillo-Martínez, A., Hernández-Rodríguez, S., Gallegos-Robles, M.A., Sánchez-Ramos, F.J. and Ortega-Morales, A.I. (2015). Presencia de Salmonella sp. en cucaracha americana Periplaneta americana L. (Blattodea: Blattidae) en Torreón, Coahuila, México. Entomología Mexicana, 2: 762-766. https://www.researchgate.net/publication/327381839 [ Links ]
De los Santos, V.A.A., Hernández, A.A.M., Eslava, C.C.A., Landa, S.P., Mora, A.G. and Luchansky, J.B. (2012). Biofilm production and resistance to disinfectants in Salmonella strains isolated from prickly pear, water and soil. Revista Mexicana de Ciencias Agrícolas, 3(6): 1063-1074. http://www.redalyc.org/articulo.oa?id=263123222001 [ Links ]
DGE (Dirección General de Epidemiología). (2017). Anuario de Morbilidad, 1984 -2017. http://www.epidemiologia.salud.gob.mx/anuario/html/anuarios.html [ Links ]
Donlan, R.M., & Costerton, J.W. (2002). Biofilms: survival mechanism of clinically relevant microorganisms. Clinical Microbiology Reviews, 15: 167-193. https://cmr.asm.org/content/15/2/167.short [ Links ]
Escartin, E.F., Castillo, A., Hinojosa-Puga, A. and Saldaña-Lozano, J. (1999). Prevalence of Salmonella in chorizo and its survival under different storage temperatures. Food Microbiology, 16: 479-486. https://doi.org/10.1006/fmic.1999.0258 [ Links ]
EFSA (Autoridad Europea de Seguridad Alimentaria). (2015). The European Union summary report on trends and sources of zoonoses, zoonotic agents and food borne outbreaks. EFSA Journal, 13: 3991. https://doi.org/10.2903/j.efsa.2015.3991 [ Links ]
Estrada-Acosta, M., Jiménez, M., Chaidez, C., León-Félix, J. and Castro-del Campo, N. (2014). Irrigation water quality and the benefits of implementing good agricultural practices during tomato (Lycopersicum esculentum) production. Environmental Monitoring and Assessment, 186: 4323-30. https://doi.org/10.1007/s10661-014-3701-1 [ Links ]
Estrada-Acosta, M.D., Ramirez, K., Medrano-Félix, J.A., Castro-Del Campo, N., López-Moreno, H.S., Jimenez- Edeza, M., Martínez-Urtaza, J. and Chaidez, C. (2017). Effect of river water exposition on adhesion and invasion abilities of Salmonella Oranienburg and Saintpaul. International Journal of Environmental Health Research, 28(1): 43-54. https://doi.org/10.1080/09603123.2017.1415308 [ Links ]
Estrada-García, T., López-Saucedo, C., Zamarripa-Ayala, B., Thompson, M.R., Gutierrez-Cogco, L. and Mancera-Martínez, A. (2004). Prevalence of Escherichia coli and Salmonella spp in street-vended food of open markets (tianguis) and general hygienic and trading practices in Mexico City. Epidemiology and Infection, 132: 1181-1184. https://doi.org/10.1017/S0950268804003036 [ Links ]
Félix-Fuentes, A., Campas-Baypoli, O.N. and Meza-Montenegro, M. (2005). Calidad sanitaria de alimentos disponibles al público de Ciudad Obregón, Sonora, México. Revista Salud Pública y Nutrición, 6: 1-14. http://respyn.uanl.mx/index.php/respyn/article/view/149/131 [ Links ]
Fernández-Escartín, E. & Torres-Vitela, M.R. (1996). Contaminación del ceviche de pescado por Salmonella en Guadalajara, Jalisco, México. Boletín de la Oficina Sanitaria Panamericana, 120: 198-203. http://iris.paho.org/xmlui/handle/123456789/15493 [ Links ]
Figueroa, A.G.A., González, R.M., Molina, G.A., Yáñez, G.R., Espinoza, N.J. and Serna, E.M.C. (2005). Identificación de Salmonella spp en agua, melones cantaloupe y heces fecales de iguanas en una huerta melonera. Medicina Interna de México, 21: 255-258. http://www.medigraphic.com/pdfs/medintmex/mim-2005/mim054c.pdf [ Links ]
Gallegos-Robles, M.A., Morales-Loredo, A., Álvarez-Ojeda, G., Vega-P, A., Chew-M, and Velarde, S. (2008). Identification of Salmonella Serotypes isolated from Cantaloupe and Chile Pepper Production Systems in Mexico by PCR-Restriction Fragment Length Polymorphism. Journal of Food Protection, 77: 2217-2222. https://doi.org/10.4315/0362-028X-71.11.2217 [ Links ]
Gallegos-Robles, M.A., Morales-Loredo, A., Álvarez-Ojeda, G., Osuna-García, J.A., Martínez, I.O. and Morales-Ramos. (2009). PCR Detection and Microbial isolation of Salmonella spp from Fresh beef and Cantaloupes. Journal of Food Science, 74(1): M37-M40. https://doi.org/10.1111/j.1750-3841.2008.01006.x [ Links ]
García-Gómez, R., Chávez-Espinoza, J., Mejía-Chávez, A. and Durán-de-Bazúa, C. (2002). Microbiological determinations of some vegetable from the Xochimilco zone in Mexico City, Mexico. Revista Latinoamerica de Microbiología, 44: 24-30. http://www.medigraphic.com/pdfs/lamicro/mi-2002/mi021e.pfd [ Links ]
Gómez-Aldapa, C.A., Rangel-Vargas, E., Bautista-De León, H. and Castro-Rosas, J. (2014). Presence of non-0157 Shiga toxin-producing Escherichia coli, enterotoxigenic E. coli, enteropathogenic E. coli and Salmonella in fresh beetroot (Beta vulgaris L.) juice from public markets in Mexico. Journal of the Science of Food and Agriculture, 94: 2705-2711. https://doi.org/10.1002/jsfa.6614 [ Links ]
González-Cortés, A., Bessudo, D., Sánchez-Leyva, R., Fragoso, R., Hinojosa, M. and Becerril, P. (1974). Water-borne transmission of chloramphenicol-resistant Salmonella Typhi in Mexico. The lancet, 302: 605-607. http://doi.org/10.1016/S0140-6736(73)92427-6 [ Links ]
Grabert, K. (1968). Incidencia de Salmonella, Shigella y Coli enteropatógeno en el perro en la ciudad de México. Técnica Pecuaria, 28-30. http://cienciaspecuarias.inifap.gob.mx/index.php/Pecuarias/article/view/1972/3532 [ Links ]
Gunn, J.S., Marshall, J.M., Baker, S., Dongol, S., Charles, R.C. and Ryan, E.T. (2014). Salmonella chronic carriage: epidemiology, diagnosis and gallbladder persistence. Trends in Microbiology, 22: 648-655. https://doi.org/10.1016/j.tim.2014.06.007 [ Links ]
Gutiérrez-Cogco, L., Montiel-Vázquez, E., Aguilera-Pérez, P. and González-Andrade, M.C. (2000). Serotipos de Salmonella identificados en los servicios de salud en México. Salud Pública de México, 42(6): 490-495.https://www.scielosp.org/scielo.php?pid=S0036-36342000000600004&script=sci_arttext&tlng=es [ Links ]
Herman, K.M., Hall, A.J. and Gould, L.H. (2015). Outbreaks attributed to fresh leafy vegetables, United States, 1973-2012Epidemiology and Infection, 143(14): 3011-3021. https://doi.org/10.1017/S0950268815000047 [ Links ]
Hernández, S.J.S., Zúñiga, E.A., Sánchez, O.I., Castro, R.J., Román, G.A.D. and Santos, L.E.M. (2007). Microbiological conditions during the slaughter process at a municipal slaughterhouse in Hidalgo, Mexico. Veterinaria México, 38(2): 187-195. http://www.medigraphic.com/pdfs/vetmex/vm-2007/vm072epdf [ Links ]
Hernández-Domínguez, C., Hernández-Anguiano, A.M., Chaidez-Quiroz, C., Rendón-Sánchez, G. and Suslow, T.V. (2008). Detection of Salmonella and fecal coliforms in water for agricultural use destined to melon “Cantaloupe”. Agricultura Técnica en México, 34(1): 75-84. http://www.scielo.org.mx/pdf/agritm/v34n1a9.pdf [ Links ]
INEGI (Instituto Nacional de Estadística y Geografía). (2017). Anuario estadístico y geográfico de los Estados Unidos Mexicanos. http://internet.contenidos.inegi.org.mx/contenidos/Productos/prod_serv/contenidos/espanol/bvinegi/productos/nueva_estruc/AEGEUM_2017/702825097912.pdf [ Links ]
Jensen, P.Ø., Givskov, M., Bjarnsholt, T. and Moser, C. (2010). The immune system vs. Pseudomonas aeruginosa biofilms. FEMS Immunology and Medical Microbiology, 59(3): 292-305. https://doi.org/10.1111/j.1574-695X.2010.00706.x [ Links ]
Jiang, C., Shaw, K.S., Upperman, C.R., Blythe, D., Mitchell, C. and Murtuguddes. (2015). Climate change, extreme events and increased risk of salmonellosis in Maryland, USA: Evidence for coastal vulnerability. Enviroment International, 83: 58-62. https://doi.org/10.1016/j.envint.2015.06.006 [ Links ]
Jiménez, M., Martínez-Urtaza, J. and Chaidez, C. (2011). Geographical and Temporal Dissemination of Salmonellae Isolated from Domestic Animal Hosts in the Culiacan Valley, Mexico. Microbial Ecology, 61: 811-820. https://doi.org/10.1007/s00248-010-9792-5 [ Links ]
Jiménez, M., Martinez-Urtaza, J., Rodriguez-Alvarez, M.X., Leon-Felix, J. and Chaidez, C. (2014). Prevalence and genetic diversity of Salmonella spp. in a river in a tropical environment in Mexico. Journal Of Water And Health, 12(4): 874-884. https://doi.org/10.2166/wh.2014.051 [ Links ]
Ledeboer, N.A. & Jones, B.D. (2005). Exopolysaccharide Sugars Contribute to Biofilm Formation by Salmonella enterica Serovar Typhimurium on HEp-2 Cells and Chicken Intestinal Epithelium. Journal of Bacteriology, 187: 3214-3226. http://doi.org/10.1128/JB.187.9.3214-3226.2005 [ Links ]
Lee, J.S., Mogasale V.V., Mogasale, V. and Lee, K. (2016). Geographical distribution of typhoid risk factors in low and middle income countries. BMC Infectious Diseases, 16: 732. http://doi.org/10.1186/S12879-016-2074-1 [ Links ]
Levantesi, C., Bonadonna, L., Briancesco, R., Grohmann, E., Toze, S. and Tandoi, V. (2012). Salmonella in surface and drinking water: ocurrence and water- mediated transmission. Food Research International, 45: 587-602. http://doi.org/10.1016/j.foodres.2011.06.037 [ Links ]
López-Cuevas, O., León-Félix, J., Jiménez-Edeza, M. and Chaidez-Quiroz, C. (2009). Detección y resistencia a antibióticos de Escherichia coli y Salmonella en agua y suelo agrícola. Revista Fitotecnia Mexicana, 32: 119-126. http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S0187-73802009000200007 [ Links ]
Lugo-Melchor, Y., Quiñones, B., Amézquita-López, B.A., León-Félix, J., García-Estrada, R. and Chaidez, C. (2010). Characterization of Tetracycline Resistance in Salmonella enterica Strains Recovered from Irrigation Water in the Culiacan Valley, Mexico. Microbial Drug Resistance, 16(3): 185-190. https://doi.org/10.1089/mdr.2010.0022 [ Links ]
Mancera, M.A., Vázquez, N.J., Ontiveros, C.M.L., Durán, V.S., López, H. D. and Tenorio, G.V.R. (2005). Identificación de Salmonella Enteritidis en huevo para consumo en la ciudad de México. Técnica Pecuaria en México, 43(2): 229-237. https://cienciaspecuarias.inifap.gob.mx/index.php/Pecuarias/article/view/1377 [ Links ]
Martínez, B.C., Gallegos, D.C., Bär, W., Márquez de Bär, G., Fernández, C.R. and Ruiz, R.G. (1999). Reptiles “mascotas”: una fuente potencial de infecciones por Salmonella. Enfermedades Infecciosas y Microbiología, 19: 266-269. http://www.medigraphic.com/cgi-bin/new/resumen.cgi?IDARTICULO=27108 [ Links ]
Martínez-Urtaza, J., Liebana, E., Garcia-Migura, L., Perez-Piñeiro, P. and Saco, M. (2004). Characterization of Salmonella enterica serovar Typhimurium from marine environments in coastal waters of Galicia (Spain). Applied and Environmental Microbiology, 70: 4030-4034. http://doi.org/10.1128/AEM.70.7.4030-4034.2004 [ Links ]
Medrano-Félix, J.A., Chaidez, C., Mena, K.D., Soto-Galindo, M.S. and Castro-del Campo, N. (2018a). Characterization of biofilm formation by Salmonella enterica at the air-liquid interface in aquatic environments. Environmental Monitoring and Assessment, 190: 221. https://doi.org/10.1007/s10661-018-6585-7 [ Links ]
Medrano-Félix, J.A., Castro-del Campo, N., Peraza, G.F.J., Martínez-Rodríguez, C.I. and Chaidez, C. (2018b). Carbon source utilization-based metabolic activity of Salmonella Oranienburg and Salmonella Saintpaul in river water. Water and Enviromental Journal, 32: 118-124. https://doi.org/10.1111/wej.12306 [ Links ]
Miranda, J.M., Mondragón, A.C., Martínez, B., Guarddon, M. and Rodríguez, J.A. (2009). Prevalence and Antimicrobial Resistance Patterns of Salmonella from Different Raw Foods in Mexico. Journal of Food Protection, 72(5): 966-971. http://doi.org/10.4315/0362-028X-72.5.966 [ Links ]
Morales-Hernández, l., Hernández-Anguiano, A.M., Chaidez-Quiroz, C., Rendón-Sánchez, G. and Suslow, T.V. (2009). Detection of Salmonella spp. on cantaloupe melón production unit and packaging facility. Agricultura Técnica en México, 35: 135-145. http://www.redalyc.org/articulo.oa?id=60812688001 [ Links ]
Nayarit-Ballesteros, N., Rubio-Lozano, M.S., Delgado-Suárez, E., Méndez-Medina, D., Braña-Varela, D. and Rodas-Suárez, O. (2016). Perfil de resistencia a antibióticos de serotipos de Salmonella spp aislados de carne molida en la Ciudad de México. Salud Pública de México, 58: 371-377. https://doi.org/10.21149/spm.v58i3.7897 [ Links ]
Olarte, J. & Galindo, E. (1973). Salmonella Typhi resistant to Chloramphenicol, Ampicillin, and Other Antimicrobial Agents: Strains isolated During an Extensive Typhoid Fever Epidemic in Mexico. Antimicrobial agents and chemotherapy, 4: 597-601. http://doi.org/10.1128/AAC.4.6.597 [ Links ]
Paniagua-Contreras, G.L., Monroy-Pérez, E. and Vaca-Pacheco, S. (2008). Prevalencia de Salmonella spp en diarrea de niños de una zona urbana del Estado de México. Revista Médica del Hospital General de México, 71(4): 192-198. http://www.medigraphic.com/pdfs/h-gral/hg-2008/hg084d.pdf [ Links ]
Painter, J. A., Hoekstra, R. M., Ayers, T., Tauxe, R. V., Braden, C. R., Angulo, F. J. and Griffin, P. M. (2013). Attribution of Foodborne Illnesses, Hospitalizations, and Deaths to Food Commodities by using Outbreak Data, United States, 1998-2008. Emerging Infectious Diseases, 19(3): 407-415. http://dx.doi.org/10.3201/eid1903.111866 [ Links ]
Pérez-Montaño, J.A., González-Aguilar, D., Barba, J., Pacheco-Gallardo, C. and Campos-Bravo, C.A., García, S., et al. (2012). Frequency and antimicrobial resistance of Salmonella serotypes on beef carcasses at small abattoirs in Jalisco State, Mexico. Journal of Food Protection, 75(5): 867-73. https://doi.org/10.4315/0362-028X.JFP-11-423 [ Links ]
Quiñonez-Ramírez, E., Vázquez-Salinas, C., Pedroche, F.F., Moreno-Sepúlveda, L. and Rodas-Suárez, O.R. (2000). Presencia de los géneros Vibrio y Salmonella, y detección de coliformes fecales en almejas del Golfo de México. Hidrobiológica, 10: 131-138. http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S0188-88972000000200008 [ Links ]
Quiroz-Santiago, C., Rodas-Suárez, O.R., Vázquez, Q.C.R., Fernández, F.J., Quiñonez-Ramírez, E.I. and Vázquez-Salinas, C. (2009). Prevalence of Salmonella in vegetables from Mexico. Journal of Food Protection, 72: 1279-1282. https://doi.org/10.4315/0362-028X-72.6.1279 [ Links ]
Reller, M. E., Tauxe, R.V., Kalish, L. A. and Molbak, K. (2008). Excess salmonellosis in women in the United States: 1968- 2000. Epidemiology and Infection, 136: 1109-1117. https://doi.org/10.1017/S0950268807009594 [ Links ]
Rubio, L.M.S., Martínez, B.J.F., Hernández, C.R., Bonilla, C.C., Méndez, M.R.D. and Núñez, E.J.F. (2013). Detección de Listeria monocytogenes, Salmonella y Yersinia enterocolitica en carne de res en puntos de venta en México. Revista Mexicana de Ciencias Pecuarias, 4: 107-115. http://www.scielo.org.mx/pdf/rmcp/v4n1/v4n1a9.pdf [ Links ]
Silva-Hidalgo, G., Ortiz-Navarrete, V.F., Alpuche-Aranda, C.M., Rendón-Maldonado, J.G., López-Valenzuela, M. and Juárez-Barranco, F. (2012). Non-typhi Salmonella serovars found in Mexican zoo animals. Research in Veterinary Science, 93: 1132-1135. https://doi.org/10.1016/j.rvsc.2012.03.005 [ Links ]
Silva-Hidalgo, G., López-Valenzuela, M., Cárcamo-Aréchiga, N., Cota-Guajardo, S., López-Salazar, M. and Montiel-Vázquez, E. (2016). Identification of bapA in strains of Salmonella enterica subsp. enterica Isolated from Wild Animals Kept in Captivity in Sinaloa, Mexico. Veterinary Medicine International, 2016: 1-4. http://dx.doi.org/10.1155/2016/3478746 [ Links ]
Simental, L. & Martínez-Urtaza, J. (2008). Climate Patterns Governing the Presence and Permanence of Salmonellae in Coastal Areas of Bahia de Todos Santos, México. Applied and Enviromental Microbiology, 74: 5918-5924. http://doi.org/10.1128/AEM.01139-08. [ Links ]
Steenackers, H., Hermans, K., Vanderleyden, J. and De Keersmaecker, S.C.J. (2012). Salmonella biofilms: An overview on occurrence, structure, regulation and eradication. Food Research International, 45: 502-531. https://doi.org/10.1016/j.foodres.2011.01.038 [ Links ]
Talavera, R.M., Varela, G.J.A., Reyes, R.N.E., Lagunas, B.S., Valladares, C.B., Alonso, F.M.U. and Velázquez O.V.V. (2011). Resistencia antibiótica de genotipos de cepas de Salmonella spp de cerdos sacrificados en rastros del Estado de México. Veterinary Mexico, 42: 269-276. http://www.scielo.org.mx/scielo.php?pid=S030150922011000400002&script=sci_arttext [ Links ]
Tamagnini, L.M. & Paraje, M.G. (2015). ¿Qué son las bacterias viables no cultivables? Revista de la Facultad de Ciencias Exactas, Físicas y Naturales, 2(2): 99-102. https://revistas.unc.edu.ar/index.php/FCEFyN/article/viewFile/10745/12674 [ Links ]
Torres, V.M.R., Navarro, H.V., Villarruel, L.A., Olea, R.M.A. (2011). Prevalence of Salmonella and Staphylococcus aureus in chorizo and longaniza. NACAMEH, 5: S96-S107. https://dialnet.unirioja.es/servlet/articulo?codigo=4024306 [ Links ]
Varela-Guerrero, J., Talavera-Rojas, M., Gutiérrez-Castillo, A.C., Reyes-Rodríguez, N.E. and Vázquez-Guadarrama, J. 2013. Phenotypic-genotypic resistance in Salmonella spp. isolated from cattle carcasses from the north central zone of the State of Mexico. Tropical Animal Health and Production, 45: 995-1000. https://doi.org/10.1007/s11250-012-0323-x [ Links ]
Vázquez-Garcidueñas, M.S., Romero-Pérez, N.L., Figueroa-Aguilar, G.A., Jaime-Sánchez, J.L. and Vázquez-Marrufo, G. (2014). Investigation of a food-borne Salmonella Oranienburg outbreak in a Mexican prison. Journal of Infection in Developing Countries, 8(2): 143-153. http://doi.org/10.3855/jidc.3367 [ Links ]
Winfield, M.D. & Groisman, E.A. (2003). Role of nonhost Environments in the Lifestyles of Salmonella and Escherichia coli. Applied and Environmental Microbiology, 69: 3687-3694. http://doi.org/10.1128/AEM.69.7.3687-3694.2003 [ Links ]
WHO (World Health Organization). (2016). Antimicrobial resistance - World Health Organization. http://www.who.int/en/news-room/fact-sheets/detail/antimicrobial-resistance [ Links ]
WHO (World Health Organization). (2018a). Salmonella (non-typhoidal). http://www.who.int/en/news-room/fact-sheets/detail/salmonella-(non-typhoidal) [ Links ]
WHO (World Health Organization). (2018b). Climate change and human health. http://www.who.int/globalchange/summary/en/index1.html [ Links ]
Wu, X., Lu, Y., Zhou, S. and Chen, L., Xu, B. (2016). Impact of climate change on human infectious diseases: Empirical evidences and human adaptation. Environment International, 86: 14-23. https://doi.org/10.1016/j.envint.2015.09.007 [ Links ]
Zaidi, M.B., McDermont,P.F., Fedorka-Cray, P., Leon, V., Canche, C. and Hubert, S.K. (2006) Nontyphoidal Salmonella from Human Clinical Cases, Asymptomatic Children, and Raw Retail Meats in Yucatan, Mexico. Clinical Infectious Diseases, 42: 21-28. https://doi.org/10.1086/498508 [ Links ]
Zaidi, M.B., Leon, V., Canche, C., Perez, C.; Zhao, S., Hubert, S.K., Abbott, J, Blickenstaff, K. and McDermott, P. (2007). Rapid and widespread dissemination of multidrug-resistant blaCMY-2 Salmonella Typhimurium in Mexico. Journal of Antimicrobial Chemotherapy, 60: 397-401. https://doi.org/10.1093/jac/dkm168 [ Links ]
Zaidi, M.B., Calva, J.J., Estrada-García, M.T., León, V., Vázquez, G. and Figueroa, G. (2008). Integrated Food Chain Surveillance System for Salmonella spp in Mexico. Emerging Infectious Disease, 14: 429-435. http://doi.org/10.3201/ eid1403.071057 [ Links ]
Zaidi, M.B., Campos, F.D., Estrada-García, T., León, M., Chim, R. and Calva, J. (2012). Burden and Transmission of Zoonotic Foodborne Disease in a Rural Community in Mexico. Clinical Infectious Disease, 55: 51-60. http://doi.org/10.1093/cid/cis300. [ Links ]
Zhang, Y., Bi, P. and Hiller, J.E. (2010). Climate variations and Salmonella infection in Australian subtropical and tropical regions. Science of The Total Environment, 408: 524-530. https://doi.org/10.1016/j.scitotenv.2009.10.068 [ Links ]
Cite this paper/Como citar este artículo: Contreras-Soto, M. B., Medrano-Félix, J. A., Ibarra-Rodríguez, J. R., Martínez-Urtaza, J., Chaidez, Q. C., Castro-del Campo, N. (2019). The last 50 years of Salmonella in Mexico: Sources of isolation and factors that influence its prevalence and diversity. Revista Bio Ciencias 6(2), e540. doi: http://dx.doi.org/10.15741/revbio.06.02.02
Recibido: 09 de Julio de 2018; Aprobado: 05 de Octubre de 2018











 texto en
texto en 


