Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista bio ciencias
versão On-line ISSN 2007-3380
Revista bio ciencias vol.6 Tepic Jan. 2019 Epub 02-Out-2020
https://doi.org/10.15741/revbio.06.e595
Artículos Originales
Especies de Lasiodiplodia en lima ‘Persa’ (Citrus latifolia Tanaka) en Morelos, México
1 Facultad de Ciencias Agropecuarias, Universidad Autónoma del Estado de Morelos (UAEM), Av. Universidad No. 1001, Col Chamilpa, Cuernavaca, Morelos, México. C.P. 62209.
2 Escuela de Estudios Superiores de Xalostoc, Universidad Autónoma del Estado de Morelos (UAEM), Av. Nicolás Bravo S/N, Parque Industrial Cuautla, Xalostoc, Ayala, Morelos, México. C.P. 62717.
3 Centro Nacional de Investigación Disciplinaria en Conservación y Mejoramiento de Ecosistemas Forestales-INIFAP, Coyoacán. México, D. F.
4 Centro de Investigaciones Biológicas. Universidad Autónoma del Estado de Morelos (UAEM), Av. Universidad No. 1001, Col Chamilpa, Cuernavaca, Morelos, México. C.P. 62209.
5 Instituto Nacional de Investigaciones Forestales Agrícolas y Pecuarias (INIFAP), Campo Experimental Zacatepec, Carretera Zacatepec-Galeana Km. 0.5, Zacatepec, Morelos, México.
6 INIFAP-Campo Experimental Iguala. Carretera Iguala - Tuxpan Km. 2.5, Tuxpan, Iguala de la Independencia, Guerrero, México. C.P. 40000.
Muestras de árboles de lima ‘Persa’, de 5 a 8 años de edad, con síntomas de gomosis, canceres y muerte descendente de ramas fueron colectadas en huertos de los municipios de Tlaltizapán, Ayala, Coatlán del Río y Amayuca en el estado de Morelos, México, durante agosto del 2015. Las muestras fueron procesadas mediante procedimientos de rutina, sembradas en papa-dextrosa-agar y purificadas mediante puntas de hifa. Los patógenos aislados fueron inoculados en plantas sanas para evaluar su patogenicidad. Se realizó la identificación morfológica y molecular y se construyó un árbol filogenético bayesiano con agrupamiento de clados. Con base a caracteres morfológicos se identificó a Lasiodiplodia sp. El análisis molecular determinó 16 aislados, que se agruparon en 4 clados y tres especies, Lasiodiplodia citricola, L. pseudotheobromae y L. theobromae, las cuales son consideradas como nuevos reportes causando enfermedad en lima ‘Persa’ para el Estado de Morelos, México.
Palabras clave: Identificación molecular; Lasiodiplodia theobromae; Lasiodiplodia pseudotheobromae; Lasiodiplodia citricola
Samples of ‘Persian’ lime trees, from 5 to 8 years old, with symptoms of gummosis, cankers and dieback of branches were collected in orchards in the municipalities of Tlaltizapan, Ayala, Coatlan del Rio and Amayuca in the state of Morelos, Mexico, during August 2015. The samples were processed by routine procedures and sown in potato dextrose-agar and purified by means of hypha tips. The isolated pathogens were inoculated in healthy plants in order to evaluate their pathogenicity. Morphological and molecular identification was carried out and a Bayesian phylogenetic tree was constructed with clades grouping. Based on morphological characters, Lasiodiplodia sp. was identified. The molecular analysis allows to determine 16 isolates, which were grouped into 4 clades and three species, Lasiodiplodia citricola, L. pseudotheobromae and L. theobromae, which are considered as new reports, causing disease in ‘Persian’ lime for the State of Morelos, Mexico.
Key words: Molecular identification; Lasiodiplodia theobromae; Lasiodiplodia pseudotheobromae; Lasiodiplodia citricola
Introducción
La producción de cítricos en México, en especial de lima ‘Persa’ (Citrus latifolia Tanaka) se ha orientado al consumo fresco y es líder en exportación de frutos a escala mundial (Schwentesius et al., 2005).
La producción nacional en México en el 2016, fue de 5.75 millones de toneladas con un valor de $16,781 millones de pesos, para ese año la superficie plantada de cítricos fue de 533,749 ha, de las cuales 180,252 ha fueron de limón mexicano (Citrus aurantifolia (Christm) Swing), 178,877 ha de toronja (Citrus paradisi Macf.) y 335,610 ha de naranja ‘Valencia’ (Citrus sinensis (L.) Osbeck) (SIAP, 2017).
En el estado de Morelos, México en el 2010 se tenían huertas de cítricos establecidas en 28 de los 33 municipios, con 1000 ha de superficie plantada, con alta producción y calidad de exportación. Actualmente se cuenta con una superficie establecida de 612 ha, en 17 municipios, donde se destaca el limón y la naranja ‘Valencia’, con 32 y 66 % de la superficie total respectivamente, y un rendimiento de 12.11 y 7.76 t ha-1 respectivamente, siendo ambas especies las de mayor importancia económica en el estado de Morelos, el resto de la superficie está compuesta por otros cítricos (SIAP, 2017; CESVMOR, 2018). La presencia generalizada de árboles de lima ‘Persa’ con síntomas de gomosis, cáncer, muerte regresiva de ramas y muerte de árboles ha provocado una reducción en la productividad de los huertos y el aumento de los costos de producción. Las recomendaciones técnicas en el Estado se centran en el uso de fungicidas específicos para el control de varias especies de Phytophthora, agente causal de la gomosis de cítricos en otros estados de la República Mexicana (Acosta-Pérez et al., 2012), sin embargo, los resultados no son satisfactorios. Los síntomas observados en árboles de lima ‘Persa’ coinciden con los causados por Lasiodiplodia spp en varias especies de árboles (Abdollahzadeh et al., 2010). El primer reporte de este fitopatógeno fue realizado por Álvarez (1976) en el cultivo de cacao (Theobroma cacao). Reportes recientes indican que este patógeno se ha aislado de diversas especies de cítricos, en California, USA, se aisló de limón ‘Eureka’, naranja ‘Valencia’, naranja ‘Washington Navel’, naranja navel ‘Fukomoto’, toronja, mandarina ‘Satsuma’ y limón ‘Meyer’ (Adesemoye et al., 2014). También se ha reportado que especies de Lasiodiplodia son comunes en los trópicos, causando enfermedades en plantas, en pre y postcosecha y que su actividad celulolítica permite la penetración y colonización de la planta de manera similar a la de un hongo que causa pudrición blanda, utilizando el almidón y otros sacáridos presentes en el sustrato inicial de la madera (Muñoz et al., 2015). Las importaciones de otros frutales hospederos de este patógeno de otros países tales como la nuez y el nogal inglés (Li et al., 1995), uva (Linaldeddu et al., 2015; Rodríguez et al., 2015), naranja dulce, encino, retama: Fabaceae (Linaldeddu et al., 2015), naranjo agrio (Alves et al., 2008), coco (Dugan et al., 2015), mango (Johnson et al., 1992; Marques et al., 2013), papayo (Netto et al., 2014), álamo, sauce (Hashemi & Mohammadi, 2016) guapuruvú (Schizolobium parahyba var. Amazonicum) (Mehl et al., 2014), berenjena (Solanum melongena) (Woodward et al., 2005), níspero (Eriobotrya japonica) (González et al., 2017), yaca (Artocarpus heterophyllus) (Ni et al., 2008), palma (Dypsis lutescens) (Pereira et al., 2015), kenaf (Norhayati et al., 2016), Jatropha (Machado et al., 2014) y varias especies introducidas de árboles forestales, de otros países han facilitado la dispersión de este patógeno (Segura-Contreras et al., 2015).
Actualmente se tienen reportes en México en una gran diversidad de cultivos por toda la República Mexicana tales como: Jamaica (Hibiscus sabdariffa L.) en Guerrero (Aparicio-Apolinar et al., 2016), papaya (Carica papaya L.) en la costa central del Estado de Veracruz (Becerra-Leor et al., 2014), Heliconias en Tabasco (Ortiz-García et al., 2014), semillas de Jatropha curcas de colecciones de Puebla, Veracruz y Chiapas (Nolasco-Guzmán et al., 2014), sábila Aloe vera en Tamaulipas (Zúñiga-Estrada & Yáñez, 2016), aguacate (Persea americana), mango (Mangifera indica), coco (Cocos nucifera), yuca (Manihot esculenta), caña de azúcar (Sacharum officinarum), cítricos (Citrus spp.), café (Coffea arabica), ñame (Dioscorea alata), camote (Ipomea batata), maíz (Zea mays), ciruela (Prunnus domestica), agave (Agave sisalana), pinos (Pinus caribaeae) de las principales zonas productoras (Agroecología, 2018; Picos-Muñoz et al., 2015).
El impacto de las especies de hongos de la familia Botryosphaeriaceae sobre los cítricos no se ha explorado ampliamente, por lo que, es probable que la diversidad de hongos, de esta familia en cítricos no se conoce bien y, además esté causando más daño de lo que se pensaba anteriormente (Adesemoye et al., 2014).
La mayoría de las descripciones de este fitopatógeno se han basado en sus caracteres morfológicos, y es a partir de la década pasada que se ha complementado la identificación con técnicas de biología moleculares (Picos-Muñoz et al., 2015).
Según estudios realizados por Phillips et al. (2013), debido a la presencia cosmopolita, el amplio número de hospederos y la variabilidad morfológica de este patógeno, existen varias especies crípticas, que afectan las plantaciones citrícolas en México, las cuales están separadas generalmente por el tamaño y forma del conidio. Por tal motivo el objetivo de este trabajo fue aislar, identificar y diagnosticar molecularmente, el agente causal de la muerte descendente de ramas y gomosis en las plantaciones de lima ‘Persa’ en el Estado de Morelos, México.
Material y Métodos
Lugar del estudio
El estudio se realizó en el mes de agosto del 2015, en el estado de Morelos, México, ubicado geográficamente a los 22°51′43″ NL, 102°36′38″ WL y 2,309 m.a.m.s.l., en huertos comerciales de lima ‘Persa’.
Las plantaciones evaluadas se ubican en los municipios de Tlaltizapán, Ayala, Coatlán del Río y Amayuca, las cuales fueron seleccionadas a partir del padrón de productores de lima ‘Persa’ del Comité Estatal de Sanidad Vegetal.
Colecta del material biológico
El muestreo en las plantaciones se realizó acorde con la propuesta de Acosta-Pérez et al. (2012), se colectaron 46 muestras, procedentes de ramas primarias, secundarias y troncos de árboles de cinco a ocho años de edad, con exudados de goma, canceres y muerte descendente de ramas, las que fueron transportadas en bolsas de polietileno y conservadas a 6 °C, hasta su procesamiento en el laboratorio de Fitopatología de la Escuela de Estudios Superiores de Xalostoc de la UAEM, Xalostoc, Morelos, México (EESuX-UAEM).
Aislamiento del hongo
Las muestras fueron seccionadas en fracciones de 0.5 cm2, incluyendo la zona de avance de la enfermedad. Los fragmentos se desinfestaron por inmersión en una solución de hipoclorito de sodio (NaClO) al 2 % por 2 min y tres lavados con agua destilada estéril por 2 min cada uno, se secaron en papel absorbente estéril para eliminar el exceso de agua.
Posteriormente, se incubaron cuatro fragmentos de tejido por placa de Petri, que contenían medio de cultivo de agar de dextrosa de patata (PDA) bajo luz blanca a 24 °C ± durante tres días. Una vez que se produjo el crecimiento del hongo, las colonias se purificaron mediante la técnica de punta hifa, en medio de cultivo PDA para aumentar la colonia.
Los aislados monospóricos, fueron resembrados en medio de cultivo jugo de vegetales V8-Agar (Ortiz, 1996), posteriormente se almacenaron en tubos de ensayo con medio PDA cubiertos con aceite (Crous et al., 2006).
Identificación y caracterización morfológica de los aislados
La identificación de los aislados se realizó con la ayuda de las claves y descripciones taxonómicas de Phillips et al. (2013), basado en los caracteres morfológicos de las ascosporas, estados conidiales, pigmentación, grosor de la pared y forma de los conidios, dimensiones, septación, presencia de parafisos en los picnidios y estriaciones. La observación se llevó a cabo con un estereoscopio de campo claro, modelo Olympus®.
Pruebas de patogenicidad
Para comprobar la patogenicidad del hongo se realizaron los postulados de Koch y la técnica descrita por Agrios (2005).
Se inocularon tallos de plantas certificadas de lima ‘Persa’ de un año de edad, a una altura de 15 cm del suelo, los tallos se desinfectaron con hipoclorito de sodio (NaClO) al 1.5 %. Luego, con un bisturí se realizó una incisión a la corteza del tallo, de aproximadamente 7 mm. Posteriormente se inocularon con discos de 0.5 cm de aislado del hongo. La inoculación se realizó mediante la colocación de micelio con medio de cultivo en las aberturas de los tallos, el testigo solo recibió medio de cultivo sin él patógeno. Para favorecer la infección, el área inoculada se cubrió con algodón humedecido con agua destilada estéril y se resguardó con Parafilm®. Las plantas fueron mantenidas individualmente bajo condiciones a campo abierto. Se realizaron evaluaciones visuales cada 15 d por 3 meses, registrando la evolución de los síntomas de la enfermedad en las plantas inoculadas.
Al detectar los síntomas de la enfermedad, se procedió a hacer el reaislamiento, con el fin de verificar el hongo inoculado corresponde al hondo aislado originalmente.
Identificación molecular
La identificación molecular se realizó en el laboratorio de Fitopatología del Centro Nacional de Investigación Disciplinaria en Conservación y Mejoramiento de Ecosistemas Forestales Coyoacán, Ciudad de México, México.
La extracción del DNA genómico (DNAg), se llevó a cabo empleando el método AP descrito por Sambrook & Russell (2012), utilizando el micelio del cultivo monospórico de los aislamientos obtenidos. La calidad del mismo se evaluó por electroforesis horizontal en gel de agarosa al 1% (Ultrapure, Gibco, USA) y las bandas se visualizaron en un transiluminador (Gel Doc 2000, BIO RAD®, USA). La concentración de DNA se cuantificó con un espectrofotómetro NanoDrop 2000 (Thermo Scientific®).
Los cebadores empleados para la identificación molecular de los hongos fueron ITS5 (5’-TCC TCC GCT TAT TGA TAT GC-3’) e ITS4 (5’-GGA AGT AAA AGT CGT AAC AAG G-3’) de los genes ribosomales (rDNA).
El análisis de las muestras fue corroborado mediante PCR según el protocolo descrito por Ahrens & Seemüller (1992), con modificaciones en los componentes: agua ultrapura estéril (16.25 μL), solución amortiguadora TBE IX (2.5 μL), MgCl2 a 2.5 mM (1.25 μL), dNTPs a 0.2 mM (0.5 μL), iniciadores universales ITS5 e ITS4 a 20 mol (1 μL de cada uno), DNA polimerasa (Promega®) a 1U (0.5 μL) y 80 ng (2 μL) de DNA g extraído previamente según Sambrook & Russell (2012).
Análisis filogenético
Los fragmentos de DNA amplificados por PCR fueron purificados empleando el kit Wizard (Promega®, USA), siguiendo el protocolo recomendado por el fabricante. El producto de PCR purificado fue secuenciado por Macrogen Inc. (Seoul, Korea del Sur).
Las secuencias fueron ensambladas, y editadas con la opción CAP (Contig Assembly Program) del Software BioEdit v7.0.9.1 (Hall, 1999). Las secuencias de hongos fueron comparadas y depositadas en la base de datos del GenBank (NCBI, 2016).
Los árboles filogenéticos fueron construidos con los datos de las terminaciones de ITS5 e ITS4, además se analizaron con el software MEGA (Molecular Evolutionary Genetics Analisys) versión 7.0.14 (Swofford, 2003; Tamura et al., 2007), alineadas con ClustalW 1.8.1 (Thompson et al., 1994) y comparados con las secuencias de genes homólogos depositados en la base de datos de la National Center for Biotechnology Information (NCBI, 2016).
Después de analizar la congruencia entre los conjuntos de datos, se hizo un análisis de máxima parsimonia (MP), se realizaron los análisis filogenéticos en PAUP (Phylogenetic Analysis Using Parsimony) versión 4.0b10 (Swofford, 2002). Los árboles filogenéticos se obtuvieron con la función de búsqueda heurística con 1,000 repeticiones de adición aleatoria, bisección y reconstrucción de árboles (TBR) como algoritmo de intercambio de ramas y los espacios o datos faltantes fueron considerados deleciones completas. Los niveles de confianza de los puntos de ramificación de árboles fueron determinados mediante el análisis de bootstraping con 1,000 repeticiones (Kimura, 1980; Felsenstein, 1985; Hillis & Bull, 1993). La secuencia HQ231345 de Phoma tracheiphila de GenBank se utilizó como el taxón fuera de grupo.
Resultados y Discusión
Identificación y caracterización morfológica de los aislados
De los 46 árboles muestreados de lima ‘Persa’, se obtuvieron 16 aislados con síntomas en ramas primarias y secundarias con muerte descendente y exudados gomosos. Solo tres de ellos fueron identificados morfológicamente según las claves taxonómicas reportadas por Phillips et al. (2013).
En la observación al microscopio estereoscópico, se identificaron colonias con crecimiento de micelio gris ahumado, luego gris olivo a gris verdoso a azul pizarra oscuro y la presencia de picnidios a los 30 d de incubación. Así como conidios producidos y formados por una sola célula, de forma ovalada, hialinos y de doble pared con 17-26 X 12 μM de tamaño en colonias inmaduras. En colonias maduras, los conidios mostraron una coloración marrón oscura, dos células y estriaciones y 18-28 X 11-16 μM de tamaño, siendo similar con estructuras reproductivas asexuales del género Lasiodiplodia, pared verruculosa <2 μM, 1-septo con estriaciones longitudinales, lo que concuerda con lo reportado por Phillips et al. (2013).
Resultados similares fueron obtenidos por Slippers et al. (2004) y Alves et al. (2008), logrando colonias en medio de cultivo moderadamente densas, con micelio aéreo que inicialmente fueron blancas tornándose gris-olivo a los 7 d hasta llegar a un color azul pizarra oscuro.
Damm et al. (2007) y Netto et al. (2014) confirmaron que la principal característica que distingue al género Lasiodiplodia de otros géneros cercanamente relacionados es la presencia de picnidios, parafisos y estriaciones longitudinales en conidios maduros, lo cual coincide con lo observado en este estudio.
Las dimensiones de los conidios y los parafisos, concuerdan con las planteadas por Pitt & Hocking (2009), Phillips et al. (2013) y Muñoz et al. (2015) para este género.
Varela et al. (2013) lo describen como el agente causal de la muerte de plantas de naranjo agrio injertadas con diferentes especies de cítricos en viveros, así como la muerte de ramas en árboles de limón mexicano y de la pudrición del pedúnculo en frutos de naranja.
La confusión en el diagnóstico del agente patógeno se debe a que produce síntomas muy similares a los que provoca Phytophthora parasitica, como la formación de gomosis, manchas foliares, pudriciones de frutas, muerte regresiva, canceres perennes y la muerte de árboles, todos estos asociados a diversas especies de hongos de la familia Botryosphaeriaceae (Muñoz et al., 2015).
Debe señalarse que hasta la fecha no había sido informada esta enfermedad para el estado de Morelos sobre lima ‘Persa’, razones que justificarían la inefectividad de su manejo en campo.
Se identificaron y caracterizaron morfológicamente tres especies dentro del género Lasiodiplodia. La primera mostró colonias con conidióforos ausentes, células conidiógenas holoblásticas, discretas, hialinas, lisas, de pared delgada, cilíndricas, proliferando recurrentemente con 1-2 anillos, 11-16 X 3-5 μM. Conidio inicialmente hialino, aséptico, elipsoide a ovoide, con contenido granular, ambos extremos redondeados ampliamente, pared <2 μM, pigmentada, verruculosa, ovoide, 1-septado con estriaciones longitudinales, correspondientes a la especie L. citricola (Figura 1).

Figura 1 Rama enferma en cámara húmeda (A), cepa aislada de cinco días de edad (B), formación de picnidios después de 7 días en rama bajo cámara húmeda (C), picnidio y conidios de L. citricola bajo microscopio compuesto a 10X y (D) corte de picnidio donde se observan parafisos, conidio en crecimiento e inmaduro (E).
Otra de las especies identificada y caracterizada fue L. pseudotheobromae, observándose conidios elipsoidales, ápices y bases redondeadas, más anchas en su medio a dos tercios superiores, de paredes gruesas, hialinas y sin septos.
La tercera especie correspondió a L. theobromae (Pat.) Griffon and Maubl., observándose células conidiógenas hialinas, de pared delgada, lisas, cilíndricas a sub-obpiriformes, holoblásticas, discretas, determinadas o indeterminadas y proliferantes por una o dos anillos distintos, o proliferando al mismo nivel, dando lugar a espesamientos periclinales, el conidia es subovoide a elipsoide ovoide, ápice redondeado ampliamente, afilando a la base truncada, más ancho en el tercio medio a tercero superior, paredes gruesas, contenido granular, inicialmente hialino y aceptado, permaneciendo hialina, llegando a ser marrón oscuro y 1-septado solamente. Luego produce la descarga de los conidiomas, con depósitos de melanina en la superficie interior de la pared dispuesta longitudinalmente dando una apariencia estriada a los conidios.
L. theobromae (Pat.) Griffon y Maubl., es cosmopolita y tiene un amplio rango de hospederos (aproximadamente 500), incluidos monocotiledóneas, dicotiledóneas y gimnospermas, es un hongo pleomórfico y ubicuo, el cual ha tenido más de un sinónimo (Abdollahzadeh et al., 2010; Wang et al., 2011, citados por Picos-Muñoz et al., 2015). Aunque tiene una amplia distribución geográfica, es común en los trópicos y subtrópicos. Este patógeno de plantas no especializado ha sido reportado causando numerosas enfermedades, incluida la muerte regresiva, pudrición de raíz, pudrición de frutos, mancha foliar y escoba de bruja entre muchos otros. También llega a ser un endófito (Mohali et al., 2005). En vista de su ocurrencia generalizada, gran cantidad de hospederos y su conocida morfología y variabilidad, es posible que L. theobromae se componga de una cantidad de especies crípticas (Punithalingam, 1980).
Lasiodiplodia pseudotheobromae es una especie emparentada con L. theobromae y se ha descrito recientemente en cultivares de Acacia, Citrus aurantium, Coffea, Gmelina y Rosa (Alves et al., 2008). Se ha reportado en diferentes especies del género Citrus, pero hasta el momento no se había reportado en lima ‘Persa’. Slippers & Wingfield (2007), reportaron a estos fitopatógenos como especies endófitas de huertos comerciales de cítricos en México.
Pruebas de patogenicidad
Las pruebas de patogenicidad realizadas en las plantas certificadas de lima ‘Persa’, revelaron que los hongos aislados en este estudio son patógenos para este cultivo, observándose los primeros síntomas a los 22 días después de la inoculación (DAI), confirmándose que las especies del género Lasiodiplodia identificadas están asociadas a esta enfermedad y que son capaces de ocasionar clorosis en hojas, muerte descendente de ramas primarias y secundarias, la formación de canceres y exudados gomosos.
Las hojas de las plantas enfermas se tornaron cloróticas, a diferencia de las del testigo. Esta sintomatología se hizo más notoria en algunas plantas, además los tallos mostraron heridas de color café. En las plantas inoculadas, no se observaron daños característicos en tallos.
A los 29 DAI, las plantas mostraron hojas con clorosis marcada en el nervio central, mientras que en las plantas sanas los tallos y hojas no mostraron daño.
A los 36 DAI se apreció una clorosis marcada con respecto a las demás observaciones, encontrándose hojas amarillentas, pero poco enrolladas, y la formación de una herida en la base del tallo (pudrición) de algunas plantas.
A los 42 DAI, algunos árboles mostraron la formación de un cáncer bien definido en la base del tallo. Los árboles testigo (sin inocular), no evidenciaron síntomas de ninguna enfermedad y se mantuvieron sanos hasta el final de las evaluaciones. Moriwaki et al. (2003) reportaron que las pruebas de patogenicidad y caracteres morfológicos permiten separar taxonómicamente las especies del género Lasiodiplodia. Estos resultados demuestran la importancia y actual vigencia de las herramientas tradicionales en la separación de especies en la taxonomía de hongos. Siendo importante para los productores de lima ‘Persa’, que exportan en gran parte esta fruta. Los caracteres morfológicos y moleculares mostraron a las mismas especies de Lasiodiplodia inoculadas.
Identificación molecular
Los estudios realizados por PCR empleando los cebadores ITS5/ITS4 mostraron la amplificación de fragmentos de DNA de 500 pb a partir de micelio fúngico de las muestras para todos los aislados.
El análisis de secuencia mostró que los hongos corresponden a las especies Lasiodiplodia citricola, L. pseudotheobromae y L. theobromae. Sin embargo, 13 de los aislamientos obtenidos, no se lograron alinear a ninguna especie del género Lasiodiplodia.
Estos resultados sugieren que estas especies que prevalecen en las huertas de cítricos constituyen una amenaza a la sanidad de las plantaciones (Timmer et al., 2002), por lo que se deben implementar tratamientos de control adecuados, ya que está siendo un problema de importancia económica en la región, debido a la alta susceptibilidad de la lima ‘Persa’ a esta enfermedad.
Bautista-Cruz et al. (2018) reportaron estudios sobre la filogenia, distribución y patogenicidad de especies de Lasiodiplodia asociadas con canceres y síntomas de muerte regresiva de lima ‘Persa’ en los estados de Veracruz y Puebla, México, donde aislaron especies de Lasiodiplodia pseudotheobromae, y L. theobromae, lo cual coincide con los resultados obtenidos, lo que sugiere que ambas especies se están propagando en las plantaciones citrícolas del México, lo que constituye una amenaza para la producción a nivel nacional.
Guajardo et al. (2018) reportan a L. theobromae por primera vez en Chile causando gomosis en limón. El monitoreo constante de este patógeno es importante para que no se extienda su diseminación en los principales huertos comerciales de cítricos en México. Adesemoye et al. (2014) reportaron otras especies de la familia Botryosphaeriaceae en lima ‘Persa’ en California, Estados Unidos, tales como Diplodia seriata y Dothiorella viticola.
Otros estudios de diversidad genética afirman que las dos especies crípticas de Lasiodiplodia (L. theobromae y L. pseudotheobromae), no han sido encontradas y estudiadas en el mismo hospedero, por lo que se desconoce de alguna posible hibridación entre ambas (Begoude et al., 2010). Por otro lado, Al-Sadi et al. (2013) encontraron un moderado nivel de diversidad genética en poblaciones de tres especies de Lasiodiplodia provenientes de distintos hospederos y orígenes geográficos, y un alto número de genotipos no especificados.
Los marcadores moleculares se han empleado recientemente para examinar genes y genotipos, modos reproductivos y especiación de un número de hongos, incluyendo Botryosphaeria spp. y sus anamorfos (Burgess et al., 2006).
Las 15 secuencias obtenidas fueron depositadas en GenBank con números de accesos para: Lasiodiplodia citricola (KY271187), Lasiodiplodia pseudotheobromae (KY284596), Lasiodiplodia theobromae (KY284589, KY284587) y Lasiodiplodia spp. (KY284584, KY284595, KY284594, KY290890, KY284592, KY284593, KY284588, KY284591, KY284586, KY284585 y KY284590), respectivamente.
Análisis filogenético
El árbol filogenético obtenido del análisis del DNA de los 16 aislamientos del género Lasiodiplodia se muestra en la Figura 2.
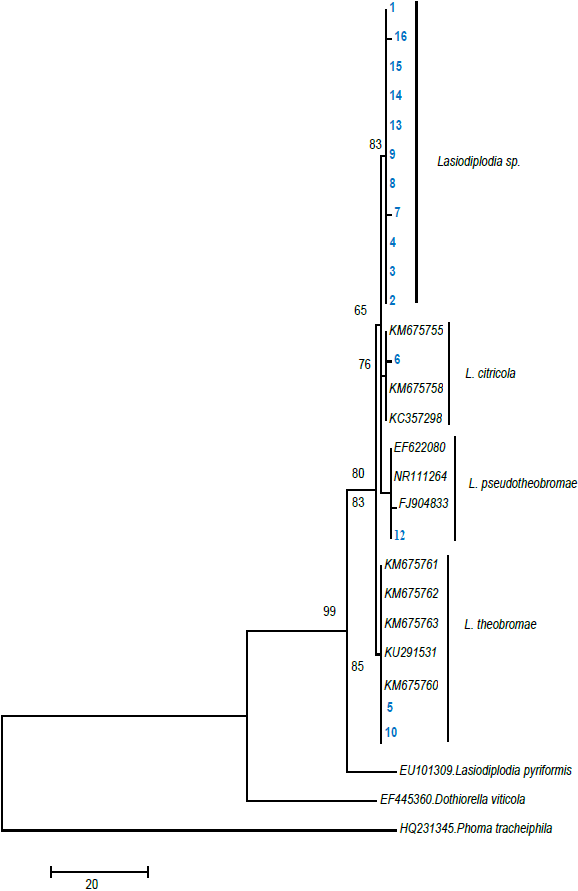
Figura 2 Árbol filogenético basado en el análisis bayesiano y máxima parsimonia utilizando los primer ITS 5 e ITS 4.
Los agrupamientos de las especies no cambiaron, manteniendo una topología similar de los cladogramas. En el análisis bayesiano, la posición de los clado (agrupamientos) de las especies de Lasiodiplodia en relación entre sí fueron diferentes, pero dentro de cada especie la topología fue similar al árbol de máxima parsimonia (TreeBASE 11035). En este estudio se identificaron cuatro clados, estos fueron: Clado 1 (Lasiodiplodia spp.), Clado 2 (L. citricola), Clado 3 (L. pseudotheobromae) y Clado 4 (L. theobromae). Las especies Dothiorella viticola (EF445360) y Phoma tracheiphila (HQ231345) se encontraron representadas fuera de grupo.
Diferentes estudios consideran que un soporte superior o igual al 70 % brinda una información confiable sobre el posicionamiento de cada especie en el cladograma (Arnold et al., 2007), lo cual coincide con los resultados obtenidos en el presente estudio.
En consecuencia, la formación del árbol permitió la conformación de agrupaciones, las cuales confirman la presencia de tres nuevas especies del género Lasiodiplodia afectando las plantaciones de lima ‘Persa’ en el estado de Morelos, México.
Conclusiones
Los síntomas observados en plantaciones de lima ‘Persa’, con muerte descendente y exudados de goma, procedentes de los municipios: Tlaltizapán, Ayala, Coatlán del Río y Amayuca, en el estado de Morelos, se obtuvieron 16 aislados, entre los que se identificaron tres especies correspondientes al género Lasiodiplodia: L. citricola, L. pseudotheobromae y L. theobromae. Asimismo, se confirmó su morfología, patogenicidad y análisis molecular a partir de los genes ITS 5 e ITS 4, con una distancia genética hasta del 99 %, fueron agrupadas en cuatro clados según el árbol filogenético y ubicadas en el GenBank, considerándose como nuevos reportes de agentes patogénicos fungosos en lima ‘Persa’ para el Estado de Morelos, México.
REFERENCIAS
Abdollahzadeh, J., Javadi, A., Mohammadi G, E., Zare, R. and Phillips, A. J. L. (2010). Phylogeny and morphology of four new species of Lasiodiplodia from Iran. Molecular Phylogeny and Evolution of Fugin 25: 1-10. https://doi.org/10.3767/003158510X524150 [ Links ]
Acosta-Pérez, J. A., Ortiz-García, C. F., Zaldívar-Cruz, J. M., Rodríguez-Cuevas, M., Bautista-Muñoz, C. C. and Castillo-Aguilar, de la C. (2012). Identificación del agente causal e importancia de la gomosis en la zona citrícola de Huimanguillo, Tabasco, México. Universidad y ciencia, 28(3): 245-258. http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S0186-29792012000300004&lng=es&tlng=es [ Links ]
Adesemoye, A. O., Mayorquin, J. S., Wang, D. H., Twizeyimana, M., Lynch, S. C. and Eskalen, A. (2014). Identification of species of Botryosphaeriaceae causing bot gummosis in citrus in California. Plant Disease. 98: 55-61. http://dx.doi.org/10.1094/PDIS-05-13-0492-RE [ Links ]
Agrios, G. N. (2005). Introduction to plant pathology. Elsevier Academic Press Publication. ISBN: 978-0-12-044563-9. https://doi.org/10.1016/C2012-0-01423-8 [ Links ]
Agroecología. (2018). Pudrición negra de los frutos y cáncer del tronco, ramas y raíces. http://www.fundesyram.info/biblioteca.php?id=3788 [ Links ]
Ahrens, U. & Seemüller, E. (1992). Detection de DNA of plant pathogenic mycoplasma like organisms by polymerase chain reaction that amplifies a sequence of the 16S rRNA gene. Phytopathology 82: 828-832. https://www.apsnet.org/publications/phytopathology/backissues/Documents/1992Articles/Phyto82n08_828.pdf [ Links ]
Al-Sadi, A. M., Al-Wehaibi, A. N., Al-Shariqi, R. M., Al-Hammadi, M. S., Al-Hosni, I., Al-Mahmooli, I. H. and Al-Ghaithi A. G. (2013). Population genetic analysis reveals diversity in Lasiodiplodia species infecting date palm, citrus, and mango in Oman and the UAE. Plant Disease 97(10): 1363-1369. https://doi.org/10.1094/PDIS-03-13-0245-RE [ Links ]
Álvarez, M. G. (1976). Primer catálogo de enfermedades de plantas Mexicanas. Fitofilo 71: 169 https://biblat.unam.mx/es/revista/fitofilo [ Links ]
Alves, A., Crous, P. W., Correia, A. and Phillips, A. J. L. (2008). Morphological and molecular data reveal cryptic speciation in Lasiodiplodia theobromae. Fungal Diversity 28: 1-13. http://www.fungaldiversity.org/fdp/sfdp/28-1.pdf [ Links ]
Aparicio-Apolinar, E., Gallardo-Huertas, A. F., Valle-de la Paz, M., Gijón-Hernández, A. R. and Guillén-Sánchez, D. (2016). Identificación molecular de hongos asociados al manchado del cáliz en Jamaica (Hibiscus sabdariffa L.) en Tecoanapa y Mecatepec, Guerrero, México. En Revista Mexicana de Fitopatología. Mexican Journal of Phyto-pathology. Fully Bilingual. ISSN-2007-8080. Volumen 34: 45 p. [ Links ]
Arnold, A., Henk, D., Eells, R., Lutzoni, F. and Vilgalys, R. (2007). Diversity and phylogenetic affinities of foliar fungal endophytes in loblolly pine inferred by culturing and environmental PCR. Micología, 99(2): 185-206. https://doi.org/10.1080/15572536.2007.11832578 [ Links ]
Bautista-Cruz, M. A., Almaguer-Vargas, G., Leyva-Mir, S. G., Colinas-León, M. T., C. Correia, K., Camacho-Tapia, M., Robledo-Yenera L., Michereff S.J. and Tovar-Pedraza, J. M. (2018). Phylogeny, distribution, and pathogenicity of Lasiodiplodia species associated with cankers and dieback symptoms of Persian lime in Mexico. Plant Disease, (ja). https://doi.org/10.1094/PDIS-06-18-1036-RE [ Links ]
Becerra-Leor, E. N., Rosas-González, X. and Silva-Rosales, L. (2014). Enfermedades presentes en el cultivo de papaya (Carica papaya L.) en la costa central del Estado de Veracruz durante los años 2012-2013. Revista Mexicana de Fitopatología. Vol. 32 (Suplemento). S59. [ Links ]
Begoude, B. A. D., Slippers, B., Wingfield, M. J. and Roux, J. (2010). Characterization of Botryosphaeriaceae and Cryphonectriaceae associated with Terminalia spp. in Africa, PhD Thesis, University of Pretoria. 268 p. https://repository.up.ac.za/bitstream/handle/2263/28515/Complete.pdf?sequence=6 [ Links ]
Burgess, T. I., Barber, P. A., Mohali, S., Pegg, G., de Beer, W. and Wingfield, M. J. (2006). Three new Lasiodiplodia spp. From the tropics, recognized based on DNA sequence comparisons and morphology. Mycologia 98(3): 423-435. https://www.ncbi.nlm.nih.gov/pubmed/17040071 [ Links ]
CESVMOR. (Comité Estatal de Sanidad Vegetal del Estado de Morelos A.C.) (2018). Comité Estatal de Sanidad Vegetal del Estado de Morelos. Campaña contra huanglongbing de los cítricos. Primer reporte de enero del 2018. https://cesvmor.org.mx/informes-mensuales-sanidad-vegetal-2018/ [ Links ]
Crous, P. W., Slippers, B., Wingfield, M. J., Rheeder, J., Marasas, W. F., Philips, A. J., Alves A., Burgess T., Barber P. and Groenewald, J. Z. (2006). Phylogenetic lineages in the Botryosphaeriaceae. Studies in Mycology, 55: 235-253. https://doi.org/10.3114/sim.55.1.235 [ Links ]
Damm, U., Crous, P. W. and Fourie, P. H. (2007). Botryosphaeriaceae as potential pathogens of Prunus in South Africa, with descriptions of Diplodia africana and Lasiodiplodia plurivora sp. nov. Mycologia 99(5): 664-680. https://www.ncbi.nlm.nih.gov/pubmed/18268901 [ Links ]
Dugan, F. M., Lupien, S. L., Osuagwu, A. N., Uyoh, E. A., Okpako, E. and Kisha, T. (2015). New Records of Lasiodiplodia theobromae in seeds of Tetrapleura tetraptera from Nigeria and fruit of Cocos nucifera from Mexico. In: 2015 Journal of phytopathology. https://doi.org/10.1111/jph.12384 [ Links ]
Felsenstein, J. (1985). Confidence limits on phylogenies: An approach using the bootstrap. Society for the Study of Evolution, 39(4): 783-791. https://doi.org/10.2307/2408678 [ Links ]
Guajardo, J., Riquelme, N., Tapia, L., Larach, A., Torres, C., Camps, R. and Besoain, X. (2018). First Report of Lasiodiplodia theobromae Causing Bot Gummosis in Citrus limon in Chile. Plant Disease, 102(4): 818 https://doi.org/10.1094/PDIS-09-17-1517-PDN [ Links ]
González, D. E., Alves, A., León, M., and Armengol, J. (2017). Characterization of Botryosphaeriaceae species associated with diseased loquat (Eriobotrya japonica) in Spain. Plant Pathol. 66:77-89. DOI:10.1111/ppa.12556. [ Links ]
Hall, T. A. (1999). BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. In Nucleic acids symposium series. Vol. 41: pp. 95-98. [London]: Information Retrieval Ltd., c1979-c2000. Oxford University Press, http://jwbrown.mbio.ncsu.edu/JWB/papers/1999Hall1.pdf [ Links ]
Hashemi, H. & Mohammadi, H. (2016). Identification and characterization of fungi associated with internal wood lesions and decline disease of willow and poplar trees in Iran. Forest Pathology, 46: 341-352. https://doi.org/10.1111/efp.12269 [ Links ]
Hillis, D. M. & Bull, J.J. (1993). An empirical test of bootstrapping as a method for assessing confidence in phylogenetic analysis. Systematic Biology 42(2): 182-192. https://doi.org/10.1093/sysbio/42.2.182 [ Links ]
Johnson, G. I., Mead, A. J., Cooke, A. W. and Dean, J. R. (1992). Mango stem end rot pathogens-Fruit infection by endophytic colonization of the inflorescence and pedicel. Annals of Applied Biology. 120: 225-234. https://doi.org/10.1111/j.1744-7348.1992.tb03420.x [ Links ]
Kimura, M. (1980) A Simple Method for Estimating Evolutionary Rate of Base Substitutions through Comparative Studies of Nucleotide Sequences. Journal of Molecular Evolution. 16(2): 111-120. https://doi.org/10.1007/BF01731581 [ Links ]
Li, H. Y., Cao, R. B., and Mu, Y. T. (1995). In vitro inhibition of Botryosphaeria dothidea and Lasiodiplodia theobromae, and chemical control of gummosis disease of Japanese apricot and peach trees in Zhejiang Province, China. Crop Protection 14:187-191. DOI: 10.1016/0261-2194(95)00011-A. [ Links ]
Linaldeddu, B. T., Deidda, A., Scanu, B., Franceschini, A., Serra, S., Berraf, T. A., Zouaoui, B. M., Ben, J. M. L. and Phillips, A. J. L. (2015). Diversity of Botryosphaeriaceae species associated with grapevine and other woody hosts in Italy, Algeria and Tunisia, with descriptions of Lasiodiplodia exigua and Lasiodiplodia mediterranea sp nov. Fungal Diversity. 71(1): 201-214 https://doi.org/10.1007/s13225-014-0301-x [ Links ]
Machado, A. R., Pinho, D. B. & Pereira, O. L. (2014). Phylogeny, identification and pathogenicity of the Botryosphaeriaceae associated with collar and root rot of the biofuel plant Jatropha curcas in Brazil, with a description of new species of Lasiodiplodia. Fungal Diversity. 67(1): 231-247 https://doi.org/10.1007/s13225-013-0274-1 [ Links ]
Marques, M. W., Lima, N. B., De MoraisJs M.A., Barbosa G. M.A., Souza B.O., Michereff S.J., Phillips A.J.L. and Marcos, A. (2013). Species of Lasiodiplodia associated with mango in Brazil. Fungal Diversity 61(1): 181-193 https://doi.org/10.1007/s13225-013-0231-z [ Links ]
Mehl, J. W. M., Slippers, B., Roux, J. and Wingfield, M. J. (2014). Botryosphaeriaceae associated with die-back of Schizolobium parahyba trees in South Africa and Ecuador. Forest. Pathology. 44: 396-408. https://doi.org/10.1111/efp.12116 [ Links ]
Mohali, T., Burgess, I. S., and Wingfield, M., J. (2005). Diversity and host association of the tropical tree endophyte Lasiodiplodia theobromae revealed using simple sequence repeat markers. Forest Pathology. 35: 385-396. https://doi.org/10.1111/j.1439-0329.2005.00418.x [ Links ]
Moriwaki, J., Sato, T. and Tsukiboshi, T. (2003). Morphological and molecular characterization of Colletotrichum boninense sp. Nov. Japan. Mycoscience 44: 47-53. https://doi.org/10.1007/S10267-002-0079-7 [ Links ]
Muñoz, P., García-Estrada, R., León-Félix, J., Sañudo-Barajas, A. and Allende-Molar, R. (2015). Lasiodiplodia theobromae en cultivos agrícolas de México: Taxonomía, hospedantes, diversidad y control. Revista Mexicana de Fitopatología. 33(1): 54-74. http://www.redalyc.org/articulo.oa?id=61240687004 [ Links ]
NCBI. (El Centro Nacional para la Información Biotecnológica) (2016). National Center for Biotechnology Information. https://www.ncbi.nlm.nih.gov/. [ Links ]
Netto, M. S. B., Assuncao, I. P., Lima, G. S. A., Marques, M. W. , Lima, W. G., Monteiro, J. H. A., de Queiroz, B. V., Michereff, S. J., Phillips, A. J. L. and Camara, M. P. S. (2014). Species of Lasiodiplodia associated with papaya stem-end rot in Brazil. Fungal Diversity. 67(1): 127-141. http://dx.doi.org/10.1007/s13225-014-0279-4 [ Links ]
Ni, H. F., Chen, R. S., Chang, S. F. and Yang, H. R. (2008). First report of Lasiodiplodia fruit rot of Jack fruit in Taiwan. Plant pathology. Disease Notes 92(7): 1137. http://dx.doi.org/10.1094/PDIS-92-7-1137A [ Links ]
Nolasco-Guzmán, V., Tovar-Pedraza, J. M., Cabrera-Huerta, E., Ayala-Escobar, V., Calyécac-Cortero, H. G., and Miranda-Rangel, A. (2014). Diversidad de hongos en semillas de Jatropha curcas de colecciones de Puebla, Veracruz y Chiapas, México. Revista Mexicana de Fitopatología. Vol. 32 (Suplemento). S64. [ Links ]
Norhayati, M., Erneeza, M. H. and Kamaruzaman, S. (2016). Morphological, pathogenic and molecular characterization of Lasiodiplodia theobromae: A causal pathogen of black rot seed Kenaf (Hibiscus cannabinus L.). International Journal of Agriculture and Biology 18(1): 80-85. https://doi.org/10.17957/IJAB/15.0065 [ Links ]
Ortiz, G. C. F. (1996). Etude de la diversite genetique de populations de Phytophthora pathogenes du cacaoyer (Theobroma cacao L.) et du cocotier (Cocos nucifera L.) (No. 04; TESIS.).129 p. http://catalogue-bibliotheques.cirad.fr/cgibin/koha/opac-detail.pl?biblionumber=19334 [ Links ]
Ortiz-García, F., Ramos-Hernández, E., Teran-Villanueva, N. and Saldaña-y-Hernández, M. I. (2014). Enfermedades de las Heliconias en plantaciones de Tabasco, México. Revista Mexicana de Fitopatología. Vol. 32 (Suplemento). S63. [ Links ]
Pereira, de M. M., Evando, A. B. J. J., Silva, M. K., Intra, M. S. and Liparini, P. O. (2015). First report of Lasiodiplodia theobromae causing stipe canker on ornamental palm (Dypsis lutescens) in Brazil. Plant Disease. Volume 0, Number. http://dx.doi.org/10.1094/PDIS-08-16-1188-PDN [ Links ]
Picos-Muñoz, P. A., García-Estrada, R. S., León-Félix, J., Sañudo- Barajas, A. and Allende-Molar, R. (2015). Lasiodiplodia theobromae en Cultivos Agrícolas de México: Taxonomía, Hospedantes, Diversidad y Control. Revista Mexicana de Fitopatología. 33(1): 54-74. http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S0185-33092015000100054 [ Links ]
Phillips, A. J. L., Alves, A., Abdollahzadeh, J., Slippers, B., Wingfield, M. J., Groenewald, J. Z. and Crous, P. W. (2013). The Botryosphaeriaceae: genera and species known from culture. Studies in Mycology. 76: 51-167. https://doi.org/10.3114/sim0021 [ Links ]
Pitt, J.I. & Hocking, A.D. (2009). Fungi and Food Spoilage. 3 ed. Springer. pp 125-127.ISBN:978-0-387-92207-2. https://www.springer.com/br/book/9780387922065 [ Links ]
Punithalingam, E. (1980). Plant diseases attributed to Botryodiplodia theobromae. Pat. J. Cramer, Vaduz. 121 pp. ISBN 3768212564, 9783768212564 [ Links ]
Rodríguez, G. E., Maldonado, E. and Alves, A. (2015). Identification and pathogenicity of Lasiodiplodia theobromae causing dieback of table grapes in Peru. European Journal of Plant Pathology. 141: 477. https://doi.org/10.1007/s10658-014-0557-8 [ Links ]
Sambrook, J. & Russell, D. W. (2012). Molecular cloning. A laboratory manual. Third Edition. 1. 1.32-1.34. Cold Spring Harbour Laboratory Press. New York. https://www.sigmaaldrich.com/catalog/product/sigma/m8265?lang=es®ion=MX [ Links ]
Schwentesius, R. R. & Manuel, Á. G. C. (2005). Limón Persa. Tendencias en el mercado mexicano. Ed. CIESTAAM/ UACh y Banco Mundial, México, D.F. 158 pp. ISBN: 968-02-0167-8. http://www.concitver.com/archivosenpdf/Limonpersa2005.pdf [ Links ]
Segura-Contreras, S., Rodríguez-Espejo, M. and Chico-Ruiz, J. (2015). Actividad antifúngica del extracto etanólico de las hojas de Schinus molle sobre el crecimiento de Lasiodiplodia theobromae en condiciones de laboratorio. Revista Científica de la Facultad de Ciencias Biológicas. Universidad Nacional de Trujillo. Trujillo. Perú. ISSN: 2313-3171. REBIOL 2015; 35(2): 47-52. http://www.revistas.unitru.edu.pe/index.php/facccbiol/article/view/1075 [ Links ]
SIAP. (El Servicio de Información Agroalimentaria y Pesquera) (2017). Atlas Agroalimentaria Anual. in: Secretaria de Agricultura, Ganadería, Desarrollo Rural, Pesca y Alimentación, México 236 p. https://www.gob.mx/siap/prensa/atlas-agroalimentario-2017 [ Links ]
Slippers, B., Crous, P. W., Denman, S., Coutinho, T. A., Wingfield, B. D. and Wingfield, M. J. (2004). Combined multiple gene genealogies and phenotypic characters differentiate several species previously identified as Botryosphaeria dothidea. Mycologia. 96(1): 83-101. https://www.ncbi.nlm.nih.gov/pubmed/21148832 [ Links ]
Slippers, B. & Wingfield, M. J. (2007). Botryosphaeriaceae as endophytes and latent pathogens of woody plants: diversity, ecology and impact. Fungal biology reviews. 21(2-3): 90-106. https://doi.org/10.1016/j.fbr.2007.06.002 [ Links ]
Swofford, D. L., (2002). (PAUP). Phylogenetic Analysis Using Parsimony (and other methods). Version 4.0b10. Sunderland, MA, USA: Sinauer Associates. http://citeseerx.ist.psu.edu/viewdoc/download?doi=10.1.1.458.6867&rep=rep1&type=pdf [ Links ]
Swofford, D. L. (2003). PAUP*. Phylogenetic Analysis Using Parsimony (*and other methods) Version 4. Sunderland, Massachusetts: Sinauer Associates. https://doi.org/10.1111/j.0014-3820.2002.tb00191.x [ Links ]
Tamura, K., Dudley, J., Nei, M. and Kumar, S. (2007). MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Molecular Biology and Evolution. 24: 1596-1599. https://www.ncbi.nlm.nih.gov/pubmed/17488738 [ Links ]
Thompson, J. D., Higgins, D. G. and Gibson, T. J. (1994). CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research. 22(22): 4673-4680. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC308517/ [ Links ]
Timmer, L. W., Garnsey, S. M. and Graham, J. H. (2002). Plagas y enfermedades de los cítricos. The American Phytopathological Society. Ed. Mundi-Prensa. Madrid. 90 pp. [ Links ]
Varela, F. S. E., Orozco, S. M., Torres, A. R. I. and Silva, A. G. L. (2013). Guía técnica para la identificación y manejo de plagas y enfermedades en cítricos. Universidad Autónoma de Tamaulipas. ISBN: 9786077654643. 428 p. [ Links ]
Woodward, J. E., Langston, Jr. D. B., Brock, J. H., Kemerait, Jr. R. C. and Brenneman, T. B. (2005). First demonstration of Koch’s postulates for Lasiodiplodia theobromae fruit spot on eggplant (Solanum melongena). Plant Disease. 89(6): 687.1 https://doi.org/10.1094/PD-89-0687A [ Links ]
Zúñiga-Estrada, L. & Yáñez-Morales, M. de J. (2015). Fungal organisms associated to the yellowing and drying of aloe. Revista Mexicana de Fitopatología. 33 (Suplemento). 2015. S96. [ Links ]
Como citar este artículo: Valle-de la Paz, M., Guillén-Sánchez, D., Gijón-Hernández, A. R., Alía-Tejacal, I. , López-Martínez, V., Juárez-López, P., Martínez-Fernández, E., Hernández-Arenas, M. and Ariza-Flores, R. (2019). Species of Lasiodiplodia in lima ‘Persa’ (Citrus latifolia Tanaka) in Morelos, México. Revista Bio Ciencias 6, e595. doi: https://doi.org/10.15741/REVBIO.06.e595
Recibido: 26 de Octubre de 2018; Aprobado: 27 de Junio de 2019











 texto em
texto em 


