Introducción
La cuenca del río Cupatitzio forma parte de la Meseta Purépecha, ubicada dentro del Eje Neovolcánico Transversal en el estado de Michoacán (Bravo et al., 2009). Se considera una de las áreas de mayor interés silvícola de la parte centro-norte del estado, con una influencia hidrológica y climática única tanto para la región central, como para la Depresión del Balsas (Conanp, 2006). Es una de las zonas de mayor importancia para la recarga de los mantos acuíferos que dan origen a un número superior de 35 manantiales y a los volúmenes del río Cupatitzio (González, 2010; Conagua, 2015). Así mismo, provee diversos servicios ecosistémicos, como la generación de energía eléctrica, de agua potable y de riego para cultivos frutícolas y de hortalizas (Conanp, 2006; Conagua, 2015).
A pesar de la importancia que tiene la cuenca para la región, las especies de plantas que conforman la cubierta vegetal de la zona de recarga hídrica no han sido descritas completamente (Bello y Madrigal, 1996), ni existen estudios básicos sobre la ecología y la genética de poblaciones o de comunidades, y, por lo tanto, no se han desarrollado propuestas confiables para la restauración y conservación de este sistema hidrológico. El estado de conservación de sus bosques no es bueno, pues los frecuentes incendios y el cambio de uso de suelo, principalmente para huertos de aguacate y zonas urbanas, influyen indudablemente en la pérdida de la vegetación nativa, particularmente de especies del género Pinus que son características de los bosques de coníferas del lugar (Bravo et al., 2009).
Los marcadores moleculares son una de las herramientas que se usan para evaluar la diversidad genética de las especies, información que es útil para establecer criterios de manejo y conservación (Brown et al., 1989; Frankham, 2010). Los microsatélites de núcleo (SSRn), son marcadores eficaces para este fin, son codominantes y altamente polimórficos, por lo que se puede obtener información genética detallada de las poblaciones e individuos (por ejemplo, los niveles de heterocigosis y endogamia, las tasas de entrecruzamiento y migración, análisis de paternidad, la certificación de cruzas controladas, análisis de pedigrí, entre otros) (Rajora et al., 2000; Rajora y Mosseler, 2001; Delgado y Piñero, 2008; Ellstrand, 2014). Sin embargo, en la actualidad existen pocos trabajos publicados formalmente que usen SSRn en pinos mexicanos (Dvorak et al., 2009; Delgado et al., 2011, 2013; Alfonso et al., 2014; Villalobos et al., 2014), y no se ha consignado ningún estudio enfocado a cuencas hidrográficas que use estas herramientas moleculares para el rescate de especies vegetales. El conocimiento de la diversidad genética de las especies que conforman los bosques forestales es esencial para su conservación efectiva y sostenible, lo cual impacta directamente en el mantenimiento integral de los sistemas hídricos asociados (FAO, 2009; Potter et al., 2017).
En este orden de ideas, en el presente estudio se planteó como hipótesis que las especies de pino distribuidas en la cuenca del río Cupatitzio contienen bajos niveles de variación genética con una estructura genética marcada entre las poblaciones. Los objetivos fueron: i) estimar los niveles de variación y estructura genética, el índice de endogamia y los tamaños efectivos de las poblaciones de cuatro especies del género Pinus distribuidas en la cuenca del río Cupatitzio; y ii) analizar las relaciones genéticas de las poblaciones y su asociación con la distribución geográfica.
Con base en los resultados se proponen núcleos de mayor diversidad genética, así como puntos estratégicos para la restauración y conservación de la cubierta vegetal de pinos en la cuenca.
Materiales y Métodos
Área de estudio
La cuenca del río Cupatitzio forma parte de la región hidrológica del río Balsas (RH18), dentro de la sub-región hidrológica Tepalcatepec-Infiernillo (I) (INEGI, 1985; DOF, 2011). Se localiza en la parte centro occidente de Michoacán; la integran los municipios Uruapan, Nuevo Parangaricutiro, Gabriel Zamora, Francisco Javier Múgica y Parácuaro (DOF, 2011). Se ubica entre los 18º49'58" y 19º36'11" N y 101º59'30" y 102º13'16" O, con una superficie aproximada de 2 659.03 km² (Conagua, 2009) (Figura 1).

ID de las poblaciones como en el Cuadro 1.
Figura 1 Ubicación geográfica de las poblaciones de Pinus colectadas en la cuenca del río Cupatitzio en Michoacán.
La vegetación predominante es el bosque de coníferas con tres tipos: bosque de pino, bosque de pino-encino y bosque mesófilo de montaña (Bello y Madrigal, 1996). Los suelos son de tipo Andosol en la parte alta de la cuenca, Luvisol y Acrisol en la parte media y Regosol y Vertisol en la parte baja (Inegi, 1985). El clima corresponde al clima templado, tipo semicálido húmedo con abundantes lluvias en verano (A)C(m)(w) (Inegi, 2010).
El área de estudio se dividió en tres zonas geográficas debido a las diferentes especies de pino: la parte alta o zona de recarga de la cuenca, con tres poblaciones; Cherán (CH1), representada por Pinus montezumae Lamb.; Quinceo (Q2) y San Lorenzo (SL3), representadas por Pinus pseudostrobus Lindl. La parte media, que incluye a la Reserva del Parque Nacional Barranca del Cupatitzio, con tres poblaciones: reserva alta (RA4), reserva media (RM5) y reserva baja (RB6), representadas por Pinus douglasiana Martínez. La tercera zona, la parte baja de la cuenca, incluye los sitios de La Pinera (LP7) y Tzaráracua (TZ8), representados por Pinus oocarpa Schiede, con un total de ocho poblaciones muestreadas (Cuadro 1 y Figura 1). Cabe mencionar que la localidad de Cherán no está contemplada dentro de la cuenca del Cupatitzio; sin embargo forma parte de la zona alta de recarga de los mantos acuíferos, por lo que se consideró importante incluirla en el estudio.
Cuadro 1 Ubicación geográfica de los sitios de colecta de las poblaciones en estudio del género Pinus.
| Zona/Especie | Sitio | ID | Ubicación geográfica | Altitud (msnm) |
|
|---|---|---|---|---|---|
| Norte | |||||
| P. montezumae | Cherán | CH1 | 19°39´5.53” N | 101°58´7.17” O | 2 956 |
| P. pseudostrobus | Quinceo | Q2 | 19°35´53.9” N | 102°02´11.7” O | 2 810 |
| P. pseudostrobus | San Lorenzo | SL3 | 19°31’35.4” N | 102°07´51.3” O | 2 246 |
| Centro | |||||
| P. douglasiana | Reserva alta | RA4 | 19°26’24.7” N | 102°06'58.1” O | 2 066 |
| P. douglasiana | Reserva media | RM5 | 19°26’25.3” N | 102°06'51.2” O | 2 035 |
| P. douglasiana | Reserva baja | RB6 | 19°25’59.7” N | 102°06’9.30” O | 1 834 |
| Sur | |||||
| P. oocarpa | La Pinera | LP7 | 19°22’59.46” N | 102°3'37.9” O | 1 656 |
| P. oocarpa | Tzaráracua | TZ8 | 19°21’03.4” N | 102°04'30.8” O | 1 481 |
Para cada especie se recolectó material foliar de cinco a 19 árboles por población, con base en la densidad del arbolado, con un total de 102 muestras, que se guardaron en bolsas de plástico y almacenaron en ultra-congelador a -80° C, hasta su uso.
Datos moleculares
La extracción de ADN se hizo con el método CTAB mini-prep para pinos (Delgado et al., 2013). Se ensayaron cinco microsatélites de núcleo (SSRn) aislados de Pinus taeda L. (Karhu, 2001), de los cuales se seleccionaron cuatro que fueron polimórficos y reproducibles (PtTX3013, PtTX3025, PtTX2146, PtTX2123). La amplificación de los microsatélites fue acorde con Elsik et al. (2000), con modificaciones en la concentración de cloruro de magnesio (4mM) (Delgado et al., 2013). Los fragmentos amplificados se separaron por electroforesis en geles de poliacrilamida (7 % 8M de Urea; Buffer TBE al 0.5 %), corridos en cámaras de secuenciación manuales a 60 Volts, durante 2-3 h a partir del tamaño de los fragmentos (Karhu, 2001). Para observarlos, los geles fueron revelados con nitrato de plata (Delgado et al., 2013), y su tamaño se determinó por medio de un marcador de 10 pb (Invitrogen TM), como referencia.
Análisis de datos
Los estimadores de la variación genética que se obtuvieron fueron las siguientes: porcentaje de loci polimórficos (% P), número total de alelos (A), promedio de alelos por locus (N A ), heterocigosis observada (H o ) y esperada (H E ) (Nei, 1987) con la fórmula:
El índice de fijación o de endogamia (F IS ) que se obtuvo como:
La desviación del equilibrio Hardy-Weinberg, se estimó con el método no paramétrico de cadenas de Marco y Monte Carlo (MCMC) con 100 000 permutaciones (Guo y Thomson, 1992). Los análisis fueron obtenidos con el programa Arlequin v. 3.5.1.2 (Excoffier y Lischer, 2010). Se hicieron pruebas exactas de desequilibrio de ligamiento para cada par de loci para las poblaciones de cada especie y una global con el método exacto de Fisher, obtenido con el programa GENEPOP v. 4.4 (Rousset, 2008). Para los cuatro SSRn utilizados se determinó la frecuencia de alelos nulos con el programa FreeNa (Chapuis y Estoup, 2007).
El tamaño efectivo (N e ), o sea, el número de individuos que contribuyen a la reproducción (Hartl y Clark, 1997), se estimó de manera indirecta de acuerdo con la fórmula:
modificada para microsatélites (Excoffier y Lischer, 2010). Dado que el parámetro θ= 4N e µ (Kimura, 1968), y al asumir una tasa de mutación (µ) de 10-3 por generación, el N e se estimó como θ/4X10-3 (Boys et al., 2005; Delgado et al., 2011; 2013; Rebolledo et al., 2018).
La estructura genética se determinó con un análisis de varianza molecular (AMOVA), entre y dentro de las poblaciones de cada especie, con el uso de los modelos de mutación de alelos infinitos (IAM) (Weir, 1996) y de paso a paso (SMM) (Slatkin, 1995). La significancia estadística se obtuvo con 1 000 permutaciones no paramétricas (Excoffier y Lischer, 2010). El parámetro M, que es la tasa de migración (M= Nm) entre pares de poblaciones, se obtuvo con la formula M= (1/F ST -1)/4 (Slatkin, 1993). Todas las estimaciones se hicieron con el programa Arlequín ver. 3.5.1.2 (Excoffier y Lischer, 2010).
Para analizar las relaciones genéticas de las poblaciones se construyó un dendrograma de Neighbor-joining, con base en las distancias genéticas estandarizadas (Da) (Nei et al., 1983). La robustez de la topología se evaluó con valor de bootstrap de 10 000 (Takezaki y Nei, 1996), con el uso del programa Poptree2 (Takezaki et al., 2010). La asociación entre la estructura geográfica y genética de las poblaciones se obtuvo mediante un análisis de agrupamiento Bayesiano con el programa BAPS (Corander et al., 2008). Este método usa información de la distribución espacial de las poblaciones para definir la estructura genética más probable. Se asumieron de 1-8 grupos (K), con 10 réplicas por K con 10 000 iteraciones, precedidas de 10 000 iteraciones iniciales.
Finalmente, se hizo un análisis de aislamiento por distancia entre pares de poblaciones y su distribución geográfica, mediante una prueba de Mantel con 1 000 permutaciones (IBD) (Bohonank, 2002). Para el análisis se usaron los valores pareados del estadístico F ST (Slatkin, 1995) y las distancias geográficas absolutas en kilómetros, obtenidas a través de la transformación de Mercator con el programa Quantum GIS Geographic Information System (v. 2.16 Nødebo) (QGIS Development Team, 2016).
Resultados y Discusión
Diversidad y estructura genética
Se registtó un total de 37 alelos en un intervalo de 13 (PtTX2146) a siete (PtTX3013) por locus. P. montezumae y P. pseudostrobus presentaron el mayor número de alelos (18 y 17 respectivamente), seguidos por P. douglasiana (14) y P. oocarpa (13). El número total de alelos entre las poblaciones de cada especie fue difrente: las de LS3 de P. pseudostrobus con 23 y RB6 de P. douglasiana con 19 tuvieron el mayor número; Q2 y RA4 de las mismas especies, el número menor, con 11 y nueve alelos respectivamente (Figura 2, Cuadro 2). Se detectaron de uno a cuatro alelos únicos (presentes en una sola población), con frecuencias entre 0.033-0.150. La población RB6 de P. douglasiana tuvo tres alelos únicos, CH1 de P. montezumae, Q2 y SL3 de P. pseudostrobus y TZ8 de P. oocarpa presentaron dos alelos, mientras que el resto de las poblaciones no se identificó a ninguno.

Figura 2 Frecuencias alélicas para los cuatro loci analizados en las poblaciones del género Pinus de la cuenca del río Cupatitzio, Michoacán, México.
Al comparar los resultados obtenidos con los de otros estudios realizados con SSRn (Delgado et al., 2011, 2013; Karhu et al., 2006), el número de alelos fue superior al de los de una población de P. pseudostrobus (19) y de P. montezumae (18) distribuidas en Michoacán (Delgado et al., 2013), las dos únicas poblaciones de P. caribae var. hondurensis (Sénécl.) W. H. Barret & Golfari para México (17) (Delgado et al., 2011) y cinco poblaciones de P. radiata D. Don en California, Estados Unidos de América y Baja California, México (8) (Karhu et al., 2006). P. oocarpa fue la única especie con un valor promedio menor a los registrados, con exepción del estudio de P. radiata (Karhu et al., 2006). Estas cifras indican que a pesar de que las poblaciones de la cuenca del Cupatitzio están fragmentadas, aún conservan variantes alélicas, por lo que se podría considerar que la deriva génica no ha actuado de manera importante en la fijación o pérdida de esta forma de diversidad en las poblaciones.
La H o en promedio fue menor a la H E para las cuatro especies analizadas. Los promedios de H o en P. pseudostrobus y P. douglasiana fueron los más altos (H o =0.378 y 0.368, respectivamente), mientras que los valores más bajos se calcularon para P. montezumae (H o = 0.338) y P. oocarpa (H o = 0.275). En contraste los valores de H E difirieron entre especies; P. pseudostrobus presentó el valor más alto, seguido de P. montezumae, P. oocarpa y P. douglasiana (Cuadro 2). Lo anterior muestra que los valores se desvían del equilibrio Hardy-Weinberg, y fueron significativos para las cuatro especies (p<0.05) (Cuadro 2).
Cuadro 2 Estimados de la variación genética de ocho poblaciones del género Pinus, distribuidas en la cuenca del río Cupatitzio, Michoacán, México.
| Zona/especie | Sitio | N | A | N a | H O | H E | F IS | N e |
|---|---|---|---|---|---|---|---|---|
| Alta P. montezumae |
CH1 | 18 | 18 | 4.5 (2.060) |
0.338 (0.143) |
0.512** (0.294) |
0.339* | 835 |
| P. pseudostrobus | Q2 | 10 | 11 | 3.0 (0.500) |
0.300 (0.180) |
0.359 (0.216) |
0.164 | 360 |
| P. pseudostrobus | SL3 | 16 | 23 | 5.7 (2.082) |
0.449 (0.214) |
0.674* (0.127) |
0.333** | 2104 |
| Promedio | 4.4 (1.546) |
0.362 (0.179) |
0.515* (0.227) |
1099 | ||||
| RA4 | 10 | 9 | 2.5 (0.250) |
0.189 (0.156) |
0.240* (0.136) |
0.218* | 183 | |
| Media | RM5 | 14 | 15 | 4.0 (2.217) |
0.337 (0.314) |
0.364 (0.229) |
0.074 | 369 |
| P. douglasiana | RB6 | 19 | 19 | 5.0 (2.217) |
0.580 (0.302) |
0.615* (0.094) |
0.057 | 1439 |
| Promedio | 3.833 (1.561) |
0.368 (0.257) |
0.406* (0.153) |
664 | ||||
| Baja | LP7 | 5 | 9 | 3.0 (0.000) |
0.300 (0.246) |
0.466** (0.111) |
0.393** | 627 |
| P. oocarpa | TZ8 | 10 | 17 | 4.5 (1.708) |
0.250 (0.254) |
0.516** (0.308) |
0.544** | 818 |
| Promedio | 3.75 (1.708) |
0.275 (0.250) |
0.491** (0.316) |
722 | ||||
| Promedio general |
15.1 | 4.02 (1.605) |
0.341 (0.228) |
0.469** (0.232) |
0.286* |
N = Número de individuos; A = Número total de alelos; N a = Promedio de alelos por locus; H o = Heterocigosis observada; H E = Hetorocigosis esperada; F IS = Índice de endogamia; N e = Tamaño efectivo. En paréntesis se indica la desviación estándar. **P=< 0.000, *P=< 0.05.
A nivel de poblaciones, el promedio de la H o también fue menor a la H E . Los valores más altos se presentaron en las poblaciones SL3 (H o = 0.449; H E = 0.674) de P. pseudostrobus y RB6 (H o = a 0.580; H E = 0.615) de P. douglasiana, mientras que los más bajos se obtuvieron para la población de RA4 (H o = 0.189; H E = 0.240) de P. douglasiana y P7 de P. oocarpa con una H o mucho menor (0.250) que la H E (0.516) (Cuadro 2). La mayoría de los valores se desvían significativamente del equilibrio Hardy-Weinberg (p<0.05), lo que sugiere diferentes intensidades de endogamia.
En general, los resultados reflejan una heterogeneidad en la cantidad de variación genética dentro de las diferentes poblaciones y zonas. No obstante, los valores se encuentran dentro de los reportado para otras especies de pino, como P. oocarpa (H E = 0.642) (Dvorak et al., 2009), P. patula Schiede. ex. Schl. & Cham y P. tecunumanii Eguiluz et Perry (H E = 0.567 y 0.646 respectivamente) (Dvorak et al., 2009), P. pseudostrobus y P. montezumae (H E = 0.277 y 0.274 respectivamente)(Delgado et al., 2013), P. mugo Turra (H E = 0.440) (Zukowska y Wachowiak, 2017), P. sylvestris L. (H E = 0.772) (Bilgen y Kaya, 2017) y P. caribaea var. hondurensis (H E = 0.575) (Rebolledo et al., 2018), lo que indica una diversidad genética intermedia y suficiente para el mantenimiento y posible adaptación de las especies a cambios ambientales.
Endogamia y tamaño efectivo
El promedio del índice de endogamia (F IS ) fue alto y significativo (F IS = 0.286), por lo que se desvía del equilibrio Hardy-Weinberg (Cuadro 2). Las poblaciones que presentaron los mayores niveles fueron LP7 (F IS = 0.393**, P= 0.000) y TZ8 (F IS = 0.544**, P= 0.000), ambas de P. oocarpa. Estos resultados sugieren que las poblaciones de la cuenca del Cupatitzio no se encuentran en equilibrio genético, debido probablemente al efecto de la reproducción entre parientes cercanos. Los valores son relativamente mayores a los obtenidos con el uso de SSRn en otras especies de pino (P. patula, F IS = 0.148, P. tecunumanii, F IS = 0.109) (Dvorak et al., 2009); P. caribaea var. hondurensis, F IS = 0.163 (Delgado et al., 2011). Esto muestra que a pesar de que las poblaciones contienen importantes niveles de variación genética (como mayor númerode alelos o niveles altos de H E ), existen procesos como la endogamia que hacen que las frecuencias de los genotipos y de los alelos se desvíen del equilibrio genético (Hartl y Clark, 1997; Holsinger, 2000).
Los valores altos de endogamia, y por lo tanto, el deficit de heterócigos observados en las población, se pueden deber a la presencia de alelos nulos (alelos no expresados) que dan lugar a valores erroneos que desvían a las poblaciones de la panmixia (Shinde et al., 2003). En el análisis para la detección de alelos nulos, únicamente el locus PtTX3025 en la población TZ8 de P. oocarpa mostró una frecuencia superior a 0.200 (0.295). No obstante, para esta especie, también se detectaron valores de endogamia significativos (F IS = 0.150, P< 0.05) con el uso de SSRn (incluyendo el locus PtTX3025) para 51 poblaciones distribuidas en México, Guatemala, Honduras y Nicaragua (Dvorak et al., 2009). La población TZ8 registró un coeficiente de endogamia significativo (F IS = 0.204, P<0.05), por lo que es pertinente considerar que la desviación de la panmixia de este locus esta determinada por el efecto de la endogamia y no por la frecuencia de alelos nulos. Asi mismo, no se reconocieron evidencias de desequilibrio de ligamiento para ninguno de los pares de loci analizados en cada especie o con la prueba global (P>0.05), lo que indica que los utilizados se segregan de manera independiente, y por lo tanto, no sobreestiman los valores de F IS obtenidos.
El tamaño efectivo (N e ), nuevamente fue mayor en P. pseudostrobus, seguido de P. montezumae, P. oocarpa y P. douglasiana. Sin embargo, los valores a nivel de las poblaciones de cada especie fueron contrastantes; la población SL3 de P. pseudostrobus presentó el mayor N e (2 104), mientras que la poblacion Q2, presentó uno de los valores más bajos (N e =360). En P. douglasiana la población RB6 tuvo un valor alto (N e = 1 439), y en la población RA4 se obtuvo el menor valor (N e =183). En las dos poblaciones de P. oocarpa, con los niveles de endogamia más altos, los estimados de N e fueron intermedios (Cuadro 2). Por ello se podría considerar que los estimados de N e resultantes, son suficientes para lograr mantener la variabilidad genética de las especies en la cuenca del Cupatitzio.
Actualmente existen pocos estudios que han estimado el N e con el uso de SSRn en pinos, como es el caso de Pinus resinosa Ait, distribuida en Estados Unidos de América, con poblaciones fragmentadas (N e entre 62-222) (Boys et al., 2005), de P. caribaea var. hondurensis (N e entre 402-868) (Delgado et al., 2011), con solo dos poblaciones fragmentadas y restringidas al sureste de México, así como de algunas poblaciones de esta variedad y de P. caribaea var. caribaea Morelet, y P. caribaea var. bahamensis (Griseb.) W. H. Barret, distribuidas en la cuenca del Caribe (N e entre 161-537) (Rebolledo et al., 2018).
Al comparar estos resultados con los obtenidos en el estudio, se observa que la mayoría de los valores son relativamente superiores. Por ello es muy probable que la fragmentación de algunas de las poblaciones como RB6 (N e = 1 439) sea muy reciente y que los efectos de la deriva genética y/o de la endogamia aún no se vean reflejados en la disminución de su variabilidad genética, y por ende en los estimados de sus tamaños efectivos.
Estructura genética y flujo genético
La estimación de la diferenciación genética entre las poblaciones de P. pseudostrobus y P. douglasiana fue más alta y significativa con el estadístico F ST (0.152 y 0.131, respectivamente) que con R ST , aunque para P. pseudostrobus ambos índices fueron significativos (P<0.05) (Cuadro 3). Es probable que para dichas especies la migración haga una mayor contribución a la diferenciación genética de las poblaciones en comparación con la mutación, la cual no sigue un modelo estricto (Crow y Aoki, 1984; Balloux y Goudet, 2002). Para P. oocarpa el estadístico F ST fue inferior (0.094) a R ST (0.295), si bien ambos fueron significativos (P<0.01). En este caso, el efecto de la mutación no es despreciable con respecto a la migración y podría ser la causa más probable de la diferenciación observada (Balloux y Goudet, 2002) (Hardy et al., 2003).
Cuadro 3 Análisis de AMOVA obtenido con los modelos de mutación de alelos infinitos (IAM; arriba de la diagonal) y paso a paso (SMM; debajo de la diagonal) para las poblaciones de Pinus pseudostrobus Lindl., Pinus douglasiana Martínez y Pinus oocarpa Schiede estudiadas en la Cuenca del río Cupatitzio.
| Recurso de variación | df | Componentes de variación | Porcentaje de variación | Promedio de Fst/Rst sobre los loci |
Valor de P |
|---|---|---|---|---|---|
| P. pseudostrobus | |||||
| Entre poblaciones | 1 | 5.677/56.592 | 15.241/12.091 | 0.152***/0.121* | 0.000/0.015 |
| Dentro de poblaciones | 50 | 46.526/411.456 | 84.759/87.901 | ||
| Total | 51 | 1.267/20964.1 | |||
| P. douglasiana | |||||
| Entre poblaciones | 2 | 0.138/3.655 | 13.061/1.948 | 0.131***/0.019 | 0.000/0.194 |
| Dentro de poblaciones | 87 | 0.872/184.913 | 86.939/97.105 | ||
| Total | 89 | 1.004/198.433 | |||
| P. oocarpa | |||||
| Entre poblaciones | 1 | 0.1096/37.074 | 9.408/29.591 | 0.094*/0.295*** | 0.015/0.000 |
| Dentro de poblaciones | 28 | 1.055/88.214 | 90.592/70.409 | ||
| Total | 29 | 1.1649/125.288 | |||
La significancia estadística fue obtenida con 1 000 permutaciones no paramétricas (Excoffier and Lischer, 2010). *P ≤ 0.05; ** P≤ 0.01; ***P≤ 0.001.
En la actualidad no existe un consenso sobre la exactitud de las estimaciones con ambos modelos de mutación, pero se ha probado, experimental y empíricamente, que depende en gran medida de los tamaños de las poblaciones, del tamaño de muestra y del número de loci analizados (Gaggiotti et al., 1999; Balloux y Goudet, 2002), con una mayor sensibilidad del estadístico R ST a los tamaños de muestra pequeños o al uso de pocos loci (< 8), debido a que sus estimaciones presentan una varianza más alta que con F ST (Golstein et al., 1995; Slatkin 1995; Balloux y Goudet, 2002; Hardy et al., 2003). Para este último estadístico, el número de poblaciones no tiene que ser infinito, ni necesariamente muy grande para que la estructura genética, y, por ende, los estimados de Nm, sean más acertados (Niegel, 2002).
Para la estimación de la estructura genética de especies de pinos mexicanos mediante el uso de SSRn en particular, se ha utilizado el estadístico F ST , con el que resultaron valores entre 0.059 en P. patula y 0.131 en P. oocarpa (Dvorak et al., 2009; Alfonso et al., 2014); en el presente estudio se obtuvieron valores superiores, excepto para P. oocarpa. Así, el Nm estimado para esta última especie es relativamente más alto (Nm= 2.41) comparado con P. pseudostrobus y P. douglasiana (Nm= 1.40 y Nm= 1.66 respetivamente).
En trabajos para especies de pinos mexicanos, los valores de Nm calculados con SSRn varían entre 1.81 en P. patula y 13.2 en P. caribaea var. hondurensis (Dvorak et al., 2009; Delgado et al., 2011). No obstante, está documentado que unidades mayores a 1.0 de Nm son suficientes para homogeneizar las frecuencias alélicas entre las poblaciones de algunas especies de plantas e impedir la diferenciación genética (Hartl y Clark, 1997).
Las coníferas, en general, tienen mecanismos de dispersión por viento, tanto de polen como de semillas, por lo que el estimado del flujo genético generalmente es superior a 1.5 unidades (Hamrick et al., 1995). Aunque también tienen capacidad para autofecundarse al ser la mayoría de las especies monoicas y auto-compatibles (Williams, 2008). Por lo tanto, se puede considerar que, a pesar de que algunas de las poblaciones muestran niveles importantes de endogamia, tienen la posibilidad de intercambiar variantes alélicas (por los valores de Nm obtenidos), lo que impide una separación genética marcada, además de que contienen alelos que se comparten entre las poblaciones de cada zona, como alelos diferenciales entre las poblaciones de las especies de interés.
Relaciones genéticas y aislamiento por distancia
El análisis de Neighbor-Joining formó dos grupos: el primero representado por las poblaciones distribuidas en la zona baja de la subcuenca (P7 y TZ8), de P. oocarpa; y el segundo, ubicado en la parte más basal del dendrograma, conformado por la población de P. montezumae (CH1) y una de las dos poblaciones de P. pseudostrobus (SL3) de la zona alta de la subcuenca. Las poblaciones más derivadas o recientes están integradas por las poblaciones de la zona media, representadas por P. douglasiana y una población de P. pseudostrobus (Q2) (Figura 3).

En la base de las ramas se indican los porcentajes de robustez de la topología obtenida con 1 000 réplicas de re-muestreo.
Figura 3 Dendrograma de Neighbor-Joining, basado en las distancias genéticas (Da) de Nei et al. (1983) entre las poblaciones del género Pinus de la cuenca del río Cupatitzio.
Este resultado es muy similar al obtenido con el análisis de agrupamiento Bayesiano BAPS, el cual formó cuatro grupos (K4), con la máxima probabilidad marginal (LogML = -960.15, P= 1.000) (Figura 4). El primer grupo representado por la población CH6 de P. montezumae; el segundo por la población Q2 de P. pseudostrobus; un tercer grupo formado por la población SL3 de P. pseudostrobus y las tres poblaciones de P. douglasiana; y el cuarto grupo formado por P. oocarpa. Solo difirieron en el segundo grupo formado por la población Q2, que en el dendrograma corresponde a la población más derivada.
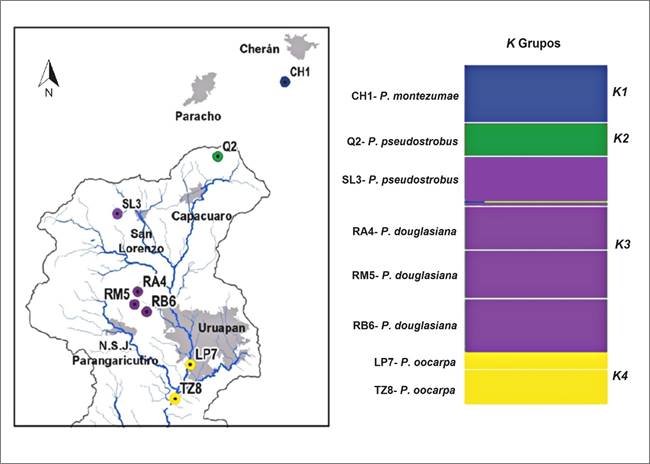
Figura 4 Agrupamiento Bayesiano BAPS, obtenido para las poblaciones del género Pinus distribuidas en la cuenca del río Cupatitzio. En barras de color se representan los 4K grupos seleccionados con la partición más óptima de los datos (P= 1.000).
Ambos tipos de análisis muestran una tendencia de agrupar a las poblaciones con su distribución geográfica, las cuales están representadas por P. montezumae y P. pseudostrobus en la parte alta de la cuenca, por P. douglasiana en la parte media y P. oocarpa en la parte baja.
Esta asociación se verificó parcialmente con el análisis de aislamiento por distancia (IBD), con una correlación moderada pero significativa (r 2 = 0.443, P<0.026), en la que 44.3 % de las diferencias genéticas observadas son atribuibles a la distancia geográfica entre las poblaciones.
Se ha demostrado que el IBD es más evidente en algunas especies de pino que tienen una distribución fragmentada, los cuales se han obtenido en varias especies analizadas con microsatélites de cloroplasto (SSRcp) (Delgado y Piñero, 2008; Moreno y Piñero, 2009; Jardón et al., 2011).
Solo se han consignado tres trabajos con el uso de SSRn en pinos, que muestran una asociación baja pero significativa: Pinus pinaster Ait en la cuenca del mediterráneo en Europa (r=0.360) (Mariette et al., 2001); P. resinosa que se ubica en el Noreste de EUA (r= 0.381) (Boys et al., 2005); y en el complejo de pinos caribeños para la cuenca del Caribe (r= 0.263) (Rebolledo et al., 2018). Estos resultados son similares al obtenido en el presente estudio, por lo que una gran parte ((60 %) de la distribución de la variación genética podría estar vinculada a otros factores, como la fragmentación espacial de las poblaciones, demarcada tanto por la geomorfología natural de la cuenca, como por al cambio de uso de suelo, de forestal a huertos de aguacate y de urbanización (Bravo et al., 2009). Dicha relación también está influenciada por las diferencias genéticas intrínsecas de los cuatro taxa estudiados, lo que se observa en los análisis de agrupamiento Bayesiano y en el dendrograma obtenido.
Recomendaciones para la conservación
Primero, se sugiere hacer actividades de conservación in situ de las poblaciones SL3 de P. pseudostrobus y RB6 de P. douglasiana, distribuidas en las zonas alta y media de la cuenca respectivamente, ya que fueron las poblaciones que presentaron los niveles mayores de H E y de variantes alélicas, las cuales podrían contener el acervo genético representativo de ambas especies. La población RB6 de P. douglasiana se encuentra dentro del área de Reserva del Parque Nacional Barranca del Cupatitzio, lo que ha ayudado a su conservación, mientras que la población SL3 de P. pseudostrobus, que se distribuye en la comunidad de San Lorenzo, requiere de acciones mediatas para delimitarla de manera formal, la cual podría ser declarada por las autoridades competentes como una Unidad de Manejo Forestal (UMAFOR; Conafor, 2009), de tal manera que ambas poblaciones funcionen como reservorios de biodiversidad y como puentes de conectividad con otras poblaciones o fragmentos (Aavick et al., 2014).
Así mismo, se recomienda la colecta de semillas de estas dos poblaciones ya que contienen la mayor variabilidad genética. Este tipo de semillas se debe aprovechar para la producción de planta a gran escala con fines de restauración o de reforestación; particularmente para el caso de la Meseta Purépecha, donde están inmersas las poblaciones mencionadas.
Por otra parte, las poblaciones RA4 (zona media) de P. douglasiana, TZ8 (zona baja) de P. oocarpa y de Q2 (zona alta) de P. pseudostrobus, presentaron los niveles de variación genética menor, y para dos de ellas se obtuvieron los niveles mayores de endogamia (RA4 y TZ8). Demográficamente, para la población de P. douglasiana de la parte alta de la Reserva RA4, se observó poca regeneración y el menor número de árboles adultos (<15), probablemente como consecuencia delos incendios frecuentes que han ocurrido en la zona. La población TZ8 de P. oocarpa, también está representada por pocos árboles adultos (<30), con nula regeneración; en este caso, se debe principalmente a los asentamientos urbanos, al estar el sitio inmerso dentro de la región sur-oriente de la ciudad de Uruapan. Es necesario realizar actividades de restauración y repoblación con base en la introducción de planta obtenida de árboles de P. pseudostrobus, P. douglasiana y P. oocarpa nativos de la región, para aumentar su tamaño censal y disminuir el efecto de la endogamia a mediano plazo.
En general, se deben implementar estrategias de conservación y restauración enfocadas al incremento del tamaño de las poblaciones, para que la diversidad genética se mantenga y con ello, la representación biológica, evolutiva y ecológica de las especies de pino de los bosques templados de la cuenca del río Cupatitzio.
Conclusiones
Las cuatro especies de pino estudiadas contienen niveles intermedios de variación genética; las poblaciones SL3 de P. pseudostrobus y RB6 de P. douglasiana presentan los mayores valores. Sin embargo, la mayoría de las poblaciones se desvían del equilibrio Hardy-Weinberg, por lo que muestran niveles importantes de endogamia. Los tamaños efectivos y los estimados del flujo genético son intermedios, pero suficientes para impedir una diferenciación genética marcada entre las poblaciones, la cual fue moderada pero significativa. Los análisis de agrupamiento Bayesiano y de las distancias genéticas, agrupan a las poblaciones acorde con su distribución geográfica e identidad taxonómica. Es necesario que dentro de los planes de conservación y restauración se consideren poblaciones de las cuatro especies, con la priorización mínimamente de las poblaciones que contienen los mayores niveles de variación genética, para que asegure su permanencia en estos bosques de pino, que son esencial para el buen funcionamiento del sistema hidrológico de la cuenca del río Cupatitzio.











 texto en
texto en 


