Introducción
El uso de especies forestales introducidas ofrece, en la mayoría de los casos, ventajas respecto a las nativas, con respecto a la velocidad de crecimiento y a los turnos de aprovechamiento, que son más breves; ambos criterios son fundamentales para desarrollar proyectos forestales viables, desde el punto de vista financiero. Los taxa de Eucalyptus son utilizados para obtener madera en ciclos cortos, pero las plantaciones son afectadas por patógenos e insectos; el hongo Botyosphaeria es causante de enfermedades severas en este tipo de árboles.
En México existen plantaciones comerciales de eucalipto de rápido crecimiento (Eucalyptus camaldulensis Dehnh., E. globulus Labill., E. grandis W. Hill, E. nitens Deane & Maiden, E. pellita F. Muell., E. urophylla S. T. Blake y el híbrido E. urophylla x grandis (E. urograndis ) (Velázquez et al ., 2013) establecidas en el Estado de México, Guerrero, Michoacán, Oaxaca, Tabasco y Veracruz.
De ese conjunto de especies destacan E. nitens y E. globulus en ambientes de mayor altitud con tendencia a heladas y E. urophylla en el trópico húmedo (Conafor, 2012). Entre 2010 y 2012 se hicieron plantaciones con los primeros dos taxa en el estado de Michoacán, en las cuales se registró mortalidad en árboles con edades de tres a cuatro años; los organismos involucrados en los tejidos muertos o enfermos fueron hongos pertenecientes a la familia Botryosphaeriacea (Botryosphaeriales, Ascomycetes) (De la Mora et al ., 2014). En el sur de Veracruz, en plantaciones de E. urophylla y E. grandis se informó de la existencia de B. rhodina (Berk. & M. A. Curtis, 1970) como causante de muerte descendente; los árboles afectados mostraron enredamiento de raíz, por lo que se presume que los hongos tuvieron un rol de patógeno oportunista (Cibrián, 2013).
Los hongos de esta familia tienen amplia distribución en el mundo y afectan a un grupo diverso de plantas dicotiledóneas, monocotiledóneas y gimnospermas (Smith et al., 2001; Mohali et al., 2006; Pérez et al., 2009, Pérez et al ., 2010). Los géneros Botryosphaeria y Neofusiccocum son patógenos endófitos, oportunistas, que inducen cancros en tallo y ramas, muerte descendente, pudrición de frutos, pudrición del cuello y damping-off en plántulas (Mohali et al ., 2006; Slippers y Wingfield, 2007).
Originalmente del género Neofusicoccum se habían reconocido 13 especies, pero el número crece conforme se hacen estudios adicionales (Sakalidis et al., 2011). Las técnicas moleculares basadas en marcadores de ADN y secuenciación son un gran apoyo para la taxonomía de los organismos. En particular los marcadores ISSR se han usado con mucho éxito por los resultados que ofrecen y la relativa facilidad con la que se obtienen. Para fines de identificación molecular, la comparación de secuencias o regiones de genes asociados a filogenia (por ejemplo, las zonas ITS) ha revolucionado la taxonomía actual de muchos organismos eucariontes y son ampliamente utilizadas (Jin et al., 2003; Awasthi et al., 2004).
El objetivo del presente trabajo fue caracterizar, comparar e identificar molecularmente aislamientos de Botryosphaeria sp., asociados a cancros y secamiento de yemas aislados de Eucalyptus sp., debido a la trascendencia de dichos hongos en las plantaciones forestales, urbanas, de restauración, agroforestales y comerciales.
Materiales y Métodos
Recolección de muestras
Se trabajó con 14 muestras de eucaliptos con síntomas de cancros, de las cuales se consideraron solo aquellas con características de Botryosphaeria sp. Nueve se obtuvieron de parcelas de la empresa Forestaciones Operativas de México S. A. de C. V. (REXCEL) (EFOMSA) y cinco de parcelas de la empresa Forestales PROTEAK durante 2013 y 2014 (Cuadro 1).
Aislamiento y purificación de los aislamientos de hongos
Las muestras se desinfestaron con hipoclorito de sodio comercial (3 %) por 5 minutos; se enjuagaron en tres ocasiones con agua destilada estéril por 5 min cada vez; se secaron con papel absorbente estéril y se sembraron tres secciones de cada muestra en medio de cultivo papa-dextrosa-agar 2 % (PDA, Bioxon ) e incubaron a 25 ± 2 °C (consideradas como repeticiones, lo que hizo un total de 42 muestras), que se mantuvieron rn rsas condiciones durante 10 días. La purificación de cada aislamiento con características de Botryosphaeria sp. se realizó mediante punta de hifa, para lo cual, una vez desarrollado el micelio en PDA, una sección de aproximadamente 1 mm fue sembrada en cajas Petri con medio Agar-Agua (AA) y mantenido 24 h a la misma temperatura. El AA es un medio pobre en el cual el micelio crece en forma muy rala; el objetivo de utilizar este medio fue para tener mayor precisión al tomar la fracción de la hifa. Al mismo tiempo, otra sección de la misma, de aproximadamente 1 mm, se transfirió a medio de cultivo de Extracto de Malta-Agar 2 % (EMA) y se mantuvo un periodo de 20-30 días para su desarrollo en las condiciones de temperatura antes mencionadas.
Caracterización morfológica
A partir de los aislamientos desarrollados en el medio de cultivo EMA, se evaluaron la coloración de micelio y el tipo de crecimiento del hongo. En los aislamientos AG-C I 114, ED-PM I 43 y FR-M a los 25 días se observó la presencia de picnidios negros, globulares, cubiertos de micelio de color gris, pero sin la formación de conidios. Estas muestras se procesaron de acuerdo a Slippers y Wingfield (2007), quienes señalan que para inducir la formación de conidios en picnidios se debe cultivar el hongo en medio AA o EMA suplementado con acículas de pino o pequeños trozos de tejido del hospedante, a 25 °C. La muestra FR-M (parcela 4) se utilizó para la identificación de dichas estructuras, a través de preparaciones microscópicas con un microscopio compuesto Carl Zeiss y el programa HardWare Setup , Microscope: Leica DM2500 , Test None. Image-Source: Leica DCF/DVM/ICD/IC3D Camera (Firewire A/B). En el caso del género, se realizó con las claves de Barnett y Hunter (1998) y de Sivanesan (1984) y para la especie se hizo con base en su morfología, de acuerdo a Smith et al. (2001).
Caracterización e identificación molecular
Con la finalidad de comparar la variabilidad genética de las 42 muestras del hongo en estudio, se obtuvieron huellas de ADN tipo ISSR. El ADN se extrajo de los cultivos monoconidiales purificados en EMA con el protocolo CTAB I, a partir de las recomendaciones de Weising et al. (2005). La calidad del ADN se verificó en gel de agarosa 1.2 % en amortiguador TAE 1X (Tris-base, ácido acético glacial, EDTA 0.5 M pH 8.0). El ADN de alto peso molecular se visualizó en un transiluminador modelo Universal Hood II (Bio-Rad) y su concentración se cuantificó en un espectrofotómetro NanoDrop (Lambda Bio 12 Perkin Elmer ).
Desarrollo de Marcadores ISSR
Se utilizaron los iniciadores (AC)8YG, (GA)8YC, (GACA)4, (GGAT)4 y (GATA)4 para caracterizar los aislamientos. Las reacciones de PCR se llevaron a cabo en un volumen final de 25 μL. La mezcla de reacción contenía Buffer 1X, MgCl2 2.5 mM, Taq ADN polimerasa 1.5 U (Promega), dNTP 200 μM, Iniciador 20 pM, ADN 100 ng y agua.
Condiciones de termociclaje
Las muestras se amplificaron en el termociclador Gene Amp PCR System 9700 (Applied Biosystems ) bajo el siguiente programa: un ciclo a 95 °C, 3 min; 35 ciclos [95 °C, 30 seg; 56 °C, 45 seg; 72 °C, 2 min] y un ciclo de extensión final a 72 °C durante 10 min. La temperatura de alineamiento para los iniciadores (GACA)4, (GGAT)4 y (GATA)4 fue de 48 °C. Para la separación de los fragmentos amplificados se trabajó en una cámara vertical C.B.S. Scientific CO® modelo MVG-216-33 con una fuente de poder E-C Apparatus Corporations ®, Modelo EC-105. El gel de acrilamida fue de 6 %, y se corrió a 245 voltios por 2.5 h, aproximadamente. Se utilizaron 2 μL del marcador de peso molecular 100 pb ADN (Promega) y 3 μL del marcador de 1 kb O´GeneRuler ™ (Fermentas) con fines de ubicación de bandas.
A partir de los perfiles de ADN observados en los geles de acrilamida, se determinó el porcentaje de bandas mono y polimórficas y con los datos se elaboró una matriz binaria. Se construyó un dendrograma con el programa Free-Tree versión 0.9.1.50 y el coeficiente Nei and Li/Dice mediante el método de agrupamiento UPGMA; el remuestreo bootstrapping fue con 100 repeticiones. Para visualizar el dendrograma se usó el programa Tree View 1.6.6, en el que se apreció la distancia genética de las muestras evaluadas.
Secuenciación de la región ITS
Para la identificación molecular se utilizó un representante de los 14 aislamientos iniciales de los eucaliptos enfermos, debido a que los perfiles de las tres muestras consideradas como repeticiones fueron iguales. Se amplificó la zona ITS (espaciadores transcritos internos) con los iniciadores universales 5HP (5´-GGA AGG AGA AGT CGT AAC AAG G-3´) y NL 4 (5´-GGT CCG TGT TTC AAG ACG G-3´). Las condiciones de reacción de PCR fueron: Buffer 1X, MgCl2 2.5 mM, Taq ADN polimerasa 1.5 U (Promega), dNTP 200 μM, Iniciador 10 pM de c/u, ADN 100 ng y agua. El programa de termociclaje consistió de un ciclo a 95 °C por 4 min; 35 ciclos [94 °C, por 1 min; 58 °C, por 1 min; 72 °C por 2 min] y un ciclo de extensión final de 72 °C por 10 min. Los ampliaciones se visualizaron en geles de agarosa 1.2 %. Las imágenes fueron documentadas con apoyo del programa Quantity One (Bio-Rad ® ).
Los productos ITS se limpiaron con EXO-SAP, previo a la secuenciación. Una vez obtenidas las secuencias, se editaron con los programas FinchTV y BioEdit y se alinearon con ClustalW . Posteriormente, fueron comparadas con algunas registradas en el GenBank (outgroup ) mediante el programa BLAST (http://www.ncbi.nlm.nih.gov/). El alineamiento de todas las secuencias se realizó en el programa MEGA 6.05, así como el árbol filogenético construido con el método estadístico de Máxima Parsimonia (MP), para ello se emplearon 1 000 repeticiones Bootstrap . Las secuencias obtenidas se subieron a la base de datos NCBI.
Resultados y Discusión
Caracterización fenotípica
El micelio de algunos de los aislamientos (AG-C I 55, ED-PM I 143, MAR, MI-T I 2, VC1, FR-M y AG-C) presentó color blanco de aspecto algodonoso a los cinco días, y a los 10 días se tornó gris oscuro a negro, además de aéreo y abundante; estas muestras registraron mayor velocidad de crecimiento. Las AG-C I 114, JA-S, RA-A, VC2, VC3, VC4 y VC5 fueron más lentas al colonizar las cajas Petri; el color de su micelio fue negro y estuvo adherido al medio de cultivo.
A los 25 días se observaron pseudotecios de color café oscuro a negro, inmersos en el medio de cultivo EMA en la muestra FR-M, así como la presencia de picnidios, conidios y ascas maduras con ascosporas (Figura 1). Características morfológicas que coinciden con los resultados de Smith et al. (2001) para Botryosphaeria eucalyptorum Crous, H. Smith et M. J. Wingf. sp. nov.
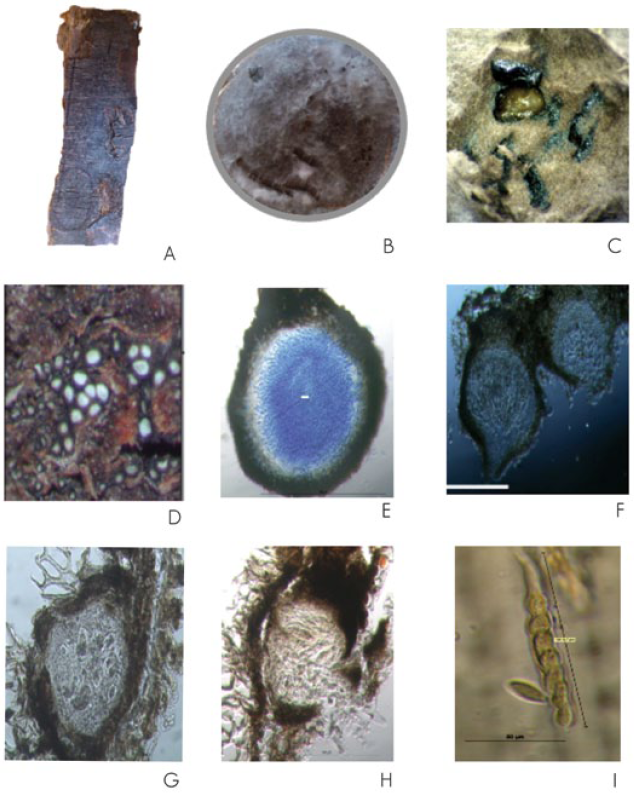
Figura 1 A) Parte de árbol enfermo utilizada; B) Neofusicoccum sp. en EMA a los 25 días; C) Aspecto de ascomas; D) Corte longitudinal de picnidios; E) Picnidio y conidio; F-H) Pseudotecios; I) Asca con ocho ascosporas, observadas a 60X con el microscopio compuesto.
Otro carácter que se ha considerado para la clasificación de las especies del género Botryosphaeria es la presencia de las formas teleomórficas (sexual) y anamórficas (asexual). Sin embargo a la fecha, la taxonomía de las especies depende principalmente de los anamorfos, debido a que algunas de las características utilizadas para su identificación como el color de los conidios, la existencia de septos, o la morfología del estroma tienen gran plasticidad dentro del taxon (Denman et al., 2000).
ISSR
Todos los iniciadores utilizados fueron capaces de generar perfiles de huellas de ADN; en particular, (AC)8YG y (GACA)4 resultaron los más informativos, porque amplificaron una cantidad de bandas superior y también mostraron mayor polimorfismo respecto al resto de los considerados; por esas razones se eligieron para hacer los agrupamientos (Figura 2). El caso contrario se verificó para (GA)8YC, (GGAT)4 y (GATA)4 que amplificaron pocas bandas y, por lo tanto, se decidió no incorporarlos al análisis de datos. Con los dos iniciadores seleccionados se visualizaron fragmentos de diferentes pesos moleculares, y para la contabilidad de las bandas de ADN solo se escogieron las de 300 pb hasta 1 500 pb, ya que fueron las de mejor consistencia y reproducibilidad en los geles de acrilamida. Cabe aclarar que las reacciones de PCR y los geles de acrilamida se corrieron al menos tres veces de manera independiente, sin observar variación en los resultados.

Figura 2 Huellas de ADN de aislamientos de Botryospaheria obtenidas con el primer (GACA)4 separadas en gel de acrilamida 6 %. Carril 1 y 44 marcador de peso molecular de 100 pb y 1 ksb Fermentas®.
Con los datos derivados de los geles y con base en el peso molecular de los fragmentos, se elaboró una matriz binaria en la que se codificó con el dígito 1 las bandas presentes y con 0 a las ausentes. Con el fin de establecer grupos genómicos con los patrones electroforéticos ISSR y correlacionarlos de forma visual con los diferentes micelios observados en los aislamientos, se construyó un dendrograma de forma radial (Figura 3), en el que se reconocen, principalmente, cuatro grupos: el grupo 1 representado por MAR- Neofusicoccum sp., VC2-Neofusicoccum sp. y VC1-Diplodia corticola A. J. L. Phillips, A. Alves & J. Luque 2004; el grupo II, que contiene a VC3-N . parvum (Pennycook & Samuels) Crous, Slippers & A. J. Phillips, 2006, FR-M Neofusicoccum sp. y VC5 Neofusicoccum sp.; el grupo III conformado por JA-S Neofusicoccum sp., AG-C I 55 N. parvum, AG-C I 114 Neofusicoccum sp., VC4-Neofusicoccum sp., ED-PM I 43-N. parvum ., MI-T I 2 N. eucalyptorum (Crous, H. Sm. ter & M. J. Wingf.) Crous, Slippers & A. J. Phillips, 2006, y AG-C N. parvum ; y el grupo IV, que incluye solamente a RAA- Lasiodiplodia pseudotheobromae A. J. L. Phillips. A. Alves & Crous, 2008. Como se aprecia, el color de los micelios de los aislamientos al interior de cada grupo es variable, e incluso al interior de la misma especie puede ser diferente, como es el caso de Neofusicoccum sp.

Figura 3 Dendrograma generado con perfiles ISSR obtenidos con los iniciadores (GACA)4 y (AC)8YG, que expresa las relaciones genómicas del género Botryospheria . Se muestra la variabilidad en color y forma de crecimiento de los micelios de los diferentes aislamientos.
Los perfiles de huellas de ADN de los distintos aislamientos revelaron alta relación genómica entre ellos. En el dendrograma respectivo no se separaron claramente las especies, quizá debido a la cantidad de fragmentos implicados en el análisis (24 totales). Sin embargo, en los tres grupos mayoritarios (I, II y III) se mezclaron diferentes especies del género. Es importante resaltar que la técnica ISSR, que se ha considerado en varias investigaciones como una opción robusta, permite estimar de forma rápida la variabilidad inter o intraespecífica de los organismos que se comparan, porque este tipo de marcadores se basa en la amplificación de fragmentos de ADN localizados entre dos regiones de microsatélites idénticos, las cuales son altamente conservadas entre las especies.
Análisis de secuencias
Los iniciadores 5HP y NL4 amplificaron fragmentos de aproximadamente 800 pb que corresponden a las regiones ITS de interés. Las secuencias se compararon con algunas secuencias cercanas reportadas en la base de datos del banco de genes del NCBI (National Center for Biotechnology Information, HYPERLINK "http://www.ncbi" www.ncbi.nih.gov) por medio del programa BLAST (Basic Local Alignment Search Tool, http://www.ncbi.nlm.nih.gov/BLAST/). La comparación con las accesiones de referencia mostró que la mayoría de los aislamientos pertenecían a Neofusicoccum sp; pero también se identificaron a D. corticola , L. pseudotheobromae, N. eucalyptorum y N. parvum. El número de accesión asignado por el NCBI a cada una de las 14 secuencias de los organismos en este estudio se indica en el Cuadro 2.
Cuadro 2 Identificación de secuencias.
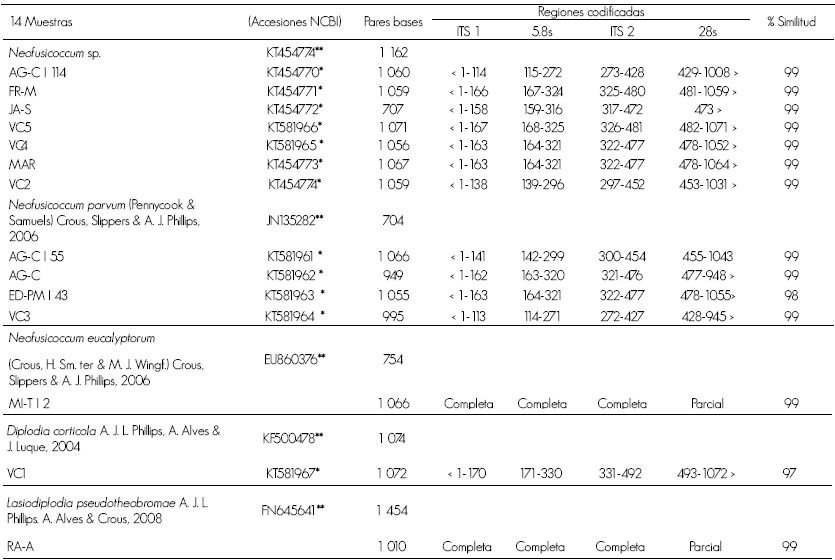
*Número de accesión de aislamientos del presente estudio; **Accesiones del GenBank usadas como referencia.
En el estado de Michoacán se hizo la identificación morfológica y molecular de N. parvum y N. eucalyptorum (B. eucalyptorum ) en aislamientos de E. nitens y E. globulus asociados a muerte descendente. Las secuencias genéticas de N. eucalyptorum (números de accesión en NCBI: KC4 79184 y KC4 799188) y N. parvum (KC4 79185, KC4 79186 y KC4 79187) tuvieron 100 % de similitud con las correspondientes en el presente estudio, lo que es el primer regostro de estas dos especies en México responsables de la enfermedad (De la Mora et al ., 2014). Por lo tanto, la investigación que aquí se documenta, confirma la existencia de N. parvum y N. eucalyptorum aislados de árboles de eucalipto afectados con cancro.
Por otro lado, se ha consignado la existencia de Lasiodiplodia theobromae (Pat.) Griffon & Maubl., 1909 y N. parvum en frutos de mango y se les ha asociado con la muerte descendente y la pudrición del pedúnculo de ese fruto en México (Sandoval et al., 2013). Dicho estudio es la primera exploración de especies de la familia Botryosphaeriacea en tal cultivo. L. theobromae , N. mangiferum y N. parvum se asocian, regularmente, con enfermedades en las regiones productoras de mango en el mundo (Sakalidis et al., 2011). L. pseudotheobromae fue reclasificada por Alves et al. (2008) como especie críptica de L. theobromae y la consideraron un taxón nuevo; desde entonces se ha reconocido en hospedantes con importancia comercial, como Citrus spp., Coffea sp., Mangifera spp. y Rosa sp. (Zhao et al., 2010).
Como ya se mencionó, hay complicaciones para la correcta identificación y diferenciación entre especies de Botryosphaeria . Los métodos moleculares son de gran apoyo para este propósito, principalmente mediante la secuenciación de ADN y la caracterización basada en marcadores moleculares que se acercan al conocimiento de la variabilidad genómica de los organismos de interés. Pero la descripción morfológica es necesaria para complementar la identificación de dichos taxa (Denman et al., 2000; Phillips et al., 2008) y, con base en sus rasgos aparentes, se han reconocido otras nuevas. La tendencia de este incremento se debe al hallazgo y descripción de especies del género encontradas en huéspedes diferentes, respecto a otro ya estudiado (Phillips et al ., 2007).
Se ha cuestionado la existencia de tantas especies, y, por lo tanto, que el número pueda ser menor, sobre todo al comparar sus secuencias de ADN. Punto de vista que se basa en el hecho de que las mutaciones de nucleótidos son las responsables del enorme polimorfismo biológico que favorece la evolución; y dado que las mutaciones suceden generalmente al azar, estas podrán tener o no impacto sobre la estructura final del organismo. Sin embargo, cuando las mutaciones afectan genes codificantes de proteínas, causan polimorfismos morfológicos un ejemplo de lo anterior es la variación en la coloración y forma del micelio de Botryosphaeria , y quizá ese sea el principal problema que ha dado lugar a la consideración de variados taxa, aun cuando las secuencias de ADN sugieren lo contrario, como fue también el caso del presente estudio.
Análisis filogenético
El árbol filogenético construido con las secuencias ITS relacionó a los aislamientos más parecidos con base en su similitud (Figura 4). En el filograma se observan dos grupos cuya diferencia es de alrededor de 0.03 de distancia relativa; el grupo mayor se divide en dos subgrupos: el primero de ellos contiene a VC3, VC4 ,VC2, FR-M, AG CI 114, JA-S, VC5, MAR, ED PM I 43 y AG C que se agruparon con las accesiones JN135282 y KJ657705 que corresponden a N. parvum y N . sp., respectivamente; el segundo subgrupo con los asilamientos AG C155 y MI T I 2 agrupados con la accesión EU860376 perteneciente a N. eucalyptorum ; y finalmente el grupo menor conformado por los aislamientos VC1 y RA A que se agruparon con FN645641 que comprende a L. pseudotheobromae .

Figura 4 Árbol filogenético por Máxima Parsimonia obtenido de secuencias de la región ITS de Neofusicoccum sp., D. corticola ., L. pseudotheobromae ., N. eucalyptorum y N. parvum identificadas en el presente estudio. Las accesiones JN135282, KJ657705, EU860376, GQ505452, KF500478 y FN645641 representan a especies de referencia.
Conclusiones
Los aislamientos caracterizados con la técnica de ISSR del género Botryosphaeria mostraron poca variabilidad genómica entre sí, aun cuando estuvieron involucradas cinco especies.
Las diferencias a nivel de nucleótidos observadas en los aislamientos respecto a las especies utilizadas como outgroups , fueron de 1-3 %
Las especies identificadas y la incidencia de las asociadas a los cancros y secamientos de las muestras de eucaliptos enfermos que se obtuvo en las 14 muestras analizadas fueron Neofusicoccum sp. 50 %; Neofusicoccum parvum 29 %; Neofusicoccum eucalyptorum , 7 %; Diplodia corticola 7 % y Lasiodiplodia pseudotheobromae 7 %.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses.
Contribución por autor
Dany Gabriel Santiago-Santiago: realización de colectas, desarrollo de protocolos en laboratorio y escritura del manuscrito; Ernestina Valadez Moctezuma y David Cibrián Tovar: negociación del financiamiento para la ejecución del proyecto, supervisión del trabajo en todos los aspectos y conclusión del escrito final.











 texto en
texto en 



