Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ciencias forestales
versión impresa ISSN 2007-1132
Rev. mex. de cienc. forestales vol.6 no.30 México jul./ago. 2015
Ensayo Técnico
La genómica en la investigación científica y en la gestión de la vida silvestre en México
The use of Genomics in scientific research and management of wildlife in Mexico
Julio César Canales-Delgadillo1, Leonardo Chapa Vargas2, Mauricio Cotera Correa3 y Laura Magdalena Scott-Morales3
1 Ducks Unlimited de México A. C. Correo: jccanales@dumac.org
2 Instituto Potosino de Investigación Científica y Tecnológica, A. C.
3 Facultad de Ciencias Forestales Universidad Autónoma de Nuevo León.
Fecha de recepción: 6 de octubre de 2014
Fecha de aceptación: 12 de febrero de 2015.
Resumen
México alberga una diversidad biológica excepcional que lo coloca entre los principales países megadiversos, pues posee tres de las 34 ecorregiones del mundo y zonas consideradas áreas silvestres a nivel mundial, como los desiertos de Chihuahua, Sonora y California; su importancia radica en que reúne alrededor de 70 % de su hábitat original en buenas condiciones y una densidad poblacional humana menor a 5 habitantes km-2. El uso de la genómica como herramienta en la investigación científica en este país tuvo sus inicios a finales de 1930 con trabajos encaminados al mejoramiento genético de cultivos comerciales y a entender los fundamentos ecológicos de la variación genética en Drosophila pseudooscura, pero hasta los años 80 y 90 comenzó el estudio de la flora y la fauna bajo esa perspectiva. Sin embargo, a pesar del potencial que las técnicas genómicas ofrecen para mejorar el desarrollo de estrategias y políticas de gestión que aseguren la producción de alimentos y la preservación de especies, no han sido extensamente utilizadas. Se presenta una revisión de las áreas del conocimiento en la vida silvestre en las que la genómica ha sido incorporada para abordar poblaciones naturales y se discuten los aspectos en los que puede incidir dentro del manejo y conservación de taxa de importancia biológica y comercial.
Palabras clave: ADN, conservación, genómica, manejo, marcadores moleculares, vida silvestre.
Abstract
Mexico has a unique biodiversity that places it within the list of megadiverse countries; it has three of the 34 ecoregions of the world and sites that are considered wilderness areas worldwide. The use of Genomics as a tool for research in Mexico began in the late 1930s with work aimed at the genetic improvement of commercial crops and to understand the ecological foundations of the genetic variation in Drosohpila pseudooscura, however, it wasn't until the decades of 1980-1990s that these tools were used for the study of natural populations of flora and fauna with purposes of conservation and management. Nonetheless, the potential that genomic tools have to improve the strategies and policies of management to ensure food production and conservation of wildlife in Mexico, these have not been widely applied. In this paper the areas of knowledge in wildlife where genomics have been applied in the study of natural populations of flora and fauna in Mexico were reviewed, and the practical applications of genomics for management and conservation of species of biological and commercial concern were discussed.
Key words: DNA, conservation, Genomics, management, molecular markers, wildlife.
Introducción
La excepcional riqueza de vertebrados y plantas vasculares con la que cuenta México ha valido para que sea reconocido como uno de los países megadiversos en el mundo; posee tres de las 34 ecorregiones del planeta: los bosques de pino-encino de las Sierras Madre, la ecorregión de Mesoamérica y la provincia florísitica de California (Myers et al., 2000; Conabio,2000). Además, los desiertos de Chihuahua, Sonora y California son considerados áreas silvestres, cuya importancia radica en reunir alrededor de 70 % de su hábitat original en buenas condiciones y una densidad poblacional humana menor a 5 habitantes km-2 (Conabio, 2006).
Si bien en los últimos años se han realizado grandes esfuerzos para caracterizar la biota mexicana, aún existen varios grupos taxonómicos como peces, anfibios y plantas vasculares que requieren investigaciones precisas sobre los procesos ecológicos que los afectan, de sus tamaños poblacionales y del estado de la diversidad genética en cada taxon (Llorente y Ocegueda, 2008). La diversidad genética de las poblaciones está influida por factores naturales como incendios, inundaciones, etcétera, pero también por el desarrollo de actividades humanas tales como cambios de uso de suelo, contaminación ambiental y alteración del hábitat, lo que la reduce y modifica las frecuencias alélicas, que es el principio de la pérdida de variabilidad genética (Bolger et al., 1991; Soulé y Mills, 1992; Pimm y Raven, 2000).
La destrucción de hábitat afecta el comportamiento de las especies, propicia cambios en el apareamiento y activa la selección sexual no azarosa, lo que rompe uno de los principios primordiales de la selección natural y acelera la erosión genética (Freeland, 2005). La pobre variación genética, a su vez, induce la disminución de las tasas de supervivencia de las crías así como de la eficacia de la respuesta inmune y, en última instancia, favorece la extinción de los taxa por una reducción de la adaptabilidad evolutiva ante cambios ambientales (Johannesson y André, 2006).
La evaluación de los factores genéticos y ambientales que inciden en la demografía de las poblaciones se lleva a cabo a través del uso de marcadores moleculares en las áreas de investigación, gestión y conservación. A pesar del gran potencial para diseñar estrategias de preservación de especies con importancia biológica y económica, en México la aplicación de dichas metodologías ha sido limitada, en parte, porque la información está dispersa, lo que sugiere que existe la necesidad de contar con un referente acerca de las técnicas disponibles para el estudio genético de las poblaciones naturales en el país.
En esta revisión se hace una síntesis de los estudios realizados con especies mexicanas y sobre temas relevantes para la conservación de recursos naturales que han utilizado técnicas genómicas y metagenómicas, desde los inicios de la disciplina de la genética de conservación en el país hasta la fecha. Se describen brevemente las técnicas moleculares más avanzadas para la generación de datos útiles en la planeación del manejo, conservación y aproximación a la vida silvestre. Y, por último, se presenta una integración de las áreas del conocimiento relacionadas a lo anterior en las que la genómica puede tener aplicaciones prácticas.
Se espera que a partir de la lectura de este documento, quienes pretendan abordar tópicos referentes a los aspectos genéticos de la vida silvestre en México obtengan los elementos necesarios para iniciar sus investigaciones, independientemente del nivel de experiencia sobre el particular.
La genómica en el estudio de poblaciones naturales
El uso de la genómica en México se inició entre 1930 y 1970 con los trabajos realizados por Edmundo Taboada y Theodosius Dobzhansky, cuyo objetivo primordial era el mejoramiento genético del maíz y del trigo, además de entender los fundamentos ecológicos de la variación genética en poblaciones naturales de Drosophila pseudoscura Frolova & Astaurov, 1929, respectivamente (Piñeiro et al., 2008a). A partir de entonces, los grupos dedicados a la genómica comenzaron a diversificar sus líneas de investigación y en la década de 1980 incluyeron aplicaciones biotecnológicas, de regulación y expresión genética en bacterias y plantas, así como de genética de poblaciones naturales de flora y fauna.
Coello et al. (1993) abordaron la genética de la conservación en México; utilizaron aloenzimas para evaluar la variabilidad genética de una saprófita rara, Lacandonia schismatica E.Martínez & Ramos, misma que, al ser casi nula, se le relacionó con el tan reducido tamaño poblacional de la especie. Los autores argumentaron que para preservar la población existente era necesario mantener el hábitat en condiciones ideales y con esto establecieron uno de los primeros antecedentes del uso de información genética como parte de un plan de conservación.
En los años siguientes el interés por conocer el estado de la diversidad genética de otros taxa y creció, y con ello, el número de trabajos realizados. Sin embargo, este esfuerzo estaba lejos de ser representativo de la biota mexicana. Una recopilación del número de publicaciones sobre variación genética en especies mexicanas demostró que hasta 2008, tales documentos por grupo taxonómico representaban más de 1 %, respecto al número de especies registradas en el país. Se resaltó que solo 11 de microorganismos habían sido investigadas hasta entonces (Piñeiro et al., 2008b), lo que pone en evidencia el rezago en relación a otras naciones.
En la última década, la creciente disponibilidad de marcadores moleculares, el acceso a bases de datos internacionales (GenBank, EMBL), además de la creciente oferta de equipos y materiales para estudios genéticos a costos más accesibles han propiciado un aumento en el número de taxa estudiados en México; dichos trabajos se refieren a mejoramiento genético de cultivos y ganado (Lozoya et al., 2010; Barrios et al., 2011; Parra et al., 2011), diversidad y flujo genético entre poblaciones silvestres (Lozano et al., 2009; Peñaloza et al., 2010; Wegier et al., 2011; Canales et al., 2012), dispersión de parásitos de importancia para la salud humana, animal y vegetal (Rosenthal, 2009; Jourdie et al., 2010; Lefévre et al., 2011), nuevos registros de marcadores moleculares (Solórzano et al., 2009; Canales et al., 2010), biogeografía (McCormack et al., 2008; Taylor et al.,2011; Bryson et al., 2011) y comportamiento (Reyes et al., 2009;Canales et al., 2012), entre otros.
Aplicaciones de los marcadores moleculares en el estudio de la vida silvestre
Tradicionalmente las investigaciones relativas a las interacciones entre organismos silvestres y su medio físico se basaban en datos morfológicos, fisiológicos y de comportamiento con los que podía construirse un perfil fenotípico que era usado para estimar la variabilidad genética de una población (Avise, 2004; Freeland, 2005). Sin embargo, estos métodos tendían a sobreestimar la variación por la plasticidad fenotípica, que se refiere a la condición en la que un mismo genotipo origina diferentes fenotipos, debido a la influencia de factores ambientales (Freeland, 2005).
En la actualidad, el estudio de algunos aspectos sobre la vida silvestre tales como el comportamiento, parentesco o estructura poblacional involucran el uso de la información contenida en los ácidos nucleicos y proteínas de los individuos, que es factible analizar a través de marcadores moleculares, lo que hace posible obtener resultados más objetivos y confiables (Lowe et al., 2004).
Existen dos tipos de marcadores moleculares con diversas aplicaciones (Cuadro 1), los codominantes que facilitan la identificación de loci homocigotos y heterocigotos; y los dominantes, que favorecen la generación simultánea de datos provenientes de múltiples loci. Gracias a los avances tecnológicos recientes, la mayor parte de los marcadores moleculares solo necesitan pequeñas cantidades de ADN para generar resultados, situación que en muchas ocasiones permite realizar muestreos no invasivos de los organismos (Taberlet et al., 1999). El costo de los marcadores es variable; de acuerdo a las características de cada uno, a los reactivos que requieren y a las condiciones o tecnología necesaria para utilizarlos suelen ser muy baratos (+) o muy caros (++++) (Cuadro 1).
Cuadro 1. Comparación de marcadores moleculares utilizados en investigación y manejo de la vida silvestre.
Table 1. Comparison of molecular markers used in research and wildlife management.
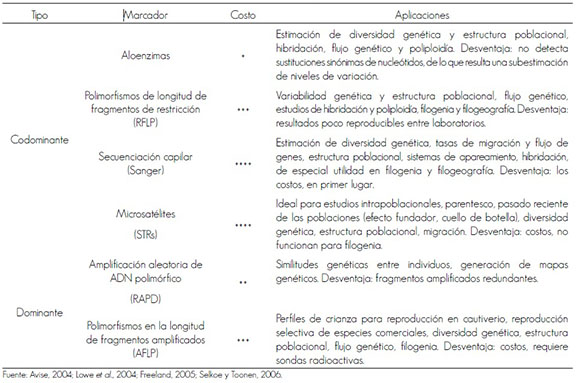
Hacer clic para agrandar
Eguiarte et al. (2013) describen las ventajas y desventajas que ofrece el uso de estos marcadores. A diferencia de los basados en proteínas o en PCR, las plataformas de secuenciación masiva o Next Generation Sequencing (next-gen) con los que se cuenta en la actualidad (Ion Torrent, 454, SOLiD, Illumina, PacBio) hacen posible secuenciar secciones mucho más amplias de un genoma, e incluso genomas completos en periodos más cortos y a precios mucho más accesibles. La ventaja principal de esta tecnología es que no se requiere conocer el genoma de los organismos que se analicen, además de que la cantidad de información generada favorece aproximaciones mucho más detalladas que con el uso de marcadores convencionales (Eguiarte et al., 2013; Escalante et al., 2014), pues, en particular, estiman la estructura y el flujo genético, los tamaños efectivos de las poblaciones, los patrones de selección natural y la endogamia; además, ofrecen la posibilidad de hacer comparaciones de expresión genética relacionadas con factores ambientales o historias de vida. Escalante et al. (2014) explican de forma pormenorizada el funcionamiento técnico de esas plataformas y brindan información sobre las tasas de error, así como de la cantidad de datos que se puede generar con cada una; Glenn (2011) consigna los costos de estos últimos, en función de las diferentes técnicas de secuenciación usadas (capilar Sanger y next-gen).
Finalmente, hay métodos que utilizan células individuales y no marcadores moleculares para evaluar la integridad del ADN como un indicador de salud ambiental. Un ejemplo es el ensayo de electroforesis de microgeles o ensayo cometa, el cual es de utilidad en el estudio de estrés ecológico sobre los organismos que habitan sitios expuestos a químicos genotóxicos (González et al., 2012).
La genómica en el estudio de la vida silvestre
Entre humanos y naturaleza no existe un beneficio mutuo debido a la influencia que las actividades antrópicas tienen sobre la transferencia de energía de un nivel trófico a otro y que impactan la biodiversidad, el flujo de carbono en los ecosistemas y la provisión de servicios ambientales (Haberlt et al., 2007). Los estudios genómicos ayudan a comprender las relaciones entre organismos o genes con su medio; además, contribuyen al diseño de estrategias que aseguran la producción de alimentos y la conservación de la flora y la fauna (Atlas et al., 2010; Bonilla,2012; O'Neill et al., 2012; Ambriz, 2012). A continuación se abordan algunas áreas en las que la genómica tiene aplicaciones encaminadas a la conservación, protección y manejo de poblaciones naturales y a la caracterización de organismos bioindicadores para la detección de patógenos y contaminantes de importancia tanto biológica, como comercial.
Biopiratería y seguridad alimentaria
La recolección y transformación de material biológico contribuyen al bienestar social, así como al desarrollo y avance de la ciencia, en particular, la medicina. Sin embargo, cuando estas actividades se realizan de forma ilegal, los efectos son contraproducentes. La biopiratería es la obtención clandestina de materiales biológicos que atenta contra la soberanía de las naciones, impacta negativamente la economía de las comunidades rurales y propicia la desaparición de especies (Robinson, 2010). Este problema se agrava por la falta de entrenamiento del personal en las aduanas u otras instituciones encargadas de la regulación de comercio o protección de la vida silvestre en la identificación de especies. El arresto de una persona bajo sospecha de traficar con ellas se hace más difícil si se interceptan cargamentos en los que solo se almacenan partes de plantas o animales; por lo tanto, es necesario contar con métodos que faciliten la identificación de organismos o sus componentes.
Se ha propuesto la caracterización genómica de los organismos para ayudar en el control de su tráfico ilegal. Hasta ahora, el gen mitocondrial citocromo C oxidasa subunidad 1 (CO1) es la mejor opción para identificar animales; la tasa de mutación del ADN mitocondrial (mtDNA) es menor a la de otros marcadores y se hereda exclusivamente por vía materna. Así, se espera que la secuencia de CO1 funcione como un detector de especies, en forma análoga al código de barras (Avise, 2004; CBOL, 2012). A pesar de que ya existen algunos, aún faltan tecnologías que permitan la secuenciación in situ de muestras que deban reconocerse.
El gen CO1 no funciona como un marcador adecuado para plantas; sin embargo ya se han efectuado pruebas de dos genes de ADN cloroplástico (matK y rbcL) en dos regiones que prometen ser buenos marcadores para código de barras en vegetales (CBOL, 2012). No obstante, que parece ser un método conveniente a nivel de especie, se ha sugerido que debe ser regionalizado, ya que a escalas geográficas muy extensas la variabilidad intraespecífica de las secuencias puede aumentar en más de 1 % (Bergsten et al., 2012), lo que afecta la precisión. En México ha sido utilizado para estudiar la diversidad de organismos como sanguijuelas (Oceguera et al., 2010), mariposas (Prado et al., 2011), plantas medicinales (Schwarzbach y Aguilar, 2012), murciélagos (Hernández et al., 2012) y avispas (Zaldívar et al., 2010).
La genómica también ha contribuido al reforzamiento de las estrategias para la implementación de la seguridad alimentaria mundial. Los recursos disponibles para la investigación genética han acelerado el descubrimiento de nuevos métodos para mejorar e incrementar la producción de cultivos comerciales en todo el mundo (Varshney et al., 2010).
Recientemente en México se ha determinado que la diversidad genética de razas nativas de maíz es más alta de lo que se creía. El teocintle, por ejemplo, es un reservorio que se ha usado desde tiempo atrás para mejorar los cultivos y crear nuevas variedades. Por lo tanto, se asume que la introducción de maíz transgénico no es necesaria, y existe preocupación por la posible pérdida de la variabilidad genética de estas razas; la probabilidad de transferencia de genes desde plantas transgénicas a las variedades nativas, además de los posibles daños ecológicos y el riesgo que ello implica para la producción de alimentos (Acevedo et al., 2011; Wang et al., 2013).
Una alternativa para asegurar la preservación de la diversidad genética de los cultivos nativos es la fundación de bancos de germoplasma. Con la creación del Centro Nacional de Recursos Genéticos (CNRG) del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP), cuyos objetivos consisten en ser la fuente de germoplasma para el mejoramiento genético de cultivares, fortalecer la seguridad y la autosuficiencia alimentaria de México, y preservar la identidad genética de las especies nativas (Sagarpa, 2010, 2012) se han cimentado las bases para la conservación de los recursos agroalimentarios y de importancia biológica. Este es un hecho de suma importancia, si se toma en cuenta que México es el centro de origen de varias especies comestibles cultivadas como maíz, frijol, aguacate y jitomate (Perales y Aguirre, 2008).
Monitoreo de contaminación y patógenos
Los efectos tóxicos de los contaminantes sobre la estructura de las poblaciones pueden ser estudiados bajo la perspectiva de la genómica. Por ejemplo, la minería es considerada una de las actividades que genera más problemas de este tipo debido a la disposición incorrecta de los desechos, lo que parece ser un factor detonante de estrés ecológico en mamíferos. Se ha demostrado que la acción del plomo y el arsénico inciden de forma negativa sobre la distribución, flujo y diversidad genética en roedores (Mussali et al., 2012).
La acumulación de residuos tóxicos en el suelo y el subsuelo es un problema recurrente para organismos detritófagos y de hábitos subterráneos; lo anterior quedó de manifiesto con los resultados de un ensayo cometa en lombrices de tierra recolectadas en la zona industrial aledaña al cauce del río Coatzacoalcos (Espinosa et al., 2010), los cuales revelaron daño a los micronúcleos de sus células, indicativo de deterioro estructural del ADN. Estos trabajos evidencian cómo ciertas características de las poblaciones, o de sus individuos, se utilizan para cuantificar los efectos de los factores ambientales sobre la fauna terrestre, y los cambios en conductas, patrones demográficos y tamaños poblacionales que originan.
La descripción molecular de comunidades bacterianas de sistemas acuáticos asociados a sitios impactados por actividades mineras o industriales ha proporcionado información muy valiosa para la identificación de organismos biomonitores (Harwood et al., 2009; Mondragón et al., 2011; Paul et al., 2013). La caracterización genética y de las rutas metabólicas de especies bacterianas capaces de reducir u oxidar ciertos metales y otros contaminantes es uno de los objetivos primordiales de la investigación biotecnológica en México.
Conservación y manejo de especies
En la actualidad muchos países fundamentan sus decisiones de conservación y manejo de especies en estudios genéticos; toman en cuenta su historia evolutiva, así como la estructura y las tasas de flujo genético entre poblaciones. Cuando se trabajan especies con historia divergente es recomendable que los esfuerzos orientados en este sentido consideren la trayectoria de cada población a fin de mantener su identidad genética.
Las estimaciones de ese tipo son útiles para lograr un control efectivo, ya que indican los patrones de dispersión tanto en flora como en fauna, información vital para preservar la diversidad de genes. Así, las recomendaciones de manejo para seis grupos de nutrias (Lontra longicaudis Olfers, 1818) en los sistemas fluviales de Chiapas se basaron en la baja diferenciación genética y altos niveles de dispersión de los grupos estudiados y se les consideró como una sola población (Ortega et al., 2012).
Una de las prácticas de manejo que involucra a poblaciones naturales es la traslocación de individuos entre áreas, que debe realizarse correctamente para no inducir endogamia. En la zona costera de Yucatán y Belice existían sospechas de hibridación entre el cocodrilo de Morelet (Crocodylus moreletii Duméril & Bibron, 1851) y el cocodrilo americano (C. acutus Cuvier, 1807); mediante el uso de STRs, González et al. (2012) no registraron evidencia de introgresión en los haplotipos de las dos especies, pero sí bajos niveles de diversidad genética, y señalaron que habría de cuidarse a C. moreletii para no arriesgar la prevalencia de su identidad genética.
Los métodos moleculares son útiles en el monitoreo de presencia y dispersión de patógenos, lo que es de especial relevancia en el sector productivo alimentario. Por ejemplo, la detección del virus del síndrome de manchas blancas (WSSV) en granjas camaroneras de Guasave, Sinaloa evitó pérdidas económicas importantes (Vázquez et al., 2010). La investigación se realizó con protocolos basados en PCR con muestras provenientes de las branquias y tubo digestivo de ostión (Crassostrea gigas Thunberg, 1793). Los mismos métodos se aplican para evaluar la presencia y dispersión de parásitos hemosporidios de aves en zonas semiáridas de México, lo que puede ayudar a detectar amenazas para la industria avícola.
Las estrategias de manejo para especies longevas de distribución restringida también es factible desarrollarlas a partir de estudios genéticos. Se ha sugerido para algunas especies vegetales de este tipo, que, mientras haya adultos reproductivos, la recolección de semillas tiene un impacto mínimo en la diversidad genética. Un ejemplo es el exceso de heterocigocidad en poblaciones de Dioon caputoi De Luca, Sabato & Vázq. Torres, una cícada severamente amenazada del centro de México (Cabrera et al., 2008). Se ha recomendado evitar la extracción no controlada de ejemplares adultos y llevar a cabo su propagación por medio de semillas.
Los estudios de genética poblacional son aplicables a especies de interés comercial, como Agave cupreata Trel. & Berger y A. potatorum Zucc., ambas importantes en la industria mezcalera del sur de México. Aguirre y Eguiarte (2013) demostraron que no existe erosión genética aparente en los grupos analizados de los dos taxa, por lo que se recomendó la reproducción por medio de semillas y no de micropropagación
Ciencias forenses y vida silvestre
Se ha estimado que el tráfico ilegal de especies vivas o de alguna de sus partes produce alrededor de US$15 mil a 50 mil millones anuales en los Estados Unidos de América (Wallace y Ross, 2012). En México no existen cifras oficiales derivadas de estas actividades, pero unos especialistas consideran que las ganancias son comparables con las del tráfico ilegal de drogas (Alvarado, 2012). Los métodos generales de la genómica aplicados en investigación forense pueden ser extendidos y ajustados para recabar y examinar evidencia de crímenes contra la vida silvestre. A través de la investigación científica y los recorridos de campo, el Departamento de Pesca y Servicios de la Vida Silvestre de los Estados Unidos de América (FWS) colabora para condenar sospechosos de caza fortuita (Wallace y Ross, 2012).
A la fecha, los agentes encargados de otorgar protección al ambiente en México realizan funciones de verificación aleatoria del cumplimiento de la normatividad y fungen solamente como asesores técnicos ante las autoridades ministeriales en los casos en proceso (Calvillo, 2010), lo que limita la aplicación de la justicia sobre crímenes cometidos contra la biodiversidad. Sin embargo, ya se han hecho propuestas para implementar medidas que emulen los procedimientos del FWS, no solo en la capacitación de los agentes de campo, sino en el empleo de peritos especializados en la aplicación de técnicas moleculares y forenses para el análisis de evidencia en delitos ambientales. La genética forense también es útil en el estudio de las relaciones ecológicas entre organismos.
Las técnicas basadas en PCR han sido utilizadas para la identificación de especies de depredadores y sus presas mediante rastros indirectos (excretas, contenidos estomacales, pelo),lo que facilita abordar especies elusivas o de gran tamaño como los pumas (Puma concolor Linnaeus, 1771), jaguares (Panthera onca Linnaeus, 1758)) o coyotes (Canis latrans Say, 1823). En la Huasteca de San Luis Potosí se incorporaron estos métodos para examinar muestras fecales para identificar dos taxones de felinos y sus principales presas. Los investigadores determinaron con éxito alrededor de 50 % de las muestras recolectadas para asignarlas a P. concolor (12 presas y a P. onca (9 presas) (Rueda, 2010).
Se ha descubierto que es posible partir de fragmentos de ADN contenidos en el intestino de moscas carnívoras para calcular la riqueza de especies en sitos de difícil acceso e incluso se ha podido detectar la presencia de mamíferos considerados raros mediante esta metodología (Yong, 2013).
La metagenómica en la aproximación a la vida silvestre está proporcionando luz sobre los procesos ambientales que influyen en el establecimiento y diversificación de diferentes tapetes microbianos o biofilms en lugares con características biológicas muy particulares como son los embalses de Cuatro Ciénegas en Coahuila, lo que da a conocer la historia evolutiva de ese grupo de organismos, y contribuye a comprender los mecanismos del desarrollo de la vida en el planeta (Bonilla et al., 2012).
Conclusiones
Las ciencias genómicas ofrecen una amplia gama de opciones para el estudio, manejo y conservación de la flora y la fauna silvestres. Los resultados de los trabajos expuestos hacen evidente que la inclusión de información generada a través del uso de marcadores moleculares podría mejorar los programas de manejo de especies de importancia comercial y en cautiverio, como es el caso de la repatriación de animales decomisados hacia sus lugares de origen.
Con base en los análisis filogenéticos, de parentesco, de variabilidad y similitud genética se podría lograr una mayor certidumbre a fin de evitar la erosión genética, la endogamia y la exogamia en poblaciones naturales.
El diseño de estrategias para la vida silvestre debe apoyarse en información generada con técnicas moleculares que ayuden a comprender a detalle las relaciones filogenéticas entre grupos de organismos o de especies para preservar la variabilidad y la identidad genética de las poblaciones y de los taxa, en particular de aquellas que están bajo algún tipo de amenaza, las nativas y las de alto valor comercial y alimenticio.
Como centro de origen de muchas especies cultivables, en México se debe preservar íntegramente el reservorio genético de estas y de las naturales, tanto de plantas como de animales que habitan en el territorio nacional. Las ciencias genómicas ofrecen la oportunidad de generar tal conocimiento.
En materia legal, los métodos relacionados a las mismas son herramientas de enorme potencial para la aplicación de justicia, regulación y protección de la soberanía de México sobre sus recursos naturales.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses.
Contribución por autor
Julio César Canales-Delgadillo: revisión sobre el tema, investigación bibliográfica y elaboración del manuscrito; Leonardo Chapa Vargas, Mauricio Cotera Correa y Laura Magdalena Scott-Morales: revisión del manuscrito y aportaciones para su enriquecimiento.
Referencias
Acevedo, F., E. Huerta, C. Burgeff, P. Koleff and J. Sarukhán. 2011. Is transgenic maize what Mexico really needs? Nature Biotechnology 29: 23-24. [ Links ]
Aguirre D., X. and L. Eguiarte. 2013. Genetic diversity, conservation and sustainable use of wild Agave cupreata and Agave potatorum extracted for mezcal production in Mexico. Journal of Arid Environments 90: 36-44. [ Links ]
Alvarado M., I. 2012. Delincuencia organizada ambiental en México, una nueva manifestación criminal del tráfico de especies. Revista Criminalidad 54(1): 283-311. [ Links ]
Ambriz, M. P. 2012. Secuenciación y análisis del genoma mitocondrial de subespcies de venado cola blanca (Odocoileus virginianus). Tesis de Maestría. CBG-IPN. Tamaulipas, México. 54 p. [ Links ]
Atlas, R., C. Rubin, S. Maloy, P. Daszak, R. Colwell and B. Hyde. 2010.One Health—Attaining optimal health for people, animals, and the environment. Microbe 5 (9): 383-389. [ Links ]
Avise, J. C. 2004. Molecular markers, natural history and evolution. Sinauer Associates, Inc. Sunderland, MA, USA. pp. 55-98. [ Links ]
Barrios G., E., C. López, J. Kohashi, J. Acosta, J. Gallegos, S. Miranda y N.Mayek. 2011. Avances en el mejoramiento genético del frijol en México por tolerancia a temperatura alta y a sequía. Revista Fitotecnia Mexicana 34(4): 247-255. [ Links ]
Bergsten, J., D. T. Bilton, T. Fujisawa, M. Elliott, M. T. Monaghan, M. Balke,L. Hendrich, J. Geijer, J. Herrmann, G. N. Foster, I. Ribera, A. N. Nilsson, T. G. Barraclough and A. P. Vogler. 2012. The effect of geographical scale of sampling on ADN barcoding. Systematic Biology 61(5): 851-869. [ Links ]
Bolger D. T., A. C. Alberts and M. E. Soulé. 1991. Occurrence patterns of bird species in habitat fragments: sampling, extinction, and nested species subsets. The American Naturalist 137 (2): 155-166. [ Links ]
Bonilla R., G., M. Peimbert, L. D. Alcaraz, I. Hernández, L. E. Eguiarte, G. Olmedo Á. and V. Souza. 2012. Comparative metagenomics of two microbial mats at Cuatro Ciénegas Basin II: community structure and composition in oligotrophic environments. Astrobiology 12: 659-673. [ Links ]
Bryson Jr., R. W., R. W. Murphy, A. Lathrop and D. Lazcano V. 2011. Evolutionary drivers of phylogeographical diversity in the highlands of Mexico: a case study of the Crotalus triseriatus species group of montane rattlesnakes. Journal of Biogeography 38 (4): 697-710. [ Links ]
Cabrera T., D., J. González A. and A. P. Vovides. 2008. Heterozygote excess in ancient populations of the critically endangered Dioon caputoi (Zamiaceae, Cycadales) from central Mexico. Botanical Journal of the Linnean Society 158: 436–447. [ Links ]
Calvillo D., G. 2010. La investigación criminal forense en apoyo al manejo sustentable de la biodiversidad. Derecho Ambiental y Ecología 6(35): 43-51. [ Links ]
Canales D., J., L. Scott M. and J. Korb. 2012. The influence of habitat fragmentation on genetic diversity of a rare bird species that commonly faces environmental fluctuations. Journal of Avian Biology 43(2): 168-176. [ Links ]
Canales D., J., L. Scott M., O. Niehuis and J. Korb. 2010. Isolation and characterization of nine microsatellite loci in the endangered Worthen's Sparrow (Spizella wortheni). Cons.ervation Genetics Resources DOI:10.1007/s12686-010-9203-8.
Coello G., A. Escalante and J. Soberón. 1993. Lack of genetic variation in Lacandonia schismatica (Lacandoniaceae: Triuridales) in its only known locality. Annals of Missouri Botanical Garden 80: 898-901. [ Links ]
Comisión Nacional para el Conocimiento y Uso de la Biodiversidad (Conabio). 2000. Estrategia nacional sobre biodiversidad de México. Semarnat. México, D. F., México. 194 p. [ Links ]
Comisión Nacional para el Conocimiento y Uso de la Biodiversidad (Conabio). 2006. Capital natural y bienestar social. Semarnat. México, D. F.,México. 32 p. [ Links ]
Consortium for Barcode of Life (CBOL). 2012. http://www.barcodeoflife.org (2 de octubre de 2014).
Eguiarte L., E., J. A. Aguirre L., L. Jardón B., E. Aguirre P. y V. Souza. 2013. Genómica de poblaciones: Nada en evolución va a tener sentido si no es a la luz de la genómica, y nada en genómica tendrá sentido si no es a la luz de la evolución. Revista Especializada de Ciencias Químico-Biológicas 16 (1): 42-56. [ Links ]
Escalante A. E., E. Jardón B., S. Ramírez B. and L. E. Eguiarte. 2014. The study of biodiversity in the era of massive sequencing. Revista Mexicana de Biodiversidad 85: 1249-1264. DOI: 10.7550/rmb.43498. [ Links ]
Espinosa R., G., C. A. Ilizaliturri, D. González M., R. Costilla, F. Díaz B., M. C. Cuevas, M. A Martínez and J. Mejía S. 2010. ADN damage in earthworms (Eisenia spp.) as an indicator of environmental stress in the industrial zone of Coatzacoalcos, Veracruz, Mexico. Journal of Environment Science and Health, Part A 45-1: 49-55. [ Links ]
Freeland, J. R. 2005. Molecular ecology. John Wiley & Sons, Ltd. Chichester,West Sussex, UK. 388 p. [ Links ]
Glenn, T. 2011. Field guide to next generation dna sequencers. Molecular Ecology Resources DOI:10.1111/j.1755-0998.2011.0324.x.
González M., D., G. Espinosa R., C. Ilizaliturri H., S. J. Mejía, Y. Jasso P. y F. Díaz B. 2012. Ensayo cometa en fauna terrestre. In: Cuevas D., M.,G. Espinosa R., C. Ilizaliturri H. y C. A. Mendoza (eds.). Métodos ecotoxicológicos para la evaluación de suelos contaminados por hidrocarburos. Semarnat-INE. México, D.F., México. pp. 107-125. [ Links ]
González T., R., D. Rodríguez, A. González R., M. R. J. Forstner, L. D. Densmore III and V. H. Reynoso. 2012. Testing for hybridization and assessing genetic diversity in Morelet's crocodile (Crocodylus moreletii)populations from central Veracruz. Conservation Genetics 13:1677-1683. [ Links ]
Haberlt, H., K. H. Erb, F. Krausmann, V. Gaube, A. Bondeau, C. Plutzar, S. Gingrich, W. Lucht and M. Fisher K. 2007. Quantifying and mapping the human appropriation of net primary production in earth's terrestrial ecosystems. Proceedings of the National Academy of the United States of America 104 (31): 12942-12947. [ Links ]
Harwood, V. J., M. Brownell, S. Wang, J. Lepo, R. D. Ellender, A. Ajidahun and C. Flood. 2009. Validation and field testing of library-independent microbial source tracking methods in the Gulf of Mexico. Water Research 43(19): 4812-4819. [ Links ]
Hernández D., A., J. A. Vargas, N. Martínez M., B. K. Lim, M. D. Engstrom and J. Ortega. 2012. ADN barcoding and genetic diversity of phyllostomid bats from the Yucatan Peninsula with comparisons to Central America. Molecular Ecology Resources 12: 590–597. [ Links ]
Johannesson, K. and C. André. 2006. Life on the margin: genetic isolation and diversity loss in a peripheral marine ecosystem, the Baltic Sea. Molecular Ecology 5(8): 2013–2029. [ Links ]
Jourdie, V., N. Álvarez, J. Molina O., T. Williams, D. Bergvinson, B. Benrey, T. C. J. Turlings and P. Franck. 2010. Population genetic structure of two primary parasitoids of Spodoptera frugiperda (Lepidoptera), Chelonus insularis and Campoletis sonorensis (Hymenoptera): to what extent is the host plant important? Molecular Ecology 19 (10):2168-2179. [ Links ]
Lefèvre T., A. J. Williams and J. C. de Roode. 2011. Genetic variation in resistance, but not tolerance, to a protozoan parasite in the monarch butterfly. Proceedings of Biological Science 278 (1706): 751-759. [ Links ]
Llorente B., J. y S. Ocegueda. 2008. Estado del conocimiento de la biota. In: Capital natural de México. Vol. I: Conocimiento actual de la biodiversidad. Conabio. México, D. F., México. pp. 283-322. [ Links ]
Lowe, A., S. Harris and P. Ashton. 2004. Ecological genetics. Design, analysis and application. Blackwell Publishing Science. New York, NY, USA. pp. 36-44. [ Links ]
Lozano F., S., I. Fernández S., M. L. Muñoz, J. García R., K. E. Olson, B. J. Beaty and W. C. Black N. 2009. The Neovolcanic axis is a barrier to gene flow among Aedes aegypti populations in Mexico that differ in vector competence for dengue 2 virus. PLos Neglected Tropical Diseases 3(6): e468. [ Links ]
Lozoya S., H., C. R. Belmar D., J. Bradeen y J. Helgeson. 2010. Caracterización de poblaciones de Phytophthora infestans (Mont de Bary) obtenidas de Solanum transgénicas y de híbridos somáticos resistentes. Interciencia 35(10): 784-788. [ Links ]
McCormack, J. E., A. T. Peterson, E. Bonaccorso and T. B. Smith. 2008. Speciation in the highlands of Mexico: genetic and phenotypic divergence in the Mexican jay (Aphelocoma ultramarina). Molecular Ecology 17 (10):2505-2521. [ Links ]
Mondragón V., A., D. F. Llamas P., G. E. González G., A. R. Márquez G., R. Padilla N., M. de Jesús Durán A. and B. Franco. 2011. Identification of Enterococcus faecalis bacteria resistant to heavy metals and antibiotics in surface waters of the Mololoa River in Tepic, Nayarit, Mexico. Environmental Monitoring Assessment 183(1-4): 329-340. [ Links ]
Mussali G., P., E. Tovar S., M. Valverde, L. Valencia C. and E. Rojas. 2012. Evidence of population genetic effects in Peromyscus melanophrys chronically exposed to mine tailings in Morelos, Mexico. Environmental Science Pollution Research DOI10.1007/s11356-012-1263-8.
Myers, N., R. A. Mittermeier, C. G. Mittermeier, G. A. B. da Fonseca and J. Kent. 2000. Biodiversity hotspots for conservation priorities. Nature 403: 853-858. [ Links ]
Oceguera F., A., V. León R. and M. E. Siddall. 2010. DAN barcoding reveals Mexican diversity within the freshwater leech genus Helobdella (Annelida: Glossiphoniidae). Mitochondrial ADN 21(1): 24-29. [ Links ]
O'Neill, E. M., R. Schwartz, C. T. Bullock, J. S. Williams, H. B Shaffer, X. Aguilar-Miguel, G. Parra-Olea and D. W. Weisrock. Parallele tagged amplicon sequencing reveals major lineages and phylogenetic structure in the North American tiger salamander (Ambystoma tigrinum) species complex. Molecular Ecology 22(1):111-129. [ Links ]
Ortega J., E. Navarrete y J. E. Maldonado. 2012. El muestreo no invasivo de las nutrias neotropicales, una especie amenazada, revela altos niveles de dispersión en el sistema fluvial del Lacantún, en Chiapas, México. Animal Biodiversity and Conservation 35(1): 59-69. [ Links ]
Parra B., G. M., A. M. Sifuentes R., X. F. de la Rosa y W. Arellano V. 2011. Avances y perspectivas de la biotecnología genómica aplicada a la ganadería en México. Tropical and Subtropical Agroecosystems 14: 1025-1037. [ Links ]
Paul, J. H., D. Hollander, P. Coble, K. L. Daly, S. Murasko, D. English and C. W. Kovach. 2013. Toxicity and mutagenicity of Gulf of Mexico waters during and after the deepwater horizon oil spill. Environmental Science and Technology 47(17): 9651-9659. [ Links ]
Peñaloza R., J. M., A. González R., L. Mendoza C., H. Caron, A. Kremer and K. Oyama. 2010. Interspecific gene flow in a multispecies oak hybrid zone in the Sierra Tarahumara of Mexico. Annals of Botany 105 (3): 389-399. [ Links ]
Perales, H. R y J. R. Aguirre. 2008. Biodiversidad humanizada. In: Soberón, J., G. Halffter y J. Llorente-Bousquets (comps.). Capital natural de México, Vol. I: Conocimiento actual de la biodiversidad. Conabio. México, D. F., México. pp. 565-603. [ Links ]
Pimm, S. L. and P. Raven. 2000. Biodiversity: extinction by numbers. Nature 403:843-845. [ Links ]
Piñeiro D., A. Barahona, L. Eguiarte, A. Rocha O. y R. Salas L. 2008a. La variabilidad genética de las especies: aspectos conceptuales y sus aspectos y perspectivas en México. In: Soberón, J., G. Halffter y J. Llorente-Bousquets (comps.). Capital natural de México, Vol. I: Conocimiento actual de la biodiversidad. Conabio. México, D. F., México. pp. 415-435. [ Links ]
Piñeiro D., D. 2008b. La diversidad genética como instrumento para la conservación y el aprovechamiento de la biodiversidad: estudios en especies mexicanas. In: Soberón, J., G. Halffter y J. Llorente-Bousquets (comps.). Capital natural de México, Vol. I: Conocimiento actual de la biodiversidad. Conabio. México, D. F., México. pp. 437-494. [ Links ]
Prado, B. R., C. Pozo, M. Valdez M. and P. D. N. Hebert. 2011. Beyond the colours: discovering hidden diversity in the Nymphalidae of the Yucatan Peninsula in Mexico through ADN Barcoding. PLoS ONE 6(11):e27776. doi:10.1371/journal.pone.0027776. [ Links ]
Reyes, D., R. E. Alcalá, D. Arias and M. Osorio B. 2009. Genetic structuring at a fine scale in the Russet-Crowned motmot (Momotus mexicanus) in a tropical dry forest in central Mexico. Western North American Naturalist 69 (2): 171-174. [ Links ]
Robinson, D. F. 2010. Confronting biopiracy: challenges cases and international debates. Earthscan Books, Ltd., London, UK. 189 p. [ Links ]
Rosenthal, B. M. 2009. How has agriculture influenced the geography and genetics of animal parasites? Trends in Parasitology 25 (2): 67-70. [ Links ]
Rueda Z., R. P. 2010. Determinación de la dieta de jaguar (Panthera onca) y puma (Puma concolor) en el municipio de Tamasopo, San Luis Potosí. Tesis de Maestría. Colegio de Postgraduados. Montecillo, Edo. de Méx., México. 81 p. [ Links ]
Secretaría de Agricultura, Ganadería, Recursos Hidráulicos, Pesca y Alimentación (Sagarpa). 2010. http://www.sagarpa.gob.mx/agricultura/Documents/SistNacRecGen.pdf (9 de enero de 2014).
Secretaría de Agricultura, Ganadería, Recursos Hidráulicos, Pesca y Alimentación (Sagarpa). 2012. http://www.sagarpa.gob.mx/saladeprensa/boletines2/paginas/2012B198 (9 d enero de 2014)
Schwarzbach, A. and H. Aguilar. 2012. The use of ADN barcoding for identification of medicinal plant products: an example from plants used in the Southern Texas-Mexico border region. BMC Complementary and Alternative Medicine (Suppl 1). p. 37. DOI:10.1186/1472-6882-12-S1-P37. [ Links ]
Selkoe K. A. and R. J. Toonen. 2006. Microsatellites for ecologists: a practical guide to suing and evaluating microsatellite markers. Ecology Letters 9: 615-629. [ Links ]
Solórzano S., A. C., A. Cortés, P. Ibarra, P. Dávila and K. Oyama. 2009. Isolation, characterization and cross-amplification of polymorphic microsatellite loci in the threatened endemic Mammillaria crucigera(Cactaceae). Molecular Ecology Resurces 9 (1): 156-158. [ Links ]
Soulé M. E. and I. S. Mills. 1992. Conservation genetics and conservation biology: a troubled marriage. In: Sandlund, O. T., K. Hindar and A. H. D. Brown (eds.). Conservation of biodiversity for sustainable development. Scandinavian University Press. Oslo, Norway. pp. 55-69. [ Links ]
Taberlet, P., L. P. Waits and G. Luikart. 1999. Noninvasive genetic sampling:look before you leap. Trends in Ecology and Evolution 14: 323–327. [ Links ]
Taylor S. A., L. Maclagan, D. J. Anderson and V. L. Friesen. 2011. Could specialization to cold-water upwelling systems influence gene flow and population differentiation in marine organisms? A case study using the blue-footed booby, Sula nebouxii. Journal of Biogeography 38:883–893. [ Links ]
Varshney R. K., J. C. Glaszmann, H. Leung and J. M. Ribaut. 2010. More genomic resources for less-studied crops. Trends in Biotechnology 28(9): 452-460. [ Links ]
Vázquez B., C., P. Álvarez R., C. Escobedo F., G. Anguiano V., M. Duran A., V. Serrano P. and C. M. Escobedo B. 2010. Detection of white spot syndrome virus (WSSV) in the Pacific oyster Crassostrea gigas. Journal in Invertebrate Pathology 104(3): 245-247. [ Links ]
Wallace, J. R. and J. C. Ross. 2012. The application of forensic science to wildlife evidence. In: Huffman, J. E. and J. R. Wallace (eds.). Wildlife forensics:methods and applications. Wiley-Blackwell, Ltd. London, UK. pp. 35-50. [ Links ]
Wang, W., H. Xia, X. Yang, T. Xu, H. J. Si, X. X. Cai, F. Wang, J. Su, A. A. Snow and B. R. Lu. 2013. A novel 5-enolpyruvoylshikimate-3-phosphate (EPSP) synthase transgene for glyphosate resistance stimulates growth and fecundity in weedy rice (Oryza sativa) without herbicide. New Phytologist. DOI:10.1111/nph.12428.
Wegier A., A. Piñeyro N., J. Alarcón, A. Gálvez M., E. R. Álvarez B. and D. Piñeiro. 2011. Recent long-distance transgene flow into wild populations conforms to historical patterns of gene flow in cotton (Gossypium hirsutum) at its centre of origin. Molecular Ecology 20: 4182-4194. [ Links ]
Yong, E. 2013. Flesh-eating flies map forest biodiversity. Nature News. Nature Publishing Group. DOI:10.1038/nature.2013.12147.
Zaldívar R., A., .J. Martínez, F. Ceccarelli, V. de Jesús B., A. Rodríguez P., A. Reséndiz F. and M. Smith. 2010. ADN barcoding a highly diverse group of parasitoid wasps (Braconidae: Doryctinae) from a Mexican nature reserve. Mitochondrial ADN 21(1): 18-23. [ Links ]














