Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ciencias forestales
versión impresa ISSN 2007-1132
Rev. mex. de cienc. forestales vol.5 no.21 México ene./feb. 2014
Articulos
Análisis de la diversidad genética del germoplasma de teca (Tectona grandis L. f.) en Ecuador
Analysis of genetic diversity of teak (Tectona grandis L. f.) germplasm in Ecuador
José Enrique Nieto-Rodríguez1, Sanjuana Hernández-Delgado2, Emmerik Motte-Darricau3 y Netzahualcóyotl Mayek-Pérez2
1 Unidad de Investigación Científica y Tecnológica de la Universidad Técnica Estatal de Quevedo.
2 Centro de Biotecnología Genómica, Instituto Politécnico Nacional. Correo-e: nmayek@ipn.mx
3 Laboratorio de Biotecnología, Universidad de Guayaquil.
Fecha de recepción: 1 de abril de 2013;
Fecha de aceptación: 20 de noviembre de 2013.
RESUMEN
La teca (Tectona grandis) es una especie originaria de Indochina que fue introducida a Ecuador en 1950, donde actualmente tiene alta demanda en el sector maderero. Existen escasos estudios locales que abordan temas de genética de poblaciones y mejoramiento en especies forestales de importancia económica en el país, lo cual es fundamental para conocer la diversidad y las relaciones entre poblaciones y fuentes de semilla, así como para identificar ejemplares que resistan condiciones adversas, ya que son los más aptos para utilizarse en la obtención de individuos con mayor productividad y rendimiento mediante herramientas biotecnológicas. El presente trabajo tuvo como objetivo analizar la diversidad y las relaciones genéticas de diez poblaciones de teca (seis de Ecuador, tres de Costa Rica y una de Honduras) con marcadores RAPD. El análisis de datos indicó la formación de dos grupos poblacionales, uno incluyó a las de Ecuador y el otro se conformó con las de Costa Rica y Honduras. Las poblaciones ecuatorianas, probablemente, provienen de Pichilingue (INIAP) o Granada, que a su vez son los más antiguas y con mayor diversidad genética en el país. Las introducciones posteriores de la especie, procedentes de Centroamérica, no resultaron emparentadas con las previamente establecidas. Los resultados evidencian la variabilidad de la teca como producto de la constante polinización libre y natural, así como la introducción de nuevas fuentes génicas.
Palabras clave: Análisis RAPD, genética de poblaciones, litoral ecuatoriano, marcadores moleculares, Tectona grandis L. f., variabilidad genética.
ABSTRACT
Teak (Tectona grandis) is a native species of Indochina that was introduced in 1950 into Ecuador, where it is currently in high demand in the timber industry. There are few local studies on population genetics and on the improvement of economically important forest species in this country, despite the fact that such studies are essential to find out about the diversity of and the relationships between populations and seed sources, as well as to identify the specimens that are resistant to adverse conditions, since these are the most suitable for the obtainment of specimens with a higher productivity and yield through the use of biotechnological tools. The purpose of this study was to analyze the genetic diversity of and relationships between ten teak populations (six from Ecuador, three from Costa Rica, and one from Honduras) with RAPD markers. The data analysis pointed at the formation of two population groups -one of which included the populations from Ecuador, while the other comprised those from Costa Rica and Honduras. The Ecuadorian populations probably come from those of Pichilingue (INIAP) or Granada, which are the oldest and the richest in genetic diversity in the country. Later introductions of the species from Central America had no kinship with those previously established. The results evince the variability of teak as the product of the ongoing free natural pollination, as well as of the introduction of new gene sources.
Key words: RAPD analysis, population genetics, Ecuatorian coastline, molecular markers, Tectona grandis L. f., genetic variability.
INTRODUCCIÓN
La teca (Tectona grandis L. f.) es un árbol originario de Indochina que se introdujo en los países tropicales de América Latina hace aproximadamente 100 años. Se estima que, actualmente, sus plantaciones en el mundo alcanzan los 3 millones de hectáreas (Centeno, 1997). En el caso que compete a Ecuador, esta especie se adaptó y adquirió gran importancia económica y ecológica por su rápido crecimiento, la calidad de madera y su gran aportación de biomasa al suelo (Hartmann et al., 1990), que se ha traducido en una alta demanda en programas de reforestación para zonas con problemas de sequía. Su madera es apreciada por sus excepcionales cualidades de dureza, resistencia, facilidad para su trabajo y que no se oxida ni corroe en contacto con metales (Gupta et al., 1980), por lo que resulta ideal para las industrias mueblera, naval y de la construcción. Es valorada por su aspecto estético, con tonos desde el dorado hasta el marrón oscuro, pasando por tonos rojizos, con fibras rectas y textura fina. Además, la elevada concentración de aceites naturales le confiere un carácter untuoso y características únicas como: resistencia al agua, a la humedad y a las condiciones de sol y lluvia (Ramos, 2000).
El primer cultivo se estableció en la Estación Experimental Tropical "Pichilingue" del Instituto Nacional de Investigaciones Agropecuarias (INIAP), localizado en Quevedo, Ecuador, cuyo germoplasma provenía de la India. A partir de esa población, se generaron los plantíos comerciales en el país; sin embargo se desconoce el origen de los demás campos forestales actuales en el litoral ecuatoriano, ya que pueden derivar de Quevedo, o de otra procedencia. La reproducción de T. grandis se realiza por semilla; es decir, por vía sexual; generalmente, los individuos que las aportan no son seleccionados ni por su origen ni por sus características físicas, lo que propicia una alta variabilidad fenotípica y genotípica en las parcelas (Dunsdon y Simons, 1996). La disponibilidad de germoplasma mejorado genéticamente y que provenga de áreas productoras de semillas o huertos semilleros en Ecuador es nula; y aún no existen programas destinados al mejoramiento de la especie.
A pesar de su importancia y potencial forestal, las investigaciones a la fecha, se concentran en técnicas de propagación vegetativa, cultivo de tejidos in vitro, modelos y densidades de plantación, estudios de mercado y biotecnología (Dalmau et al., 2004). Se necesita realizar trabajos orientados al conocimiento genético de la especie dentro del país que permitan la implementación de planes de manejo y mejora genética; asimismo, conocer los patrones de variabilidad del germoplasma permitirá determinar su potencial para la formulación y ejecución de programas destinados a su conservación y a establecer las bases para estructurar un programa integral para hacer más productivo y rentable el aprovechamiento de uno de los árboles de mayor importancia nacional y así, contribuir al desarrollo del sector.
La presente investigación tuvo como objetivo analizar la diversidad y las relaciones genéticas con fuentes de semilla de teca entre poblaciones del Ecuador y otros países de América, con base en técnicas de biología molecular, mediante RAPD (ADN Polimórfico Amplificado al Azar), que constituyen una herramienta importante para este tipo de estudios (Neale y Sederoff, 1991; Ahuja, 2001).
MATERIALES Y MÉTODOS
Material biológico
Se estudiaron 10 poblaciones de T. grandis distribuidas en distintas localidades del macrositio denominado "litoral ecuatoriano" (Cuadro 1, Figura 1), que se caracteriza por tener condiciones de bosque húmedo tropical, cálido y que se desarrolla en altitudes menores a 200 m (Nieto - Rodríguez et al., 2013). Cuatro poblaciones provenían de la región de Guayas, tres de Los Ríos y tres de Costa Rica. En cada sitio se recolectaron muestras de hojas jóvenes de 10 plantas; dicho material se almacenó y procesó en el Laboratorio del Programa de Biotecnología de la Universidad de Guayaquil, en Guayaquil, Ecuador.
A excepción del germoplasma de Pichilingue, que constituye la introducción original de teca a Ecuador, se asume que el restante del país fue propagado a partir de este mismo. La población de La María es originaria de Honduras y las tres poblaciones de Costa Rica se conservan en Quevedo, Ecuador.
La población de La María se cultivó cerca del INIAP - Pichilingue; mientras que las de Costa Rica provienen directamente de dicho país.
Evaluación de protocolos de extracción de ADN
Para la extracción y purificación del ADN se evaluaron tres protocolos: Dellaporta et al. (1983), Doyle y Doyle (1987) y Uyemoto et al. (1998). El primero consistió en macerar tejido con nitrógeno líquido (-196 °C) hasta obtener un polvo fino. Se añadió buffer de extracción (100 mM Tris - HCl, pH 9; 500 mM EDTA; 500 mM NaCl; 10 mM β-mercaptoetanol; SDS 20 %) y se incubó en baño maría a 65 °C durante 10 min. Se añadió acetato de potasio, centrifugó a 1 700 rpm por 30 min y se dejó reposar de 12 h a 20 °C. Se realizó una segunda centrifugación para formar la pastilla de ADN y retirar el isopropanol, a la que se adicionó solución para diluir (50 mM Tris-HCl, pH 8 y 10 mM EDTA); posteriormente, se aplicó fenol-cloroformo (25:1), se centrifugó y se extrajo el sobrenadante para nuevamente colocarse en isopropanol (400 mL y acetato de sodio (60 mL). Una vez formado el pellet, se retiró el resto al invertir el tubo Eppendorf y se dejó secar 15 min. Las muestras se lavaron con etanol 70 % dos veces y se resuspendieron en TE 1X.
El protocolo de Doyle y Doyle (1987) consistió en macerar de 25 a 50 mg de tejido con nitrógeno líquido y se agregaron 700 mL de buffer de extracción (2 % CTAB, 1.42M NaCl, 20mM EDTA, 100mM Tris HCl, pH 8.0 y 2% PVP 40), precalentado a 65 °C. Se añadieron 0.5 mM de ácido ascórbico modificado (0,2% de β- mercaptoetanol) para después centrifugar 2 min a 13 000 rpm., se recuperó el sobrenadante y se agregaron 4 mL de ARNasa (10 mg mL-1), se mezcló por inversión y se incubó 5 min a 65 °C. Posterior a ello, se agregaron 570 mL de cloroformo isoamílico (24:1) y se centrifugó a 13 000 rpm por 10 min a temperatura ambiente. Luego, se precipitó el ADN al adicionar 0.7 vol de isopropanol frío y se centrifugó 5 min. a 13 000 rpm. Por último, la pastilla se lavó con etanol 70 % y el ADN se resuspendió en 15 µL de TE 1X.
El protocolo de Uyemoto et al. (1998) consistió en colocar el amortiguador CTAB a 65 °C durante 15 min (2 % CTAB; 1.4 M NaCl; 20 mM EDTA, pH 8; 100 mM Tris HCl, pH 8; 0.2% de β-mercaptoetanol y agua ultrapura). En seguida, se maceraron 100 mg de tejido vegetal y se añadieron 800 mL del amortiguador CTAB para después incubar 20 min a 60 °C; se centrifugó 2 min a 13 000 rpm y se recuperó el sobrenadante, al que se agregaron 600 mL de cloroformo-isoamílico 24:1 para luego centrifugar 5 min a 13 000 rpm. El sobrenadante se transfirió a un tubo limpio al que se adicionó 1 vol de isopropanol frío; y la mezcla se colocó 10 min en hielo para precipitar el ADN. Se centrifugó 8 min a velocidad máxima, a continuación la fase acuosa se desechó. La pastilla se enjuagó con etanol 80% y se secó al ambiente, para después resuspenderla en 25 mL de TE. Finalmente, se agregó RNAsa (10 µg mL-1) y se incubó 1 h a 37 °C.
El ADN procedente de cada uno de los protocolos se visualizó en gel de agarosa 1.2 %. Para determinar la eficiencia de los procedimientos se evaluó tanto la cantidad como la calidad del ADN obtenido, con base en los valores de pureza, cantidad y absorbancia en un espectrofotómetro con luz UV Lambda 40 (Perkin Elmer®). Además, al ADN extraído se le hicieron pruebas de digestibilidad, estabilidad y amplificación por PCR con ayuda de oligonucleótidos decaméricos OPA 15 y OPC 7 de la casa Operon® (Operon Technologies) (Nieto-Rodríguez et al., 2005).
Análisis a través de RAPD
El análisis con RAPD se desarrolló de acuerdo con el protocolo descrito por Williams et al. (1990). La técnica consiste en una PCR con volumen total de reacción de 25 mL que incluye Taq DNA polimerasa, dNTPs, cloruro de magnesio y buffer para PCR. Las condiciones utilizadas para la amplificación constaron de tres segmentos: el primero de desnaturalización a 94 °C por 1 min; el segundo de 40 ciclos con desnaturalización a 94 °C por 1 min, hibridación a 36 °C por 1.30 min y polimerización a 72 °C por 2 min; así como uno final de polimerización de 10 min a 72 °C. Los fragmentos se separaron por electroforesis a 65 V por 150 min en un gel de agarosa 1.4 %. La visualización de los productos de PCR se realizó en un trans iluminador con luz UV.
Análisis estadístico
Se amplificaron quince iniciadores decaméricos (Operon Technologies) en tres muestras (INIAP - India, Santa Cruz - Costa Rica y Granada - Ecuador) y se seleccionaron los oligonucleótidos con mayor número de bandas amplificadas (OPA 12; OPA 16; OPA 15; OPC 1; OPC 4; OPC 7; OPC 8; OPC 9 y OPC 10). La lectura de los geles RAPD se efectuó de manera directa mediante la asignación del número uno a la banda con mayor peso molecular y así sucesivamente, en orden descendente hasta la banda con menor peso molecular en el gel. Para cada oligonucleótido se calcularon las bandas amplificadas, en las que se denominó como 1 a la presencia de una banda y 0 a la ausencia de la misma. Se asumió que las bandas con igual velocidad de migración en el gel en individuos diferentes eran idénticas.
Con los datos binarios se elaboraron matrices que fueron utilizadas para calcular las distancias genéticas entre poblaciones con base en el coeficiente de apareamiento simple (Nei y Li, 1979). Con estas, se construyó un dendrograma, según el método de agrupamiento de pares no ponderados con medias aritméticas (UPGMA) (Nei y Kumar, 2000). Los valores de diversidad para cada iniciador se calcularon por ecotipo, con base en la distancia de Nei (1978):

Donde:
p = Frecuencia de la banda en las poblaciones
n = Número de individuos analizados
El valor de la diversidad genética que se detectó con cada oligonucleótido fue calculado con base en la estimación de r. El valor de diversidad de todas las muestras (H) se calculó con base en el número total de marcadores e individuos analizados y al utilizar la ecuación:
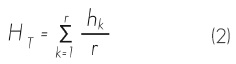
Donde:
hk = Medias ponderadas por el valor de todos los marcadores
r = Número de marcadores revelados para cada oligonucleótido
La matriz de similitud también se usó para realizar un análisis jerárquico de varianza molecular (AMOVA) (Excoffier et al., 2005) como fue descrito por Huff et al. (1993), con el programa Arlequin 2.0 (Schneider et al., 1997).
La robustez del dendrograma RAPD se evaluó mediante el remuestreo y la generación de 1 000 matrices de distancias genéticas generadas a partir de matrices de distancias reconstruidas y reducidas, como se hizo el análisis de conglomerados original (Nei y Li, 1979; Skroch et al., 1992), de modo que se estimaron los límites de confianza de cada comparación apareada (Felsenstein, 1989). La topología del dendrograma de consenso resultante fue comparada del original. El análisis de robustez se llevó a cabo con el programa FreeTree (Hampl et al., 2001) y se visualizó con el programa TreeView versión 1.6.6 (Page, 2000).
La estructura de las poblaciones se infirió con base en el algoritmo de agrupamiento con un modelo Bayesiano, para lo que se utilizó el programa de cómputo STRUCTURE versión 2.3.4 (Pritchard y Wen, 2000; Pritchard et al., 2010); el cual se corrió considerando K conglomerados genéticos, caracterizados con las matrices de frecuencias de alelos. Así, se estimó la proporción del genoma por individuo derivado de cada conglomerado genético (proporción de ascendencia). Se efectuaron 20 corridas independientes con K de 1 a 10, con 30 000 repeticiones de Cadenas de Markov - Monte Carlo (CMMC) y 300 000 periodos de rodaje (burn-in periods), así como un modelo de "mezcla" y frecuencias de alelos correlacionadas. El valor óptimo de K se calculó de acuerdo con los criterios de Evanno et al. (2005), con el programa Harvester versión 0.56.4 (Earl and von Holdt, 2012).
RESULTADOS
Evaluación de protocolos de aislamiento de ADN
El protocolo de aislamiento de ADN de Uyemoto et al. (1998) fue el único cuyo ADN se pudo fue amplificar mediante PCR. Las pruebas de amplificación con dos oligonucleótidos decaméricos RAPD (OPA 15 y OPC 7) se hicieron con ADN aislado a partir de los tres protocolos y se generaron perfiles genéticos con 9 y 11 bandas, respectivamente. La concentración del ADN aislado causó problemas en las amplificaciones preliminares debido a su alta concentración (≥ 500 ng mL-1) y, por consiguiente las muestras se diluyeron 1:20 para el análisis con RAPD. Mediante el protocolo de Uyemoto et al. (1998) se obtuvieron valores promedio de pureza de 1.40 260 x 280-1; concentración de 560 mg mL-1 y absorbancia de 0.265.
Análisis a través de RAPD
El análisis de RAPD se desarrolló con nueve iniciadores decaméricos, y generó 111 bandas en las diez poblaciones de T. grandis, dichas bandas tuvieron alta reproducibilidad y resolución, detectándose 28 productos monomórficos y 83 polimórficos (74.9 %) (Cuadro 2). El intervalo de peso molecular de los productos amplificados fue de 0.4 a 2.5 Kb y el número de productos amplificados, por población, varió de 10 a 14. El porcentaje de marcadores polimórficos correspondió a 85.7 % para el OPC 9 y de hasta 64.2 % para OPC 4.
El AMOVA indicó diferencias significativas entre las dos jerarquías analizadas: poblaciones de teca del Litoral Ecuatoriano y del extranjero. La mayor proporción de la varianza molecular se determinó entre poblaciones (Cuadro 3). El valor FST=0.56 evidenció alta diferenciación genética entre y dentro de las poblaciones analizadas. El dendrograma de poblaciones mostró la formación de dos grupos, uno incluyó aquellas que se localizan en Ecuador y el otro, a las del extranjero (Figura 2).
Los números en los nodos representan los valores de robustez (%) para 1 000 repeticiones.
El análisis con los iniciadores RAPD OPA 15 y OPA 16 produjeron altos niveles de diversidad en teca. El valor HT fue de 0.71 (Cuadro 4). Las poblaciones de El Empalme, Alba Isabel, San Nicolás, Balzar, Granada y Pichilingue son genéticamente similares y corroboran la teoría de que la primera introducción de teca al Ecuador ocurrió en Pichilingue (población 4) y de esta última, aparentemente, se originaron las otras poblaciones de establecidas a lo largo del Litoral Ecuatoriano.
Para K = 2 el análisis de conglomerados con enfoque Bayesiano discriminó las poblaciones ecuatorianas de las demás. Los resultados obtenidos con K = 2 fueron consistentes (Figura 3), de acuerdo con el cálculo de los valores de ΔK.
DISCUSIÓN
La extracción del ADN de plantas que contienen grandes cantidades de polifenoles, taninos y polisacáridos es complicada (Webb y Knapp, 1990; Varadarajaran y Prakash, 1991). En el caso de T. grandis, así como en otras especies maderables, existen altas concentraciones de metabolitos secundarios, tales como polifenoles y taninos, que inhiben la acción enzimática de polimerasas y endonucleasas de restricción, mismas que son fundamentales en los procesos de digestión y amplificación por PCR (Collins y Symons, 1992; Nieto-Rodríguez et al., 2005). Para desarrollar estrategias de genotipificación como los RAPD o el empleo de cualquier otro tipo de marcadores moleculares, es necesario contar con ADN de buena calidad que se recolecte de hojas maduras, debido a que las más jóvenes contienen concentraciones superiores de compuestos que dificultan la extracción, mientras que las de mayor edad presentan tejidos lignificados (Matthew et al., 1998). La concentración del ADN fue muy alta, por lo cual se diluyó para optimizar el proceso de amplificación. Ferreira y Grattapaglia (1998) sostienen que en el análisis RAPD uno de los factores clave es justamente este, puesto que, cuando la cantidad es muy alta, la lectura de bandas en el gel no es muy clara; por otro lado, si esta es inferior, se ocasiona la pérdida de bandas importantes.
La elección de RAPD como estrategia de análisis genético se hizo porque son marcadores moleculares apropiados para el estudio de especies en las cuales se desconoce su secuencia genómica, aparte de ser una técnica de fácil implementación que permite la detección correcta de la diversidad genética en especies forestales, tales como Pinus muricata D. Don. (Wu et al., 1999), Pilocarpus microphyllus Stapf. (Moura et al., 2005), Pinus patula Schiede ex Schltdl. et Cham. (Luna-Rodríguez et al., 2005) y Schizolobium parahybum (Vell.) Blake (Canchignia-Martínez et al., 2007), en comparación con otras técnicas de genotipificación más complejas como los AFLP (polimorfismos en la longitud de los fragmentos amplificados) y los SSR (microsatélites). Los RAPD fueron muy eficientes para detectar la variación de las accesiones analizadas, con los que se obtuvieron valores de diversidad HT ≥ 0.70, que son mayores a los registrados en P. sylvestris L. (<0.37) (Szmidt et al., 1996) y P. oocarpa Schiede ex Schltdl. (<0.40) (Díaz et al., 2001). En 15 poblaciones de S. parahybum, especie maderable nativa del Ecuador, se utilizaron diez oligonucleótidos RAPD de las series OPA y OPC y se obtuvieron de 12 a 16 productos amplificados por oligonucleótido, porcentajes de polimorfismo de 95 % y HT = 0.87 (Canchignia-Martínez et al., 2007). El alto valor de HT en este trabajo evidencia la constante recombinación genética natural que presenta Tectona grandis en Ecuador y que responde a factores bióticos y abióticos que inciden en la reproducción y diseminación de su semilla, incluido el hombre.
A pesar de que la teca es una especie exótica en Ecuador, se infiere que, con base en los resultados, la población que originó al resto del germoplasma ecuatoriano analizado fue Pichilingue (India). Las distancias genéticas entre las poblaciones fueron relativamente bajas y se agruparon de acuerdo con su origen, lo que obedece al gran parentesco de los individuos estudiados, similar a lo citado por Chung et al. (2004) en Quercus acutissima Carruth.. El germoplasma ecuatoriano se dividió en dos subgrupos, uno con las accesiones más antiguas (≥ 40 años) y genéticamente más diversas del INIAP-Pichilingue y Granada; y otro que incluyó poblaciones de Alba Isabel, El Empalme, Balzar y San Nicolás, poblaciones más recientes (≥ 25 años) y que se corroboró, son menos variables. El bajo nivel de diversidad en las poblaciones jóvenes de teca se asocia con el origen limitado del germoplasma del cual proceden, pues en este caso se utilizaron árboles con características fenotípicas superiores pero con estrecho parentesco entre sí. A partir de lo anterior se considera a Pichilingue y a Granada como los dos probables sitios de plantación inicial de teca en Ecuador, de donde posteriormente ocurrió la dispersión de la especie como cultivo comercial al resto del país.
Las locaciones de teca con fuente de semilla de Honduras y Costa Rica mostraron los niveles de diversidad genética más altos, lo cual sugiere que la variación en Ecuador también se ha enriquecido con la introducción de germoplasma nuevo. Las diferencias significativas entre los grupos considerados (ecuatorianos y no ecuatorianos) el AMOVA corrobora la variación genética significativa, producto de un origen geográfico distinto, por el alto grado de diferenciación genética. Estudios realizados en diferentes especies forestales como Cedrela odorata L. y Pinus oocarpa muestran que, por lo general, existe una relación directa entre las distancias geográficas y las genéticas de las poblaciones (Parker y Hamrick, 1996; Parker et al., 1997).
Este trabajo ofrece resultados similares a los observados en otro taxón de importancia para Ecuador en el que se analizó la diversidad genética y las relaciones entre su germoplasma, el "pachaco" (Schizolobium parahybum), especie exótica que se adaptó a las condiciones ambientales del país, y en la que se generó diversidad, producto de la polinización libre entre árboles dispersos (Canchignia-Martínez et al., 2007). La reproducción sexual ha favorecido cierta variabilidad genética que los silvicultores ecuatorianos han aprovechado de acuerdo con sus intereses, o bien, la selección natural privilegió la predominancia de los mejores individuos (Tipan, 1982). Una demanda importante y constante es la disponibilidad de semilla de plantas superiores para el establecimiento de las nuevas plantaciones forestales. Sin embargo, en general, se produce o se dispone de poca información sobre la naturaleza genética, origen o historia de la semilla para utilizarse en dichas plantaciones (Canchignia-Martínez et al., 2007).
CONCLUSIONES
Existe diferenciación genética significativa entre fuentes de semilla de T. grandis de Ecuador y las provenientes del extranjero. El germoplasma ecuatoriano es genéticamente más diverso.
Es probable que las plantaciones comerciales de teca del Litoral Ecuatoriano provengan de semilla producida por las introducciones de Pichilingue (INIAP) y Granada. Las plantaciones jóvenes establecidas con semillas de Honduras y Costa Rica son diferentes al germoplasma que ya existía en el país.
AGRADECIMIENTOS
Los autores agradecen el apoyo económico de éste trabajo por parte del Dr. E. Motte y de la Universidad Técnica Estatal de Quevedo, Ecuador. S. Hernández-Delgado y N. Mayek-Pérez son becarios del Sistema Nacional de Investigadores y de los programas de estímulos al desempeño de los investigadores (EDI) y becas de exclusividad (COFAA) del Instituto Politécnico Nacional.
REFERENCIAS
Ahuja, M. R. 2001. Recent advances in molecular genetics of forest trees. Euphytica (121): 173-195. [ Links ]
Canchignia - Martínez, H. F., S. Hernández-Delgado, M. González - Paz, E. Motte and N. Mayek - Pérez. 2007. Genetic relationships among Schizolobium parahybum (Vell.) Blake (Leguminosae) ecotypes from Ecuador and other countries. Silv. Genet. (56) : 214 -22. [ Links ]
Centeno J., C. 1997. The management of teak plantations. Mérida, Venezuela. www.tremail.nl/teakscan.dal/files/mngteak.htm. (12 de febrero de 2009). [ Links ]
Collins, G. G. and R. H. Symons. 1992. Extraction of nuclear DNA from grape vine leaves by a modified procedure. Plant Mol. Biol. Rep. (10): 233 – 235. [ Links ]
Chung, M. Y., M. G. Chung, E. R. Myers, J. M. Chung, K. J. Kim, C. W. Park, B. Y. Sun and J. H. Park. 2004. Spatial genetic structure in distributed populations of Quercus acuatissima (Fagaceae). Silv. Genet. (53): 206 - 211 [ Links ]
Dalmau, K. P., S. J. Gallardo B. e I. Rivadeneira. 2004. Proyecto del cultivo de la teca, como alternativa de forestación e inversión a largo plazo. Escuela Superior Politécnica del Litoral. Guayaquil. Ecuador. http://www.dspace.espol.edu.ec/bitstream/123456789/648/1/1195.pdf (22 de octubre de 2009). [ Links ]
Dellaporta, S. J., J. Wood and J. V. Hicks. 1983. A plant DNA minipreparation. Version II. Plant Mol. Biol. Rep. (1): 19 - 21. [ Links ]
Díaz V., L. M. Muñiz and E. Ferrer. 2001. Random amplified polymorphic DNA and amplified fragment length polymorphism assessment of genetic variation in Nicaragua populations of Pinus oocarpa. Mol. Ecol. (10): 2593 - 2603. [ Links ]
Doyle J. J. and J. L. Doyle. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem. Bull. (19): 11-15. [ Links ]
Dunsdon, A. J., and A. J. Simons. 1996. Provenance and progeny trials. In: J. L. Stewart, G. E. Allison, and A. J. Simons (eds). Gliricidia sepium. Genetic Resources for Farmers. Oxford Forestry Institute, University of Oxford. Oxford, UK. 125 p. [ Links ]
Earl, D. A. and B. M. vonHoldt. 2012. Structure Harvester: a website and program for visualizing structure output and implementing the Evanno method. Cons. Genet. Resour. (4): 359 - 361. [ Links ]
Evanno, G., S. Regnaut and J. Goudet. 2005 Detecting the number of clusters of individuals using the software Structure: a simulation study. Mol. Ecol. (14): 2611 - 2620. [ Links ]
Excoffier, L., G. Laval and S. Schneider. 2005. Arlequin ver. 3.0: An integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online (1): 47 - 50. [ Links ]
Felsenstein, J. 1989. PHYLIP manual, version 3.2. University of California Herbarium, Berkeley, CA. USA. s/p. [ Links ]
Ferreira, M. y D. Grattapaglia. 1998. Introducción al uso de marcadores moleculares en el análisis genético. 3ª ed. EMBRAPA-CENARGEN. Brasilia, Brasil. 218 p. [ Links ]
Gupta, P. K., A. L. Nadgir, A. F. Mascarenhas and V. Jaganthan. 1980. Tissue culture of forest trees: clonal multiplication of Tectona grandis L. (teak) by tissue culture. Plant Sci. Lett. (17): 259 - 268. [ Links ]
Hampl, V., A. Pavlicek and J. Flegr. 2001. Construction and bootstrap analysis of DNA fingerprinting - based phylogenetic trees with the freeware program Freetree: Application to Trichomonad parasites. Int. J. Syst. Ecol Microbiol. (51): 731 - 735. [ Links ]
Hartmann, H. T., T. E. Kester and F. T. Davies. 1990. Plant propagation: principles and practices. 5th ed. Prentice and Hall. Englewood Cliffs, NJ. USA. 647 p. [ Links ]
Huff, D., R. Peakall and E. Smouse. 1993. RAPD variation whithin and among natural populations of outcrossing buffalograss [Buchoe dayloides (Nutt.) Engelm]. Theor. Appl. Genet. (86): 927 - 934. [ Links ]
Luna-Rodríguez, M., J. López-Upton and L. Iglesias-Andreu. 2005. Morphometric and molecular (RAPD) variability in a plantation of Pinus patula in Veracruz, México. Agrociencia (39): 231 - 235. [ Links ]
Matthew, D., M. Davey, J. Brian, K. Lowe, F. Bligh, S. Roach and C. Jones. 1998. DNA isolation and AFLP genetic fingerprinting of Theobroma cacao(L.). Plant Mol. Biol. Rep. (16): 49 –59. [ Links ]
Moura E. F., J. P. Pinto, J. B. Santos and O. A. Almeira. 2005. Genetic diversity in a jaborandi (Pilocarpus microphyllus Stapf.) germplasm bank assessed by RAPD markers. Rev. Bras. Plantas Medicinais (7): 1-8. [ Links ]
Neale, D. and R. Sederoff. 1991. Paternal inheritance of chloroplast DNA and maternal inheritance of mitochondrial DNA in loblolly pine. Theor. Appl. Genet. (77): 212 – 216 [ Links ]
Nei, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics (89): 583 - 590. [ Links ]
Nei, M. and S. Kumar. 2000 Molecular Evolution and Phylogenetics. Oxford University Press, Oxford. UK. pp. 77-183. [ Links ]
Nei, M. and W. H. Li. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleasas. Proc. Natl. Acad. Sci. (76): 5269 - 5273. [ Links ]
Nieto-Rodríguez, J. E., L. Ramos y E. Motte. 2005. Extracción y purificación de ADN de Tectona grandis L. para su empleo en la técnica RAPD. Foresta Veracruzana (7):1 - 6. [ Links ]
Nieto-Rodríguez, J. E., S. Hernández-Delgado and N. Mayek-Pérez. 2013. Morphological and genetic analysis of Triplaris guayaquilensis Wedd (Polygonaceae): one native tree from Ecuador. Ciencia Forestal (23): 415 - 423. [ Links ]
Page, R. D. M. 2000. Tree View. Division of Environmental and Evolutionary Biology, Institute of Biomedical and Life Sciences. University of Glasgow. Glasgow, Scotland. http://taxonomy.zoology.gla.ac.uk/rod/treeview.html ( 12 de septiembre de 2009). [ Links ]
Parker, K. and A. Hamrick. 1996. Genetic variation in sand pine (Pinus clausa). Canadian J. For. Res. (26): 244–254 [ Links ]
Parker, A., A. Hamrick and E. Stacy. 1997. Allozyme diversity in Pinus virginiana (Pinaceae): Intraspecific and interspecific comparisons. Amer. J. Bot. (84): 1372 - 1382. [ Links ]
Pritchard, J. K. and X. Wen 2000. Inference of population structure using moltilocus genotype data. Genetics (155): 945 - 959. [ Links ]
Pritchard, J. K., X. Wen and D. Falush. 2010. Documentation for structure software: version 2.3. University of Chicago. Chicago, IL. USA. 38 p. [ Links ]
Ramos, L. 2000. Algunos avances en la morfogénesis de la teca (Tectona grandis L.). Tesis de Maestría en Ciencias. Universidad de Ciego de Ávila. Ciego de Ávila, Cuba. 55 p. [ Links ]
Schneider, S., J. M. Kueffer, D. Roessli and L. Excoffier. 1997. Arlequin version 1.1: A software for population genetic data analysis. Genetics and Biometry Laboratory. University of Geneva. Geneva, Switzerland. s/p. [ Links ]
Skroch, P., J. Tivang and J. Nienhuis. 1992. Analysis of genetic relationships using RAPD markers data. In: Crop Science Society of America (ed.). Applications of RAPD technology to plant breeding. Madison, WI. USA. pp. 26 - 29. [ Links ]
Szmidt, A., X. Wang and M. Lu. 1996. Empirical assessment of allozyme and RAPD variation in Pinus sylvestris (L.) using haploid tissue analysis. Heredity (76): 412 - 420 [ Links ]
Tipan, G. 1982. El Pachaco. Centro Forestal Conocoto del INIAP. Quito, Ecuador. 18 p. [ Links ]
Uyemoto, J. K., Y. P. Zhang and B. C. Kirkpatrick. 1998. A small-scale procedure for extracting nucleic acids from woody plants infected with various phytopathogens for PCR assay. J. Virol. Meth. (23): 312 - 316. [ Links ]
Varadarajaran, G. S. and C. S. Prakash. 1991. A rapid and efficient method for the extraction of total DNA from the sweet potato and its related species. Plant Mol. Biol. Rep. (9): 6 – 12 [ Links ]
Webb, D. M. and S. J. Knapp. 1990. DNA extraction from a previously recalcitrant plant genus. Plant Mol. Biol. Rep. (8): 180 - 185 [ Links ]
Williams, J. G. K., K. Livak, J. Rafalski and S. Tingey. 1990. DNA polymorphic amplified by arbitrary primers is useful as genetic markers. Nucl. Acids Res. (18): 341 - 345. [ Links ]
Wu, J., K. Krutovski and S. H. Strauss. 1999. Nuclear DNA diversity population differentiation and phylogenetic relationships in the California close - cone pines based on RAPD and allozyme markers. Genome (42): 893 - 908. [ Links ]














