El ganado criollo representa un recurso genético de gran importancia para el abastecimiento de alimentos y materias primas en zonas de condiciones climáticas extremas, escasos recursos alimenticios y gran incidencia de enfermedades infecciosas y parasitarias1, contribuye potencialmente a la reducción del hambre y de la pobreza, así como al desarrollo sostenible2. No obstante, la incapacidad de apreciar el valor real biológico, económico y cultural de estos animales, ha ocasionado una agresiva extensión de razas altamente seleccionadas provocando una constante erosión que pone en peligro la existencia de estos recursos, representando una pérdida irreparable de variabilidad genética, que pudiera ser de gran valor para enfrentar los efectos del cambio climático.
En el estado de Oaxaca, se cuenta con una población de ganado criollo localizado en la región Mixteca (Figura 1A), denominado Criollo Mixteco. Fenotípicamente, son animales de talla mediana, con una alzada a la cruz promedio de 1.03 ± 0.16 m y un peso promedio de 176 ± 51.48 kg (parámetros reportados para una edad de 1 a 3 años)3, su pelaje puede ser negro o colorado uniforme o en berrendo (Figura 1B). El origen del bovino Criollo Mixteco se remonta a la época de la colonia, probablemente como parte de los animales que acompañaron a los primeros españoles para la construcción de los conventos a lo largo del territorio Oaxaqueño, en lo que actualmente se conoce como la Ruta Dominica4.
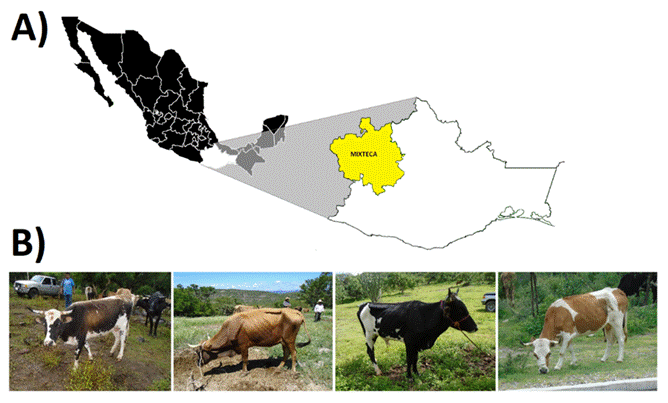
En la imagen se aprecia la coloración y características morfoestructurales del Bovino Criollo Mixteco
Figura 1 A) Ubicación geográfica de la región Mixteca de Oaxaca. B) Ejemplares de Bovino Criollo Mixteco
Desde su introducción a la región, el Criollo Mixteco fue adaptándose de forma exitosa a las condiciones geográficas y ambientales prevalecientes en la zona, la cual se caracteriza por una orografía complicada y fluctuaciones en la disponibilidad de alimentos y agua. No obstante, este ganado es capaz de ser productivo en dichas condiciones, lo que los convierte en animales idóneos para desarrollar sistemas de producción respetables con el medio ambiente, resilientes y capaces de enfrentar los cambios en el entorno, principalmente con la tendencia actual en América Latina hacia el desarrollo de sistemas de producción más intensivos y sostenibles5.
De acuerdo con la Organización de las Naciones Unidas para la Alimentación y la Agricultura (FAO), la identificación y caracterización genética del ganado criollo o local es un paso primordial en la conservación y aprovechamiento de estos recursos genéticos6. En este sentido, los marcadores moleculares microsatélites han demostrado ser una herramienta altamente efectiva para caracterizar genéticamente las poblaciones de ganado bovino criollo en América7-10, por lo que, en el presente estudio se evaluó la diversidad genética de la población de bovinos Criollos Mixtecos y sus relaciones genéticas con otras poblaciones bovinas locales y especializadas, mediante el empleo de 19 marcadores microsatélites y 32 poblaciones de referencia, con el objetivo de generar información sobre el estado de conservación de este valioso recurso genético local.
Se seleccionaron un total de 40 bovinos Criollos Mixtecos adultos (29 hembras y 11 machos), los cuales se identificaron con base en las características fenotípicas de ejemplares criollos reportadas previamente en la literatura, entre las que se encuentran el patrón de coloración, talla y parámetros zoométricos3,11, aquellos individuos que presentaban características fenotípicas propias de razas Cebú fueron descartados. Con el fin de evitar relaciones de parentesco cercano entre los individuos seleccionados, se incluyó únicamente un ejemplar por cada unidad de producción bovina muestreada, separadas geográficamente en diferentes comunidades de la Mixteca Oaxaqueña (17°48′00″ N, 97°46′00″ O), además de confirmar, mediante entrevista, la ausencia de conexiones genéticas (uso de sementales) entre las unidades de producción. El tamaño de la muestra se definió tomando como referencia la información publicada para estudios genéticos poblacionales mediante marcadores moleculares microsatélites12, así como el tamaño de muestra sugerido por la FAO para estudios de caracterización genética de poblaciones de ganado local mediante microsatélites (n=25 a 40)13.
El material biológico consistió en muestras de sangre total con anticoagulante (EDTA) obtenidas mediante punción aséptica de la vena yugular, a partir de las cuales se llevó a cabo la extracción de ácidos nucleicos, utilizando el kit ReliaPrep Blood gDNA Miniprep System (Promega), siguiendo las instrucciones del fabricante. Para el análisis genético de la población se empleó un panel de 19 marcadores microsatélites recomendados por la FAO-ISAG14 (Cuadro 1), los cuales se amplificaron mediante PCR y procesaron en un secuenciador capilar ABI377XL (Applied Biosystems), con la posterior tipificación alélica, siguiendo la metodología establecida en el Laboratorio de Mejora y Conservación de Recursos Genéticos Animales de la Universidad de Córdoba, España7.
Cuadro 1 Frecuencias alélicas observadas en la población de Bovinos Criollos Mixtecos
| BM1818 | BM1824 | BM2113 | CSRM60 | CSSM66 | |||||
|---|---|---|---|---|---|---|---|---|---|
| Alelo | Frec. | Alelo | Frec. | Alelo | Frec. | Alelo | Frec. | Alelo | Frec. |
| 260 | 0.0132 | 179 | 0.2750 | 126 | 0.0385 | 91 | 0.0125 | 179 | 0.0132 |
| 262 | 0.0395 | 181 | 0.1750 | 128 | 0.0513 | 93 | 0.1500 | 181 | 0.0921 |
| 264 | 0.3553 | 183 | 0.3875 | 130 | 0.0769 | 95 | 0.0125 | 183 | 0.1974 |
| 266 | 0.1842 | 185 | 0.0125 | 132 | 0.0256 | 97 | 0.1375 | 185 | 0.0789 |
| 268 | 0.3421 | 189 | 0.1500 | 134 | 0.0769 | 99 | 0.0250 | 187 | 0.0132 |
| 270 | 0.0526 | 136 | 0.2436 | 101 | 0.0125 | 189 | 0.2895 | ||
| 272 | 0.0132 | 138 | 0.3077 | 103 | 0.4125 | 191 | 0.0132 | ||
| 140 | 0.1026 | 105 | 0.1875 | 193 | 0.1842 | ||||
| 142 | 0.0769 | 111 | 0.0375 | 197 | 0.1184 | ||||
| 113 | 0.0125 | ||||||||
| ETH003 | ETH010 | ETH185 | ETH225 | HAUT27 | |||||
| Alelo | Frec. | Alelo | Frec. | Alelo | Frec. | Alelo | Frec. | Alelo | Frec. |
| 103 | 0.0263 | 209 | 0.1667 | 220 | 0.0286 | 139 | 0.2000 | 128 | 0.0135 |
| 109 | 0.1053 | 211 | 0.0128 | 222 | 0.0571 | 141 | 0.0125 | 140 | 0.0270 |
| 115 | 0.0658 | 213 | 0.1026 | 226 | 0.0571 | 143 | 0.1125 | 142 | 0.0811 |
| 117 | 0.3421 | 215 | 0.1154 | 228 | 0.6000 | 145 | 0.0250 | 146 | 0.0135 |
| 119 | 0.1974 | 217 | 0.3333 | 230 | 0.0143 | 147 | 0.2625 | 148 | 0.5405 |
| 123 | 0.0658 | 219 | 0.1923 | 232 | 0.1000 | 149 | 0.2875 | 150 | 0.2297 |
| 125 | 0.1711 | 221 | 0.0256 | 234 | 0.1143 | 151 | 0.0125 | 152 | 0.0676 |
| 129 | 0.0263 | 223 | 0.0513 | 236 | 0.0143 | 153 | 0.0250 | 154 | 0.0270 |
| 240 | 0.0143 | 157 | 0.0625 | ||||||
| HEL009 | ILSTS006 | INRA32 | INRA63 | MM12 | |||||
| Alelo | Frec. | Alelo | Frec. | Alelo | Frec. | Alelo | Frec. | Alelo | Frec. |
| 149 | 0.0128 | 287 | 0.0556 | 168 | 0.0152 | 175 | 0.4250 | 105 | 0.0132 |
| 151 | 0.0128 | 289 | 0.0417 | 174 | 0.0152 | 177 | 0.3250 | 109 | 0.0132 |
| 153 | 0.3077 | 291 | 0.2361 | 176 | 0.1364 | 179 | 0.0125 | 117 | 0.1579 |
| 155 | 0.0641 | 293 | 0.2778 | 178 | 0.2879 | 183 | 0.2000 | 119 | 0.1184 |
| 157 | 0.0256 | 295 | 0.1111 | 180 | 0.3485 | 185 | 0.0375 | 121 | 0.2368 |
| 159 | 0.0256 | 297 | 0.2639 | 182 | 0.0303 | 123 | 0.1184 | ||
| 161 | 0.2308 | 299 | 0.0139 | 184 | 0.1212 | 125 | 0.0526 | ||
| 163 | 0.1154 | 186 | 0.0152 | 129 | 0.0132 | ||||
| 165 | 0.0513 | 188 | 0.0303 | 131 | 0.0263 | ||||
| 167 | 0.0513 | 133 | 0.1974 | ||||||
| 169 | 0.0128 | 135 | 0.0395 | ||||||
| 171 | 0.0897 | 139 | 0.0132 | ||||||
| SPS115 | TGLA053 | TGLA122 | TGLA227 | ||||||
| Alelo | Frec. | Alelo | Frec. | Alelo | Frec. | Alelo | Frec. | ||
| 242 | 0.0375 | 151 | 0.0658 | 134 | 0.0250 | 79 | 0.0769 | ||
| 244 | 0.4875 | 153 | 0.0395 | 140 | 0.0125 | 81 | 0.0256 | ||
| 246 | 0.0625 | 157 | 0.2105 | 142 | 0.0375 | 83 | 0.1282 | ||
| 248 | 0.1875 | 159 | 0.1711 | 144 | 0.0375 | 85 | 0.3077 | ||
| 250 | 0.0250 | 163 | 0.0526 | 146 | 0.0750 | 87 | 0.0128 | ||
| 252 | 0.0875 | 165 | 0.0921 | 148 | 0.0125 | 89 | 0.0641 | ||
| 254 | 0.0250 | 167 | 0.1842 | 150 | 0.3875 | 91 | 0.0641 | ||
| 256 | 0.0875 | 169 | 0.1447 | 152 | 0.2875 | 93 | 0.0128 | ||
| 175 | 0.0263 | 154 | 0.0125 | 95 | 0.0385 | ||||
| 181 | 0.0132 | 156 | 0.0125 | 99 | 0.2692 | ||||
| 160 | 0.0625 | ||||||||
| 168 | 0.0125 | ||||||||
| 174 | 0.0250 | ||||||||
La información generada a partir de la tipificación alélica se empleó para calcular las frecuencias alélicas y genotípicas de cada marcador microsatélite utilizando el complemento MSTools para Excel (Genetics Dept, TCD, Ireland). El número de alelos por locus, número efectivo de alelos, heterocigosidad observada y esperada, contenido de información polimórfica (PIC) y el coeficiente de endogamia FIS se estimaron con el programa Popgene v 1.3215. La prueba de equilibrio Hardy-Weinberg se llevó a cabo con el programa Arlequin v 3.116.
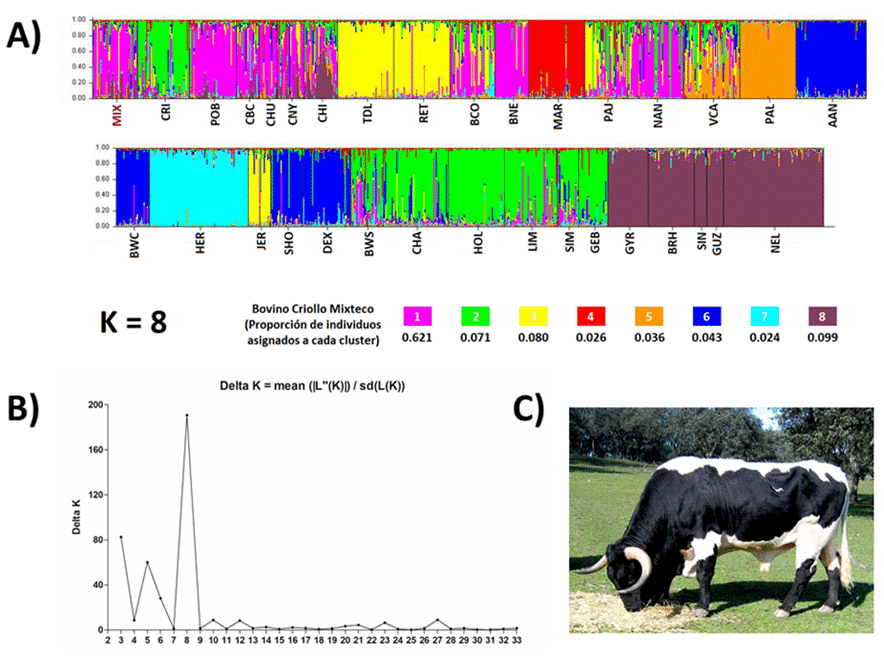
Para llevar a cabo el análisis de la estructura poblacional y las relaciones genéticas, se utilizó la información alélica de 32 poblaciones de referencia, previamente reportadas en la literatura (Figura 2)17, pertenecientes al Consorcio BIOBOVIS (https://BIOBOVIS.jimdofree.com/) de la Red de Conservación de la Biodiversidad de los Animales Domésticos Locales (CONBIAND). Las poblaciones de referencia se dividieron en cuatro grupos de acuerdo con su origen o especialización (criollos de México, ibéricos, europeos especializados y Cebú). Se estimó la distancia genética entre pares de poblaciones de Nei (DST) con el software Arlequin v3.1, con la información generada en la matriz de distancia genética se creó una representación gráfica en forma de árbol filogenético empleando el software SplitsTree v.4.14.16, con el método Neighbor Joining. Finalmente, se utilizó el programa Structure18 para inferir la estructura de la población, usando los siguientes parámetros: 100,000 iteraciones de calentamiento seguidas de 1’000,000 de iteraciones de Monte Carlo basadas en cadenas de Markov (MCMC). Un total de 32 diferentes corridas (K2 a K33) se realizaron para estimar el número más probable de cluster existentes. El valor de K óptimo se estimó mediante el método del valor modal de la distribución de Delta K, utilizando la fórmula Delta K = mean (|L’’(K)|) / sd (L(K)).

El círculo muestra la ubicación de las poblaciones criollas dentro del árbol de distancia genética (exceptuando al Criollo Lechero Tropical), revelando su posición intermedia entre las razas Bos taurus europeas y Bos indicus. Poblaciones: MIX (Bovino Criollo Mixteco), CRI (Criollo Lechero Tropical), POB (Criollo Poblano), CBC (Criollo Baja California), CHU (Criollo Chihuahua), CNY (Criollo Nayarit), CHI (Criollo Chiapas), TDL (Lidia), RET (Retinta), BCO (Berrenda en Colorado), BNE (Berrenda en Negro), MAR (Marismeña), PAJ (Pajuna), NAN (Negra Andaluza), VCA (Vaca Canaria), PAL (Palmera), AAN (Aberdeen Angus), BWC (British White Cattle), HER (Hereford), JER (Jersey), SHO (Shorthorn), DEX (Dexter), BWS (Brown Swiss), CHA (Charolais), HOL (Holstein- Fresian), LIM (Limousin), SIM (Simmental), GEB (Gelbvieh), GYR (Gyr), BRH (Brahman), SIN (Sindi), GUZ (Guzerat), NEL (Nelore).
Figura 2 Árbol de distancia genética de Nei (distancia insesgada) mediante el método Neighbor Joining
En el Cuadro 1 se muestran los resultados del cálculo de frecuencias alélicas para cada uno de los 19 loci analizados en la población de bovinos Criollos Mixtecos. En todos los loci analizados se observó variación polimórfica. En total se detectaron 168 alelos distribuidos en los 19 microsatélites, lo que representa un número medio de alelos de 8.8 ± 2.1, siendo los marcadores TGLA122 (Na= 13), MM12 (Na= 12) y HEL009 (Na= 12) los que presentaron un mayor número de alelos (Cuadro 2). En lo que respecta al número efectivo de alelos, se observó una media de 4.5 ± 1.2 alelos, mientras que el contenido de información polimórfica promedio fue de 0.7286 ± 0.0748. El valor de heterocigosidad observada fue de 0.7170 ± 0.0998 y la heterocigosidad esperada fue de 0.7700 ± 0.0682. En el Cuadro 2 se muestran los resultados de los principales parámetros de diversidad genética para cada uno de los marcadores microsatélites evaluados. Los marcadores HAUT27, ILSTS006 y TGLA227 fueron los únicos loci en desequilibrio Hardy Weinberg (P<0.05). Por otra parte, 15 de los 19 marcadores microsatélites mostraron valores de FIS positivos y los 4 restantes mostraron valores negativos; sin embargo, en su mayoría se encontraron separados del valor cero obteniéndose un valor medio de FIS de 0.058, siendo los marcadores TGLA227 y BM1818 los que mostraron mayor desviación del valor de FIS positivo y el marcador CSRM60 la mayor desviación de FIS negativo.
Cuadro 2 Parámetros de diversidad genética
| Marcador | Na | Ne | PIC | Ho | He | FIS | P value |
|---|---|---|---|---|---|---|---|
| BM1818 | 7 | 3.5479 | 0.6695 | 0.5789 | 0.7277 | 0.1938 | 0.0640 |
| BM1824 | 5 | 3.5834 | 0.6728 | 0.6500 | 0.7301 | 0.0984 | 0.7939 |
| BM2113 | 9 | 5.3462 | 0.7907 | 0.7949 | 0.8235 | 0.0222 | 0.8494 |
| CSRM60 | 10 | 4.0100 | 0.7195 | 0.8250 | 0.7601 | -0.0991 | 0.6078 |
| CSSM66 | 9 | 5.3780 | 0.7895 | 0.8684 | 0.8249 | -0.0668 | 0.4087 |
| ETH003 | 8 | 4.8456 | 0.7673 | 0.7632 | 0.8042 | 0.0384 | 0.8776 |
| ETH010 | 8 | 4.9223 | 0.7704 | 0.7436 | 0.8072 | 0.0668 | 0.1559 |
| ETH185 | 9 | 2.5574 | 0.5860 | 0.5143 | 0.6178 | 0.1555 | 0.2228 |
| ETH225 | 9 | 4.7690 | 0.7597 | 0.7750 | 0.8003 | 0.0194 | 0.9550 |
| HAUT27* | 8 | 2.7939 | 0.6022 | 0.6216 | 0.6509 | 0.0319 | 0.0042 |
| HEL009 | 12 | 5.5410 | 0.7990 | 0.8205 | 0.8302 | -0.0012 | 0.8609 |
| ILSTS006* | 7 | 4.5474 | 0.7458 | 0.8056 | 0.7911 | -0.0326 | 0.0136 |
| INRA32 | 9 | 4.1644 | 0.7244 | 0.6667 | 0.7716 | 0.1227 | 0.4309 |
| INRA63 | 5 | 3.0505 | 0.6101 | 0.6000 | 0.6807 | 0.1074 | 0.4384 |
| MM12 | 12 | 6.5045 | 0.8283 | 0.8421 | 0.8575 | 0.0049 | 0.2746 |
| SPS115 | 8 | 3.3934 | 0.6763 | 0.6750 | 0.7142 | 0.0430 | 0.7280 |
| TGLA053 | 10 | 6.8274 | 0.8366 | 0.7368 | 0.8649 | 0.1367 | 0.1248 |
| TGLA122 | 13 | 4.0455 | 0.7211 | 0.7000 | 0.7623 | 0.0702 | 0.5162 |
| TGLA227* | 10 | 4.9951 | 0.7743 | 0.6410 | 0.8102 | 0.1985 | 0.0017 |
Na= número de alelos; Ne= número efectivo de alelos; PIC= contenido de información polimórfica; Ho= heterocigosidad observada; He= heterocigosidad esperada; FIS= coeficiente de endogamia; P value = Significancia equilibrio Hardy Weinberg.
El análisis de distancia genética reveló que el Criollo Mixteco se agrupa con las poblaciones criollas de México (Figura 2), mostrando una menor distancia con los bovinos criollos de Chihuahua (D= 0.046), Baja California (D= 0.064) y Puebla (D= 0.078). Con respecto al resto de grupos poblacionales, el Criollo Mixteco presentó una menor distancia genética con la población Berrenda en Colorada (D= 0.103), perteneciente al grupo de razas locales españolas, así como con la raza Limousin (D= 0.190), perteneciente a las razas europeas especializadas. La mayor distancia genética se observó con cada una de las razas Bos indicus (Nelore 0.588 - Gyr 0.734).
Finalmente, los resultados del análisis de la estructura poblacional mediante el test de asignación con el software Structure (Figura 3A) y posterior cálculo del valor K óptimo mediante el método del valor modal de la distribución de Delta K (Figura 3B), reveló que, el K óptimo fue de 8. Las proporciones de asignación a cada cluster, para el Criollo Mixteco, se muestran en la Figura 3A. De acuerdo con los resultados de K8, se observa que, a excepción de la población Criolla Lechera, el resto de poblaciones criollas de México, en el que se incluye al Criollo Mixteco, presentan un mayor porcentaje de asignación al cluster 1, que incluye a las poblaciones locales españolas Berrenda en Negro, Berrenda en Colorado, Negra Andaluza y Pajuna (Figura 3). El resto del genoma del bovino Criollo Mixteco se distribuyó de la siguiente manera: 14.2 % con el resto de razas locales españolas, 13.8 % con razas europeas especializadas y 9.9 % con el cluster de las poblaciones Cebú.
(Fuente fotografía: Agrupación nacional de asociaciones de criadores de ganado bovino de las razas Berrenda en Negro y Berrenda en Colorado).
Figura 3 A) Resultados del análisis Bayesiano Structure para la población de Bovinos Criollos Mixtecos, empleando 32 poblaciones de referencia y un total de 8 cluster inferidos (K8). B) Estimación del K óptimo mediante el método del valor modal de la distribución de Delta K. C) Ejemplar de la raza Berrenda en Negro
Las poblaciones locales desempeñan un papel primordial en ganadería, ya que significaron la base para el desarrollo de razas especializadas y actualmente constituyen un reservorio de diversidad genética que debe ser preservado19. En este sentido, el bovino Criollo Mixteco representa un recurso ganadero local de gran valor que debe ser conservado, por lo que su evaluación genética es un punto importante para lograr este objetivo.
Los resultados de diversidad alélica, representada por el número medio de alelos observados, mostraron una similitud con los datos reportados en estudios previos en poblaciones bovinas, cuando se comparan con los valores medios por población [6.92 ± 0.99]7, [6.78 ± 1.88]8, [8.31 ± 2.10]20, sin embargo, son inferiores al compararse con el número medio de alelos detectados considerando a las poblaciones criollas como grupo [14.21 ± 3.74]8, [15.5 ± 0.9]17. La diferencia observada en el número medio de alelos al ser comparada a nivel grupal se debe a la heterogeneidad de las poblaciones criollas en América, la cual ha sido confirmada en diversos estudios empleando polimorfismos autosómicos y mitocondriales21-23. Dicha heterogeneidad es probablemente el resultado de diversos factores como diferencias en el origen de las poblaciones17, diferenciación por su localización geográfica18, así como al proceso de deriva genética y la contribución de animales de diferentes orígenes, los cuales han sido mezclados en algún momento con las poblaciones criollas, tal como se ha descrito con la introgresión de razas Cebú, africanas y británicas21,23-24. Este dato debe tomarse con cautela, ya que la heterogeneidad observada también podría reflejar el estado de amenaza de la población, por dilución a causa del mestizaje intensivo o como consecuencia del aislamiento y abandono17.
Los valores de heterocigosidad observada y esperada representan una medida de la diversidad genética en una población, sin embargo, su estimación se realiza a partir de datos diferentes, Por un lado, la heterocigosidad esperada se estima a partir de las frecuencias alélicas, mientras que la heterocigosidad observada se estima a partir de frecuencias genotípicas, por lo cual, las diferencias observadas entre ambas estimaciones pueden ser un indicador del nivel de endogamia en la población25. En el presente estudio, se observó una diferencia entre los valores de heterocigosidad, siendo la heterocigosidad observada inferior a la heterocigosidad esperada, lo que sugiere una tendencia a la endogamia, tal como pudo comprobarse posteriormente en la estimación del valor de FIS. Por otra parte, al comparar los valores de heterocigosidad esperada, así como el número efectivo de alelos, con los valores reportados para las razas criollas empleando un panel similar de microsatélites10,17,20, se observa una correspondencia en los resultados. El número efectivo de alelos representa el número de alelos esperados en una población con la misma heterocigosidad pero con frecuencias alélicas distribuidas por igual26, por lo que, si las frecuencias alélicas se encuentran muy desequilibradas y solo algunos alelos son mayoritarios, el número efectivo de alelos tenderá a ser menor27, tal como se pudo observar en la población Criolla Mixteca, en la que algunos alelos son más frecuentes en la población. La relevancia de la estimación del número de alelos y número efectivo de alelos radica en que este dato puede utilizarse como criterio de conservación, ya que la diversidad alélica puede tener implicaciones importantes en la respuesta a la selección para la adaptación a entornos cambiantes28, lo cual resulta de gran importancia al hablar de poblaciones criollas, al ser consideras reservorios de información genética para afrontar posibles alteraciones ambientales producto del cambio climático, por lo que resulta de gran importancia implementar medidas para la conservación de la diversidad alélica.
El contenido de información polimórfica superó en todos los marcadores el valor de 0.5, lo cual, de acuerdo con la escala propuesta por Botstein29, indica que los microsatélites evaluados son altamente informativos, y podrían ser empleados para posteriores estudios de monitoreo de la diversidad genética o pruebas de filiación. En lo que respecta al índice de fijación de Wright, la estimación de este parámetro proporciona una medida del grado de endogamia de los individuos con respecto a la población a la que estos pertenecen30. Aunque generalmente es positivo, el valor de FIS puede ser negativo si se evita sistemáticamente el apareamiento consanguíneo dentro de las poblaciones31. En el presente estudio los valores de FIS estimados fueron en su mayoría positivos, alejados de cero, lo que sugiere, una tendencia a la endogamia por deficiencia de heterocigotos. El resultado observado en la población, en el que la desviación de FIS con respecto al valor cero es positivo puede atribuirse a la condición de población doméstica, donde los apareamientos no son aleatorios y la proporción de machos es menor en comparación con las hembras, además de ser sistemas de producción largos con pocos animales y cuyos remplazos suelen ser obtenidos dentro de las mismas unidades de producción, lo que predispone a la consanguinidad32-33. Por otra parte, en lo que respecta a la prueba de equilibrio Hardy-Weinberg, de los 19 marcadores analizados, únicamente los marcadores HAUT27, ILSTS006 y TGLA227 se encontraron en desequilibrio; este dato incrementa la fiabilidad de los resultados obtenidos en el estudio y de igual forma, sugiere que la población no está siendo sometida a fuerzas perturbadoras que ocasionen cambios significativos en sus frecuencias genotípicas34.
Las relaciones genéticas entre poblaciones se analizaron utilizando la información genotípica de 32 razas pertenecientes al consorcio BIOBOVIS de la Red CONBIAND, las cuales fueron seleccionadas por su posible relación con el bovino Criollo Mixteco. Se emplearon 6 poblaciones criollas de México, 9 poblaciones locales españolas, las cuales podrían encontrarse dentro de las poblaciones fundadoras de los bovinos latinoamericanos, 12 razas europeas especializadas y 5 razas Cebú. Tal como puede observarse en la Figura 2, los resultados del cálculo de distancia genética muestran que el Criollo Mixteco se agrupa con las poblaciones criollas mexicanas de Chihuahua, Nayarit, Baja California y Puebla, separado de los grupos de bovinos europeos y bovinos Cebú. Esta distribución se ha reportado en trabajos previos con bovinos criollos latinoamericanos17,35 lo que demuestra que al igual que otras poblaciones criollas de América, el Criollo Mixteco presenta una identidad estrechamente relacionada con otros bovinos criollos debido a su origen compartido; dicha identidad se ha conservado a pesar de la distancia geográfica entre las poblaciones. Resulta interesante mencionar que la menor distancia genética con una población no criolla se observó con la población Berrenda en Colorado, la cual se ha descrito como una de las posibles poblaciones fundadoras de los bovinos criollos de América20. En la actualidad, en la región Mixteca, las razas Cebú han sido sustituidas por bovinos Bos taurus de mejor temperamento, ya que, de acuerdo con los productores, los cruzamientos entre bovinos de tipo Cebú y bovinos criollos generaban individuos con un temperamento difícil de manejar, mientras que los bovinos europeos al haber sido sujetos a una selección más intensiva para la docilidad y facilidad de manejo36, no presentan este problema. Probablemente, este hecho ha evitado que la introgresión de germoplasma Cebú en la población criolla se incrementara, manteniendo la distancia genética entre dichas poblaciones.
En lo que respecta a la estructura poblacional, se llevó a cabo el análisis bayesiano con el software Structure, calculando diferentes valores de K (2 - 33), con la estimación posterior del K óptimo (K= 8). Se sugirió un modelo con poblaciones K= 8, debido a que se asoció a una mayor probabilidad (Figura 3B), lo que sugiere que existen razas o grupos estrechamente relacionados7. Para un K = 8, los resultados indican que el ganado Criollo Mixteco mantiene una estructura poblacional relativamente uniforme, compartiendo un 62 % de su genoma con las poblaciones criollas de México, exceptuando al Lechero Tropical. De forma similar a lo reportado en las razas locales ecuatorianas, se observó una contribución del ganado Berrendo en Colorado y Pajuno, así como una escasa relevancia del ganado Marismeño20, lo cual sugiere que el Criollo Mixteco se integra en su origen con las poblaciones criollas latinoamericanas y refuerza la teoría de que dichas razas forman parte de las poblaciones fundadoras del ganado criollo en América Latina, ya que como se ha descrito en otros trabajos, la mayor parte del ganado español que dio origen a las poblaciones criollas en América procedía del sur de España20. Además, dicho clúster también agrupa a las razas locales españolas Berrenda en Negro (Figura 3C) y Negra Andaluza, lo que resulta interesante debido a que los patrones de coloración característicos del Criollo Mixteco son similares a los patrones de coloración de dichas poblaciones ibéricas, principalmente en la población Berrenda en Negro, cuyo porcentaje de genoma asignado a dicho cluster fue de un 87.4 %. La composición del genoma Criollo Mixteco para K = 8 mostró una heterogeneidad genética, la cual ha sido descrita previamente en otras poblaciones criollas empleando marcadores mitocondriales, autosómicos y del cromosoma Y22,24,37. Los resultados confirman que la influencia del ganado ibérico es predominante en el Criollo Mixteco, tal como sucede en las poblaciones criollas de América, las cuales conservan firmas genéticas de su ascendencia ibérica21. No obstante, también es posible inferir que pueden haber existido contribuciones recientes de germoplasma exótico, pertenecientes a bovinos de diferentes orígenes, por los porcentajes de asignación observados en menor proporción, pertenecientes a bovinos europeos y Cebú. La presencia de germoplasma Cebú puede ser el remanente de la importación de toros utilizados como sementales para hembras de razas criollas, una práctica de mestizaje indiscriminado, extensamente utilizado en diversos países de América Latina desde mediados del siglo pasado22,33, pero que, como se mencionó, actualmente no es común entre los productores de la región Mixteca. Otra de las posibles causas de la presencia de germoplasma Cebú, puede estar relacionada con el flujo de genes ancestrales entre el ganado Cebú africano y el ganado Ibérico, antes de su arribo a América, tal como ha sido sugerido en estudios previos empleando marcadores SNP y ADN mitocondrial24,38.
El bovino Criollo Mixteco presenta un nivel de variabilidad genética similar a lo reportado en estudios de poblaciones bovinas criollas de América. Además, guarda una mayor relación genética con otras poblaciones bovinas criollas de México, sin embargo, se evidencia la influencia de germoplasma exótico, en menor porcentaje, proveniente de razas Bos taurus especializadas y razas Bos indicus.











 texto en
texto en 


