Introducción
El género Bison (bisonte) es originario de Asia y la región central de Europa. Su llegada al continente americano fue a partir de las migraciones del bisonte estepario (Bison priscus) y del bisonte gigante (Bison latifrons). El actual bisonte americano (BA; Bison bison) es una población producto de los procesos de adaptación, evolución y selección natural; además, es una población alopátrica, definida por las líneas bisonte de las llanuras (Bison bison) y bisonte de las montañas (Bison athabascae). Datos históricos y arqueológicos muestran el desarrollo del BA en las praderas de Norteamérica, con estimaciones de hasta 60 millones de individuos; no obstante, la cacería para la alimentación y el comercio de pieles, fueron las principales causas de la gran disminución de esta población, colocándola en riesgo de extinción1,2,3,4.
En México, los informes históricos mencionan la presencia del BA en los estados de Chihuahua, Coahuila, Durango y Sonora; la manada de Janos-Hidalgo, fue una manada transfronteriza que se movía entre Chihuahua y Nuevo México5,6. En el rancho El Uno, ubicado en la reserva de la biosfera de Janos (Chihuahua, México), se generó una manada de conservación con 23 individuos procedentes del parque nacional Wind Cave de Estados Unidos7. El BA, como recurso genético, forma parte de la biodiversidad con sus componentes de tiempo y espacio, así como los valores de uso y opción. Los componentes de tiempo y espacio se determinan por la evolución y cambios de los niveles de riqueza, abundancia relativa y dominancia de la especie. El valor de uso se establece por los beneficios que aporta el recurso genético, el valor de opción está definido por el papel o aporte del recurso genético en la estabilidad del ecosistema8,9.
La biodiversidad de las poblaciones es la propiedad de adaptación e integración a los ecosistemas, como resultado de las fuerzas evolutivas y de la genética de las poblaciones; caso particular, los posibles efectos de selección natural, deriva y migración genética. La diversidad genética, como componente de la biodiversidad, comprende las diferencias en el material genético heredable. La variabilidad genética, es una medida de la diferenciación de los genotipos, en función del tamaño de la población y los criterios que definen la herencia del material genético. La estructura genética de una población está determinada por su historia evolutiva, expresa la cantidad de diversidad genética que alberga y cómo se reparte dentro de la población. No obstante, la pérdida de diversidad genética es el principal problema de las poblaciones en riesgo, concepto que se debe considerar en el diseño de esquemas de conservación10,11. Los objetivos del presente estudio fueron analizar la estructura y variabilidad genética, con base en marcadores genéticos de tipo SNP (Polimorfismo de Nucleótido Simple), de la manada de BA del rancho El Uno.
Material y métodos
La manada de bisontes del rancho El Uno se desenvuelve en un entorno agreste y nula proximidad con el humano, aislada y protegida de interacciones con poblaciones bovinas u otras especies que pudieran alterar su desarrollo, con instalaciones y personal especializado; cada año se realiza el conteo e identificación de la manada. Se muestrearon 174 bisontes (80 % del total), 102 hembras y 72 machos nacidos a partir de 2012; la obtención de ADN fue a partir de sangre depositada en tarjetas especializadas en el laboratorio GeneSeek de la empresa Neogen. Se analizaron los genotipos de 42,366 SNP distribuidos en los 29 cromosomas y definidos en el chip GGP Bovine 50K. En la fase de edición, se descartaron los loci con porcentaje de usables menor a 90 % (PSUM), monomórficos, con frecuencia del alelo menor (FAM) inferior a 0.01 y en desequilibrio Hardy-Weinberg (HW; P<0.05).
Posterior al proceso de edición, con el panel de SNP seleccionados como polimórficos se estimó de forma general, y por cromosoma, la heterocigosis esperada (He) y observada (Ho), el contenido de información polimórfica (CIP), el índice de fijación (FIS), el índice de Shannon (IS) y el desequilibrio de ligamiento (DL), como indicadores de variabilidad genética12,13. El DL se evaluó con base en la correlación (r2) entre frecuencias de haplotipos a través de loci14; la r2 fluctúa de cero a uno; valores cercanos a cero indican ausencia de DL y segregación independiente, conforme se acerca a 1, indica asociación no aleatoria entre loci. Adicionalmente, se estimó la relación de parentesco (Rij; %) a través de todos los individuos, así como el tamaño efectivo de población (Ne) con base en la r2 promedio ajustada aplicando el método de Waples14. Las estimaciones de r2 se realizaron con el programa FSTAT15; para He, Ho, CIP y FIS se utilizó el programa GenAlex16; LDNE17 se utilizó para el análisis de Ne y ML-Relate18 en la estimaciones de Rij.
Para determinar la estructura genética se utilizó el programa para análisis genéticos Structure19, el cual utiliza un agrupamiento bayesiano para inferir cuantas líneas o genomas (k) conforman la población a partir del análisis de genotipos utilizando marcadores genéticos. El procedimiento supone que los individuos son de ascendencia pura (k= 1) vs ascendencia de dos o más líneas (k ≠ 1), asignando proporcionalmente un genoma a cada línea. El agrupamiento bayesiano para inferir k se deriva de la distribución de probabilidad a posteriori generada por el método de Monte Carlo basado en Cadenas de Markov. Se evaluaron cinco posibles líneas, donde los individuos fueron asignados probabilísticamente. El número de líneas (k) que proporciona el mejor ajuste se deriva a partir de la verosimilitud logarítmica de cada paso en el muestreo y el valor máximo u óptimo se obtuvo con el planteamiento de Evanno et al20 y el programa Structure Harvester21.
Resultados y discusión
Como resultado del proceso de edición, se identificó y seleccionó un panel con 2,135 SNP polimórficos (5.04 % de rendimiento, con respecto al total de SNP evaluados), con un promedio de 74 SNP por cromosoma. En el proceso de edición se descartaron 40,231 SNP, distribuidos de la siguiente forma: 84.5 % fueron monomórficos, 8.5 % por PSUM, 6.3 % por FAM y 0.70 % por HW. Para el panel de SNP polimórficos, las estimaciones de IS, Ho, He, FIS y CIP fueron de 0.30, 0.187, 0.182, -0.029 y 0.152, respectivamente. Los valores de He, Ho y CIP determinan la viabilidad de un marcador genético en estudios de variabilidad genética; el número de SNP por cromosoma, así como los estimadores de variabilidad genética, se describen en el Cuadro 1. Estimaciones del IS cercanas a cero se pueden asociar con homogeneidad en la población, lo cual reduce la incertidumbre para predecir la probabilidad de asignación de un individuo a la población que pertenece. El FIS, a través de los cromosomas, presentó valores de -0.002 a -0.062; éste mide los niveles de heterocigosis y homocigosis, cuyos valores van de -1 a 1; presenta valores positivos cuando hay un déficit de heterocigotos y valores negativos cuando hay un exceso de heterocigotos, los valores cercanos a cero indican una estabilidad en la relación de homocigosis y heterocigosis. La Rij oscila de 0 a 100 %, en 174 individuos se generaron 15,051 estimaciones con un valor promedio de 7.61 %. Las estimaciones de la Rij se describen en los siguientes cinco estratos: el 45.1 % de las estimaciones fue igual a cero; el 14.5 % presentaron valores de 0.01 a 4.9; el 27.3 % de 5.0 a 19.9; el 11.8 % de 20.0 a 49.9; y el 1.3 % fue igual o mayor a 50.0. La consanguinidad (F) de un individuo es la mitad de la Rij de los padres, con las estimaciones de la Rij es posible la selección de subpoblaciones para esquemas de reproducción y conservación, con el planteamiento de mantener los niveles de F.
Cuadro 1 Número de SNP polimórficos y estimadores de variabilidad genética por cromosoma
| Cr | ni | nf | CIP | Ho | He | FIS | IS | r2 |
|---|---|---|---|---|---|---|---|---|
| 1 | 2587 | 88 | 0.155 | 0.194 | 0.186 | -0.034 | 0.304 | 0.022 |
| 2 | 2199 | 52 | 0.153 | 0.193 | 0.186 | -0.029 | 0.302 | 0.019 |
| 3 | 2072 | 56 | 0.211 | 0.263 | 0.260 | -0.011 | 0.403 | 0.023 |
| 4 | 1933 | 170 | 0.117 | 0.140 | 0.136 | -0.035 | 0.241 | 0.222 |
| 5 | 2173 | 160 | 0.105 | 0.126 | 0.122 | -0.025 | 0.215 | 0.205 |
| 6 | 2056 | 70 | 0.193 | 0.240 | 0.234 | -0.025 | 0.372 | 0.020 |
| 7 | 1858 | 232 | 0.195 | 0.240 | 0.226 | -0.062 | 0.376 | 0.324 |
| 8 | 1832 | 47 | 0.186 | 0.228 | 0.225 | -0.024 | 0.359 | 0.021 |
| 9 | 1818 | 57 | 0.230 | 0.298 | 0.289 | -0.032 | 0.437 | 0.026 |
| 10 | 1736 | 205 | 0.089 | 0.107 | 0.103 | -0.028 | 0.189 | 0.288 |
| 11 | 1766 | 52 | 0.143 | 0.174 | 0.170 | -0.019 | 0.282 | 0.019 |
| 12 | 1418 | 49 | 0.218 | 0.281 | 0.270 | -0.034 | 0.415 | 0.031 |
| 13 | 1544 | 65 | 0.151 | 0.192 | 0.187 | -0.020 | 0.295 | 0.127 |
| 14 | 1483 | 61 | 0.156 | 0.190 | 0.189 | -0.013 | 0.307 | 0.105 |
| 15 | 1395 | 59 | 0.176 | 0.221 | 0.212 | -0.034 | 0.342 | 0.022 |
| 16 | 1302 | 40 | 0.194 | 0.247 | 0.235 | -0.044 | 0.372 | 0.024 |
| 17 | 1233 | 41 | 0.195 | 0.246 | 0.239 | -0.021 | 0.375 | 0.026 |
| 18 | 1219 | 33 | 0.210 | 0.267 | 0.255 | -0.039 | 0.399 | 0.028 |
| 19 | 1218 | 65 | 0.124 | 0.147 | 0.146 | -0.016 | 0.251 | 0.210 |
| 20 | 1335 | 50 | 0.192 | 0.238 | 0.232 | -0.031 | 0.370 | 0.026 |
| 21 | 1183 | 33 | 0.185 | 0.235 | 0.228 | -0.032 | 0.358 | 0.024 |
| 22 | 1017 | 23 | 0.197 | 0.242 | 0.239 | -0.017 | 0.379 | 0.029 |
| 23 | 943 | 35 | 0.223 | 0.274 | 0.275 | -0.010 | 0.425 | 0.074 |
| 24 | 1081 | 56 | 0.162 | 0.197 | 0.198 | -0.002 | 0.317 | 0.113 |
| 25 | 749 | 115 | 0.083 | 0.099 | 0.094 | -0.031 | 0.179 | 0.504 |
| 26 | 879 | 145 | 0.096 | 0.108 | 0.107 | -0.015 | 0.204 | 0.256 |
| 27 | 724 | 26 | 0.248 | 0.313 | 0.308 | -0.024 | 0.466 | 0.036 |
| 28 | 785 | 21 | 0.209 | 0.265 | 0.263 | -0.015 | 0.401 | 0.022 |
| 29 | 828 | 29 | 0.200 | 0.254 | 0.247 | -0.025 | 0.383 | 0.026 |
Cr= cromosoma, ni= número de loci evaluados, nf= número de loci polimórficos, CIP= contenido de información polimórfica, Ho= heterocigosis observada, He= heterocigosis esperada. FIS= índice de fijación, IS= índice de Shannon, r2= correlación promedio entre frecuencias de haplotipos a través de loci.
El Ne fue de 12.5 y la r2 promedio general fue de 0.099, en el Cuadro 1 se presenta la r2 promedio por cromosoma. El análisis de estructura genética mostró que la población de estudio está conformada por tres líneas, con proporciones de 0.730, 0.157 y 0.113; en la Figura 1 se presenta la estructura y composición de la población analizada a través de las cinco líneas evaluadas. Con base en el Ne, el posible cambio o incremento en los niveles de consanguinidad, por generación, pueden ser del orden del 4.0% (ΔF = 1/(2*Ne)). En poblaciones pequeñas bajo esquemas de conservación el incremento de los niveles de F es un indicador de pérdida de variabilidad genética; sus efectos, a partir de la depresión consanguínea, trasciende en rasgos de viabilidad, supervivencia, reproducción, resistencia a enfermedades, estrés ambiental, entre otros22,23. Para el desarrollo de poblaciones en esquemas de conservación, es recomendable un Ne igual o mayor a 5010, con el objetivo de mantener el incremento de la consanguinidad igual o menor a 1 % por generación. Tokarska et al24 en una población de bisonte europeo obtuvieron estimaciones de Ne de 7.0 a 28.4 a través de cinco generaciones, observando que el aumento de la población no trascendió en el incremento del Ne; además, señalan que el Ne puede estar influenciado por el tamaño de la población fundadora y que bajos niveles de Ne se pueden asociar a deriva genética, con mayor pérdida de diversidad. El potencial evolutivo de una población depende de la variabilidad genética y del Ne; si el Ne es pequeño, la deriva genética tiene trascendencia, con posible impacto en el potencial evolutivo de la población25.
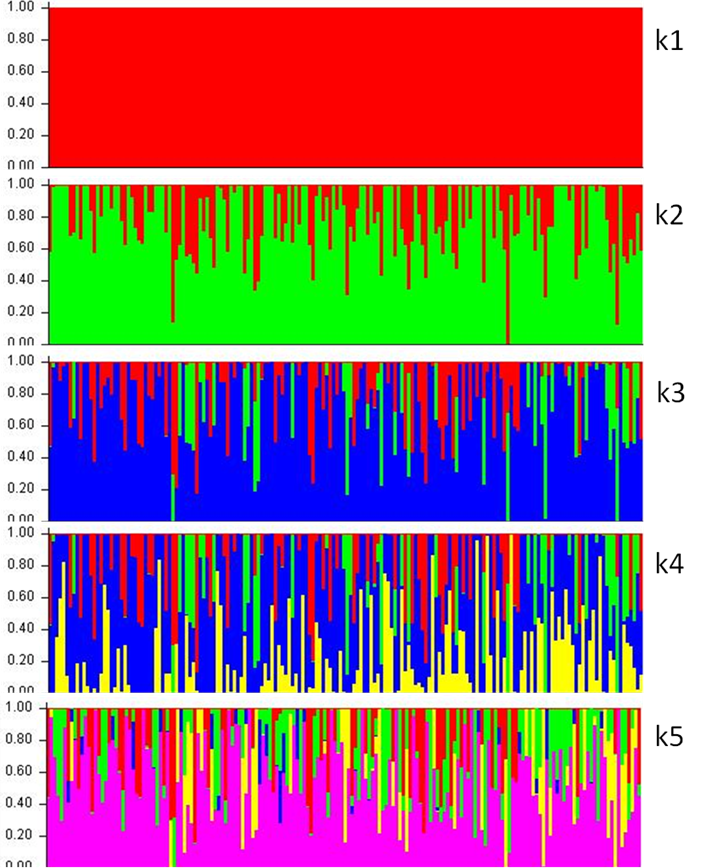
Cada línea vertical representa a un individuo y el segmento de color representa la proporción de cada grupo.
Figura 1: Análisis de la estructura de la población de bisonte americano en México, a través de cinco líneas (k= 1, 2, 3, 4, 5)
En estudios afines, utilizando el chip Bovine SNP50K, Pertoldi et al26 y Pertoldi et al27 en tres poblaciones de bisontes (una de Europa y dos de América) obtuvieron porcentajes de SNP polimórficos de 1.8, 2.6 y 2.9, con estimaciones de He de 0.135, 0.197 y 0.199. En otro trabajo28 identificaron 2.8 % de SNP polimórficos; Eugeniu et al29 reportaron un rendimiento del 9.35 % en SNP polimórficos, con estimaciones de Ho y He de 0.306 y 0.250, respectivamente. En bisonte europeo30 identificaron 1,536 SNP polimórficos, distribuidos por cromosoma en el intervalo de 8 a 136 SNP y un valor promedio de 51.2.
La actual población de BA de los Estados Unidos de América se derivó de un proceso de cuello de botella con trascendencia en su variabilidad y estructura genética31,32. El presente trabajo muestra tres líneas genéticas que definen la población de estudio; dado su origen, se puede atribuir que la línea de mayor proporción corresponde al bisonte de las llanuras; en las líneas complementarias, se puede señalar una posible contribución del bisonte de las montañas y una tercera línea generada por la separación y desarrollo de la población de estudio. La posible diferenciación de la población de estudio puede estar en función de la interacción genotipo-ambiente, así como su adaptación y contribución al ecosistema. El aislamiento genético entre subpoblaciones afecta algunos procesos demográficos y evolutivos; el reducido flujo de genes puede conducir a la acumulación de diferencias genéticas entre las subpoblaciones, debido a las normas de la genética de poblaciones33. Conjuntamente, las diferencias a través de poblaciones se pueden derivar de la diversidad genética contenida en los ancestros fundadores y su aportación relativa, así como del Ne y su evolución a través del tiempo32. La subestructura genética no siempre coincide con las diferencias morfológicas o geográficas obvias entre las subpoblaciones. Con información de marcadores genéticos se requieren estudios complementarios para contrastar la población de estudio con otras poblaciones, identificar las posibles fuentes u origen del material genético, o en su caso, definir la posible diferenciación. Halbert y Derr32, en once poblaciones de bisontes, identificaron la diferenciación genética a través de ocho clústeres; En otro trabajo33 detectaron dos subpoblaciones genéticamente distintas en el hato del parque nacional Yellowstone. Cronin et al34, analizando la estructura de 12 hatos, encontraron tres líneas o subestructuras genéticas, con una constitución promedio de 0.412, 0.303 y 0.285.
Conclusiones e implicaciones
Se identificaron tres líneas genéticas; donde, dos líneas se pueden asociar a las poblaciones de origen y una tercera por el proceso de separación y los efectos de selección natural o deriva genética. La variabilidad genética, así como los niveles de parentesco, se deben de considerar en el diseño de esquemas de reproducción y conservación.











 texto en
texto en 


