La degradación de los pastizales ocasiona una pérdida importante de productos y servicios que brindan estos ecosistemas al ser humano, como son el forraje para el ganado, captación de agua, retención de suelo y captura de carbono1. La fragmentación, expansión de especies invasoras, conversión de pastizales en áreas de agricultura, el crecimiento poblacional, entre otros, son algunas de los principales problemas que continúan afectando a los pastizales2. Para atender estos problemas en los últimos años se han realizado trabajos con la finalidad de seleccionar genotipos de diferentes especies de pastos para utilizarlos en la rehabilitación de pastizales3,4,5. Sin embargo, la selección de materiales se ha enfocado principalmente en características agronómicas y se le ha dado poca importancia a la estructura genética y al potencial de adaptación a condiciones climáticas.
La estructura genética de una especie la determina principalmente la adaptación a las condiciones ambientales de los sitios donde se distribuye. Por esta razón, conocer la estructura genética de las poblaciones puede ayudar a delimitar la distribución de los tipos genéticos y su uso potencial en programas de restauración ecológica6,7. Una de las especies más utilizadas en restauración de pastizales en el norte de México es el pasto banderita [Bouteloua curtipendula (Michx.) Torr.], debido a que se adapta a una amplia gama de climas y tiene un excelente valor forrajero. Debido a esto, en los últimos años se han realizado diversos trabajos para seleccionar genotipos sobresalientes de pasto banderita, que puedan ser utilizados para restaurar los pastizales del estado de Chihuahua8,9,10. Sin embargo, es necesario que se analice la estructura genética de las poblaciones de esta especie en el Estado y la aptitud ambiental que poseen.
Una manera de estimar áreas con aptitud ambiental potencial para distribución de una especie, es mediante el uso de modelos matemáticos, tales como el método de máxima entropía que aplica el software (MaxEnt)11. El uso de este tipo de métodos mediante la implementación de MaxEnt tiene una serie de ventajas entre las cuales destacan las siguientes: solo se requiere datos de presencia para su uso, muestra resultados confiables con un número limitados de datos, se emplean datos ambientales de tipo continuo y/o categórico, los resultados se muestran en formato de grafico (mapa) lo cual facilita su interpretación. Así mismo, dentro de las virtudes que posee el software es que permite apreciar la relevancia que tiene cada variable ambiental en la distribución de la especie mediante la prueba de Jacknife que genera11,12. Por lo antes descrito, los modelos generados a partir de la implementación de MaxEnt han sido ampliamente utilizado para estimar el nicho climático de especies de flora y fauna13,14. Así, este modelo se ha utilizado para identificar áreas con aptitud ambiental con potencial para la distribución del pasto banderita en México y Estados Unidos15,16, lo cual se considera un requisito si se desea rehabilitar un ecosistema17. No obstante, esta herramienta solo se ha utilizado a nivel especie y no para estimar áreas con aptitud ambiental de tipos genéticos que existen dentro de las especies, lo cual podría ayudar para hacer más eficientes los programas de restauración de ecosistemas. Por lo anterior, el objetivo fue evaluar la estructura genética del pasto banderita en el estado de Chihuahua, México, aplicando los modelos de MaxEnt a nivel de grupo genético e identificando las áreas con aptitud ambiental para los grupos genéticos resultantes para la especie.
La toma de muestras se realizó en 51 poblaciones, distribuidas en 29 municipios del estado de Chihuahua, situado en el norte de México. Se encuentra dentro de las coordenadas geográficas de 32 y 25 latitud norte y -103 y -109 longitud oeste. Los sitios de muestreo se ubicaron en las eco-regiones árida y semiárida del Estado, con el objetivo de captar una mayor diversidad genética posible (Figura 1).

Figura 1 Localización geográfica del material genético colectado, correspondiente a 51 poblaciones de pasto banderita (Bouteloua curtipendula), distribuidas en el estado de Chihuahua, México
La estructura genética del pasto banderita fue analizada mediante marcadores moleculares AFLP (Polimorfismos en la longitud de fragmentos amplificados). En cada sitio se colectaron hojas de plantas, las cuales fueron utilizadas para la extracción de ADN, proceso realizado con base en el método propuesto por Doyle & Doyle18. El análisis AFLP se llevó a cabo mediante el método propuesto por Vos et al19. El análisis AFLP comenzó con la digestión de 2 µl de ADN diluido por medio de las enzimas de restricción EcoRI y MseI. Posteriormente, los fragmentos de ADN digeridos fueron ligados con adaptadores para EcoRI y MseI. Después, se adhirió un nucleótido extra a los primers (EcoRI + A y MseI + A) para realizar la pre-amplificación. La amplificación selectiva se realizó mediante cuatro combinaciones de primers marcados con fluorescencia: MseI + CTG - EcoRI + AAG, MseI + CTG - EcoRI + ACT, MseI + CAG - EcoRI + AGG, MseI + CAG - EcoRI + AAC. La reacción en cadena de la polimerasa se realizó en un termo ciclador cycler (Verity Applied Biosystems 2720), con el siguiente programa: un ciclo de 94 °C por 30 s, 65 °C por 30 s, 72 °C por 1 min; 12 ciclos de 94 °C por 30 s, 65 °C por 30 s, 72 °C por 1 min; 23 ciclos de 94 °C por 30 s, 56 °C por 30 s, 72 °C por 1 min. Los productos de la amplificación selectiva (2 µl) se mezclaron con 8 µl de formamida y 1 µl de marcado Eco 700 GeneScan (Applied Biosystems). Finalmente, la separación de los fragmentos amplificados se hizo en el analizador de DNA LI-COR, cargando 0.8 µl de muestra por pozo. Se utilizaron oligos o primers, marcados con fluorescencia, a diferentes longitudes de onda (700 nm y 800 nm).
Con el patrón de bandas obtenido del análisis AFLP, se formó una matriz binaria de presencia (1) o ausencia (0) de bandas.
Para el análisis de la información, los datos binarios fueron sometidos a un análisis de estructura genética, con base en el algoritmo de agrupamiento Bayesiano, mediante el software STRUCTURE versión 2.3.420,21. El programa STRUCTURE se corrió 30 veces para cada K número de conglomerados genéticos y se analizó desde K=1 hasta K=10. En cada corrida se efectuaron 10,000 repeticiones de cadena de Markov-Monte Carlo (CMMC) y 100,000 periodos de rodaje (burn-in periods). Este análisis se realizó por medio de un modelo de mezcla y frecuencia de alelos correlacionados. Se consideró como número óptimo de K grupos al que obtuvo el valor mayor de la probabilidad posterior promedio (LK) y de ΔK, de acuerdo con los criterios propuestos por Evanno et al22. Los valores de la probabilidad posterior promedio y ΔK se obtuvieron a través del sitio web Structure Harvester23. La asociación entre las zonas climáticas del Estado y la distribución de las poblaciones integradas en los grupos genéticos se analizó mediante una prueba de independencia Ji-cuadrada (α=0.05).
Los grupos conformados en el análisis de estructura genética fueron comparados mediante un análisis de varianza molecular (AMOVA)24. Este análisis se llevó a cabo empleando el software GenAIEx versión 625. Con los estadísticos F (ΦST) obtenidos del AMOVA, se calculó el índice de flujo genético entre grupos, con la fórmula Nm= [0.25 (1- ΦST)/(ΦST)]26. Adicionalmente, los datos genéticos fueron analizados mediante el algoritmo de Monmonier, para detectar posibles barreras eco-geográficas que afecten el flujo genético entre poblaciones. Este análisis se realizó con ayuda del software Barrier versión 2.227, donde los valores de Bootstrap de cada barrera fueron calculados con 100 matrices de distancias del coeficiente Dice.
El sofware GenAIEx versión 625 fue utilizado para obtener los estadísticos de diversidad, porcentaje de loci polimórficos, promedio de alelos por locus, número de alelos efectivos, índice de información de Shannon (I) y diversidad de Nei (He), para los grupos genéticos formados por el análisis STRUCTURE. Estos índices fueron estimados con base en el supuesto de que cada locus representa un par de alelos, cuando en una banda se encuentra presencia o ausencia de fragmento AFLP. Los estadísticos de diversidad para cada población se compararon mediante la prueba Wilcoxon con corrección Bonferroni (α=0.05).
La aptitud ambiental de los grupos genéticos formados por el análisis STRUCTURE, fue determinada mediante el algoritmo de MaxEnt del software de MaxEnt 3.3.311. El modelo se corrió con las coordenadas de las poblaciones analizadas genéticamente, para cada grupo genético por separado. Del conjunto de datos, el 75 % se utilizó para probar los modelos, mientras que el 25 % de los datos restantes fueron utilizados para validar los modelos, utilizando la prueba de bootstrap, con 50 réplicas. Los modelos de aptitud ambiental generados fueron evaluados utilizando el análisis de la curva característica de operación del receptor (ROC, por sus siglas en inglés) y el área bajo la curva (AUC, por sus siglas en inglés). La puntuación del AUC es de utilidad para medir el rendimiento del modelo. Cuanto más alto sea el valor de AUC (más cerca de 1), mejor será el modelo para estimar probabilidad de presencia de la especie.
Un total de 22 variables climáticas fueron utilizadas como predictoras para modelar la distribución potencial del pasto banderita. Se emplearon 19 variables bioclimáticas: temperatura promedio anual (Bio1), rango de temperatura promedio diurna (Bio2), oscilación térmica anual (Bio3), estacionalidad de la temperatura (Bio4), temperatura máxima del mes más caliente (Bio5), temperatura mínima del mes más frío (Bio6), rango de temperatura anual (Bio7), temperatura promedio del trimestre más húmedo (Bio8), temperatura promedio del trimestre más frío (Bio9), temperatura promedio del trimestre más caliente (Bio10), temperatura promedio del trimestre más frío (Bio11), precipitación anual (Bio12), precipitación del mes más húmedo (Bio13), precipitación del mes más seco (Bio14), estacionalidad de la precipitación (Bio15), precipitación del trimestre más húmedo (Bio16), precipitación del trimestre más seco (Bio17), precipitación del trimestre más caliente (Bio18) y precipitación del trimestre más frío (Bio19)28. Adicionalmente se incluyó la radiación solar promedio anual (Rad), evapotranspiración promedio anual (Vapr) velocidad del viento promedio anual (Wind). Las variables que se utilizaron se obtuvieron de la base de datos WorldClim (https://www.worldclim.org) y se delimitaron al espacio geográfico del estado de Chihuahua con el software ArcMap 10.3. Los datos climáticos son estimaciones de interpolación del periodo 1950-2000, con una resolución espacial de 30 arco-segundos. El resultado de la aplicación de MaxEnt es un mapa logístico, que muestra la distribución potencial del pasto banderita con valores que van de 0 (inadecuado) a 1 (óptimo).
El análisis de estructura genética (STRUCTURE) dividió a las poblaciones de pasto banderita en dos grupos genéticos, ya que K=2 obtuvo los valores más altos de ΔK y LK (Figura 2).
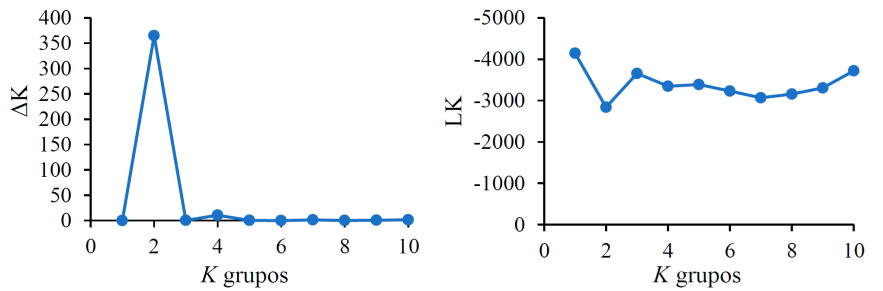
Valores desde K=1 hasta K=10, donde se muestra a K=2 como número óptimo de grupos
Figura 2 Valores de delta K (ΔK) y probabilidad posterior promedio (LK) para la estructura genética de 53 poblaciones de pasto banderita (Bouteloua curtipendula) de Chihuahua, México
La divergencia genética de estos grupos parece haber sido generada por una adaptación a las condiciones climáticas del Estado, debido a que se encontró una alta asociación entre las eco-regiones del Estado y la formación de los grupos (Ji cuadrada= 32.9; P<0.0001). El 89 % de las poblaciones integradas al Grupo 1 se ubican en la región semiárida y el 11 % en la región árida. En contraste, el 90 % de las poblaciones del Grupo 2 se encuentran en la región árida y el 10 % en la región semiárida (Figura 3B). Estos resultados concuerdan con los obtenidos en el análisis BARRIER, el cual identificó barreras genéticas que coinciden con la frontera entre las regiones árida y semiárida (Figura 3C).
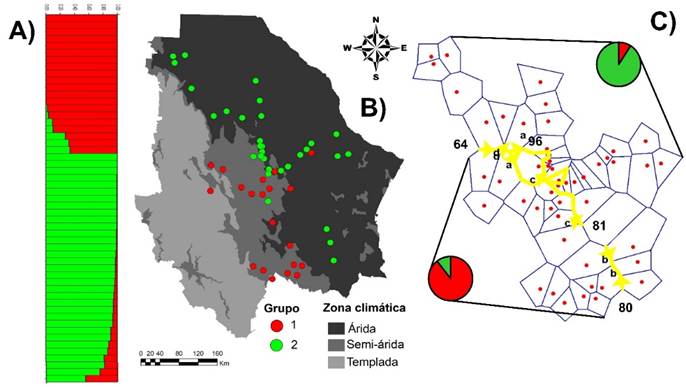
A) Análisis STRUCTURE con K= 2 realizado con base en 186 fragmentos AFLP, el color representa la proporción de la probabilidad de pertenecer a cada grupo genético. B) Estructura en contexto geográfico de cada zona climática del Estado. C) Barreras genéticas identificadas mediante análisis BARRIER, líneas amarillas representan las barreras y los números el valor de Bootstrap (1000 remuestreos). Gráficas circulares representa el porcentaje de poblaciones dentro de cada grupo, dentro de cada región.
Figura 3 Estructura genética de 53 poblaciones de pasto banderita (Bouteloua curtipendula), en el estado de Chihuahua, México
Los grupos conformados por el análisis STRUTURE fueron significativamente (P<0.0001) diferentes, de acuerdo con el AMOVA. No obstante, las diferencias entre grupos solo explicaron el 7 % de la variación global, lo que indica que existe amplia variabilidad genética dentro de grupos. En pastos, es común que las diferencias entre grupos expliquen solamente una pequeña proporción de la variación total, ya que generalmente existe un alto flujo genético entre sus poblaciones29,30. Por otra parte, la baja diferenciación genética entre poblaciones (7 %), revelada por AMOVA, parece contrastar con la marcada asociación entre las zonas climáticas del Estado y la formación de los grupos. No obstante, esto puede deberse a que un solo locus puede estar altamente relacionado con la adaptación a condiciones climáticas. Tso y Allan7 encontraron una alta relación entre la frecuencia de un solo locus con la precipitación del trimestre más seco del año (R2=0.84) y con la estacionalidad de la precipitación (R2= 0.77), al estudiar poblaciones de Bouteloua gracilis.
El intercambio genético encontrado entre grupos genéticos (F st = 3.41) puede considerarse alto, en comparación con los resultados obtenidos en otras investigaciones. Durka et al31, encontraron un flujo genético de 0.76 entre 30 poblaciones del pasto Stipa pulcherrima, distribuidas por diferentes países de Europa y Asia. Así mismo, Mitchell et al29 encontraron un F st de 0.02 entre 85 poblaciones australianas de Microlaena stipoides. Ambos estudios fueron realizados mediante marcadores AFLP.
Los grupos formados por el análisis STRUCTURE mostraron diferencias significativas (P<0.05) en los parámetros de diversidad evaluados. El Grupo 2, que se distribuye principalmente en la región árida, obtuvo los valores más altos (P<0.05) en todos los parámetros de diversidad (Cuadro 1). Este resultado es consistente con Zhang et al32, quienes encontraron que los niveles de diversidad genética de Festuca ovina son mayores en zonas áridas. Estos investigadores obtuvieron una correlación positiva entre el índice de diversidad genética de Nei y la temperatura media anual (r= 0.56) y una correlación negativa (r= -0.60) con la precipitación media anual. Resultados similares fueron también encontrados en Dactylis glomerata33. Diversos estudios señalan que las poblaciones de plantas tienden a poseer mayor diversidad en ambientes adversos34,35. Las poblaciones que se desarrollan en ambientes altamente áridos tienden a poseer mayor diversidad genética como mecanismo de adaptación a la sequía36.
Cuadro 1 Parámetros de diversidad de dos grupos genéticos de pasto banderita (Bouteloua curtipendula) del estado de Chihuahua, México
| Grupo | Porcentaje de loci polimórficos |
Promedio de alelos por locus | Número de alelos efectivos |
I | H e |
|---|---|---|---|---|---|
| Grupo 1 | 59.1 | 1.56b | 1.34b | 0.302b | 0.211b |
| Grupo 2 | 90.4 | 1.90a | 1.43a | 0.427a | 0.280a |
ab Valores en una columna con la misma letra no son estadísticamente diferentes (P<0.05; Wilcoxon con corrección Bonferroni). I= índice de información de Shannon; H e = diversidad genética de Nei.
Por otra parte, ambos grupos presentaron alto nivel de diversidad genética dentro de ellos, de acuerdo con el índice de información de Shannon (Grupo 1 = 0.302 y Grupo 2= 0.427). En comparación, Todd et al37 evaluaron 56 accesiones de Panicum virgatum y obtuvieron un valor de I de 0.317. Así mismo, en 281 cultivares de Pennisetum purpureum encontraron valores de I de entre 0.12 y 0.3438. Dado el amplio número de genotipos analizados en las investigaciones antes mencionadas, se puede considerar que la diversidad genética encontrada en este estudio fue alta para ambos grupos.
El modelo de MaxEnt se alimentó con las coordenadas de 20 poblaciones para el Grupo 1 y 32 para el Grupo 2 de acuerdo a la división resultante del análisis STRUCTURE. Este modelo mostró que el nicho climático del Grupo 1 obtuvo un valor promedio de AUC de 0.91, con una desviación estándar de 0.031. El valor promedio de AUC del nicho climático del Grupo 2 fue de 0.93, con una desviación estándar de 0.015. Por esta razón, se puede considerar que la estimación de la aptitud ambiental de ambos grupos genéticos tiene un alto grado de confiabilidad11,39. La Figura 4 muestra la aptitud ambiental de ambos grupos genéticos, donde se observa que el nicho climático de ambos grupos se diferencia claramente. El nicho climático del Grupo 1 se distribuye principalmente en el centro y sur del estado de Chihuahua (Figura 4A), mientras que el del Grupo 2 se localiza en el centro, oeste y noreste del Estado (Figura 4B). Estos resultados sugieren que los grupos genéticos divergieron por una adaptación evolutiva y que cada genotipo está adaptado a las condiciones climáticas de la región. En este sentido, los programas de restauración con pasto banderita deberían ser realizados con materiales locales de cada eco-región del estado de Chihuahua. Sin embargo, los programas de restauración de pastizales en México se han realizado con materiales extranjeros, debido principalmente a la baja disponibilidad de semilla. En el caso del pasto banderita, se han utilizado variedades provenientes de Estados Unidos. Por ejemplo, se ha utilizado la variedad “El Reno” del pasto banderita con resultados desfavorables debido a su baja adaptabilidad al clima de México. Respecto a esto, Morales et al40 analizaron 277 genotipos de pasto banderita, colectados en diferentes estados de México, y encontraron que más de la mitad de ellos presentaron mejor potencial productivo que la variedad El Reno. Otros investigadores4 encontraron que la variedad El Reno presenta baja capacidad de establecimiento y producción de forraje, en comparación con materiales nativos.

Grupo 1 (A) y Grupo 2 (B), donde el color rojo representa las área con mayor aptitud ambiental y el color azul menor aptitud.
Figura 4 Mapas del modelo MaxEnt para dos grupos genéticos de pastos banderita (Bouteloua curtipendula) identificados mediante marcadores AFLP y análisis STRUCTURE
Diversas investigaciones señalan que los programas de restauración se deben realizar con materiales locales, lo que garantiza una mayor probabilidad de éxito y además se preserva la estructura genética local41,42. Esto concuerda con los resultados del presente estudio, los cuales proponen que los programas de revegetación con pasto banderita deben ser realizados con materiales de cada eco-región del estado de Chihuahua.
La alta diversidad encontrada dentro de cada grupo genético sugiere la posibilidad de seleccionar genotipos sobresalientes para cada eco-región. En los últimos años se ha realizado un importante número de trabajos para seleccionar genotipos sobresalientes de pasto banderita9,43. No obstante, estas investigaciones han dado poca importancia a las condiciones ambientales a las que están adaptados los diferentes genotipos y se han enfocado principalmente en características productivas. Por el contrario, los resultados de la presente investigación proveen información básica sobre la aptitud ambiental potencial de las poblaciones de pasto banderita en el estado de Chihuahua, la cual podría apoyar a los programas de selección de genotipos productivos.
Las variables de mayor contribución sobre el modelo de aptitud ambiental del Grupo 1 fueron estacionalidad de la precipitación (79.5 %) y precipitación del trimestre más frío (7.4 %). Por otra parte, el modelo de aptitud ambiental del Grupo 2 estuvo influenciado principalmente por la precipitación del trimestre más frío (36.2 %), oscilación térmica anual (13.2 %), radiación solar promedio anual (8.1 %) y temperatura media del trimestre más seco (7.4 %; Cuadro 2). Estos resultados concuerdan con investigaciones previas, realizadas para modelar el nicho climático del pasto banderita en México y Estados Unidos. Respecto a esto, Martínez et al16 encontraron que la oscilación térmica y la precipitación fueron los descriptores ecológicos que más contribuyeron en la distribución potencial del pasto banderita en México. Así mismo, Martinson et al.15 concluyeron que la temperatura media anual fue el factor más importante en la distribución potencial de este pasto en Estados Unidos.
Cuadro 2 Contribución relativa (%) de variables ambientales sobre el modelo de aptitud ambiental (MaxEnt) de dos grupos genéticos de pasto banderitia (Bouteloua curtipendula)
| ID | Variable | Contribución (%) para el Grupo 1 |
Contribución (%) para el Grupo 2 |
|---|---|---|---|
| Wind | Velocidad del viento promedio anual | 0.6 | 6.7 |
| Rad | Radiación solar promedio anual | 2.7 | 8.1 |
| bio-2 | Rango de temperatura promedio diurna [media mensual (temperatura máxima - temperatura mínima)] | 2.2 | 2 |
| bio-3 | Oscilación térmica anual (Rango de temperatura promedio diurna / Rango de temperatura anual) x (100) | 1.1 | 13.2 |
| bio-9 | Temperatura media del trimestre más seco | 1.8 | 7.4 |
| bio-13 | Precipitación en el mes más húmedo | 0.1 | 4.3 |
| bio-14 | Precipitación en el mes más seco | 0.3 | 4.3 |
| bio-15 | Estacionalidad de la precipitación (coeficiente de variación) | 79.5 | 1.4 |
| bio-17 | Precipitación del trimestre más seco | 0.2 | 5.3 |
| bio-18 | Precipitación del trimestre más caliente | 0.8 | 3.4 |
| bio-19 | Precipitación del trimestre más frío | 7.4 | 36.2 |
Para un analisis más detallado, las curvas de respuestas de las variables de mayor contribución se ilustran en la Figura 5. Acorde a estas curvas, el Grupo 1 presenta mayor probabilidad de desarrollarse en áreas con más de 120 del coeficiente de la estacionalidad de la precipitación y se desarolla mejor en zonas de entre 0 y 20 mm de precipitación en el trimestre más frío (diciembre, enero y febrero). Por otra parte, el Grupo 2 también se desarolla mejor en zonas con entre 0 y 20 mm de precipitación de trimestre más frío. Además, prefiere áreas con oscilación térmica anual de entre 49 y 53, radiación solar promedio anual de entre 18,500 y 19,000 w m-2 y temperatura media del trimestre más seco de 6 a 10 °C. Los resultados de las curvas de respuesta de los grupos genéticos sugieren que el Grupo 1 no resiste periodos amplios de sequía y necesita una precipitación dispersa durante los meses cálidos del año (junio-octubre). Por otra parte, el Grupo 2 puede resistir menor oscilación térmica, mayor temperatura y mayor radiación solar, pero necesita que la precipitación se concentre en los meses más cálidos. Lo anterior resalta las diferencias evolutivas de cada grupo genético y la importancia de utilizarlos en la eco-región de donde son originarios.

Figura 5 Curva de respuesta de dos grupos genéticos de pasto banderita (Bouteloua curtipendula) de acuerdo a las variables de mayor influyencia sobre su aptitud ambietal
En conclusión, las poblaciones de pasto banderita del estado de Chihuahua están divididas en dos grupos genéticos. La distribución de estos grupos está altamente influenciada por las condiciones climáticas de las eco-regiones del Estado y cada grupo genético posee un nicho climático diferente. Se recomienda que los programas de restauración con pasto banderita se realicen con genotipos locales de cada eco-región del estado de Chihuahua y en áreas con aptitud ambiental. Así mismo, podrían utilizarse en áreas con condiciones edafoclimáticas similares a las de Chihuahua. La diversidad genética encontrada dentro de cada grupo genético representa una oportunidad para genotipos sobresalientes, los cuales puedan ser utilizados en programas de restauración de pastizales para cada eco-región. No obstante, es necesario realizar estudios que consideren proyecciones climatológicas, ya que se desconoce cómo puede afectar el cambio climático a los tipos genéticos de pasto banderita y sus repercusiones en futuros trabajos de restauración.











 texto en
texto en 


