Introducción
Salmonella es un grupo de bacterias Gram negativas que comprende >2,600 serotipos clasificados en dos especies, S. enterica (incluye seis subespecies) y S. bongori. Entre estos, Salmonella enterica subsp. enterica (>1,500 serotipos) es el principal grupo responsable de enfermedades en el hombre denominadas “salmonelosis”1. Las principales manifestaciones clínicas son fiebre entérica (tifoidea y paratifoidea) y gastroenteritis causadas por los serotipos tifoideos y no tifoideos de Salmonella, respectivamente2. La estimación global anual de la salmonelosis es de >25 millones de casos de fiebre entérica y 153 millones de casos de gastroenteritis con ~300 mil muertes, las cuales se asocian mayormente al consumo de alimentos contaminados3. Actualmente, la salmonelosis es una de las cuatro principales enfermedades de transmisión alimentaria (ETA) a nivel mundial4.
Salmonella está ampliamente distribuida en la naturaleza y puede sobrevivir en una gran variedad de alimentos (origen animal y hortalizas), los cuales se han identificado como vehículos de transmisión de la bacteria2,4,5. La naturaleza zoonótica de los serotipos no tifoideos de Salmonella señala a los animales como el principal factor de riesgo para exponer a la bacteria al medio ambiente y transferir al patógeno a los humanos durante la producción, manipulación o consumo de los alimentos6. La vigilancia de Salmonella debe basarse en métodos de detección confiables que favorezcan la inocuidad alimentaria7.
La detección de Salmonella en los alimentos puede ser compleja, debido a que las bacterias a menudo se encuentran en concentraciones bajas en los alimentos. Otro aspecto que dificulta la detección del microorganismo en un alimento es el proceso de elaboración, los microorganismos de fondo y el tipo de matriz alimentaria8. Esto puede advertir un riesgo sanitario y justificar la necesidad de métodos oportunos para la detección de patógenos relevantes como es Salmonella7.
El método de cultivo es considerado el estándar de oro para el aislamiento y la detección de microbios patógenos en los alimentos9. El método de cultivo para la detección de Salmonella implica una etapa de pre-enriquecimiento no selectivo, seguido de enriquecimiento selectivo y sembrado en agares selectivo, y posterior caracterización bioquímica y serológica de colonias presuntivas, permitiendo obtener un resultado negativo o positivo en 4 y 6-7 días, respectivamente9. El consumo de tiempo, mano de obra y reactivos que involucra este método son un inconveniente para la detección rápida de Salmonella en la industria alimentaria7.
Actualmente se han desarrollado métodos moleculares rápidos como el método de reacción en cadena de la polimerasa (PCR) y su variante en tiempo real (rtPCR), que permiten la detección de Salmonella en un tiempo corto (24-72 h) en diversos alimentos10-12. Pruebas inmunológicas, bioquímicas y biosensores también se han propuesto como métodos rápidos8. Los métodos moleculares son útiles como herramientas de detección reduciendo la mano de obra y el tiempo de respuesta en comparación con los métodos de cultivo12. Sin embargo, la precisión de los métodos moleculares puede limitarse por la presencia de sustancias inherentes del alimento (sales biliares, bilirrubina, hemoglobina, urea, polisacárido, heces y antígenos y ADN, que pueden interferir con los resultados13.
La separación inmunomagnética (IMS) se ha utilizado para el aislamiento de Salmonella en diferentes matrices alimentarias8,14-16. Las IMS mejora la sensibilidad y especificidad de detección de Salmonella en los alimentos debido a las perlas de poliestireno anti-Salmonella que capturan a la bacteria, y ésta puede ser identificada por métodos de cultivo, inmunológicos o moleculares17. Recientemente se han desarrollado métodos que combinan las tecnologías IMS y rtPCR, que prescinden de la extracción de ADN y concentran al microorganismo por IMS después de un enriquecimiento primario de <24 h10,11,18.
En México, Salmonella representa un tema concerniente para la salud pública y la industria alimentaria. La Dirección General de Epidemiología de México reporta anualmente 28,815 casos de fiebres tifoidea y 78,681 casos de otras salmonelosis a nivel nacional19. Pero, la identificación de estos casos como ETA no está esclarecida. A pesar de que, en México, Salmonella se ha identificado como agente contaminante de carne de pollo20,21, carne de puerco21, carne de res21-23, vegetales24 y huevo25.
La detección oportuna de Salmonella en la carne de res es de especial interés dado su elevada frecuencia (28.9 a 32.4 %) en el alimento21-23 y la relevancia de la producción (1,960 t) y consumo per cápita (14.8 kg) anual de carne en México26. Además, el ganado y las aves se han identificado como reservorios de Salmonella27. En México, el método de cultivo es el estándar de oro para la detección de Salmonella en los alimentos, cuyo proceso puede resultar laborioso y requerir mucho tiempo (4 a 7 días) para obtener resultados finales28. Por lo tanto, el objetivo de este estudio fue realizar una validación interna del método comercial de rtPCR-IMS para la detección de Salmonella en carne de res, y comparar su eficiencia con el método de cultivo de referencia empleado en México.
Material y métodos
Descripción del estudio
El estudio de validación interna del método de reacción en cadena de polimerasa en tiempo real acoplado a separación inmunomagnética (rtPCR-IMS) para la detección de Salmonella en carne de res se evaluó respecto al método de cultivo de referencia en México “NOM-114-SSA1-1994”28, como se especifica en el manual de validación de métodos microbiológicos alternativos propuesto por la ISO16140:200329. La validación del método de rtPCR-IMS se realizó en muestras de carne de res presuntivas y contaminadas artificialmente. Los parámetros evaluados fueron límite de detección, sensibilidad, especificidad, selectividad, y el grado de concordancia entre los métodos.
Cepas bacterianas
La cepa de Salmonella ATCC 35664 se utilizó como control de referencia para el ensayo de validación. Una colonia pura se transfirió a 30 ml de caldo de soya tripticaseína (TSB; Becton Dickinson) y se incubó durante 4 h en un baño de agua a 35± 2 °C con agitación constante. Una alícuota del cultivo se utilizó para inocular 50 ml de TSB para obtener una absorbancia (DO) de 0.1 a 600 nm. Este subcultivo se incubó en las mismas condiciones hasta obtener una DO 1.0 (λ= 600 nm). Diluciones seriadas y recuento de placa en agar entérico Hektoen (Becton Dickinson) se realizaron para estandarizar las concentraciones de 1-5, 6-10, 1-15 y 16-30 UFC·100 µl-1.
Un listado de 60 cepas clasificadas como Salmonella (n= 30) y no-Salmonella (n= 30) se usaron para el parámetro de selectividad (Cuadro 1). La selección de las cepas no-Salmonella fue con base a las características bioquímicas que comparten con Salmonella o por ser considerados agentes contaminantes de la carne. La mayoría de las cepas no-Salmonella correspondían a cultivos de colección y se obtuvieron comercialmente. Las cepas Salmonella se obtuvieron del laboratorio, cuya identificación se realizó previamente con pruebas moleculares. Todas las cepas se cultivaron en TSB a 37 ºC por 24 h.
Cuadro 1 Microorganismos utilizados para la prueba de selectividad
| Prueba de inclusividad | Prueba de exclusividad | ||||
|---|---|---|---|---|---|
| Cepas Salmonella | No | Referencia | Cepas no Salmonella | No | Referencia |
| S. Agona | 1 | CIAD A.C | Bacillus subtilis | 1 | Ambiental |
| S. Albany | 1 | CIAD A.C | Candida Albicans | 1 | ATCC 10231 |
| S. Anatum | 1 | CIAD A.C | Citrobacter freundii | 1 | LEM 04001 |
| S. B monofásica | 1 | CIAD A.C | Citrobacter freundii | 1 | LEM-04001 |
| S. C1 monofasica | 1 | CIAD A.C | Enterobacter aerogenes | 1 | LEM-04003 |
| S. Cayar | 1 | CIAD A.C | Enterobacter aerogenes | 1 | ATCC 13048 |
| S. Cholerasuis | 1 | CIAD A.C | Enterobacter cloacae | 1 | LEM-04013 |
| S. Enteritidis | 1 | CIAD A.C | Enterococcus faecalis | 1 | LIBM-01003 |
| S. F | 1 | CIAD A.C | Escherichia coli | 1 | LEM-01005 |
| S. Gaminara | 1 | CIAD A.C | Escherichia coli | 1 | ATCC 25922 |
| S. Give | 1 | CIAD A.C | Eschrichia coli O157:H7 | 1 | CENAPA700728 |
| S. Haviana | 1 | CIAD A.C | Klebsiella pneumoniae | 1 | ATCC 13883 |
| S. Infantis | 1 | CIAD A.C | Listeria innocua | 1 | ATCC 33090 |
| S. Luciana | 1 | CIAD A.C | Listeria ivanovvi | 1 | ATCC 19119 |
| S. Meliagridis | 1 | CIAD A.C | Listeria monocytogenes | 1 | Ambiental |
| S. Minnesota | 1 | CIAD A.C | Listeria monocytogenes | 1 | ATCC 7694 |
| S. Montevideo | 1 | CIAD A.C | Listeria monocytogenes | 1 | ATCC 7644 |
| S. Muenchen | 1 | CIAD A.C | Listeria monocytogenes | 1 | Ambiental |
| S. Newport | 1 | CIAD A.C | Proteus mirabilis | 1 | ATCC 12453 |
| S. Oranienburg | 2 | CIAD A.C | Proteus mirabilis | 1 | LEM-03011 |
| S. Saintpaul | 2 | CIAD A.C | Proteus vulgaris | 1 | LEM-06070 |
| S. San Diego | 1 | CIAD A.C | Pseudomonas aeroginosa | 1 | ATCC 27853 |
| S. Senftenberg | 1 | CIAD A.C | Pseudomonas aeruginosa | 1 | LEM-01002 |
| S. Sohanina | 1 | CIAD A.C | Rhodococcus equi | 1 | LEM-01019 |
| Salmonella sp | 1 | CIAD A.C | Rhodococcus equi | 1 | ATCC 6939 |
| S. Typhimurium | 1 | CIAD A.C | Shigella flexneri Gpo. B | 1 | ATCC 12022 |
| S. Thompson | 1 | CIAD A.C | Shigella flexnieri | 1 | LEM-04004 |
| S. Weltevreden | 1 | CIAD A.C | Shigella sonnei | 1 | ATCC 9290 |
| CIAD A.C | Staphylococcus aereus | 1 | ATCC 25923 | ||
| CIAD A.C | Staphylococcus epidermis | 1 | ATCC 12228 | ||
Las siglas representan: CIAD A.C. (Centro de Investigación en Alimentación y Desarrollo A.C.), CENAPA (Centro Nacional de Servicios de Diagnóstico en Salud Animal), ATCC (American Type Culture Collection), LEM (código interno), LIBM (código interno)
Colección de las muestras de carne de res
Para el estudio se recolectaron 60 muestras de carne cruda de res que consistían en carne tipo molida (n= 20), pulpa negra (n= 20) y pulpa redonda (n= 20) procedentes de tres mercados ubicados en la ciudad de Culiacán, Sinaloa, México. El número de muestras de carne se asignó de acuerdo con la recomendación mínima sugerida por la ISO16140:200329. Se tomaron muestras de 500 g de carne, las cuales se colocaron en bolsas de plástico estériles y transportadas en refrigeración al laboratorio para su posterior análisis. El estudio contempló realizar el análisis en las muestras presuntivas para simular los efectos reales de contaminación que ocurren en la naturaleza, y en muestras de carne contaminadas artificialmente.
Análisis de las muestras de carne de res presuntivas
Se homogenizaron 50 g de carne de res con 50 ml de agua destilada estéril en un triturador a 230 rpm por 2 min. Posteriormente, la muestra se dividió equitativamente en porciones de 25 ml para el método de rtPCR -IMS y NOM-114-SSA1-1994.
Análisis de las muestras de carne de res contaminadas artificialmente
Se utilizaron las muestras de carne de res clasificadas como negativas para Salmonella por ambos métodos. Una porción de 100 g de carne de res se homogenizó con 100 ml de agua destilada estéril durante 2 min a 230 rpm, y se separaron equitativamente en dos porciones de 50 ml; una porción se usó como control negativo y la porción restante para la contaminación de la carne con Salmonella. La porción contaminada se homogeneizó durante 2 min a 230 rpm, y subsecuentemente se dividió el homogenizado en dos porciones de 25 ml para su análisis por ambos métodos. Paralelamente, se incluyó un control negativo bajo las mismas condiciones. La selección del inóculo correspondió al límite de detección que permite una recuperación fraccional de la bacteria por alguno de los métodos.
Límite de detección
Para establecer el límite de detección relativa, se prepararon cinco niveles de inóculo de Salmonella: 0, 1-5, 6-10, 1-15 y 16-30 UFC·25 ml-1. A partir de las muestras de carne de res clasificadas como negativas para Salmonella, se tomaron 250 g de muestra y se homogeneizaron con 250 ml de agua destilada estéril durante 2 min a 230 rpm en un homogeneizador automático (Stomacher 400 Circulador). El homogenizado se fraccionó en 10 porciones de 25 ml para su inoculación con Salmonella: porción 1-2 (0 UFC·25 ml-1), porción 3-4 (1-5 UFC·25 ml-1), porción 5-6 (6-10 UFC·25 ml-1), porción 7-8 (1-15 UFC·25 ml-1) y porción 9-10 (16-30 UFC·25 ml-1). Los niveles de inóculo añadidos se corroboraron mediante el método de recuento en placa en agar entérico Hektoen, y los tipos de porciones se repartieron equitativamente para su análisis para ambos métodos. El límite de detección corresponderá a la concentración más pequeña del inóculo que se pueda detectar en la muestra en el 50 % de las ocasiones por los métodos29.
Prueba de selectividad
La prueba de selectividad de los métodos se realizó in vitro sin el uso de muestras de carne de res y la selección del inóculo bacteriano se realizó según las especificaciones de la ISO16140:200329. Para cada bacteria (Cuadro 1) se estandarizó un inóculo a una absorbancia de 1.0 (λ= 600 nm) en ambos ensayos. Para el ensayo de exclusividad se ajustó una concentración de 104 UFC·100 µl-1 en 225 ml de agua peptonada. Mientras que, en el ensayo de inlcusividad se ajustó la concentración de 100 UFC·100 µl-1 en 225 ml de los medios de enriquecimiento utilizados por los métodos rtPCR -IMS (agua peptonada) y NOM-114- SSA1-1994 (caldo lactosado), y la posterior adición de 100 µl de un pool de las cepas empleadas en la prueba de exclusividad. Todos los cultivos fueron evaluados por los métodos de rtPCR -IMS y NOM-114- SSA1-1994.
Método de cultivo de referencia (NOM-114-SSA1-1994)
Muestras de carne (25 ml) se enriquecieron con 225 ml de caldo de lactosa (Becton Dickinson) y se incubó a 35 ± 2 °C durante 24 ± 2 h. Posteriormente, se transfirieron alícuotas de 1 ml del cultivo a 10 ml de caldo Selenito Cistina (Becton Dickinson) y a 10 ml de caldo Rappaport Vassiliadis (Becton Dickinson), para su incubación a 35 ± 2 °C durante 24 h. Una vez completada la incubación, se inocularon alícuotas de 10 µl de los cultivos previos en agar entérico Hektoen (Becton Dickinson), Agar Xilosa Lisina Desoxicolato (Becton Dickinson) y agar Salmonella-Shigella (Becton Dickinson), y se incubaron a 35 ± 2 °C por 24 h. A partir de los agares, se seleccionaron colonias presuntivas (n= 3) de Salmonella para su identificación mediante pruebas bioquímicas primarias (agar de hierro de azúcar triple, agar de lisina y hierro y caldo de urea), API 20E (Biomeriux NC) y pruebas serológicas basadas en la detección de antígeno O polivalente (InDRE)28. Paralelamente, se incluyeron un control negativo (medio sin bacteria) y un control positivo (Salmonella ATCC 35664).
Método rtPCR-IMS
La detección de Salmonella se realizó de acuerdo con las condiciones del proveedor (http://www.biocontrolsys.com ). Todos los reactivos necesarios para la preparación de la reacción rtPCR para Salmonella (GDS Salmonella Tq 71008) están disponibles comercialmente por Assurance GDS®, BioControl System Inc. Una porción de 25 ml de muestra se enriqueció con 225 ml de agua de peptona tamponada (Becton Dickinson), y se incubó a 35 ± 2 °C por 24 h. Posteriormente, se transfirió 1 ml del enriquecimiento previo a la placa de concentración que contenía 20 µl de reactivo de concentración (perlas magnéticas recubiertas con anticuerpos anti-Salmonella), y se mezcló por 10 min en un homogeneizador automático (Vortex Mixed Biocontrol Sistema Bellevue). Las perlas magnéticas se extrajeron con una pipeta magnética (Pick Pen ™ Biocontrol System Bellevue), se lavaron con 1 ml del tampón por 7 seg, y se transfirió a una placa que contenía 35 µl del reactivo de resuspensión. Con una pipeta multicanal, se transfirieron 20 µl del tampón de resuspensión con las perlas inmunomagnéticas, y se depositaron en los tubos de PCR, que previamente contenían la sonda, los oligonucleótidos y la ADN Taq-polimerasa (5- Prime). Finalmente, los tubos se colocaron en la máquina de rtPCR. Paralelamente, se incluyeron un control negativo (medio sin bacteria) y un control positivo (Salmonella ATCC 35664). Todas las muestras analizadas por el ensayo de rtPCR-IMS se evaluaron nuevamente a partir del enriquecimiento original utilizando el método de referencia para verificar la detección de Salmonella.
Análisis estadístico
Los parámetros de validación del método rtPCR-IMS se basaron en la comparación de los acuerdos positivos (AP), acuerdos negativos (AN), desviaciones positivas (DP) y desviaciones negativas (DN) de los resultados obtenidos en la detección de Salmonella en las muestras de carne en comparación al método de NOM-114-SSA1-1994. Los parámetros se calcularon con las siguientes fórmulas:
El grado de concordancia entre los métodos (rtPCR-IMS y NOM-114-SSA1-1994) se determinó por el índice kappa y la prueba de McNemar (χ2) con significancia del 5%.
Resultados
Límite de detección
El Cuadro 2 muestra la capacidad de los métodos de rtPCR-IMS y NOM-114-SSA1-1994 para la detección de los diferentes niveles de contaminación de Salmonella en carne de res. El límite de detección para el método de rtPCR-IMS fue de 1 - 5 UFC·25 g-1. No se obtuvieron productos de PCR de las muestras no inoculadas.
Cuadro 2 Límite de detección de Salmonella en las muestras de carne mediante los métodos de rtPCR-IMS y NOM-114-SSA1-1994
| No. de réplica | rtPCR-IMS | NOM-114-SSA1-1994 | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1-5 UFC | 6-10 UFC | 11-15 UFC | 16-30 UFC | 0 UFC | 1-5 UFC | 6-10 UFC | 11-15 UFC | 16-30 UFC | 0 UFC | |
| 1 | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- |
| 2 | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- |
| 3 | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- |
| 4 | +/+/- * | +/+/+ | +/+/+ | +/+/+ | -/-/- | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- |
| 5 | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- | +/+/ - | +/+/+ | +/+/+ | +/+/+ | -/-/- |
| 6 | +/ -/ - | +/+/+ | +/+/+ | +/+/+ | -/-/- | +/+/+ | +/+/+ | +/+/+ | +/+/+ | -/-/- |
+ = Salmonella sp. fue detectada en la muestra; - = Salmonella sp. no fue detectada en la muestra.
*El ensayo fue negativo, pero la confirmación por el método de referencia fue positiva a partir del enriquecimiento original.
Sensibilidad, especificidad y exactitud
El Cuadro 3 resume los acuerdos (AP y AN), las desviaciones (DP y DN) y los parámetros de validación relativa del ensayo de rtPCR-IMS para la detección de Salmonella en las muestras de carne de res. La tasa de detección de Salmonella en las muestras presuntivas (n = 60) mediante el análisis de rtPCR -IMS (21.6%) y NOM-114-SSA1-1994 (20.0 %) no fue suficiente para calcular los parámetros de validación.
Cuadro 3 Comparación de los métodos de rtPCR-IMS y NOM-114-SSA1-1994 para la detección de Salmonella en carne de res
| Muestra | Resultados* | Y | Sensibilidad | Falso negativo | Especificidad | Falso positivo | Exactitud | χ2 | Kappa | |||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| AP | DP | AN | DN | No | (%) | (%) | (%) | (%) | (%) | |||
| Presuntivas | 5 | 7 | 41 | 7 | 14 | ND | ND | ND | ND | ND | ND | ND |
| Contaminadas** | 56 | 0 | 3 | 1 | 1 | 98.2 | 1.8 | 100 | 0 | 98.3 | 1.0 (P=0.317) | 0.85 |
*AP (acuerdo positivo): Detección del patógeno por ambos métodos. AN (acuerdo negativo): No detección del patógeno por ambos métodos. DP (desviación positiva): Detección del patógeno por el método alterno, pero no por el método de referencia. DN (desviación negativa): Detección del patógeno por el método de referencia, pero no por el método alterno. ND (no determinado).
**Los resultados corresponden a muestras de carnes inoculadas con una concentración de 1-5 UFC·25 ml-1 de Salmonella ATCC 35664.
En las 60 muestras de carne contaminadas artificialmente (1-5 UFC·25 ml-1), los ensayos de rtPCR-IMS y NOM-114-SSA1-1994 detectaron a Salmonella en 56 (93.3 %) y 59 (98.3 %) veces, respectivamente. Los métodos coincidieron en la detección de Salmonella en 59 ocasiones: 56 AP y 3 AN. Solo se obtuvo una DN (detectada por NOM-114 pero no por rtPCR-IMS). La rtPCR-IMS presentó una sensibilidad de 98.2 % (56/57), especificidad de 100 % (3/3) y exactitud de 98.3 % (59/60). Los índices de concordancia (k = 0.85 y χ2 = 1.0) y el valor discordante (Y= 1), indicaron que el ensayo de rtPCR-IMS y el método de referencia (NOM-114-SSA1-1994) coinciden en los criterios estadísticos (Cuadro 3). Todas las muestras de carne no inoculadas fueron negativas para Salmonella por ambos métodos.
Selectividad
Los métodos de rtPCR-IMS y NOM-114-SSA1-1994 presentaron 100 % de exclusividad y 100 % de inclusividad. Ninguno de los métodos reportó reacciones cruzadas. La Figura 1 muestra las amplificaciones del método de rtPCR-IMS correspondientes a la prueba de inclusividad y exclusividad.
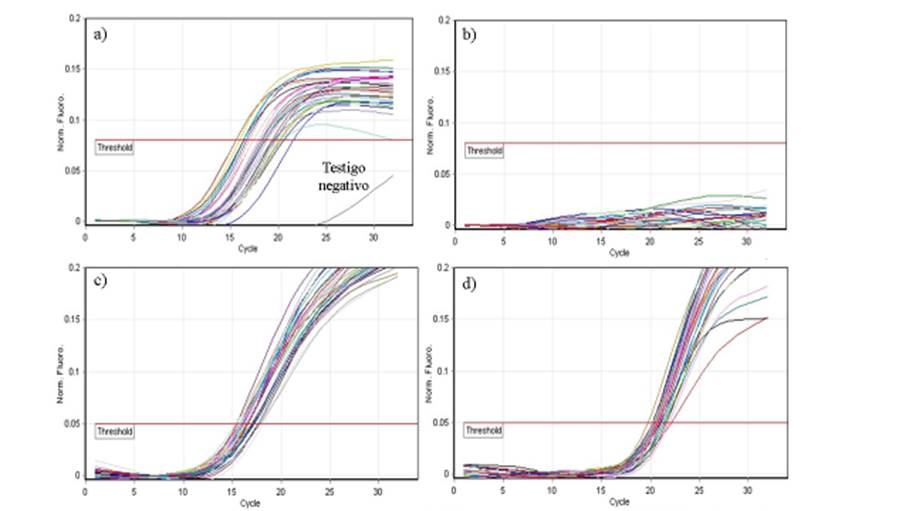
La figura a y b muestran la amplificación de las 30 cepas de Salmonella (25 serotipos diferentes) y las 30 cepas de no-Salmonella, respectivamente. La posición de las líneas respecto al umbral indican un resultado positivo (superior) o negativo (inferior). La figura c y d muestran la amplificación de los controles internos de la reacción (ICA) de las cepas incluidas en el ensayo de inclusividad (a) y exclusividad (b). Las de líneas ICA rebasaron el umbral por lo que se considera una reacción válida.
Figura 1 Amplificaciones del método de rtPCR-IMS para la prueba de inclusividad y exclusividad
Discusión
Salmonella representa una amenaza para la salud pública y la industria alimentaria a nivel mundial5, y en México no es la excepción19,20-25. Los resultados de este trabajo exhiben la alta persistencia de Salmonella en las muestras presuntivas de carne de res (20 a 21.6 %), como previamente lo han expuesto algunos estudios en México21-23. Los métodos microbiológicos convencionales sirven de base para el análisis de rutina en muchos laboratorios de seguridad alimentaria y salud pública debido a la facilidad de uso, la fiabilidad de los resultados, la alta sensibilidad y especificidad8. No obstante, el tiempo de análisis (5 a 7 días) de los métodos de cultivo se observa como una limitante. La incorporación de los métodos moleculares rápidos para la detección de Salmonella en los alimentos permite una intervención temprana y hace posible la protección preventiva del consumidor10,11,18.
El límite de detección del método de rtPCR-IMS y NOM-114-SSA1-1994 fue de 1 - 5 UFC·25 g-1 en carne de res, que corresponde a la concentración más baja evaluada. Del total de muestras inoculadas con 1 - 5 UFC·25 g-1, el método alternativo arrojó tres repeticiones negativas, de las cuáles solo dos se confirmaron como verdaderamente negativas por el método de referencia (Cuadro 2). La no detección por el método de rtPCR-IMS se puede explicar por la cantidad pequeña (1 - 5 UFC·25 g-1) de Salmonella en el enriquecimiento no selectivo, las consideraciones probabilísticas de inoculación o el efecto del enriquecimiento30. Widjojoatmodjo et al31 destacan la importancia del pre-enriquecimiento previo a la detección por PCR para aumentar su sensibilidad, dado que la mayoría de los métodos de PCR necesitan concentraciones altas de microorganismos para una adecuada detección. Similar a nuestros resultados, Notzon et al30 y O´Regan et al32 reportaron un límite de detección del método de rtPCR-IMS en carne de res de 10-100 UFC·25 g-1, y en carne de pollo de 1-10 UFC·25 g-1, respectivamente. Estos métodos emplearon un enriquecimiento de 6 h30 ó 24 h32 previo a la detección por PCR. Por otra parte, se menciona10 que la IMS es una alternativa para evitar el enriquecimiento secundario permitiendo la detección de 1-10 células en un periodo de incubación de 12 - 24 h. El límite de detección observado (1 - 5 UFC·25 g-1) con la rtPCR-IMS permitiría alinearse con los requerimientos normativos nacionales (NOM-213-SSA1-2002) que exigen la cero tolerancia de Salmonella en 25 g de carne cruda de res33.
Los datos obtenidos en las muestras presuntivas no permitieron determinar los parámetros de validación, debido a que los cálculos se realizan sobre una serie de resultados negativos obtenidos por el método de referencia, los cuales no pueden exceder el doble del número de los resultados positivos según lo estipulado en el manual de validación29. Por lo que este estudio, explica la validación del método de rtPCR-IMS para el análisis de Salmonella basado en muestras de carne de res contaminadas artificialmente. El grado de sensibilidad, especificidad, exactitud, y concordancia del método comercial de rtPCR-IMS con el método de cultivo de referencia valida su uso para el análisis de Salmonella en carne de res generando resultados idóneos en un tiempo de 24 h (Cuadro 2). Además, la tasa de falsos positivos (0 %) y falsos negativos (1.8 %) del método son bajas. Es importante señalar que los métodos moleculares no reemplazan las técnicas de cultivo, dado que los resultados positivos deben confirmarse por el método de referencia34.
Algunos estudios previos han expuesto la concordancia de protocolos de rtPCR-IMS con los métodos de cultivo de referencia para la detección de Salmonella en carne de res30 y pollo32,35, resaltando su alto grado de sensibilidad (94 - 100 %), especificidad (80 - 94 %) y exactitud (89 - 100 %). Estas características determinadas en el método comercial de rtPCR-IMS (Cuadro 3) pueden atribuirse a que las perlas inmunomagnéticas contienen antígenos que permite concentrar al microbio de interés a partir de enriquecimientos no selectivos disminuyendo el tiempo de análisis35. Además, los oligonúcleótidos empleados son capaces de detectar diferentes tipos de cepas de Salmonella (Figura 1).
La desviación negativa (DN) observada entre los métodos (Cuadro 2), puede explicarse debido a que el método de cultivo contiene diversas etapas de enriquecimiento que favorecen la recuperación de células dañadas y el crecimiento del microorganismo de interés en comparación a los métodos rápidos12. También, la presencia de Proteus, E. coli, Klebsiella aerogenes y Enterobacter en estado mucoide en el caldo de enriquecimiento pueden unirse a los anticuerpos de las perlas causando reacciones cruzadas e impidiendo la detección de Salmonella35. Ampliamente se ha descrito que el tipo de matriz y sus componentes químicos pueden afectar los resultados de los métodos moleculares12-13. En cuanto a los tres acuerdos negativos entre los métodos, puede atribuirse a la cantidad extremadamente baja del microorganismo después del enriquecimiento o que no existían células en el inóculo inicial.
El valor de McNemar (χ2 = 1.0, P = 0.317) obtenido en este estudio cumplen con el parámetro de no significancia (χ2 < 3.84)29, y demuestra que no existe diferencia entre los métodos de rtPCR-IMS y el método NOM-114-SSA1-1994 para la detección de Salmonella en carne de res. Además, el índice Kappa revela una alta concordancia (0.85 o 85%) entre los métodos. Notzon et al30 infirieron la equiparabilidad del método alterno de rtPCR-IMS con una concordancia de 85 % (k = 0.85) y 87 % (k = 0.87) para la detección de Salmonella en carne contaminadas artificial y naturalmente, respectivamente.
Un método selectivo es aquel que permite detectar al analito que se está examinando, y que puede garantizar que la señal detectada solo puede ser un producto de ese analito específico29. En este sentido el método de rtPCR-IMS fue capaz de discriminar a Salmonella, dado que detectó las 30 cepas de Salmonella correspondiente a 25 diferentes serotipos aún en presencia de otros microorganismos, y no generar interferencia con las cepas diferentes a Salmonella. Mercanoglu & Griffiths36 han reportado que la combinación de rtPCR y IMS para la detección de Salmonella presentan una selectividad del 100%, atribuyéndose esta propiedad al efecto de las perlas inmunomagnéticas y la selección de los oligonucleótidos empleados.
Conclusiones e implicaciones
Los resultados proponen a la rtPCR-IMS como un método eficiente para la detección rápida de Salmonella spp. en carne de res dado que no presentó diferencias con el método de referencia (NOM-114-SSA1-1994), aportando la ventaja de detectar al microorganismo en un tiempo corto (24 h) y en una concentración mínima (1 UFC·25 g-1) y sin causar reacciones cruzadas con otros microorganismos que se encuentran como microbiota natural en la carne de res. La incorporación de este tipo de métodos a la industria alimentaria y laboratorios microbiológicos permitirán una respuesta rápida para asegurar la inocuidad de los alimentos y prevenir el riesgo de enfermedades. Adicionalmente, se alerta a las autoridades sanitarias sobre la alta incidencia de Salmonella en la carne cruda a fin de incluir controles a lo largo de la cadena alimentaria.











 texto en
texto en 


