Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista mexicana de ciencias pecuarias
versão On-line ISSN 2448-6698versão impressa ISSN 2007-1124
Rev. mex. de cienc. pecuarias vol.12 no.3 Mérida Jul./Set. 2021 Epub 14-Mar-2022
https://doi.org/10.22319/rmcp.v12i3.5562
Articulos
Efecto de dos estrategias de agrupación de padres fantasmas en la evaluación genética de rasgos de crecimiento en el ganado Braunvieh mexicano
a Universidad Autónoma Chapingo. Departamento de Zootecnia, Posgrado en Producción Animal. Km. 38.5 Carretera México-Texcoco. 56230, Chapingo, Estado de México. México.
b University of Otago, Department of Mathematics and Statistics, Dunedin, New Zealand.
El estudio tuvo como objetivo comparar dos estrategias de agrupación de conjuntos de padres desconocidos o padres fantasmas (GPF) en la evaluación genética de rasgos de crecimiento para el ganado Braunvieh mexicano. Los datos fenotípicos incluyeron los pesos al nacimiento (PN), destete (PD) y al año (PA). El pedigrí incluyó 57,341 animales. La primera estrategia involucró 12 GPF (G12) según el año de nacimiento de la progenie del padre desconocido y el sexo del padre desconocido, mientras que la segunda involucró 24 GPF (G24) según el año de nacimiento de la progenie del padre desconocido y las cuatro vías de selección. Los modelos animales incluyeron efectos fijos y el efecto genético aditivo directo aleatorio; el PD también incluyó efectos aleatorios genéticos maternos y ambientales permanentes maternos. Las correlaciones producto-momento entre el VGE de G0 (sin GPF) y G12 fueron 0.96, 0.77 y 0.69 para PN, PD y PA, respectivamente, y entre el VGE de G0 y G24 fueron 0.91, 0.54 y 0.53, respectivamente. Las correlaciones de rango correspondientes entre G0 y G12 fueron 0.94, 0.77 y 0.72, y entre G0 y G24 fueron 0.89, 0.61 y 0.60. Las tendencias genéticas mostraron una desviación base de la tendencia genética de G0, a excepción para PN de G12. Los resultados no respaldan el uso de las dos estrategias de agrupación en la población y los rasgos estudiados, y se requiere investigación adicional. Es importante introducir GPF en el modelo, una contribución suficiente del fenotipo de los descendientes a los GPF y evitar la colinealidad entre GPF y los efectos fijos. Los grupos genéticos deben reflejar los cambios en la estructura genética de la población para los padres desconocidos, incluidas las diferentes fuentes de materiales genéticos, y los cambios realizados por selección a lo largo del tiempo.
Palabras clave Bovinos Braunvieh; VGE; Grupos genéticos; Correlación de rango; Padres desconocidos
The study aimed to compare two grouping strategies for unknown parents or phantom parent groups (PPG) on the genetic evaluation of growth traits for Mexican Braunvieh cattle. Phenotypic data included birth (BW), weaning (WW) and yearling (YW) weights. Pedigree included 57,341 animals. The first strategy involved 12 PPG (G12) based on the birth year of the unknown parent’s progeny and the sex of the unknown parent, while the second involved 24 PPG (G24) based on the birth year of the unknown parent’s progeny and 4-selection pathways. The animal models included fixed effects and the random direct additive genetic effect; WW also included random maternal genetic and maternal permanent environmental effects. Product-moment correlations between EBV from G0 (no PPG) and G12 were 0.96, 0.77 and 0.69 for BW, WW and YW, respectively, and between EBV from G0 and G24 were 0.91, 0.54, and 0.53, respectively. Corresponding rank correlations between G0 and G12 were 0.94, 0.77, and 0.72, and between G0 and G24 were 0.89, 0.61, and 0.60. Genetic trends showed a base deviation from the genetic trend of G0, except for BW of G12. The results did not support the use of the two grouping strategies on the studied population and traits, and further research is required. Introducing PPG to the model, enough phenotype contribution from descendants to PPG, and avoiding collinearity between PPG and fixed effects are important. Genetic groups should reflect changes in the genetic structure of the population to the unknown parents, including different sources of genetic materials, and changes made by selection over time.
Key words Braunvieh cattle; EBV; Genetic groups; Rank correlation; Unknown parents
Introducción
El Braunvieh mexicano es una raza de ganado de doble propósito. Desde junio de 2003, se han realizado evaluaciones genéticas nacionales para los rasgos de crecimiento de esta raza en México1. Como en cualquier población ganadera, hay padres desconocidos en el pedigrí. Se asume que los padres desconocidos no están relacionados, no son endogámicos y tienen un solo descendiente. Los padres desconocidos pueden corresponder a animales base en la primera generación o extenderse a lo largo de generaciones. Ellos afectan el progreso genético de varias formas: (i) reduciendo la intensidad de la selección para animales con padres desconocidos, (ii) la incertidumbre de linaje disminuye la precisión de las evaluaciones genéticas, (iii) la identificación incorrecta de los padres produce sesgos en estimaciones de valores genéticos (VGE) y de heredabilidad (2. La mejor predicción lineal insesgada (MPLI) regresa las predicciones de mérito genético de los animales para padres desconocidos de media cero. Dependiendo de los antecedentes genéticos y la generación a la que pertenecen los padres desconocidos, su mérito genético esperado podría ser diferente de cero. Quass3 estableció una metodología para considerar grupos de padres fantasmas (GPF) o grupos genéticos en MPLI. Aunque los GPF no son de interés per se, se consideran para facilitar el modelado y el cómputo4. Además, junto con la corrección estadística de la información de pedigrí faltante no aleatoria, los GPF permiten la estimación directa de parámetros genéticos cuantitativos5.
Debido a que no existen reglas específicas para determinar los GPF, su definición se basa principalmente en los criterios del investigador, pero generalmente incluye un componente de tiempo6. Otros factores comúnmente considerados en las estrategias de agrupación son el sexo del progenitor o la intensidad de selección4,7,8. Todos los descendientes de un individuo con GPF contribuyen a la estimación del efecto GPF5, por lo que es poco probable que tener GPF con un número igual de individuos afecte la capacidad del modelo animal para estimar el efecto GPF con una precisión aceptable. Sin embargo, cualquier estrategia para asignar padres desconocidos a GPF debe reflejar el nivel genético promedio de los padres desconocidos9.
Debido a la inclusión de GPF en el modelo, Theron et al7 observaron un cambio significativo y una reducción del sesgo en la tendencia genética de la producción de leche para las Holstein sudafricanas. Del mismo modo, se detectó una reducción en el sesgo de VGE al incluir GPF en los análisis genéticos para los pesos al destete, posdestete y al año, la circunferencia escrotal y la puntuación de musculatura en ganado Nelore10. El objetivo de este estudio fue comparar dos estrategias de agrupación de padres desconocidos a GPF en la evaluación genética de rasgos de crecimiento en el ganado Braunvieh mexicano.
Material y métodos
Datos
Los registros de pedigrí y fenotípicos del ganado Braunvieh mexicano se obtuvieron de la Asociación Mexicana de Criadores de Ganado Suizo de Registro (Ciudad de México). Los registros fenotípicos fueron pesos al nacimiento (PN), destete (PD) y al año (PA) de animales nacidos entre 1985 y 2017, en 229 ranchos en todo México. Los pesos de destete y al año se ajustaron a 240 d y 365 d de edad, respectivamente, de acuerdo con el procedimiento propuesto por la Federación para el Mejoramiento Genético de Bovinos de Carne11. Los registros fuera del rango de la media ± 3 DE para el rasgo de interés no se incluyeron en los análisis. Los registros de PD y PA fuera de 240 ± 45 d y 365 ± 45 d de edad, respectivamente, también fueron excluidos de los análisis. El pedigrí se obtuvo (padres), comenzando de animales con un fenotipo disponible (para cualquiera de los tres rasgos), y limitado a animales nacidos desde 1970. El pedigrí final incluyó 57,341 individuos, 18,689 machos, 38,652 hembras, 2,746 padres y 27,015 madres.
Los grupos contemporáneos se formaron teniendo en cuenta el hato, el año y la estación de nacimiento (lluviosa o seca). Los registros de grupos contemporáneos con menos de cuatro animales se excluyeron de los análisis. El Cuadro 1 muestra el número final de registros y la estadística descriptiva para cada rasgo.
Análisis genéticos
Los análisis genéticos comprendieron la estimación de parámetros genéticos y la MPLI12 para la población Braunvieh mexicana, utilizando los siguientes modelos de un solo rasgo:
para PN y PA, y
para PD, donde y, b, u, m, mpe y e son vectores de registros fenotípicos, efectos fijos, efectos genéticos aditivos directos, genéticos aditivos maternos, ambientales permanentes maternos y residuales, respectivamente. X, Z 1, Z 2y Z 3 son matrices de incidencia que relacionan los registros con b, u, m y mpe, respectivamente. Los efectos fijos fueron:
bPN = [sexo, pureza Braunvieh, edad de la madre, (edad de la madre)2, grupo contemporáneo de nacimiento]
bPD = [sexo, pureza Braunvieh, edad de la madre, (edad de la madre)2, grupo contemporáneo de predestete, condición de alimentación con leche]
bPA = [sexo, pureza Braunvieh, grupo contemporáneo de posdestete, alimentación posdestete]
Hubo 1,778, 1,450 y 1,038 grupos contemporáneos de nacimiento, grupos contemporáneos de predestete y grupos contemporáneos de posdestete, respectivamente. Las condiciones de alimentación con leche eran amamantamiento sin ordeña, amamantamiento con ordeña adicional y alimentación con sustituto de la leche. Los regímenes alimentarios posdestete fueron pastoreo, semiestabulación y estabulación total. Las proporciones sexuales fueron cercanas a 1. La edad de la madre en el parto tuvo un mínimo, media, DE y máximo de 1.70, 6.64, 3.04 y 17.00 años, respectivamente. La pureza de Braunvieh tuvo un mínimo, media, DE y máximo de 0.88, 0.99, 0.01 y 1.00, respectivamente. Se utilizaron los modelos oficiales para la evaluación de los rasgos estudiados en ganado Braunvieh mexicano. Las estructuras de (co)varianza fueron:
para PN y PA, y
para PD, donde A es la matriz de relación genética aditiva basada en pedigrí, I
Nd
e I
N
son matrices de identidad de orden igual al número de madres y observaciones.
Grupos genéticos
La evaluación de las estrategias de agrupación genética se realizó mediante la comparación de VGE a partir de la MPLI con y sin GPF. Los criterios utilizados para formar los grupos de padres desconocidos fueron:
1) Año de nacimiento: El año de nacimiento del padre desconocido fue cinco años antes del año de nacimiento de su progenie. Los años de nacimiento de los padres desconocidos se agruparon en seis clases: 1965-69, 1970-74, 1975-79, 1980-84, 1985-89 y 1990-96.
2) Sexo del padre desconocido.
3) Vía de selección (padre de padre, padre de madre, madre de padre, madre de madre).
Las dos estrategias de agrupación genética fueron:
G12: Clase del año de nacimiento (6 niveles) × sexo del padre desconocido (2 niveles).
G24: Clase del año de nacimiento (6 niveles) × vía de selección (4 niveles).
Los grupos genéticos basados en criterios como el sexo del ancestro faltante o las vías de selección permiten la evaluación de diferentes diferenciales de selección genética4. Asimismo, la inclusión de la categoría año de nacimiento permite modelar el mejoramiento genético a lo largo del tiempo3,7. El Cuadro 2 muestra el número de padres desconocidos en cada GPF para cada estrategia.
Cuadro 2 Criterios y frecuencia de padres desconocidos en los grupos de padres fantasmas
| Estrategia1 | Padre desconocido | Grupo de años2 | |||||
|---|---|---|---|---|---|---|---|
| 1965- 1969 |
1970- 1974 |
1975- 1979 |
1980- 1984 |
1985- 1989 |
1990- 1996 |
||
| G12 | Padre | 540 | 513 | 820 | 941 | 678 | 433 |
| Madre | 647 | 457 | 664 | 891 | 564 | 35 | |
| G24 | Padre de padre | 119 | 58 | 72 | 90 | 87 | 143 |
| Padre de madre | 421 | 455 | 748 | 851 | 591 | 290 | |
| Madre de padre | 145 | 57 | 51 | 84 | 73 | 9 | |
| Madre de madre | 502 | 400 | 613 | 807 | 491 | 26 | |
1 Grupo de padres fantasmas con niveles 12 (G12) y 24 (G24).
2 Año de nacimiento de la progenie - 5.
Las ecuaciones del modelo mixto (para PN y PA), sin incluir GPF en el modelo, fueron:
Donde
La incorporación de los efectos de GPF en el mérito genético de los animales (es decir, VGE = û + Qĝ) se puede hacer directamente en las ecuaciones del modelo mixto, utilizando la transformación de Quaas y Pollak14 que implica la absorción de las ecuaciones de GPF, lo que da3:
Este procedimiento evita el paso adicional de calcular û + Qĝ después de la Ec. [4] y la necesidad de recrear la matriz Q, que es computacionalmente costosa. La transformación de Quaas y Pollak14 no está implementada en el software MTDFREML. Por lo tanto, las Ec. [3, 4] se aplicaron para la MPLI con y sin GPF, respectivamente (términos adicionales de los efectos genéticos maternos y de los ambientales permanentes maternos se involucraron para PD). Los valores genéticos estimados que representan GPF (û + Qĝ) se obtuvieron utilizando las funciones "qmat" y "Qgpu" del paquete "ggroups" de R 15, donde se calculó la matriz de contribuciones de GPF a los individuos en un pedigrí (Q), y se agregaron contribuciones de GPF (Qĝ) al mérito genético de los animales (û), con ĝ y û obtenidos de MTDFREML13.
Comparaciones de estrategias de agrupación
Las comparaciones entre las estrategias de agrupamiento se realizaron mediante:
Las correlaciones producto-momento de Pearson y rango de Spearman entre los VGE obtenidos con y sin PPG. Las tendencias genéticas obtenidas para cada análisis, promediando los VGE por año de nacimiento.
Resultados y discusión
Hubo 3,925 animales con padre desconocido, 3,258 animales con madre desconocida y 2,430 animales con padre y madre desconocidos. Los padres desconocidos fueron asignados a 12 o 24 GPF (G12 y G24; Cuadro 2). Los componentes de varianza obtenidos con y sin GPF se muestran en el Cuadro 3. Las estimaciones de los parámetros para los rasgos estudiados bajo diferentes escenarios fueron muy similares. Por lo tanto, la elección del modelo no debe interferir con la estimación de los parámetros genéticos.
Cuadro 3 Componentes de varianza para el peso al nacimiento (PN), peso al destete (PD) y peso al año (PA) para la población Braunvieh mexicana estimados con 12 (G12), 24 (G24) y sin (G0) grupos de padres fantasmas
| Estrategia | Rasgo |
|
|
|
|
|---|---|---|---|---|---|
| G0 | PN | 2.69 | - | - | 8.54 |
| PD | 87.76 | 8.80 | 23.12 | 435.85 | |
| PA | 86.27 | - | - | 692.96 | |
| G12 | PN | 2.69 | - | - | 8.53 |
| PD | 83.14 | 8.43 | 23.06 | 436.58 | |
| PA | 81.30 | - | - | 695.01 | |
| G24 | PN | 2.71 | - | - | 8.52 |
| PD | 90.27 | 10.09 | 21.37 | 435.37 | |
| PA | 85.72 | - | - | 692.52 |
Theron et al7 reportaron que la inclusión de GPF tiene una influencia menor en la estimación de los componentes de co(varianza), asimismo, Shiotsuki et al16 mostraron que el uso de una matriz de relaciones que incluye grupos genéticos no genera diferencias en las estimaciones de la varianza contrastadas con el uso de una matriz sin grupos genéticos. En algunos estudios7,8,10 se obtuvieron componentes de varianza considerando un modelo "control", el cual no incluía grupos genéticos, y esos componentes de varianza se utilizaron para predecir valores genéticos en un modelo que incluía GPF, similar al procedimiento aplicado en esta investigación.
Las estadísticas descriptivas para VGE obtenidas con modelos que incluyen o excluyen GPF se muestran en el Cuadro 4. En un modelo animal básico, se asume la existencia de un solo grupo genético17. Dado que los valores genéticos son desviaciones de la media del grupo genético, todos los valores en la población base tienen una esperanza de cero5,17. La metodología de grupos genéticos permite asignar efectos genéticos a múltiples grupos dentro de la población base, que podrían tener una media diferente5. El VGE que incluye grupos genéticos considera que cada individuo hereda la media de los efectos en el grupo genético de sus padres más la media del valor genético de sus padres; por lo tanto, la esperanza del VGE para la población no es cero5,17 porque no se cumple con el supuesto de distribución de los valores genéticos. Asignar padres desconocidos a GPF con un promedio posiblemente distinto de cero de mérito genético cambiaría el VGE de sus descendientes. En consecuencia, la media esperada del VGE obtenido tras considerar los grupos genéticos cambia para el producto Qg3.
Cuadro 4 Estadísticas descriptivas de los valores genéticos estimados para los rasgos de crecimiento obtenidos con 12 (G12), 24 (G24) y sin (G0) grupos de padres fantasmas para el ganado Braunvieh mexicano
| Estrategia | Rasgo | Mínimo | Media ± DE | Máximo |
|---|---|---|---|---|
| G0 | PN | -5.36 | 0.03 ± 0.79 | 5.13 |
| PD | -26.67 | -0.17 ± 3.87 | 25.32 | |
| PA | -24.06 | 0.32 ± 3.39 | 24.34 | |
| G12 | PN | -5.24 | 0.31 ± 0.83 | 5.37 |
| PD | -18.73 | 14.19 ± 5.20 | 39.77 | |
| PA | -50.60 | -7.77 ± 4.81 | 18.08 | |
| G24 | PN | -3.67 | 2.06 ± 0.88 | 7.57 |
| PD | -53.15 | -10.75 ± 8.17 | 32.23 | |
| PA | -49.13 | 31.10 ± 7.27 | 82.12 |
PN= Peso al nacimiento, PD= Peso al destete, PA= Peso al año.
Las correlaciones producto-momento de Pearson y de rango de Spearman entre el VGE con y sin GPF se muestran en el Cuadro 5. Los coeficientes de correlación entre el VGE sin GPF (G0) y G12 fueron superiores a los de G0 y G24, para todos los rasgos y grupos de animales (es decir, machos y hembras, con y sin fenotipo). Las correlaciones fueron menores para los animales sin fenotipo que con fenotipo, y más bajas para las hembras que para los machos. En general, las correlaciones fueron más altas para PN que para PD, y más altas para PD que para PA (Cuadro 5).
Cuadro 5 Coeficientes de correlación de Pearson (y Spearman) entre los valores genéticos estimados sin grupos de padres fantasmas (VGE_G0), con 12 grupos de padres fantasmas (VGE_G12) y 24 grupos padres fantasmas (VGE_G24), en la población Braunvieh mexicana
| Tipo de correlación | Rasgo | ||
|---|---|---|---|
| Peso al nacimiento |
Peso al destete | Peso al año | |
| Total de animales | n =57,341 | n = 57,341 | n = 57,341 |
| r(VGE_G0, VGE_G12) | 0.959 (0.942) | 0.766 (0.766) | 0.692 (0.717) |
| r(VGE_G0, VGE_G24) | 0.912 (0.891) | 0.538 (0.610) | 0.535 (0.605) |
| Machos con fenotipo | n = 15,810 | n = 10,748 | n = 7,384 |
| r(VGE_G0, VGE_G12) | 0.988 (0.982) | 0.914 (0.886) | 0.853 (0.846) |
| r(VGE_G0, VGE_G24) | 0.975 (0.964) | 0.786 (0.763) | 0.743 (0.737) |
| Machos sin fenotipo | n = 2,879 | n = 7,941 | n = 11,305 |
| r(VGE_G0, VGE_G12) | 0.941 (0.923) | 0.796 (0.797) | 0.719 (0.760) |
| r(VGE_G0, VGE_G24) | 0.861 (0.830) | 0.606 (0.627) | 0.587 (0.636) |
| Hembras con fenotipo | n = 15,844 | n = 10,585 | n = 7,055 |
| r(VGE_G0, VGE_G12) | 0.986 (0.979) | 0.901 (0.879) | 0.844 (0.840) |
| r(VGE_G0, VGE_G24) | 0.972 (0.960) | 0.752 (0.749) | 0.710 (0.743) |
| Hembras sin fenotipo | n = 22,808 | n = 28,067 | n = 31,597 |
| r(VGE_G0, VGE_G12) | 0.895 (0.877) | 0.635 (0.659) | 0.596 (0.634) |
| r(VGE_G0, VGE_G24) | 0.799 (0.781) | 0.407 (0.491) | 0.445 (0.515) |
Se ha propuesto que los coeficientes de correlación entre el VGE inferiores a 0.90 podrían cambiar la jerarquización de los animales para la evaluación genética18. Las estimaciones de los coeficientes de correlación obtenidos aquí sugieren posibles cambios en la jerarquización principalmente para PD y PA. Petrini et al8 también señalaron cambios en el orden para PD debido a la inclusión de GPF (las estimaciones de correlación de Pearson y Spearman variaron de 0.50 a 0.70). Por otro lado, la inclusión de GPF resultó en pequeños cambios en la jerarquización de PN en este estudio. Los resultados para PN concuerdan con lo observado para la producción de leche7, PA y la ganancia de peso posdestete16, la circunferencia escrotal o la puntuación de musculatura8. Los cambios de orden se deben a los cambios que los grupos genéticos hacen en el VGE de sus descendientes.
La Figura 1 ilustra el efecto de GPF en las tendencias genéticas. Las tendencias fueron relativamente similares para machos y hembras. Para PN, G12 aumentó la pendiente de la tendencia genética, en comparación con G0; G24 también aumentó la pendiente de la tendencia genética, pero mostró una desviación base de G0. Para PD, tanto G12 como G24 mostraron grandes fluctuaciones en los primeros años. Las tendencias genéticas de G12 y G24 mostraron diferencias base positivas y negativas con G0, respectivamente (Figura 1). Para PA, la tendencia genética de G24 mostró una gran desviación base de las tendencias genéticas de G0 y G12 (Figura 1). Generalmente, si una tendencia genética no pasa de cero, indica un problema base para los VGE. Por lo tanto, G24 está descartado para PN y PA, y G12 está descartado para PD, ya que G24 no cruza cero para PN y PA, y G12 no cruza cero para PD (Figura 1). Por otro lado, se espera que una estrategia de agrupación robusta funcione bien para diferentes rasgos16, ya que una estrategia de agrupación de rasgos específica sería una complejidad adicional para las evaluaciones genéticas de rutina.

Figura 1 Tendencias genéticas de los rasgos de crecimiento por MPLI (VGE (línea sólida)), MPLI con 12 grupos de padres fantasmas (VGE_G12 (línea discontinua)) y MPLI con 24 grupos de padres fantasmas (VGE_G24 (línea punteada)), con seis clases de años de nacimiento consideradas en los grupos de padres fantasmas
Sin embargo, en la práctica, una estrategia de agrupación puede funcionar bien para un rasgo, pero no para otro rasgo, especialmente si los efectos fijos son diferentes para los dos rasgos8. Es probable que los posibles problemas con la implementación de GPF se deban a la colinealidad o confusión entre GPF y efectos fijos3,5. Una solución a este problema es considerar a los GPF como efectos aleatorios.
El efecto de la inclusión de GPF en la tendencia genética ha sido variable. Theron et al7 mostraron que la inclusión de GPF en la evaluación genética tuvo un efecto drástico en la tendencia genética de los rasgos de producción de leche, teniendo una respuesta más alta (casi el doble) cuando se incluyó GPF. Además, Shiotsuki et al16 observaron tendencias genéticas más altas para el peso posdestete y PA cuando el modelo incluyó GPF. En contraste, la inclusión de GPF en los análisis genéticos para PD, circunferencia escrotal y puntuación de musculación mostró una tendencia genética más baja que cuando no se incluyeron GPF8. Se podría inferir que la efectividad de los GPF en las evaluaciones genéticas depende de la estructura de la población, los rasgos estudiados y los criterios adoptados para definir los GPF. Se ha propuesto que la definición de los GPF debe equilibrar la complejidad de los grupos genéticos y la representación de las diferencias genéticas8. Además, los grupos genéticos deben considerar los criterios de selección adoptados por los criadores.
Se consideran dos posibles razones de los problemas observados con las tendencias genéticas: la escasez de fenotipos de la progenie que apoyan la inferencia de algunos GPF, y la posible confusión o colinealidad entre GPF y los efectos fijos en el modelo, especialmente los grupos contemporáneos. La Figura 2 muestra la frecuencia de animales, padres y madres faltantes a lo largo de los años de nacimiento, y la Figura 3 muestra la frecuencia de fenotipos por año de nacimiento. Se puede interpretar que no hubo suficientes fenotipos que respaldaran la predicción de soluciones de GPF en los primeros años (padres desconocidos nacidos antes de 1990, es decir, su progenie nacida antes de 1995). La contribución de los fenotipos disminuye a medida que aumenta el número de generaciones entre el GPF y el descendiente fenotipado, y disminuye con una menor heredabilidad.
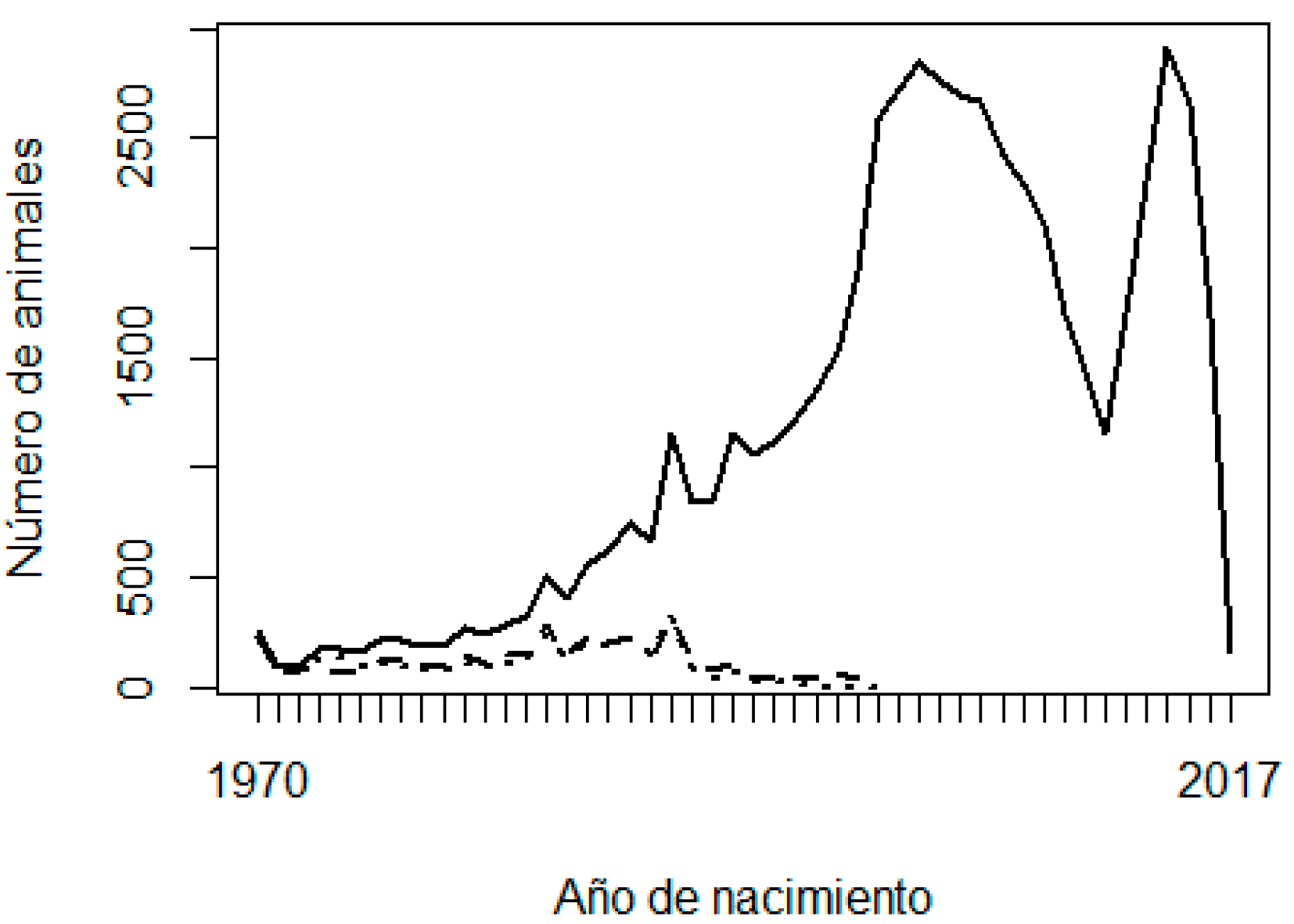
Figura 2 Número de animales (línea sólida), padres desconocidos (línea discontinua) y madres desconocidas (línea punteada) por año de nacimiento
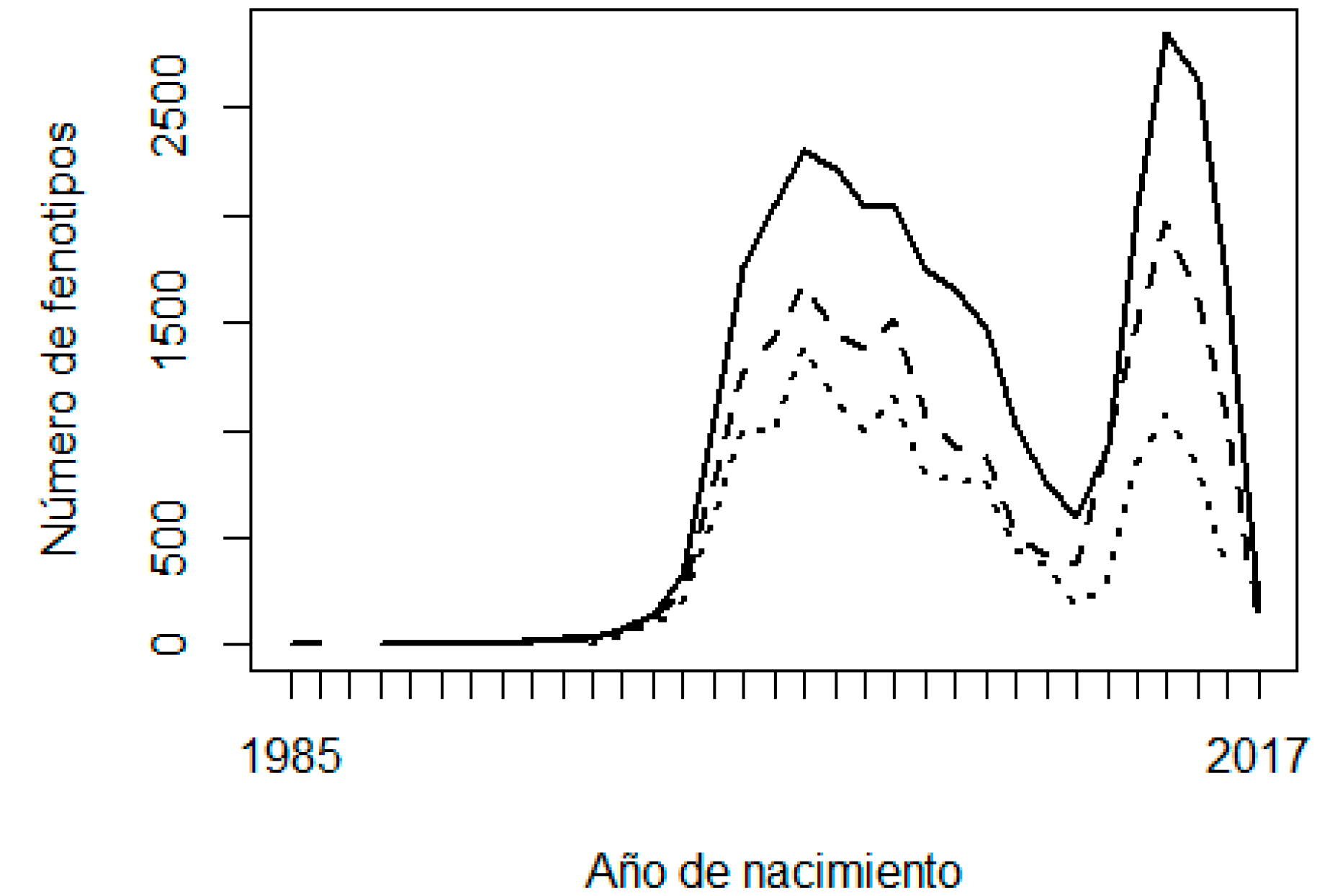
Figura 3 Número de fenotipos de peso de nacimiento (línea sólida), peso al destete (línea discontinua) y peso al año (línea punteada) por año de nacimiento
La Figura 2 muestra que la población estudiada no tenía una necesidad real de GPF en el modelo animal, o que los GPF podrían limitarse a solo unos pocos grupos, de modo que los fenotipos de generaciones distantes de descendientes podrían respaldar la estimación de esos pocos GPF. Los modelos animales con GPF son más benéficos para las poblaciones con una prevalencia más alta y amplia de información de pedigrí faltante, especialmente si diferentes antecedentes genéticos (por ejemplo, materiales genéticos importados) o diferentes estrategias/presión de selección están involucrados en diferentes grupos de animales (por ejemplo, machos vs. hembras o diferentes vías de selección). Las tendencias genéticas (Figura 1) muestran que la población no ha estado bajo una selección eficiente, y existe una oportunidad excelente para el mejoramiento genético hacia la producción sostenible en los sistemas y ambientes de producción mexicanos. Las Figuras 2 y 3 también muestran problemas de recopilación de datos entre los años 2003 y 2014, y entre 2014 y 2017. La integridad y exactitud de los datos son esenciales para evaluaciones genéticas precisas y confiables.
Como se mencionó en la subsección Datos, se obtuvo el pedigrí (parentesco) a partir de animales fenotipados. El número de animales y padres faltantes (Figura 2) fue mayor sin esta restricción. Sin embargo, en ese caso, hubo padres faltantes adicionales sin contribución del comportamiento de la progenie; por lo tanto, no hay información para hacer inferencias sobre ellos. Se recomienda extraer el pedigrí de animales fenotipados, decidir sobre la formación de grupos genéticos, luego agregar animales que no habían sido extraídos y asignar a sus padres desconocidos a los GPF existentes.
Idealmente, debería haber una conectividad de grupo fija entre diferentes GPF (es decir, como el concepto de conectividad genética entre grupos fijos (niveles de un efecto fijo)). En otras palabras, los fenotipos de diferentes grupos de efectos fijos deben aportar información a diferentes GPF. Incluso se ha recomendado formar (algunos) GPF compuestos tanto de padres y madres8. El paquete "ggroups" de R15 permite formar GPF de ambos sexos. Definiciones similares de GPF y algunos efectos fijos pueden causar colinealidad. En esa situación, el número de grupos de efectos fijos que aportan información a cada GPF disminuye. Una forma de comprobar la colinealidad dentro y entre los efectos fijos y GPF es comprobar el eigenvalor mínimo de [X
Q
p]ʹ[X
Q
p], donde Q
p es Q con filas limitadas a animales fenotipados. Los problemas de estimación de GPF en el modelo no se limitan a este estudio. Estos problemas se observan a menudo debido a efectos de confusión (colinealidad) entre GPF19. Incluso, al reducir tal confusión cambiando la composición de GPF, podría haber confusión entre GPF y otros efectos fijos. Esos problemas de estimabilidad se eliminaron y la estimación de valores genéticos parecen normales al considerar GPF como efectos aleatorios mediante la adición de
Conclusiones e implicaciones
Se probaron dos estrategias para agrupar a padres desconocidos en GPF (G12 y G24) en PN, PD y PA en ganado Braunvieh mexicano. Las dos estrategias utilizaron los criterios más comunes para definir los GPF (año de nacimiento de la progenie, sexo del padre desconocido para G12 y vía de selección para G24). Las tendencias genéticas tuvieron una desviación compensada de la tendencia genética de la MPLI sin GPF, a excepción de PN del G12. Además, la inclusión de GPF en el modelo puede haber causado colinealidad entre GPF y algunos efectos fijos. La escasez de fenotipos que respaldan las soluciones para algunos efectos de GPF fue otra razón para la falta de beneficio de las dos estrategias de agrupación en la población y los rasgos estudiados. Se recomienda definir los GPF con base en un subconjunto de pedigrí, en el que los padres están conectados a descendientes fenotipados, luego agregar el resto de animales y asignar a sus padres desconocidos a los GPF existentes, para evitar un número excesivo e innecesario de grupos genéticos. Más importante que el número de progenie por GPF o intervalos de años iguales que definen los GPF, es la cantidad de contribuciones fenotípicas para predecir los efectos de los GPF. Se recomienda tener menos traslape entre las definiciones de los GPF y los efectos fijos para reducir la colinealidad entre ellos.
Agradecimientos
Los autores agradecen al Consejo Nacional de Ciencia y Tecnología (CONACyT, México) por el apoyo financiero al primer autor durante sus estudios de doctorado. Los autores también agradecen a la Asociación Mexicana de Criadores de Ganado Suizo de Registro por permitir el uso de sus datos, y al Dr. Dale Van Vleck por brindar su ayuda y apoyo con el software MTDFREML.
REFERENCIAS
1. AMCGSR. Asociación Mexicana de Criadores de Ganado Suizo de Registro. Resumen de evaluaciones genéticas para ganado Suizo Americano 2017. Techn Bull. Mexico. 2018. [ Links ]
2. Van Vleck LD. 1970. Miss-identification in estimating the paternal sib correlation. J Dairy Sci 1970;53:1469-1474. [ Links ]
3. Quaas RL. Additive genetic model with groups and relationships. 1988;71:1338-1345. [ Links ]
4. Westell RA, Quass RL, Van Vleck LD. Genetic groups in an Animal Model. J Dairy Sci 1988;71:1310-1318. [ Links ]
5. Wolak ME, Reid JM. Accounting for genetic differences among unknown parents in microevolutionary studies: How to include genetic groups in quantitative genetics animal models. J Anim Ecol 2017;86:7-20. [ Links ]
6. Fikse F. Fuzzy classification of phantom parent group in an animal model. Genet Sel Evol 2009;41:1-8. [ Links ]
7. Theron HE, Kanfer FHJ, Rautenbach L. The effect of phantom parent groups on genetic trend estimation. South African J Anim Sci 2002;32:130-135. [ Links ]
8. Petrini J, Pertile SF, Eler JP, Ferraz JB, Mattos EC, Figuereido LG, et al. Genetic grouping strategies in selection efficiency of composite beef cattle (Bos taurus x Bos indicus). J Anim Sci 2015;93:541-552. [ Links ]
9. Pollak EJ, Quass RL. Definition of group effect in sire evaluation models. 1983;66:1503-1509. [ Links ]
10. Oliveira Junior GA, Eler JP, Ferraz JBS, Petrini J, Mattos EC, Mourão GB. Definição de grupos genéticos aditivos visando melhor predição de valores genéticos em bovinos de corte. Rev Bras Saúde Prod Anim 2013;14:277-286. [ Links ]
11. BIF. Beef Improvement Federation. Guidelines for Uniform Beef Improvement Programs. 9th ed. North Carolina State University, Raleigh, NC, USA. 2018. [ Links ]
12. Henderson CR. Best linear unbiased and prediction under a selection model. Biometrics 1975;31:423-447. [ Links ]
13. Boldman KG, Kriese LA, Van Vleck LD, Van Tassell CP, Kachman SD. A manual for use of MTDFREML, a set of programs to obtain estimates of variances and (co)variances. Washington, DC: USDA, ARS. 1995. [ Links ]
14. Quass RL, Pollack EJ. Modified equations for sire models with groups. J Dairy Sci 1981;64:1868-1872. [ Links ]
15. Nilforooshan MA, Saavedra-Jiménez LA. ggroups: a R package for pedigree and genetic groups data. Hereditas 2020;157:17. [ Links ]
16. Shiotsuki L, Cardoso FF, Silva JAIIV, Albuquerque LG. Comparison of a genetic group and unknown paternity models for growth traits in Nellore cattle. J Anim Sci 2013;91:5135-5143. [ Links ]
17. Van Vleck LD. Breeding value prediction with maternal genetic groups. J Anim Sci 1990;68:3998-4013. [ Links ]
18. Crews DH, Franke DE. Heterogeneity of variances for carcass traits by percentage Brahman inheritance. J Anim Sci 1998;73:1803-1908. [ Links ]
19. Schaeffer LR. Necessary changes to improve animal models. J Anim Breed Genet 2018;135:124-131. [ Links ]
Recibido: 27 de Noviembre de 2019; Aprobado: 23 de Noviembre de 2020











 texto em
texto em 


