La anaplasmosis es una enfermedad reconocida por su impacto mundial en el área de la salud y el sector pecuario1,2. La enfermedad es causada por bacterias del género Anaplasma, que en ganado bovino se presenta como una enfermedad debilitante que en ciertos casos puede llegar a ser mortal. La presencia de anaplasmosis puede producir pérdidas económicas en el sector ganadero por disminución de la producción lechera, retraso en el crecimiento y baja en la ganancia de peso de los animales3,4. Es importante recalcar que el verdadero impacto económico de la anaplasmosis bovina no es posible de determinar debido a la falta de datos epidemiológicos en países latinoamericanos5. El manejo de la enfermedad se vuelve complejo por la existencia de portadores asintomáticos del patógeno, los cuales actúan como reservorios, contribuyen a la propagación e incrementan el riesgo de contagio para las poblaciones ganaderas susceptibles a la enfermedad5.
Para la detección de Anaplasma spp., la reacción en cadena de la polimerasa (PCR) muestra mayor especificidad y sensibilidad en comparación con métodos convencionales como el cultivo, la serología y microscopía óptica6,7. Además, la probabilidad de que ocurra detección cruzada de otros hemoparásitos al utilizar PCR es mínima6. Un paso previo e importante para realizar la detección de un patógeno por PCR es la extracción del ADN a partir de muestras clínicas, es por esto que la eficiencia de la extracción de ADN es primordial para obtener resultados confiables y repetibles. Idealmente, para detectar Anaplasma spp. por PCR, se utiliza una muestra de sangre completa, esto es debido a la capacidad que tiene este género para parasitar diferentes células sanguíneas7. Sin embargo, diversos elementos de la sangre o fallas en el muestreo, transporte o conservación de las muestras pueden dar lugar a cambios que dificulten la extracción del material genético o inhiban la reacción de PCR8,9. Es así que en muchas ocasiones se puede observar alteraciones en la homogeneidad de las muestras, así como también la aparición de hemólisis y de coágulos, lo que dificulta la lisis celular y liberación del material genético durante su extracción. Así mismo, la sangre hemolizada contiene una mayor concentración de hemoglobina y sus derivados, lo que puede comprometer la eficacia diagnóstica de la PCR. Por esta razón, la sangre hemolizada y coagulada es generalmente descartada para las pruebas de detección molecular.
Actualmente, existen diversos kits de extracción de ADN de diferentes casas comerciales que han sido diseñados para obtener ADN de alta calidad, pero de baja concentración10, a partir de muestras complejas o de mala calidad como la sangre coagulada y hemolizada11. Sin embargo, su elevado costo limita su uso en investigaciones de bajo presupuesto, situación muy frecuente en países en vías de desarrollo. El método de extracción de ADN con resina de Chelex® 100 es sencillo y barato de ejecutar, por lo tanto, es utilizado en una gran variedad de tejidos12,13. Este método empieza con un tratamiento térmico o si es necesario la maceración del tejido, con el objetivo de lisar las células y liberar el ADN. Luego, la resina de Chelex® 100 actúa como quelante al capturar los iones de magnesio y como consecuencia evita la degradación del ADN debido a la acción de las nucleasas12. El protocolo con resina de Chelex® 100 presentado por Singh et al14 se destaca por obtener eficientemente ADN de alta cantidad y pureza. Este protocolo está diseñado para sangre seca en papel filtro, su uso no ha sido descrito para otro tipo de muestras. Es así como el presente estudio busca optimizar dicho protocolo de extracción de ADN a partir de muestras de sangre bovina hemolizada y coagulada, con el fin de que sea utilizado para la detección molecular de patógenos en animales, específicamente de Anaplasma spp. en bovinos.
Se utilizaron 40 muestras de sangre que fueron colectadas en septiembre de 2018 dentro de un proyecto más amplio denominado “Planes de Finca”15) que es impulsado por el Gobierno Autónomo Descentralizado de la Provincia de Esmeraldas (GADPE) y el Instituto Interamericano de Cooperación para la Agricultura (IICA), con la participación de la Universidad San Francisco de Quito. Las muestras provienen de bovinos mestizos Bos taurus, con al menos 50 garrapatas en su cuerpo, a los que se les extrajo 5 ml de sangre de vena coccígea o yugular en tubos con anticoagulante. Las muestras se mantuvieron en refrigeración a 4-5 °C hasta llegar al laboratorio del Hospital Veterinario Docente de la Universidad San Francisco de Quito, en donde se congelaron hasta su procesamiento.
Al descongelar las muestras, se confirmó que éstas estaban hemolizadas y con coágulos de pequeño o mediano tamaño. Se realizó la extracción de ADN en un subgrupo de 10 muestras con estas características y a las que previamente se les había confirmado positividad para Anaplasma sp. Para el análisis se utilizó el protocolo diseñado por Singh14. Debido a que este protocolo de extracción de ADN fue diseñado para muestras de sangre seca en papel filtro, se realizó un ensayo con diferentes volúmenes de sangre (5 µl, 50 µl y 100 µl). Un resumen del protocolo de Singh et al14 se puede encontrar en el Cuadro 1. Adicionalmente, para mejorar la pureza del ADN obtenido con el volumen de muestra definido en el procedimiento anterior, se adicionaron pasos al protocolo, específicamente a la fase de precipitación de proteínas. Dichos pasos se detallan a continuación: a) añadir alcohol al 75 % en proporción 1:1 con el volumen de sobrenadante recuperado en el paso 10; b) dejar reposar durante una noche a -20 oC por aproximadamente 16 h; c) centrifugar la mezcla a 13,000 rpm por 1.5 min; d) recuperar el sobrenadante; e) realizar el paso 11 según el Cuadro 1
Cuadro 1 Protocolo de extracción de ADN según Singh et al14
|
Extracción de ADN 1. Calentar a 100 oC, 300 μl de solución stock de resina de Chelex® 100 al 7% por 10 min. 2. Añadir 5 μl de sangre y calentar la mezcla por 8 min a 100 oC. 3. Mezclar con la ayuda de un vórtex por 15 s y recalentar por 7 min a 100 oC. 4. Centrifugar por 1.5 min a 12,000 rpm. 5. Recuperar el sobrenadante y descartar el pellet. Precipitación de Proteínas 6. Añadir solución stock de acetato de amonio (7.5 M) al sobrenadante recuperado previamente de forma que la solución final tenga una concentración de 2.5 M 7. Reposar la mezcla por 5 min en hielo. 8. Mezclar con la ayuda de un vórtex por 10 seg. 9. Centrifugar por 13,000 rpm por 10 min a - 4 oC. 10. Recuperar el sobrenadante en un tubo nuevo. Precipitación de ADN 11. Añadir solución stock de acetato de sodio (3 M) al sobrenadante recuperado de forma que la solución final tenga una concentración de 0.3 M. 12. Añadir 200 μl de alcohol absoluto. 13. Mezclar con la ayuda de un vórtex por 5 seg. 14. Reposar la mezcla por 4 h a -20 oC. 15. Centrifugar a 13,000 rpm por 10 min a -4 oC. Descartar sobrenadante. 16. Lavar el pellet dos veces: la primera vez con 200 μl alcohol 75 % helado y la segunda vez con 200 μl alcohol 100 % helado. Cada lavado fue seguido por una centrifugación a 13,000 rpm por 10 min a -4 oC y el descarte del sobrenadante. 17. Dejar secar el pellet al aire por 10 min. 18. Resuspender en 100 μl de 1X AE 19. Incubar por 10 min a 55 oC. |
Se comparó la cantidad y pureza del ADN entre el protocolo original y el que contenía modificaciones en la fase de precipitación de proteínas. La concentración y calidad del ADN de las muestras se midieron por espectrofotometría en un NanoVue Plus Spectrophotometer (GE Healthcare, USA). El análisis molecular de este estudio fue realizado bajo el Contrato Marco de Acceso a los Recursos Genéticos Nro. MAE-DNB-CM-2018-0106.
Para demostrar la eficiencia de las modificaciones realizadas al protocolo de Singh14, se extrajo ADN de 30 muestras de sangre hemolizada y coagulada de las que se desconocía su positividad para Anaplasma spp. La extracción de ADN fue realizada utilizando el protocolo de Singh modificado en las secciones anteriores y al que en adelante se le llamará Protocolo de Chelex Modificado (PCM). El ADN de las mismas muestras fue extraído con los kits comerciales DNeasy Blood & Tissue Kits Print Qiagen® y AccuPrep® Genomic DNA Extraction Kit Bioneer®, siguiendo las recomendaciones del fabricante para extracción de ADN a partir de sangre completa. Para confirmar la utilidad del PCM en la detección molecular de Anaplasma spp. se realizó una reacción de PCR específica para este género bacteriano detallado por Zobba et al16. Los amplicones obtenidos a partir de 13 muestras positivas (n= 40) fueron secuenciados en Functional Biosciences, Inc. (Wisconsin-USA) para confirmar la presencia de ADN de Anaplasma sp. También, se utilizó el gen constitutivo que codifica para la proteína β-Actina, siempre presente en las muestras de sangre bovina17) para descartar la posibilidad de obtener falsos negativos debido a la inhibición de la reacción de PCR. El detalle de los cebadores y el tamaño del producto de ambas reacciones de PCR están detallados en el Cuadro 2.
Cuadro 2 Secuencias de cebadores utilizados en este estudio
| Gen | Cebador | Secuencia: 5’ - 3’ | Tamaño | Ref. |
|---|---|---|---|---|
| 16S rDNA | AnaplsppF | AGAAGAAGTCCCGGCAAACT | 800 bp | (18) |
| AnaplR3 | GAGACGACTTTTACGGATTAGCTC | |||
| β-actina | XAHR 17 | CGGAACCGCTCATTGCC | 289 bp | (19) |
| XAHR 20 | ACCCACACTGTGCCCATCTA |
Todas las pruebas estadísticas se realizaron con un nivel de significancia del 95 % con el software R v.3.3.018. Se realizó la prueba de normalidad de Shapiro-Wilks y pruebas no paramétricas para el análisis de los datos del estudio. Para determinar las diferencias en la cantidad de ADN obtenida entre los volúmenes de sangre (5 µl, 50 µl y 100 µl), se utilizó la prueba de Friedman y análisis post-hoc de Wilcoxon; los valores de P fueron ajustados con la corrección de Bonferroni19. La pureza de ADN, de los ensayos de precipitación de proteínas, se comparó con la prueba no paramétrica Wilcoxon. La concordancia de los resultados de PCR entre los tres métodos de extracción fue determinada con la prueba Kappa de Cohen y la prueba McNemar20,21.
El protocolo de Singh14, se utilizó en 10 muestras de sangre hemolizada y coagulada. Para definir el volumen de muestra se probaron 5 µl, 50 µl y 100 µl de sangre líquida de cada muestra. Los resultados mostraron que la concentración de ADN difiere entre los volúmenes de sangre utilizados (Test de Friedman: χ2(2) = 16.800, P=0.017) (Figura 1), excepto entre 50 µl y 100 µl de muestra (Wilcoxon: Z= -2,293, P= 0.063). Por lo que, para los siguientes pasos, se utilizó 50 µl de muestra, con la intención de utilizar el volumen más bajo.
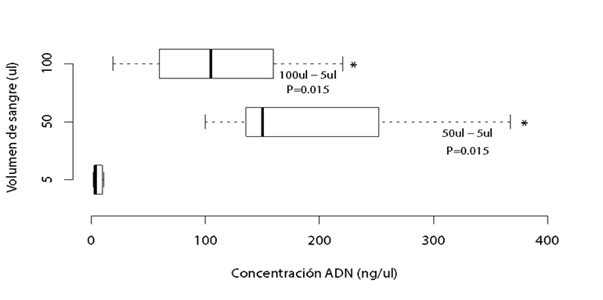
Se utilizó el método de extracción de ADN publicado por Singh et al14 (χ2(2) = 16,800; P=0.017*).
Figura 1 Concentración de ADN (ng/µl) obtenida a partir de diferentes volúmenes de sangre (n=10)
La pureza del ADN extraído con el protocolo publicado por Singh et al14, a partir de las muestras de sangre coagulada y hemolizada, arrojó un radio mediano entre A260/280 de 0,820 (n=10). Al modificar la fase de precipitación de proteínas en el protocolo PCM se logró incrementar el radio mediano entre A260/280 a 1,970 (n=10). En comparación al protocolo original, el PCM permite obtener un incremento significativo en los rangos 260/280 (Wilcoxon: Z= -2,803, P=0.005) y 260/230 (Wilcoxon: Z= -2,666, P=0.002) respectivamente (Figura 2). Es importante mencionar que la concentración de ADN en estas muestras disminuyó significativamente (Wilcoxon: Z= -2,803, P=0.005) en relación a la obtenida con el protocolo original. Sin embargo, esto no interfirió en la detección molecular de Anaplasma spp. en ninguna de las muestras. Adicionalmente, se observó que las bandas inespecíficas, observadas al amplificar las muestras extraídas con el protocolo original, desaparecían al utilizar el PCM.
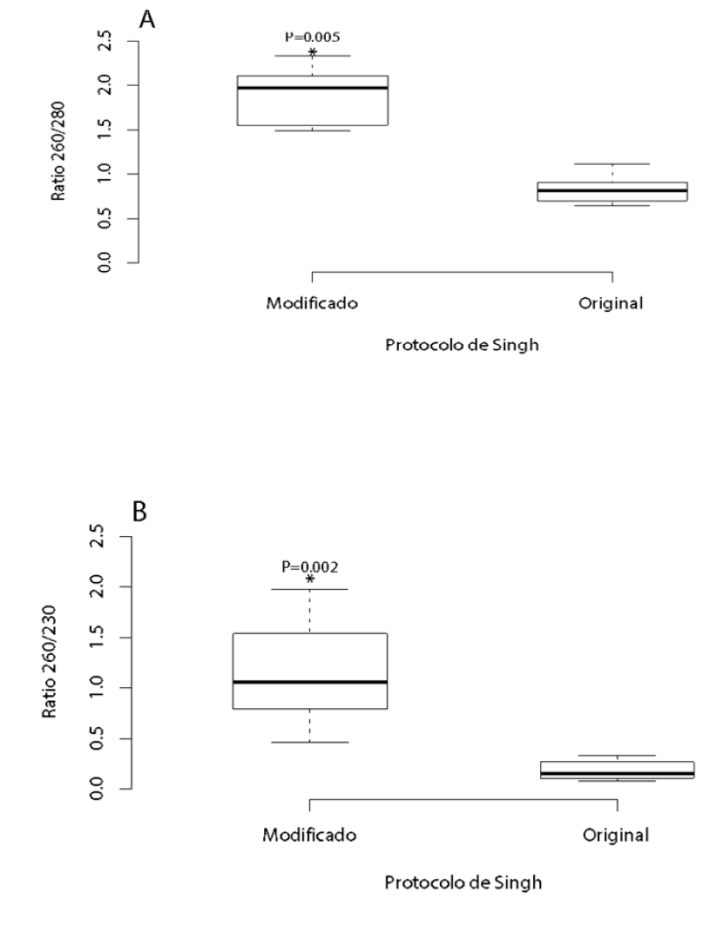
Figura 2 Comparación de A. pureza y B. cantidad de ADN obtenida con el protocolo de extracción de ADN de Singh et al14) con el PCM (n=10).
El ADN de 30 muestras de sangre coagulada y hemolizada fue extraído con dos kits comerciales y con PCM. No se observó inhibición de la reacción de PCR en las muestras de ADN obtenidas con los tres métodos de extracción. Además, se obtuvieron resultados equivalentes para la presencia de Anaplasma spp. (Cuadro 3) con índices de concordancia Kappa de Cohen de 0.72 entre el protocolo PCM y los dos kits comerciales. La prueba exacta de McNemar determinó que no existe diferencia significativa en la proporción de muestras positivas por PCR entre los kits y el protocolo PCM (McNemar's Ji-cuadrada 0.500; P=0.5)
Cuadro 3 Resultados de positividad de Anaplasma spp. en muestras de sangre extraída con diferentes métodos (n=30)
| Kit Qiagen | Kit Bioneer | ||||
|---|---|---|---|---|---|
| PCM | Positivos | Negativos | Positivos | Negativos | |
| Positivos | 25 | 0 | 25 | 0 | |
| Negativos | 2 | 3 | 2 | 3 | |
PMC= Protocolo de Chelex modificado.
La anaplasmosis bovina es una enfermedad que tiene impacto negativo sobre el sector pecuario1,2. Esta enfermedad es causada por especies del género Anaplasma y produce anemia, debilidad, reducción en el crecimiento y en la producción lechera, abortos e incluso puede producir la muerte en ganado bovino infectado22. Desafortunadamente, en países de América Latina y el Caribe se desconoce la verdadera prevalencia de anaplasmosis en ganado bovino. Esta situación se agrava debido a que el 80 % de los productores ganaderos de estos países son pequeños agricultores familiares que viven en zonas rurales y marginales23. En estas zonas, la vacunación y control de las enfermedades infecciosas se encuentran limitados a la disponibilidad de recursos económicos, que en la mayoría de casos son escasos24. Esto es relevante en países en vías de desarrollo, en el que los insumos de laboratorio pueden costar de 2 a 10 veces más que en países desarrollados25. Como consecuencia, es importante contar con técnicas que permitan detectar con precisión la presencia de Anaplasma spp. y que al mismo tiempo sean asequibles, tanto para las entidades de control como para los pequeños y grandes ganaderos.
La detección molecular de patógenos por PCR a partir de muestras clínicas se ha convertido en una metodología muy utilizada para el diagnóstico y monitoreo de enfermedades infecciosas26. Esta técnica también permite la posterior caracterización del patógeno mediante la secuenciación de su material genético, información que puede ser de gran utilidad para entender la epidemiología de una enfermedad. Un paso muy importante, previo a la detección molecular, es la extracción del material genético a partir de las muestras. Este procedimiento está estrechamente relacionado a la colección, transporte y almacenamiento de las muestras. Todos estos pasos deben asegurar que el material genético del patógeno permanezca intacto hasta que se lleve a cabo el procedimiento de extracción. Desafortunadamente, las condiciones no siempre son idóneas, por ejemplo, cuando se necesita obtener muestras de animales que viven en áreas rurales de difícil acceso, es complicado asegurar una óptima toma y conservación de la muestra. En dichas condiciones es muy frecuente que las muestras muestren hemolisis y coágulos, lo que dificulta su posterior análisis al inhibir la reacción de detección molecular del patógeno.
Los resultados demuestran que las modificaciones adicionadas al protocolo de extracción de ADN14 permiten obtener ADN, a partir de sangre hemolizada y coagulada, que puede ser utilizado para la detección molecular de Anaplasma spp. Al extraer ADN a partir de 50 µl de muestra con el PCM, se observó que los valores de pureza, en los radios 260/280 y 260/230, eran superiores en comparación a los resultados obtenidos con el protocolo publicado por Singh14. Una limitación del protocolo modificado es la baja cantidad de ADN que se obtiene en relación con los kits comerciales, sin embargo, no se encontraron diferencias significativas entre la positividad de Anaplasma spp. obtenida a partir del ADN extraído con PCM y los kits comerciales utilizados. Así mismo, el índice de concordancia Kappa de 0.72 indica que existe una alta concordancia más allá del azar entre los resultados obtenidos con los métodos de extracción20. Es así que la estandarización del volumen de sangre y la modificación al protocolo de referencia14, permitieron tener resultados equivalentes entre la extracción del material genético con PMC y los kits comerciales. En el futuro, sería interesante probar este método de extracción para la detección de otros patógenos que pueden encontrarse en la sangre de bovinos y de otros animales.
Adicionalmente, el protocolo modificado permite obtener ADN de Anaplasma spp. a partir de muestras de sangre bovina a un costo considerablemente menor al de los kits comerciales. Para ilustrar esto, se consideraron los valores de los reactivos a proveedores autorizados en el Ecuador. Es así que, para extraer ADN a partir de 250 muestras, tomando en cuenta todos los reactivos utilizados para el PCM, se requiere alrededor de 60 dólares americanos. En contraste con el costo de los kits comerciales utilizados en esta investigación, que llega a ser hasta 10 veces mayor: Bionner® y Qiagen® para 250 reacciones el costo es aproximadamente $600 y $2,000 respectivamente, sin contar con el precio del equipo automatizado necesario para utilizar el kit de Bionner®.
La mayoría de reactivos que se utilizan para realizar investigación en países como Ecuador, Colombia, México, entre otros, no son fabricados en Latinoamérica. Debido a esto, los valores de importación y de comisiones de los proveedores locales incrementan enormemente el costo de análisis de muestras. Lamentablemente, la falta de dinero es la mayor limitante para realizar estudios de investigación en nuestros países. Por lo tanto, es importante ser creativos y buscar métodos confiables que abaraten los costos de investigación y el monitoreo de enfermedades infecciosas. Solo así se podrá entender la epidemiología de estas enfermedades, y como consecuencia aplicar planes control/monitoreo efectivos, que beneficien a la producción animal y a las comunidades humanas expuestas.
Esta investigación adapta un protocolo de bajo costo que utiliza resina de Chelex® 100 para la extracción de ADN a partir de sangre hemolizada y coagulada. Este protocolo permite obtener ADN a partir del cual se puede realizar la detección molecular de Anaplasma spp. por PCR con resultados equivalentes a los obtenidos con kits comerciales. El protocolo propuesto es ideal para realizar monitoreo de Anaplasma spp. en bovinos con un presupuesto limitado. Es importante generar protocolos alternativos de bajo costo para la detección y análisis molecular de patógenos, que permitirán realizar investigación con menores recursos, principalmente en países en vías de desarrollo. De esta manera se podrán generar resultados actualizados para que las autoridades del sector ganadero y de salud pública actúen de manera eficaz e informada. Tomando en cuenta las ventajas económicas que presenta el Protocolo de Chelex Modificado (PCM), sugerimos comprobar su utilidad para la detección de otros patógenos y su eficiencia de extracción en otros tipos de muestra.











 texto en
texto en 


