En la estación experimental ‘La Cumaca’ de la Facultad de Ciencias Veterinarias de la Universidad Central de Venezuela, se lleva a cabo un programa general de mejoramiento genético para características productivas de la raza Brahman, representando una importante fuente de genes para esta población1.
En la metodología aplicada para la realización de evaluaciones genéticas existen diferentes elementos críticos. En primer lugar, el PA548, con base en los cuales se estiman los valores genéticos (VG) de los animales puede estar sesgado, ya que se asume que el crecimiento es lineal. Resultados publicados2,3,4,5 en animales B. indicus, demuestran que existen variaciones en la forma de crecimiento a través de la trayectoria de las pruebas de comportamiento. En segundo lugar, generalmente el sexo del animal se considera como efecto fijo en el modelo, lo cual asume implícitamente que los componentes de (co)varianza son los mismos en ambos sexos. Este enfoque puede incorporar otra fuente de sesgo sobre las estimaciones de los VG, disminuyendo su precisión y por consiguiente, afectándose la marcha del programa de mejora6,7,8. Los elementos expuestos, pueden afectar las correlaciones genéticas del mismo rasgo entre ambos sexos (rHM), pudiéndose manifestar efectos de interacción genotipo-sexo.
El PV se ha expresado generalmente a un punto fijo, aunque parece muy lógico examinar estas relaciones a lo largo de la edad. Si la información está disponible, los componentes de (co)varianza pueden estimarse mediante modelos multivariados (MT) o, preferiblemente, por medio de regresión aleatoria (RA). Se han publicado resultados comparando modelos MT y RA para PV en ganado Bos indicus2,4, que demuestran las ventajas de RA. Sin embargo, en este enfoque longitudinal no se han examinado las relaciones entre los sexos de los animales. Por lo anterior, se requieren más evidencias, particularmente si se toma en cuenta posibles relaciones entre PV y comportamiento reproductivo (CR) de las hembras.
Existe consenso sobre la importancia del CR para la economía del rebaño de carne. Sin embargo, la aparente baja h2 de la mayor parte de los rasgos reproductivos9,10,11 ha sido un factor limitante para su utilización como criterio de selección directa. Como alternativa, se han publicado resultados sobre la circunferencia escrotal (CE) o el PV medido en los machos jóvenes, y su respuesta correlacionada con el CR de las hembras medido por servicios por gestación; días al parto y TG al primer servicio. Aun cuando los resultados son alentadores12,13, estos se han expresado a una edad fija, manteniéndose la interrogante de la evolución de estas tendencias a lo largo de la edad hasta el primer parto.
Los objetivos de este estudio fueron: estimar las heredabilidades y las correlaciones genéticas entre el PA548 y la TG de las novillas en primera temporada de reproducción, mediante un MT; estimar los componentes de (co)varianza genética del PV de ambos sexos a lo largo de la edad mediante RA; comparar los valores genéticos para cada ith edad (VGi), estimados según RA, con los VG según MT.
La Estación Experimental La Cumaca, ubicada a 472 msnm cerca de la ciudad de San Felipe, estado Yaracuy, Venezuela, tiene una extensión de 433 ha, de las cuales 300 ha son cultivadas con pastos guinea, estrella, swazzi, pará y alambre. La precipitación anual promedio es de 1,650 mm con una temperatura promedio de 24 a 31.9 ºC y una humedad relativa promedio de 84 %14. Cuenta con un rebaño de ganado Brahman puro, registrado, de alrededor de 180 vacas en producción.
El peso vivo ajustado a 548 días de edad (PA548) de 3,120 animales nacidos entre febrero del 2000 y junio del 2011 se editó eliminando aquellos registros con incoherencias en el pedigrí, ausencia o problemas en la fecha de nacimiento. Finalmente, quedaron disponibles los registros de 2,777 animales (1,377 hembras y 1,400 machos). Estos animales eran hijos de 984 madres (729 en el vector de datos) y 107 sementales (48 en el vector de datos). El archivo de pedigrí incluyó 3,977 animales. Un total de 94,752 registros de PV individual, provenientes de 1,776 hembras y 1,864 machos, nacidos entre febrero del 1978 y junio del 2011 fueron utilizados. Estos animales eran hijos de 1,291 madres (929 en el vector de datos) y 128 sementales (58 en el vector de datos), y el archivo de pedigrí contó con 4,070 animales. Estos datos fueron editados, eliminado aquellos registros con incoherencias en el pedigrí, ausencia o problemas en la fecha de nacimiento y datos registrados a menos de 30 o más de 570 días de edad. Se eliminaron datos fuera del rango de ±3.2 desviaciones estándar dentro de clases de edades de 30 días de rango. Finalmente, quedaron disponibles un total de 53,258 datos individuales de 1,737 hembras y 1,803 machos.
Se corrieron varios modelos utilizando el procedimiento GLM de SAS15. El Cuadro 1 presenta algunos indicadores de los datos estudiados.
Cuadro 1 Indicadores de peso vivo de animales Brahman en la estación experimental La Cumaca, Venezuela
| Característica | Tamaño de muestra en hembras |
Hembras | Tamaño de muestra en machos |
Machos |
|---|---|---|---|---|
| Peso al nacer | 1,776 | 29.5 ± 4.5 | 1864 | 31.9 ± 4.9 |
| Peso al destete | 1,639 | 165.8 ± 26.2 | 1630 | 177.3 ± 28.3 |
| Peso a 365 días | 1,396 | 209.9 ± 29.8 | 1410 | 231.8 ± 34.9 |
| Peso a 450 días | 1,378 | 235.6 ± 32.0 | 1392 | 271.2 ± 38.9 |
| Peso a 548 días | 1,340 | 290.0 ± 34.1 | 1385 | 326.3 ± 42.2 |
| Número de registros de peso vivo | 25,781 | 25781 | 27477 | 27477 |
| Tasa de Gestación | 1,377 | 0.67 ± 0.37 |
Tres bloques de análisis fueron realizados utilizando el programa Asreml316:
Bloque 0. Modelo multivariado (MU) para PA548 y TG.
Donde:
yies un vector correspondiente a PA548 y TG analizados al mismo tiempo;
bies un vector de efectos fijos de la jk th combinación del sexo-año-mes (con 275 niveles para PA548 y 124 para TG).
aies un vector aleatorio correlacionado debido al efecto genético aditivo del i th animal con datos y sus antecesores sin registros (4,070 niveles) para PA548 y TG;
eiles un vector residual aleatorio correlacionado entre los rasgos 1 y 2;
X y Z son matrices de incidencia que conectan los efectos fijos y aleatorios con el vector de observaciones.
En este modelo se asume que:
Var
En la cual G
i
=
Accij =
Donde: Pev es la varianza del error de predicción (valor propio para cada animal para el carácter estudiado), y
Para el análisis de PV a lo largo de la trayectoria de la edad se aplicó RA, utilizando varios modelos y sin considerar los efectos maternos, los cuales variaron en el orden de ajuste del polinomio para los efectos aleatorios, así como las estimaciones de componentes de (co)varianza total o intra sexo del animal. En total se realizaron dos bloques de modelos:
Bloque 1- Se asume que los componentes de (co)varianza son los mismos en ambos sexos.
Bloque 2- Se asume que los componentes de (co)varianza no son los mismos en ambos sexos.
En ambos bloques Yijkl representa las diferentes estimaciones de PV en el l ak ésimo animal, del j ésimo sexo. Los efectos fijos (fixed
i) fueron año-mes de control con 674 niveles: sexo- edad al parto de la vaca con 18 niveles representados en todos los modelos, de manera que los resultados pueden compararse aplicando los criterios informativos LogL; BIC y AIC. Los seis modelos solo se diferencian en el orden de ajuste del polinomio de Legendre (
donde A es el numerador de la matriz de parentesco entre los animales con datos y sus antecesores sin registros (n= 4,070 animales totales). Ip es la matriz de identidad para los efectos aleatorios de ambiente permanente individual (dimensión p=3540 para el bloque 1 con varianza
La versión empleada del asreml produce automáticamente el análisis de componentes principales de esta matriz, la cual facilita la interpretación de las trayectorias de las (co)varianzas estimadas. En este caso, KG es una matriz simétrica conformada por cuatro sub matrices con los mismos componentes que contiene las (co)varianzas para efectos genéticos en las hembras (Kh); machos (Km) y sus covarianzas (Khm) con sus respectivas varianzas del intercepto (
En general, los parámetros genéticos de h2 y rg se estiman aplicando fórmulas clásicas17. Los estimados de VG del PV para cada sexo se estiman mediante la solución del mejor modelo donde, para el kth animal se tendrá:
y donde
Los parámetros genéticos resultantes del bloque 0 se presentan en el Cuadro 2, donde se incorporaron las correlaciones entre los VG estimados por MU y MT.
Cuadro 2 Componentes de (co)varianza y heredabilidad del peso vivo ajustado a 548 días y la tasa de gestación al primer parto (Modelos del bloque 0)
| Parámetro genético | Peso vivo (kg) | Tasa de gestación (d) |
|---|---|---|
| Varianza genética-MU | 369.9 | 0.017588 |
| Varianza genética-MT | 375.3 | 0.02335 |
| Heredabilidad- MU | 0.337±0.11 | 0.084±0.03 |
| Heredabilidad- MT | 0.349±0.10 | 0.109±0.02 |
| Corr genética- Peso y tasa de gestación | 0.309±0.11 | |
| Precisión, % del valor genético (VG) MU | 63.5±12.1 | 34.2±8.9 |
| Precisión, % del valor genético MT | 64.1±12.1 | 39.6±9.8 |
| Corr entre valores genéticos MU x MT | 0.996 | 0.8971 |
| Corr VG tasa de gestación MU x VG peso MU | 0.286 | |
| Corr VG gestación MU x VG peso MT | 0.570 | |
Para PA548 no hubo diferencias entre MU y MT. Para TG, el MT incrementó la h2, mejorando las precisiones de los VG. La rg entre ambos caracteres fue positiva (rg =0.309), indicando ausencia de antagonismo en la mejora de los dos rasgos. Las correlaciones entre los VG según ambos modelos fueron superiores a 0.897, de lo que se infiere que no existirán cambios en el orden de mérito por ambos procedimientos. El modelo MT tiene ventajas adicionales, manifestadas en una mayor correlación con el VG para TG, así como mayor precisión en los estimados de VG para este último rasgo, cuyo valor de h2 fue bajo.
La bondad de ajuste de los seis modelos de los bloques 1 y 2 se determinó mediante los criterios LogL, AIC y BIC, los tres coincidieron en que el polinomio de tercer orden para el efecto genético es el de mejor ajuste a los datos. En particular, los modelos del bloque 2 presentan mejores resultados, que demuestran que existe una significativa variación entre sexos para los componentes genéticos de (co)varianza.
La h2 del PV a lo largo de la edad para ambos sexos, así como la correlación genética entre ellos se presentan en la Figura 1. Las tendencias de h2 presentan ligeros aumentos conforme avanza la edad, siendo superiores para las hembras. Las rg reflejan un patrón inverso con valores entre 0.25 a 0.35. Por otro lado, la distribución de frecuencia de los VG para PA548, estimados según el modelo del bloque 0 y según RA se muestran en la Figura 2, en donde puede observarse una superposición en los tres estimados de VG. El análisis de componentes principales de la matriz KG del modelo 6 seleccionado, indica que el primer (vp1) y segundo vector propio (vp2) explicaron el 57 y 31% de la variación genética, respectivamente. Los VG de los mejores 200 animales del MU (método oficial actualmente en uso) y de la RA a lo largo de la edad y para cada sexo, se muestran en la Figura 3. En ella se visualiza que la tendencia en todos los machos es positiva, mientras que en las hembras se pueden encontrar animales con VG negativos.
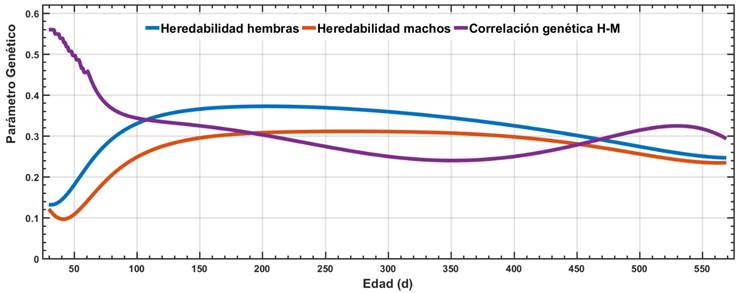
Figura 1 Estimados de heredabilidad y correlación genética para el peso vivo de hembras y machos a lo largo de la escala de edad en animales Brahman (modelo 6)
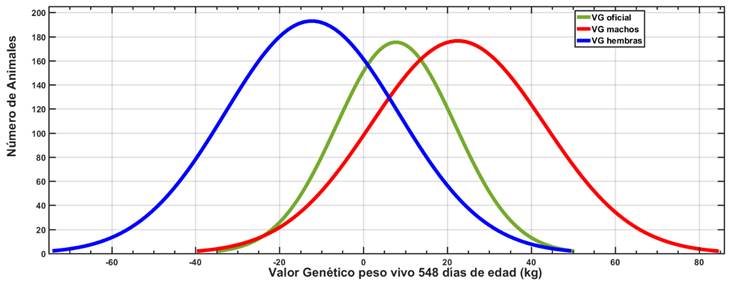
Figura 2 Distribución de frecuencia de los valores genéticos para peso a 548 días de edad según modelo actualmente en uso y mediante regresión aleatoria (modelo 6)
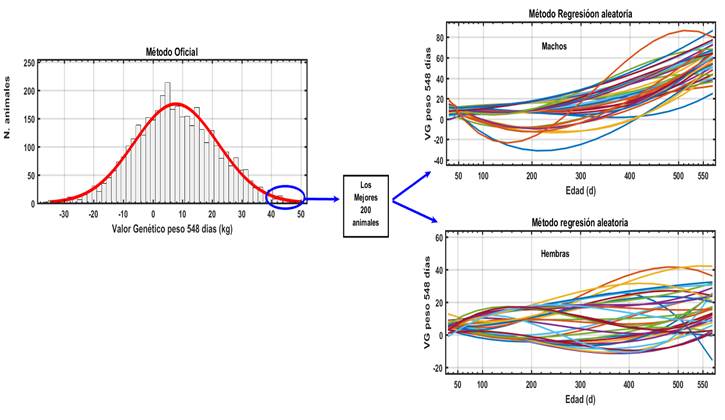
Figura 3 Evolución de los Valores genéticos de animales Brahman de cada sexo a lo largo de la trayectoria de edad, seleccionados según modelo actual de evaluación
La evolución del mérito respecto al año de nacimiento de los animales se muestra en la Figura 4. El progreso genético anual fue de 0.933 ± 0.021 kg/año para PA548 el carácter principal en el programa de mejora aplicado, mientras que para TG fue de 0.354 ± 0.010 %/año.

Figura 4 Evolución del Mérito genético para peso vivo y tasa de gestación en animales Brahman de la estación experimental La Cumaca
Los valores de h2 para PA548 coinciden con varias referencias en este tipo de animales publicados en Venezuela19,20, así como con los publicados en ganado B. indicus en diferentes países de América Latina21. El progreso genético para PA548, fue inferior a lo publicado por otros autores20,22.
Los estimados de h2 con MU para TG fueron bajos, lo cual es coherente con la mayor parte de las publicaciones sobre caracteres reproductivos10,23. Sin embargo, los niveles de h2 para TG con MT aumentaron (h2=0.109 vs 0.087), incrementándose la precisión promedio de los VG en 15.7 %. Los niveles de rg entre ambos rasgos sugieren ausencia de antagonismo, de manera que un proceso de selección por PV y TG pueden llevarse a la práctica, enfoque ya sugerido en otras publicaciones11,12,24. La mayor variabilidad genética (Figura 2) y diferentes niveles de h2 y rg para PA548 a lo largo de la trayectoria de la edad-sexo (Figura 1) indican que la manifestación de este rasgo no debe considerase como expresión del mismo carácter en ambos sexos. Lo anterior coincide con otros resultados publicados7,8.
El dimorfismo sexual (DS) muy evidente en estos datos se ha estudiado en detalle en el contexto evolutivo de las poblaciones, existiendo debate sobre la importancia de la heterogeneidad de varianzas entre sexos y sus efectos sobre la especialización y adaptabilidad de las poblaciones25, mientras que existen planteamientos previos sobre cambios en DS como respuesta correlacionada a la selección por fertilidad. Relacionado con este último punto de vista, los resultados de este trabajo permiten presentar un enfoque novedoso ya que disponemos de los VG en cada sexo para PA548M y PA548H (resultados de la solución del modelo seis, bloque dos), de manera que se puede estimar un DS de origen genético como DSg= VGPA548M - VGPA548H y estos estimados de Dsg se pueden relacionar con los VG de los mismos animales para TG (solución del modelo MT del bloque 0). Los resultados del análisis indicaron que una ecuación cuadrática (TG= 0.574 + 0.1045*DSg + 0.000765*DSg2 - 0.0000244*DSg3 y R2= 96.1 %) fue la de mejor ajuste a los datos, manifestándose un incremento del orden de +1.2% en TG por cada 10 kg de DSg, con un punto máximo cuando 40>DSg<60 con TG= +4.4 %. Por el contrario, cuando -10>DSg<0, la TG fue de -1.4 %. Estos resultados son alentadores, pero se requieren mayores investigaciones sobre esta temática que pueden tener una importante aplicación práctica en los sistemas de producción del ganado de carne.
En este trabajo se detecta una amplia variabilidad genética tanto en PV como en TG de animales del tipo Brahman. Se sugiere utilizar modelos MT, que posibilita incrementos sustanciales en los valores de h2 y en la precisión de los VG estimados, particularmente en TG. El análisis de RA ha indicado que tanto los niveles de h2 como de las rg entre el PV de hembras y machos varían a lo largo de la trayectoria de la edad, de manera que no se deben considerar como expresiones del mismo rasgo. Finalmente, y como tendencias adicionales, se ha podido identificar una importante variabilidad genética en dimorfismo sexual, la cual está relacionada con la TG, aunque esta sugerencia requiere de otras investigaciones con mayor número de animales.











 texto en
texto en 


