Introducción
La lechería bovina mexicana se desarrolla especialmente en una provincia biogeográfica denominada Altiplano central mexicano1. En el interior de esta región, las unidades de producción lechera (UPL) enfrentan problemas de insuficiencia en la producción, rentabilidad económica y de inocuidad de los productos2. Dentro de los problemas de inocuidad se ha resaltado la presencia de aflatoxinas (AF) porque ocasionan un fuerte impacto económico asociado a la contaminación de las cosechas agrícolas, al deterioro de la salud de los animales, la disminución de su productividad, así como a la contaminación de alimentos de origen animal3. Las AF tienen propiedades hepatotóxicas, nefrotóxicas e inmunosupresoras, y son consideradas como los carcinógenos naturales más potentes que se conocen4. Las AF se han cuantificado en piensos, leche y productos lácteos destinados para la alimentación de la población humana mexicana5-7. Lo cual sugiere que la presencia de AF en el pienso de las vacas es un problema usual en las UPL que induce a la contaminación de la leche con los metabolitos de AF.
Cuando las vacas lecheras ingieren productos agrícolas contaminados con AF, los mecanismos enzimáticos bioactivan la micotoxina mediante la formación de un epóxido que reacciona con las estructuras celulares y ácidos nucleicos dañando su integridad4. El epóxido reactivo puede también neutralizarse mediante su conjugación con glutatión o su excreción como un metabolito que se elimina en la leche principalmente como aflatoxina M1 (AFM1); también AFM1 es considerada como un agente tóxico y carcinogénico para los humanos4.
Las AF son metabolitos secundarios de varios hongos filamentosos del género Aspergillus, los cuales se encuentran distribuidos mundialmente y contaminan una gran variedad de productos agrícolas, especialmente los cereales8. También A. flavus ha sido identificado en México, tanto en suelo agrícola y como en grano de maíz9,10. La reproducción asexual del género Aspergillus ha sido descrita previamente11,12, así como también su filogenia, morfología y ciclo biológico8,13. Aunque Aspergillus flavus es considerado la especie con mayor capacidad de producción de aflatoxinas14, también hay cepas de A. flavus sin capacidad aflatoxigénica, así como otras especies de Aspergillus que producen AF (A. parasiticus, A. nomius, A. pseudonomius, A. arachidicola, A. bombycis, A. pseudotamarii, A. minisclerotigenes y A. togoensis)15. La capacidad aflatoxigénica se expresa principalmente bajo condiciones de estrés ambiental16,17, siempre y cuando su genotipo incluya la información involucrada en la cadena metabólica de producción de AF18,19. Una estrategia para la identificación molecular de las diferentes especies del género Aspergillus es el empleo del gen de la calmodulina (CaM), los fragmentos correspondientes a la región de los espaciadores internos de la transcripción (ITS) y el gen regulador de la ruta biosintética de aflatoxinas (aflR)8,20.
El objetivo de este trabajo fue caracterizar morfológica, molecular y aflatoxigénicamente aislados de Aspergillus flavus obtenidos de unidades de producción lechera del altiplano central mexicano.
Material y métodos
Lugar de estudio
El estudio se realizó con un diseño descriptivo, longitudinal y no experimental. Se seleccionó una unidad de producción lechera con el método no probabilístico por conveniencia y se le dio seguimiento por 24 meses. La UPL se ubicó en el Altiplano Central Mexicano (21°48’N, 102°03’ O; 1986-2008 msnmm), con clima templado-seco y semicálido-semiseco, con lluvias en verano, temperatura promedio de 18.4 º C, precipitación pluvial anual de 518.4 mm y altitud máxima de 2,300 msnm.
En el periodo de observación, la UPL contó con un promedio de 2,749 vacas Holstein, alojadas en estabulación libre dentro de corrales limitados con cercas metálicas, áreas sombreadas y comederos al libre acceso. El proceso de ordeño de las vacas se realizó con equipo automatizado obteniendo una producción promedio diario por vaca de 28.1 litros. La producción láctea se destinó a plantas agroindustriales de la región. Las dietas alimenticias se elaboraron como una ración total mezclada (RTM) en una fábrica de piensos, donde el ensilaje de maíz y el concentrado se incorporaron en carros mezcladores. La RTM se formuló para satisfacer los requerimientos nutricionales de las vacas lecheras. El maíz para ensilaje se obtuvo directamente de las áreas agrícolas de la UPL, mientras que el concentrado proteico-energético se adquirió en la Asociación Local de Ganaderos Lecheros de Aguascalientes; empleando como principales ingredientes canola, soya, maíz rolado, sorgo, pasta de soya, grano de maíz seco destilado, semilla de algodón, heno de alfalfa y avena, así como premezcla de vitaminas y minerales.
Obtención y manejo de muestras
Se obtuvieron un total de 288 muestras alimenticias (1.0 kg): ración total mezclada (RTM), alimento concentrado y ensilaje de maíz; se secaron en una estufa con circulación forzada de aire (OF-22G JEOI-TECH, Lab Companion, Corea). Las muestras se pulverizaron (500-800 µm) en un molino universal de funcionamiento continuo (MF series 10 Basic, IKA®-Werke, Alemania) y se almacenaron en refrigeración (4-5 °C) hasta su procesamiento (< 7 días). Durante el ordeño (matutino y vespertino) se obtuvieron un total de 288 muestras de leche cruda, directamente del tanque recolector (500 ml), por cada lote productivo (altas, medias y bajas). Las muestras se transportaron en refrigeración y se conservaron en congelación (-20°C) hasta su procesamiento (< 7 días).
Con ayuda de un sacabocado para suelo, se muestrearon cinco parcelas agrícolas, eligiendo cuatro puntos de muestreo sobre la superficie de cada parcela y tomando cinco sub-muestras de 100 g en cada punto, a 3-30 cm de profundidad. Las cinco sub-muestras se reunieron en una bolsa con cierre hermético. Finalmente se tamizaron (500-800 µ) y se conservaron en refrigeración hasta su procesamiento.
Cuantificación de aflatoxinas
Las muestras de alimento se analizaron de acuerdo con el método oficial 990.33 de la AOAC21, empleando columnas de fase sólida (SPE; SupelcleanTM LC-18 SPE tube, Sigma-Aldrich, EUA). El eluato extraído de las muestras derivatizadas con ácido trifluoroacético se analizó mediante HPLC (Límite mínimo de detección 2 µg/kg) con detector de fluorescencia (Bomba binaria Varian Pro Star; FP detector 2020, Varian Associates Inc., Victoria, Australia), columna C18 y guarda columna (LC-18 y LC-18; Thermo Fisher Scientific, Massachusetts, EUA). Los datos de cuantificación se obtuvieron a través del software Galaxie (Ver. 1.9.302.530) y las concentraciones de AF se calcularon mediante curvas estándar de AF purificadas (Sigma-Aldrich, St. Louis, MO, USA). La AFM1 se cuantificó en la leche cruda con la técnica de ELISA competitivo empleando un kit comercial (Ridascreen fast® aflatoxin M1 R-1121, R-Biopharm, Alemania; rango de detección 0.005-0.08 µg/kg). De acuerdo a las instrucciones del fabricante, las muestras se homogeneizaron y se centrifugaron. La absorbancia se midió a 450 nm en un lector de microplacas de ELISA (BioTek Instruments, Inc., USA). Los resultados se interpretaron con base a la curva de calibración realizada con AFM1 purificada (Sigma-Aldrich, St. Louis, MO, USA).
Aislamiento y caracterización fúngica
Los hongos se aislaron empleando la técnica de vaciado en placa con cuatro diluciones seriadas en agua peptonada estéril22; las muestras se sembraron en agar papa y dextrosa (PDA), extracto de malta con rosa de Bengala y Czapeck; se incubaron (28 °C) en oscuridad durante 7 días. Se aislaron y purificaron las colonias y se identificaron las características morfológicas microscópicas congruentes con la descripción del género13. Los aislados congruentes con A. flavus8,13) se sometieron al proceso de fijación con glutaraldehído (2%), deshidratación con alcoholes graduales y acondicionamiento con un secador de punto crítico (Samdri 795, Tousimis Research Rockville, Meryland) y un metalizador (Desk II, Denton Vacuum, EUA); se digitalizaron en un microscopio electrónico de barrido (JSM-5900 LV, JEOL, EUA) y se realizaron diez mediciones de cada estructura (estípite, vesícula, espora, esclerocio y fiálide) mediante un software (JEOL Scanning Electron Microscope). Las estructuras morfológicas de los aislados identificados se compararon contra cepas conocidas de A. flavus, denominadas AF-36, AF-Cuatitlán (AF-C) y AF-Tamaulipas, (AF-T)14,23. Las cepas obtenidas en este estudio fueron registradas en el NCBI (National Center for Biotechnology Information) con las claves de accesión respectivas.
La capacidad aflatoxigénica de los aislados se caracterizó mediante cromatografía en capa fina (TLC)24; se usaron placas de sílica gel sin indicador de fluorescencia (Z265829, Sigma-Aldrich, EUA) activadas en una estufa de alta temperatura (OF-22G JEOI-TECH, Lab Companion, Corea). Las placas con estándares purificados de AF (A6636-50MG, Sigma Aldrich, EUA) se colocaron dentro de una cámara cromatográfica con fase móvil cloroformo:acetona:isopropanol (85:10:5, v/v/v) durante hora y media. Finalmente, las placas secas se visualizaron en un transiluminador. También se utilizó la técnica de vapores de amonio en agar coco de acuerdo a la metodología descrita anteriormente25. Se inocularon aislados monospóricos en agar coco estéril y se dejaron incubar en oscuridad (30 °C, 5-7 días). Posteriormente, se agregó hidróxido de amonio (200 µl; J.T. Baker, México) al 25% en la tapa de las cajas Petri y se observó la distribución y la intensidad del color. La presencia de color se tomó como indicativo de producción de AF.
Análisis molecular
La extracción de ADN genómico de cultivos monospóricos de Aspergillus spp. se realizó siguiendo métodos estandarizados previamente26. La calidad de ADN obtenido se visualizó con electroforesis (45 min a 85 volts) en gel de agarosa (1%) con amortiguador TAE 1X y se cuantificaron comparando contra concentraciones conocidas de ADN de fago λ (Thermo Fisher Scientific, MA USA). La visualización del ADN se realizó en un fotodocumentador (Bio-Rad Molecular Imagen®- Gel Doctm XR, CA EUA) con el software Quantity One versión 4.6.7.
Siguiendo los protocolos descritos previamente27,28, se realizó la amplificación de un fragmento correspondiente a la región de espaciadores internos de la transcripción (ITS; ITS1-5.8S-ITS2 RNAr) con los iniciadores universales ITS1 (5´-TCCGTAGGTGAACCTGCGG-3´) e ITS4 (5´-TCCTCCGCTTATTGATATG -3´); se amplificó el gen de la calmodulina (CaM) con los iniciadores CMDA7F (5´-GCCAAAATCTTCATCCGTAG-3´) y CMDA8R (5´-ATTTCGTTCAGAATGCCAGG-3´) y se amplificó el gen regulador de la ruta biosintética de aflatoxinas (aflR) empleando los iniciadores aflR-F (5´-GGGATAGCTGTACGAGTTGTGCCAG-3´) y aflR-R (5´-TGGKGCCGACTCGAGGAAYGGGT-3´) de Eurofins Genomics, Lousville KY, USA. Para la amplificación fue utilizada la enzima Go-Taq polimerasa (Promega, Madison, WI USA) y un termociclador (modelo 9700 Applied Biosystems). Los productos de PCR obtenidos (ITS, calmodulina y aflR) se separaron por electroforesis en gel de agarosa (1%) y para observarlos se emplearon como intercalantes los reactivos SYBR® Gold y Orange DNA Loading Dye (Thermo Fisher Scientific, MA USA); las bandas resultantes se observaron en un fotodocumentador de imágenes (BIO-RAD Molecular Imagen®- GEL DOCTM XR CA, EUA) con el software Quantity One (versión 4.6.7.). Se incluyeron escaleras marcadoras de peso molecular (Axygen Biosciences, CA, EUA). Los productos de PCR se purificaron con el reactivo ExoSAP-IT® PCR Product Cleanup (Afflymetrix, Thermo Fisher Scientific Inc. Santa Clara, California, EUA). Los productos de PCR purificados se secuenciaron en cadenas forward y reverse con el método dideoxi29. Las muestras se inyectaron en un secuenciador (ABI 3730XL Genetic Analyzers) y las secuencias resultantes se registraron en un electroferograma. Los electroferogramas fueron visualizados con el software Chromas Lite y se compararon con los registros del NCBI usando la herramienta BLAST (Basic Local Alignment Search Tool).
Análisis estadístico
Los datos de cantidad de ración total mezclada, producción lechera, concentración de AF en pienso y leche, así como de las mediciones de las estructuras de cada aislado se sometieron a un análisis de varianza de una vía (ANDEVA) considerando separadamente como factores la época del año y el nivel de producción de leche (alta, media o baja) en el cual se tuvieron lotificadas las vacas. Para determinar las diferencias entre las medias y los intervalos de confianza al 95% se realizó el procedimiento de diferencia honestamente significativa (HSD) de la prueba de Tukey; P<0.05 fue considerado como significativo para todos los análisis estadísticos.
Resultados
Frecuencia de Aspergillus spp
Se identificaron un total de 283 aislados fúngicos, 56.8 % provinieron de muestras del pienso de las vacas lecheras y el resto de suelo agrícola (Cuadro 1). Un total de 88 aislamientos (31.1 %) mostraron características morfológicas correspondientes a las descritas para el género Aspergillus; En la Figura 1 se observan los conidióforos, los conidios y las métulas características. También se encontraron aislamientos con morfología compatible con los géneros Penicillium, Fusarium, Rhizopus, Mucor, Cladosporium, Trichoderma, Alternaria, Curvularia y Bipolaris. Las proporciones respectivas de cada género se muestran en el Cuadro 1.
Cuadro 1 Géneros fúngicos identificados en muestras mensuales * de una unidad productora lechera en el altiplano central mexicano
| Género | Suelo | Ensilaje de maíz | Alimento concentrado | Ración total mezclada | Total | |||||
|---|---|---|---|---|---|---|---|---|---|---|
| No. | % | No. | % | No. | % | No. | % | No. | % | |
| Muestras | 40 | -- | 96 | -- | 96 | -- | 96 | -- | 328 | -- |
| Aspergillus | 30 | 24.6 | 16 | 42.1 | 19 | 32.8 | 23 | 35.4 | 88 | 31.1 |
| Penicillium | 23 | 18.9 | 3 | 7.9 | 4 | 6.9 | 9 | 13.8 | 39 | 13.8 |
| Fusarium | 20 | 16.4 | 3 | 7.9 | 11 | 19.0 | 2 | 3.1 | 36 | 12.7 |
| Rhizopus | 15 | 12.3 | 4 | 10.5 | 7 | 12.1 | 8 | 12.3 | 34 | 12.0 |
| Mucor | 15 | 12.3 | 6 | 15.8 | 4 | 6.9 | 9 | 13.8 | 34 | 12.0 |
| Cladosporium | 5 | 4.1 | 4 | 10.5 | 10 | 17.2 | 10 | 15.4 | 29 | 10.2 |
| Trichoderma | 10 | 8.2 | 0 | 0.0 | 0 | 0.0 | 0 | 0.0 | 10 | 3.5 |
| Alternaria | 4 | 3.3 | 2 | 5.3 | 1 | 1.7 | 2 | 3.1 | 9 | 3.2 |
| Curvularia | 0 | 0.0 | 0 | 0.0 | 2 | 3.4 | 1 | 1.5 | 3 | 1.1 |
| Bipolaris | 0 | 0.0 | 0 | 0.0 | 0 | 0.0 | 1 | 1.5 | 1 | 0.4 |
| Total | 122 | 100 | 38 | 100 | 58 | 100 | 65 | 100 | 283 | 100 |
* Muestras de ingredientes alimenticios: 24 meses, por cuadruplicado. Muestras de suelo: 5 parcelas, por cuadruplicado, 2 temporadas.

Páneles: A) Conidióforo: Cc = Cabeza conidial, Ee = Estípite, Pe = Célula pie, Mo = Micelio. B) Cabeza conidial uniseriada: Co = Conidio o espora; Fe = Fiálide, Va = Vesícula. C) Cabeza conidial biseriada: Co = Conidio, Fe = Fiálide, Va = Vesícula, Me = Métula.
Figura 1 Estructuras morfológicas del género Aspergillus
Solamente seis aislamientos mostraron una morfología coincidente con A. flavus y provinieron del alimento concentrado (AC1, AC2 y AC3) y del ensilaje de maíz (EM1, EM2, EM3); al ser cultivados en PDA, mostraron una coloración verde olivo con la periferia blanquecina. La mayoría de los aislados (5/6) mostraron presencia de esclerocios de color marrón oscuro, mientras que sólo uno no presentó estas estructuras. Microscópicamente, los conidióforos de A. flavus presentaron cabezas conidiales irradiadas y uniseriadas, el estípite con paredes rugosas, la vesícula esférica, los conidios tenían forma globosa con irregularidades en la superficie y el micelio septado y blanquecino (Figura 2).

Páneles: A) Cc = cabeza conidial, Ee = estípite. B) Va = vesícula. C) conidióforo. Fe = fiálides, Co = conidio. D) Cc = conidio, Fe = fiálide. E) Mo = micelio, So = septo. F) Esclerocio = Eo.
Figura 2 Estructuras morfológicas de Aspergillus flavus
Los seis aislados de A. flavus provenientes de pienso mostraron diferencias significativas en la morfometría de sus estructuras al ser comparadas contra las cepas control (AF-C y AF-T; Cuadro 2). Estas cepas presentaron cabezas conidiales radiadas, con estípite rugosa y vesícula de forma esférica, mientras que AF-C sólo presentó fiálides (uniseriadas) y AF-T fiálides y métulas (biseriadas); la forma de las conidias era globosa con irregularidades en la superficie. Cuatro aislados se clasificaron como cepas L (esclerocios largos > 400 µm), uno como cepa S (esclerocio corto <400 µm) y otro sin esclerocio.
Cuadro 2 Comparación de las dimensiones de las estructuras morfológicas de aislados de Aspergillus flavus obtenidos de ensilaje de maíz (EM) y alimento (AC) integral de vacas lecheras, así como de las cepas Cuautitlán (C) y Tamaulipas (T)
| Aislado | Estípite | Vesícula | Espora | Media | Esclerocio | Media | Fiálide | |||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Media | LI | LS | Media | LI | LS | Media | LI | LS | LI | LS | LI | LS | ||||||||||||
| AF-C | 237 | c | 183 | 291 | 33.6 | c | 27.5 | 39.7 | 3.3 | b | 3.1 | 3.4 | 418 | cd | 353 | 483 | 5.9 | ab | 4.9 | 6.9 | ||||
| AF-T | 646 | a | 564 | 727 | 76.6 | a | 67.7 | 85.5 | 2.8 | c | 2.7 | 2.9 | 453 | bc | 361 | 545 | 6.4 | ab | 4.9 | 7.8 | ||||
| AF-AC1 | 313 | ab | 241 | 386 | 54.3 | b | 46.3 | 62.2 | 3.1 | b | 3.0 | 3.3 | 592 | ab | 516 | 667 | 5.1 | b | 4.4 | 5.8 | ||||
| AF-AC2 | 373 | ab | 292 | 454 | 58.2 | b | 49.3 | 67.0 | 3.0 | bc | 2.8 | 3.1 | 428 | cd | 352 | 503 | 5.1 | b | 4.3 | 5.8 | ||||
| AF-AC3 | 441 | ab | 347 | 534 | 47.8 | ab | 37.5 | 58.0 | 3.1 | b | 2.9 | 3.3 | -- | -- | -- | 6.6 | ab | 5.5 | 7.7 | |||||
| AF-EM1 | 331 | ab | 255 | 407 | 50.5 | b | 42.1 | 58.8 | 3.5 | a | 3.4 | 3.7 | 268 | d | 193 | 344 | 4.8 | b | 4.1 | 5.5 | ||||
| AF-EM2 | 218 | c | 115 | 320 | 38.8 | ab | 27.6 | 50.1 | 3.2 | b | 3.1 | 3.4 | 672 | a | 597 | 748 | 5.4 | ab | 4.1 | 6.6 | ||||
| AF-EM3 | 320 | b | 239 | 401 | 47.3 | ab | 38.4 | 56.2 | 3.6 | a | 3.5 | 3.8 | 561 | ab | 505 | 617 | 6.9 | a | 6.0 | 7.9 | ||||
-- Sin esclerocio- LI, LS Límite inferior y superior de los intervalos de confianza.
abcd Medias de columnas con diferente literal muestran diferencias estadísticas significativas (P<0.05).
Los aislados identificados como A. flavus se analizaron por la técnica de TLC y de vapores de amonio en agar coco para identificar la presencia de aflatoxina (Figura 3). Cinco aislamientos (AF-AC1, AF-AC2, AF-AC3, AF-EM2 y AF-M3) tuvieron una reacción positiva a la presencia de AF y el otro (AF-EM1) fue negativo a la producción de aflatoxina en ambas técnicas. Estos resultados coincidieron con la amplificaron del gen aflR.
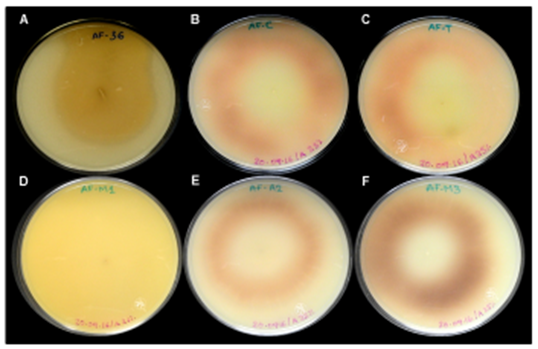
Páneles: A) AF-36, no aflatoxigénico (AF-). B) AF-Cuatitlán y C) AF-Tamaulipas, aflatoxigénico (AF+). D) AF-EM1, AF- aislado de ensilaje de maíz. E) AF-AC2, AF+ aislado de alimento concentrado. F) AF-EM3, AF+ aislado de ensilaje de maíz.
Figura 3 Intensidad de color en medio agar coco al contacto con vapores de amonio de aislamientos Aspergillus flavus a los 4 días de crecimiento
Análisis molecular
De los 88 aislados identificados morfológicamente como Aspergillus spp., el 49 % amplificó para el gen calmodulina y el 31 % amplificó para el ITS (Figura 4). Los análisis de las secuencias obtenidas mostraron que las especies fueron A. oryzae (45.5 %), A. niger (10.2 %), A. ochraceus (3.4 %), A. pseudodeflectus (4.5 %), A. ustus (10.2 %), A. flavus (6.8 %), A. versicolor (5.6 %), A. nidulans (5.6 %), A. sublatus (4.5 %) y A. sydowii (3.4 %).
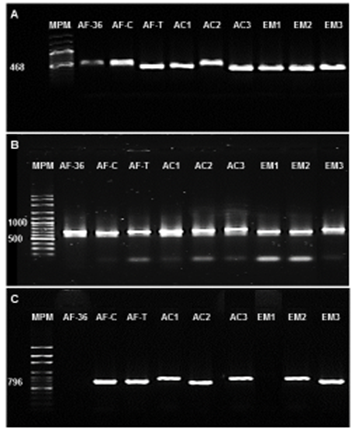
Carriles: MPM) Marcador de peso molecular (100pb). Controles: A. flavus no aflatoxigénico (AF-36) y aflatoxigénicos (Cuautitlán, AF-C; Tamaulipas, AF-T). A. flavus aislados de ensilaje de maíz (EM1, EM2 y EM3) y de alimento concentrado (AC1, AC2 y AC3) destinado a las vacas lecheras
Figura 4 Electroforesis en gel de agarosa al 1% de los productos de PCR amplificados del gen calmodulina (Panel A), región de espaciadores internos de la transcripción 5.8S rDNA-ITS2 (Panel B) y gen regulador de la ruta biosintética de aflatoxinas, aflR (Panel C)
Los cinco aislados locales identificados como A. flavus aflatoxigénico amplificaron el fragmento de 796 pb para el gen aflR. El análisis de las secuencias de las cepas testigo (AF-36, AF-C y AF-T) y de los seis aislados de A. flavus (AF-AC1, AF-AC2, AF-AC3, AF-EM1, AF-EM2 y AF-EM3) mostraron tener un porcentaje de identidad mayor al 90% con aislamientos de A. flavus registrados en la base de datos del NCBI (Cuadro 3).
Cuadro 3 Identidad de los aislados de Aspergillus flavus obtenidos de las dietas alimenticias para vacas lecheras en Aguascalientes, México, al compararlos con secuencias existentes en el Centro Nacional para la Información en Biotecnología (NCBI)a
| ID del Aislado | Origen | Iniciadorb | Clave de accesión | Coincidencia (%) |
|---|---|---|---|---|
| Control- AF-36 | Control | ITS1 | LN482513.1 | 99 |
| ITS4 | KX550912.1 | 98 | ||
| Control+ C (AHY255) | Maíz (Cuatitlán) | CMDA7F | AY974341.1 | 98 |
| CMDA8R | AY974340.1 | 96 | ||
| ITS1 | LC105688.1 | 100 | ||
| ITS4 | KT254587.1 | 99 | ||
| AFLRF | FN398160.1 | 100 | ||
| AFLRR | L32576.1 | 100 | ||
| Control+ T (AHY256) | Maíz (Tamaulipas) | ITS1 | KX015990.1 | 100 |
| ITS4 | KF221065.1 | 99 | ||
| AFLRF | XM_002379905.1 | 100 | ||
| AFLRR | AF441435.2 | 99 | ||
| AC1 (AHY257) | Alimento concentrado | ITS1 | KF434090.1 | 100 |
| ITS4 | KF434090.1 | 99 | ||
| AFLRF | AF441434.1 | 100 | ||
| AFLRR | XM_002379905.1 | 99 | ||
| AC2 (AHY258) | Alimento concentrado | CMDA7F | AY974341.1 | 99 |
| ITS1 | KM285408.1 | 100 | ||
| ITS4 | EF409812.1 | 95 | ||
| AFLRF | XM_002379905.1 | 100 | ||
| AFLRR | FN398160.1 | 93 | ||
| AC3 (AHY259) | Alimento concentrado | CMDA7F | XM_002374071.1 | 99 |
| CMDA8R | XM_002379905.1 | 99 | ||
| ITS1 | KF434090.1 | 99 | ||
| ITS4 | KT356196.1 | 97 | ||
| AFLRF | AF441435.2 | 99 | ||
| AFLRR | AF441434.1 | 99 | ||
| EM1 (AHY195) | Ensilaje de maíz | CMDA7F | AY510451.1 | 95 |
| CMDA8R | XM_002374071.1 | 100 | ||
| ITS1 | HQ010119.1 | 99 | ||
| ITS4 | KX641192.1 | 99 | ||
| EM2 (AHY203) | Ensilaje de maíz | ITS1 | KT356196.1 | 98 |
| ITS4 | LN482513.1 | 100 | ||
| AFLRF | MH752566.1 | 100 | ||
| AFLRR | KY769955.1 | 100 | ||
| EM3 (AHY204) | Ensilaje de maíz | CMDA7F | XM_002374071.1 | 100 |
| CMDA8R | XM_002374071.1 | 100 | ||
| ITS1 | KX572367.1 | 99 | ||
| ITS4 | KT356196.1 | 99 | ||
| AFLRF | L32577.1 | 100 | ||
| AFLRR | MH752564.1 | 100 |
aNCBI (National Center for Biotechnology Information): https://blast.ncbi.nlm.nih.gov/
b CMDA = calmodulina; ITS = espaciadores internos de la transcripción; AFLR = regulador de la ruta biosintética; R = reversa; F = hacia adelante.
Aflatoxinas detectadas en pienso y en leche
De las 288 muestras obtenidas de alimento, 286 (99.3 %) presentaron niveles detectables de AF (18.5 ± 3.7 µg/kg); de las cuales el 10.4 % sobrepasaron los límites máximos permisibles por la legislación mexicana (20 µg/kg). En las 183 muestras de leche cruda, la presencia de AFM1 (0.021 µg/kg) se detectó en el 39.9 % y el 12.0 % rebasó el límite máximo permisible utilizado como norma por la agroindustria que adquiría la leche cruda (0.050 µg/kg). La mayor incidencia de AF en pienso se presentó en verano y otoño (P<0.01) en comparación con primavera e invierno (17.4ª ± 3.2 y 14.8 ab ± 1.6 vs 8.1 bc ± 0.5 y 5.9 c ± 0.5, µg/kg, respectivamente). A su vez, la concentración de AFM1 se correlacionó de manera directa con la concentración de AF en la RTM en un modelo doble cuadrado (P<0.01; R2= 30.5 %). También la presencia de AFM1 en leche se correlacionó significativamente con el nivel de producción de leche y con el consumo de RTM; en promedio, las vacas consumieron diariamente 621 µg de aflatoxinas en 42 kg de RTM, produjeron diariamente 26.2 L de leche con una carga total de 0.603 µg de AFM1, lo que representó una gran capacidad de las vacas lecheras para metabolizar las AF y solamente eliminaron una fracción mínima (0.09 %; Cuadro 4); en general, los lotes productivos de vacas con alta producción se expusieron a la ingestión de mayores cantidades de AF (658 µg/vaca/día) y la presencia y eliminación de AFM1 en la leche de las vacas altas productoras fue mayor al lote de vacas de mediana y de baja producción (0.22 µg/vaca/día).
Cuadro 4: Exposición promedio (± EE) de las vacas lecheras a la contaminación natural por aflatoxinas en la ración total mezclada (RTM y eliminación promedio de AFM1 por lote productivo
| Lote | Vacas (No) |
Producción de leche (kg/vaca/día) |
RTM (kg/vaca/día) |
Ingestión AF (µg/vaca/día) |
Eliminación AFM1 | ||||
|---|---|---|---|---|---|---|---|---|---|
| (µg/kg) | (µg/vaca/día) | (%) | |||||||
| Altas | 1760 | 32.0a | 44.7a | 658a | ± 97.0 | 0.024a | ± 0.005 | 0.72 | 0.11 |
| Medias | 606 | 20.2b | 36.4c | 546a | ± 83.4 | 0.022a | ± 0.005 | 0.44 | 0.08 |
| Bajas | 388 | 14.8c | 38.4b | 568a | ± 82.6 | 0.015b | ± 0.004 | 0.22 | 0.04 |
| Total | 2754 | 28.0 | 42.0 | 621 | ± 92.0 | 0.021 | ± 0.005 | 0.58 | 0.09 |
ab Literales diferentes indican diferencia significativa entre lotes productivos P<0.05.
Discusión
En este estudio longitudinal de dos años de duración se demostró la presencia de Aspergillus flavus en pienso de las vacas lecheras del Altiplano Central Mexicano. Estos aislados mostraron características morfológicas, toxicológicas y moleculares congruentes con las cepas que poseen la capacidad de producir aflatoxinas. Además, se corroboró la acumulación de AF en el pienso y en la leche que produjeron las vacas. Aunque ya se contaba con información de la existencia en México de poblaciones indígenas de A. flavus con y sin capacidad de producir aflatoxinas, en nuestro conocimiento, este estudio agrega por primera vez la identificación molecular, toxicológica y morfométrica de A. flavus a la cuantificación de aflatoxinas en la lechería, lo cual es de importancia para la producción animal y la salud pública.
Frecuencia de Aspergillus spp. y A. flavus
Aspergillus fue el género micótico con mayor ocurrencia en la UPL (31 %) seguido de Penicillium (13.8 %) y Fusarium (12.7 %). Estos géneros ya han sido identificados en México en maíz amarillo almacenado9 y en híbridos de maíz destinados al consumo humano y animal30. Se ha sugerido que la presencia frecuente de hongos micotoxigénicos y sus toxinas en maíz es debida al manejo inadecuado del cultivo y a la falta de aplicación efectiva de estrategias de control de la infección5,31.
En este estudio, A. flavus se aisló de pienso para vacas lecheras en una proporción del 6.8 % del total de los Aspergillus identificados (6/88). En suelo agrícola destinado a la producción alterna de maíz forrajero con avena no se identificó esta especie. En México, A. flavus ha sido identificado en suelo para cultivos de maíz de grano10 en granos de maíz comercial32. Lo anterior sugiere que las poblaciones fúngicas de esta especie se encuentran distribuidas en las zonas agrícolas de México.
En este estudio, el análisis morfométrico mostró que la tercera parte de los aislados de A. flavus contenía abundantes esclerocios cortos (< 400 µm) por lo que se clasificaron como cepas S, mientras que el resto fueron denominadas como cepas L con escasos esclerocios, pero de tamaño mayor (>400 µm). Ya se señalado la importancia de esta morfología y su asociación con la aflatoxigenicidad de las cepas S, mientras que las cepas L contienen tanto aislados atoxigénicos como toxigénicos33,34.
Análisis molecular
Todas las 25 secuencias obtenidas de los aislados identificados morfológicamente como A. flavus mostraron un gran porcentaje de identidad (> 90%) con secuencias registradas de cepas A. flavus. También se obtuvieron fragmentos amplificados para el gen CaM de 468 pb, para el gen aflR de 796 pb y para los ITS con un rango de 600-800 pb (ITS1-5.8S-ITS2). Se ha señalado que la región rADN de los espaciadores internos de la transcripción es el código de barras oficial de ADN para hongos porque es el marcador secuenciado con mayor frecuencia y es una herramienta de gran utilidad para la descripción de especies de A. flavus8. También se ha referido que el gen CaM es capaz de distinguir entre casi todas las especies de Aspergillus8. Por otra parte, el gen aflR es necesario para la transcripción de la mayoría de los genes involucrados en la activación de las reacciones enzimáticas necesarias para la formación de las aflatoxinas35-37. Con base a lo anterior este trabajo integra la identificación molecular a la identificación morfológica de seis aislamientos de A. flavus obtenidos de la cadena productiva lechera en la región del Altiplano Central Mexicano.
Capacidad aflatoxigénica
Los seis aislados identificados como A. flavus se evaluaron con TLC y vapores de amonio en agar coco para identificar su capacidad de producir aflatoxinas, cinco se clasificaron como aflatoxigénicos y uno se clasificó como negativo a la producción de aflatoxinas. Estos resultados mostraron un comportamiento comparable a los obtenidos en otros estudios realizados en México10 en que identificaron aislamientos de A. flavus relacionados genéticamente con A. flavus AF-3632. Esta cepa no aflatoxigénica ha sido empleada en el desarrollo de estrategias de control biológico para reducir la producción de aflatoxinas de cepas aflatoxigénicas en campos productores de algodón15. Lo anterior sugiere que las poblaciones mayoritarias de A. flavus aflatoxigénicas se distribuyen en ecosistemas agrícolas alrededor del mundo y que coexisten con cepas sin capacidad de producir aflatoxinas.
Aflatoxinas detectadas en pienso y en leche
El 99.3 % (286/288) de las muestras de alimento de las vacas lecheras presentaron niveles detectables de AF (18.5 ± 3.7 µg/kg) de las cuales el 10.4 % sobrepasaron los límites máximos permisibles por la legislación mexicana (20 µg/kg); además el 39.9 % (73/183) de las muestras de leche cruda mostraron la presencia de AFM1. Proporciones comparables de contaminación con AF han sido detectadas en piensos balanceados producidos en México5, en la ración total mezclada de establos de México38; así como en muestras de alimento de origen asiático39. En México se han reportado la incidencia de AFM1 en leche cruda de vaca6,7; también en diversos países se ha identificado incidencia de AFM1 en la leche destinada al consumo humano3,40-42. Lo anterior sugiere que la contaminación por aflatoxinas de alimento destinado al consumo humano y animal es una problemática mundial de salud pública.
En este estudio se mostró una asociación entre el nivel de producción de leche de las vacas con la cantidad y de la concentración de AFM1 eliminada en la leche, de tal forma que las vacas altas productoras consumieron una mayor cantidad de alimento y se expusieron a cantidades más grandes de aflatoxinas en su pienso; por consiguiente, fue mayor la cantidad total, así como la concentración de AFM1 en la leche cruda (P<0.05). Se ha señalado que la tasa de eliminación de AFM1 en leche se ve influenciada por la cantidad de pienso ingerido al día por cada animal, la cantidad de leche que produce al día y la etapa de lactación en la que se encuentra43.
Conclusiones e implicaciones
En este estudio se demostró morfológica, toxicológica y molecularmente la ocurrencia de A. flavus en ensilaje de maíz y en la ración total mezclada, lo cual condujo a la contaminación por aflatoxinas de casi la totalidad de las dietas de las vacas lecheras, así como a la presencia residual de AFM1 en la leche cruda. Las vacas lecheras lograron metabolizar y eliminar por vías diferentes a la leche más del 99 % de las aflatoxinas presentes en su dieta.











 texto en
texto en 


