Introducción
Desde el inicio del proceso de domesticación, hace 10,000 años aproximadamente, el ganado caprino ha estado en estrecha relación con la especie humana1,2. Se considera uno de los primeros animales de granja en ser domesticado y ha participado en acontecimientos como la revolución agrícola del Neolítico, el desarrollo del comercio y las migraciones humanas3. Estos acontecimientos implicaron algunos mecanismos básicos de la evolución, tales como; migración de animales, selección, deriva génica e incluso mutación, lo que ayuda a explicar en gran medida la elevada capacidad de adaptación del ganado caprino a diferentes ecosistemas y las más de 300 razas que existen en la actualidad1,4.
Desde su llegada al continente americano en 1493, los colonizadores españoles se encargaron de propagar la especie caprina, así como de capacitar a pobladores nativos en su manejo y en la selección de los mejores animales. Con el paso del tiempo, estas acciones dieron origen a nuevas poblaciones, las cuales estaban mejor adaptadas a las condiciones ambientales locales; poblaciones denominadas criollas5.
Actualmente en México existen poco más de 8.7 millones de cabras6, y al igual que en otros países en vías de desarrollo, éstas representan una oportunidad de subsistencia para las personas que habitan en zonas áridas y semiáridas, donde la vegetación es escasa y las condiciones del agostadero son pobres7,8. Aun cuando se distribuyen por todo el país, se han agrupado en tres regiones principales; el mosaico mixteco, el del centro o bajío y el del norte o lagunero9. Particularmente, en el mosaico del centro del país se encuentra la Cabra Criolla Negra (CCN), la cual se distribuye principalmente en los estados de Querétaro y Guanajuato. La principal finalidad zootécnica de la CCN es la producción de leche, la cual presenta un mayor contenido de sólidos totales y un excelente rendimiento quesero con respecto a otras razas criadas en México10,11,12. Esta cabra era considerada como raza Granadina por sus características morfológicas y por su origen, sin embargo, los más de 500 años de evolución independiente han hecho que se diferencie genéticamente de esta raza. Pese lo anterior, no existen trabajos previos que estudien el estado genético de la CCN. Las importaciones de animales de razas mejoradas se han incrementado en los últimos años y se vienen realizando cruzamientos de forma indiscriminada, lo que pone en riesgo el estado de salud genética de la población13.
La evaluación de la diversidad genética dentro y entre las razas permite conocer la estructura de la población y hace posible establecer estrategias de conservación, mejoramiento genético y utilización sostenible de los recursos genéticos14. Los marcadores moleculares del tipo microsatélite han demostrado ser de gran utilidad para los estudios de caracterización genética dentro y entre poblaciones, esto debido a su codominancia genética, abundancia, distribución aleatoria a través del genoma, alta reproducibilidad, neutralidad con respecto a la selección y alto nivel de polimorfismo14,15. En los últimos años, se han llevado a cabo numerosos estudios de diversidad genética en varias especies de ganado utilizando estos marcadores, convirtiéndose en los marcadores genéticos de elección para muchas aplicaciones moleculares, algunas de estas son: diversidad genética16,17, estructura poblacional18,19, filogenia20, evaluación de paternidad21, etc. El objetivo del presente estudio fue evaluar la diversidad genética y la estructura poblacional de la Cabra Criolla Negra a través de marcadores microsatélites.
Material y métodos
Muestras biológicas
Se colectaron muestras de pelo de 45 animales pertenecientes a tres rebaños de CCN utilizando el método no probabilístico de oportunidad y ubicados en tres municipios del estado de Querétaro; Cadereyta de Montes (n= 7), El Marqués (n= 11) y San Juan del Río (n= 27). Los animales se muestrearon utilizando el siguiente criterio de inclusión; animales no emparentados, mayores a un año de edad, de capa negra con orejas rígidas o semipendulantes. En estas poblaciones no se cuenta con información genealógica y debido a esto se tomó en cuenta la información de parentesco entre los animales proporcionada por los propietarios.
Se incluyó la información de 25 microsatélites localizados en 455 individuos de 13 poblaciones caprinas (Retinta, Verata, Blanca Serrana, Celtibérica, Malagueña, Murciano-Granadina, Florida, Payoya, Serrana, Formentera, Saanen, Alpina y Anglonubia) integradas en el proyecto de Biodiversidad Caprina Iberoamericana (BioGoat)22.
Análisis molecular
La extracción del ADN se realizó a partir de las muestras de pelo utilizando resina quelante Chelex® 100 (Bio Rad Laboratories, Inc. USA)23. Se utilizaron 30 microsatélites recomendados por el comité mixto ISAG/FAO para el análisis de la diversidad genética en animales domésticos14 de los cuales, 25 marcadores se encontraron en común con las 13 poblaciones utilizadas del proyecto BioGoat. La amplificación de los marcadores se realizó por reacción en cadena de la polimerasa (PCR), para lo cual se utilizaron cebadores marcados con fluorescencia24. Los amplicones obtenidos como resultado de la PCR fueron separados por electroforesis capilar (ABI PRISM 3130 Genetic Analyser; Applied Biosystems) de acuerdo con las recomendaciones del fabricante. El tamaño de los alelos se determinó utilizando el estándar de tamaño interno GeneScan-400HD ROX (Applied Biosystems) mientras que los genotipos fueron obtenidos por medio del software GENOTYPER 2.5.1. Se incluyeron muestras de referencia en cada prueba como método de comprobación del genotipado.
Análisis estadístico
El número total de alelos por marcador (NA) se calculó por conteo directo de los alelos obtenidos para cada uno de estos, mientras que, el número medio de alelos (NMA) se calculó como la sumatoria del NA dividida entre los 30 marcadores utilizados. La heterocigosis observada (Ho) fue resultado de dividir el número de individuos heterocigotos en cada marcador entre el total de individuos positivos al mismo marcador. La heterocigosis esperada (He) se estimó utilizando la fórmula de Nei25. El contenido de información polimórfica (CIP) que representa un indicativo de la calidad de los marcadores utilizados26 se estimó utilizando el complemento para Microsoft Excel 2010 MICROSATELLITE TOOLKIT27. El número efectivo de alelos (NEA), el cual hace referencia a los alelos con capacidad de pasar a la siguiente generación28, se realizó a través del software POPGENE v. 1.32. La prueba exacta del equilibrio Hardy-Weinberg, misma que considera el déficit de heterocigotos se realizó mediante el software GENEPOP v.4.229. Esta prueba se llevó a cabo para los 30 marcadores utilizados en la CCN ocupando el método en cadena de Markov (desmemorización 5,000, 100 lotes y 10,000 interacciones por lote).
El coeficiente de endogamia de los individuos respecto a las subpoblaciones (FIS), respecto al total de la población (FIT), de las subpoblaciones comparado con el total de la población (FST) y el coeficiente de diferenciación genética (GST) se calcularon con un intervalo de confianza de 95%25,30. La estimación de estos parámetros se realizó mediante el software GENETIX v. 4.0531.
Se calculó la matriz de distancias genéticas de Reynolds32 que es la distancia mínima de Nei normalizada con una valoración de heterocigosis en la población fundadora utilizando el software POPULATIONS v.1.2.28. Se obtuvieron Split Graphs mediante el algoritmo “NeighborNet” utilizando el programa SPLITSTREE433.
El origen de la estructura genética de las poblaciones incluidas en el estudio se determinó mediante técnicas de clúster (K), que representan el número de poblaciones y usa un algoritmo bayesiano que emplea un modelo basado en el método de Cadenas de Markov de Monte Carlo (CMMC), que estima la distribución a posteriori de cada coeficiente de mezcla de cada individuo, el cual se realizó utilizando el software STRUCTURE v.2.3.434. Se utilizó un periodo de “burn-in” de 50,000 iteraciones y 200,000 repeticiones de CMMC, los resultados se visualizaron mediante el programa DISTRUCT35. Para estimar el K óptimo, se fijaron valores de K2 a K15, el análisis se realizó con 15 repeticiones para cada valor de K siguiendo la metodología de Evanno36 y utilizando el software STRUCTURE HARVESTER37.
Resultados
Variación genética en la población Criolla Negra
Se obtuvieron 243 alelos para los 30 marcadores utilizados. El NMA fue de 8.1 alelos por locus en esta población (Cuadro 1). El NA más alto por marcador (13) lo presentaron MM12 y SRCRSP23, seguido por los marcadores BM6526 y HSC con 12 alelos. El marcador con menor NA fue MAF209 con 2 alelos. Adicionalmente, el NEA con mayor valor correspondió a HSC (NEA= 9.14) y el de menor valor fue para MAF209 (NEA= 1.25), este resultado pudiera verse afectado por la proporción de marcadores polimórficos, el número de alelos por marcador y sus frecuencias, así como por el tamaño de la muestra. La heterocigosis esperada (He) promedio en la población fue de 0.71, este valor varió entre 0.20 para MAF209 y 0.90 para HSC. El promedio de heterocigosis observada (Ho) fue de 0.62 y el rango 0.18 y 0.93 para los mismos marcadores que mostraron valores extremos para He.
Cuadro 1 Microsatélites analizados, número de alelos detectados (NA), número efectivo de alelos (NEA), heterocigosis esperada (He) y observada (Ho), contenido de información polimórfica (CIP) y las desviaciones del equilibrio Hardy-Weinberg (EHW)
| Microsatélite | NA | NEA | He | Ho | CIP | EHW P-val |
|---|---|---|---|---|---|---|
| BM1329 | 8 | 3.44 | 0.72 | 0.67 | 0.68 | 0.20 |
| BM1818 | 8 | 4.63 | 0.79 | 0.71 | 0.76 | 0.12 |
| BM6506 | 9 | 3.26 | 0.70 | 0.55 | 0.66 | 0.02 |
| BM6526 | 12 | 4.93 | 0.81 | 0.86 | 0.78 | 0.90 |
| BM8125 | 6 | 3.64 | 0.73 | 0.62 | 0.68 | 0.02 |
| CRSM60 | 8 | 4.32 | 0.78 | 0.71 | 0.73 | 0.03 |
| CSRD247 | 6 | 3.25 | 0.70 | 0.77 | 0.66 | 0.97 |
| CSSM66 | 11 | 7.06 | 0.87 | 0.34 | 0.84 | 0.00 |
| ETH010 | 4 | 2.69 | 0.63 | 0.56 | 0.56 | 0.19 |
| ETH225 | 4 | 1.41 | 0.29 | 0.25 | 0.26 | 0.22 |
| HAUT27 | 8 | 4.11 | 0.77 | 0.71 | 0.72 | 0.28 |
| HSC | 12 | 9.14 | 0.90 | 0.93 | 0.88 | 0.31 |
| ILSTS011 | 6 | 2.87 | 0.66 | 0.59 | 0.59 | 0.33 |
| INRA063 | 5 | 2.46 | 0.60 | 0.64 | 0.51 | 0.72 |
| MAF065 | 11 | 5.78 | 0.84 | 0.87 | 0.81 | 0.24 |
| MAF209 | 2 | 1.25 | 0.20 | 0.18 | 0.18 | 0.43 |
| McM527 | 8 | 5.33 | 0.82 | 0.82 | 0.79 | 0.58 |
| MM12 | 13 | 7.00 | 0.87 | 0.75 | 0.84 | 0.03 |
| OarFCB011 | 10 | 5.73 | 0.83 | 0.75 | 0.80 | 0.12 |
| OarFCB048 | 10 | 7.14 | 0.87 | 0.78 | 0.84 | 0.01 |
| OarFCB304 | 10 | 3.72 | 0.74 | 0.61 | 0.69 | 0.00 |
| SPS115 | 3 | 2.02 | 0.51 | 0.27 | 0.44 | 0.00 |
| SRCRSP08 | 10 | 3.76 | 0.74 | 0.70 | 0.69 | 0.23 |
| TGLA122 | 8 | 2.20 | 0.55 | 0.49 | 0.52 | 0.02 |
| SRCRSP05 | 7 | 3.09 | 0.68 | 0.59 | 0.63 | 0.05 |
| SRCRSP23 | 13 | 8.49 | 0.89 | 0.68 | 0.87 | 0.00 |
| SRCRSP24 | 10 | 3.59 | 0.73 | 0.56 | 0.69 | 0.00 |
| ILSTS019 | 6 | 2.27 | 0.57 | 0.51 | 0.53 | 0.04 |
| INRA005 | 5 | 2.23 | 0.56 | 0.45 | 0.51 | 0.03 |
| INRA006 | 10 | 5.29 | 0.82 | 0.77 | 0.79 | 0.18 |
| Promedio | 8.1 | 4.20 | 0.71 | 0.62 | 0.66 |
P>0.05= No significativo
El valor promedio del CIP para la población de CCN fue de 0.66. En este caso, el marcador menos informativo (CIP <0.25) fue MAF209 (CIP= 0.18), seguido de ETH225 y SPS115 con valores de 0.26 y 0.44, mientras que los 27 marcadores restantes fueron más informativos (CIP >0.5). Finalmente, se observaron desviaciones significativas del equilibrio de Hardy Weinberg (EHW) en 14 de los 30 microsatélites probados en la población (P≤0.05).
Diferenciación genética entre poblaciones
El coeficiente de endogamia (FIS, FIT y FST) y el coeficiente de diferenciación genética (GST) se estimaron para cada uno de los 25 microsatélites compartidos entre la CCN y las otras 13 razas incluidas en el proyecto BioGoat22 con un intervalo de confianza del 95% (Cuadro 2). El valor promedio para FIS fue de 0.067, presentando valores negativos de FIS en los marcadores BM8125 y MAF209 (-0.002 y -0,006) indicando en estos un exceso de heterocigotos18,19. De los marcadores utilizados 11 presentaron un valor de FIS superior a 0.05. Los valores de GST fueron similares a los obtenidos para, FST. El marcador que representa un valor más alto fue BM6526 (0.114), en contraste con el MAF209 que fue el más bajo (0.037).
Cuadro 2 Coeficiente de diferenciación genética y coeficiente de endogamia para cada microsatélite compartido entre la cabra criolla negra y las razas Retinta, Verata, Blanca serrana, Celtibérica, Malagueña, Murciano granadina, Florida, Payoya, Serrana, Formentera, Saanen, Alpina, Anglonubia
| Microsatélite | GST | FIS | FIT | FST |
|---|---|---|---|---|
| BM1329 | 0.081 | 0.024 | 0.078 | 0.056 |
| BM1818 | 0.077 | 0.024 | 0.085 | 0.062 |
| BM6506 | 0.074 | 0.044 | 0.094 | 0.053 |
| BM6526 | 0.114 | 0.045 | 0.111 | 0.069 |
| BM8125 | 0.076 | -0.002 | 0.065 | 0.067 |
| CRSM60 | 0.041 | 0.064 | 0.091 | 0.029 |
| CSRD247 | 0.082 | 0.026 | 0.095 | 0.071 |
| CSSM66 | 0.083 | 0.271 | 0.316 | 0.062 |
| ETH010 | 0.058 | 0.032 | 0.076 | 0.047 |
| ETH225 | 0.057 | 0.015 | 0.058 | 0.044 |
| HSC | 0.068 | 0.095 | 0.141 | 0.050 |
| ILSTS011 | 0.058 | 0.068 | 0.114 | 0.049 |
| INRA063 | 0.051 | 0.164 | 0.195 | 0.038 |
| MAF065 | 0.073 | 0.025 | 0.084 | 0.060 |
| MAF209 | 0.037 | -0.006 | 0.018 | 0.024 |
| McM527 | 0.081 | 0.087 | 0.151 | 0.070 |
| MM12 | 0.060 | 0.059 | 0.098 | 0.042 |
| OarFCB011 | 0.084 | 0.065 | 0.134 | 0.073 |
| OarFCB048 | 0.060 | 0.058 | 0.103 | 0.049 |
| SPS115 | 0.097 | 0.187 | 0.257 | 0.086 |
| SRCRSP08 | 0.106 | 0.049 | 0.143 | 0.099 |
| TGLA122 | 0.091 | 0.070 | 0.146 | 0.082 |
| Promedio | 0.073 | 0.067 | 0.121 | 0.058 |
FIS= coeficiente de endogamia de los individuos respecto a las subpoblaciones, FIT= coeficiente de endogamia respecto al total de la población, FST= coeficiente de endogamia de las subpoblaciones comparado con el total de la población, GST= coeficiente de diferenciación genética.
Distancia genética entre poblaciones y su representación gráfica
Las distancias genéticas entre las 14 razas comparadas se presentan en el Cuadro 3. La menor distancia genética se presentó entre la CCN y la raza Murciano-Granadina (0.133). La mayor distancia genética se obtuvo con la raza Anglonubia (0.420). Con el objetivo de facilitar la interpretación de los valores obtenidos en la matriz de distancias genéticas, se construyó un dendrograma Neighbor-Net para representar gráficamente estos valores (Figura 1).
Cuadro 3: Matriz de distancias genéticas de Reynolds entre las 14 razas caprinas incluidas en el estudio
| RET | VERA | BLANCA | CELTIB | MALAG | MG | FLO | PAY | SER | FOR | SAAN | ALP | ANG | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| VERA | 0.025 | ||||||||||||
| BLANCA | 0.025 | 0.032 | |||||||||||
| CELTIB | 0.025 | 0.036 | 0.027 | ||||||||||
| MALAG | 0.021 | 0.025 | 0.023 | 0.021 | |||||||||
| MG | 0.044 | 0.045 | 0.031 | 0.034 | 0.041 | ||||||||
| FLO | 0.023 | 0.023 | 0.028 | 0.026 | 0.015 | 0.041 | |||||||
| PAY | 0.034 | 0.044 | 0.047 | 0.036 | 0.046 | 0.061 | 0.038 | ||||||
| SER | 0.034 | 0.038 | 0.035 | 0.028 | 0.024 | 0.048 | 0.027 | 0.045 | |||||
| FOR | 0.071 | 0.094 | 0.081 | 0.078 | 0.083 | 0.099 | 0.083 | 0.081 | 0.107 | ||||
| SAAN | 0.071 | 0.061 | 0.070 | 0.069 | 0.070 | 0.069 | 0.061 | 0.077 | 0.078 | 0.119 | |||
| ALP | 0.063 | 0.056 | 0.052 | 0.062 | 0.055 | 0.076 | 0.049 | 0.064 | 0.073 | 0.119 | 0.071 | ||
| ANG | 0.124 | 0.125 | 0.130 | 0.151 | 0.132 | 0.146 | 0.126 | 0.178 | 0.149 | 0.221 | 0.161 | 0.142 | |
| CCN | 0.039 | 0.052 | 0.045 | 0.044 | 0.046 | 0.038 | 0.048 | 0.067 | 0.053 | 0.102 | 0.083 | 0.073 | 0.130 |
RET= Retinta, VERA= Verata, BLANCA= Blanca serrana, CELTIB= Celtibérica, MALAG= Malagueña, MG= Murciano granadina, FLO= Florida, PAY= Payoya, SER= Serrana, FOR= Formentera, SAAN= Saanen, ALP= Alpina, ANG= Anglonubia, CCN= Criolla negra.
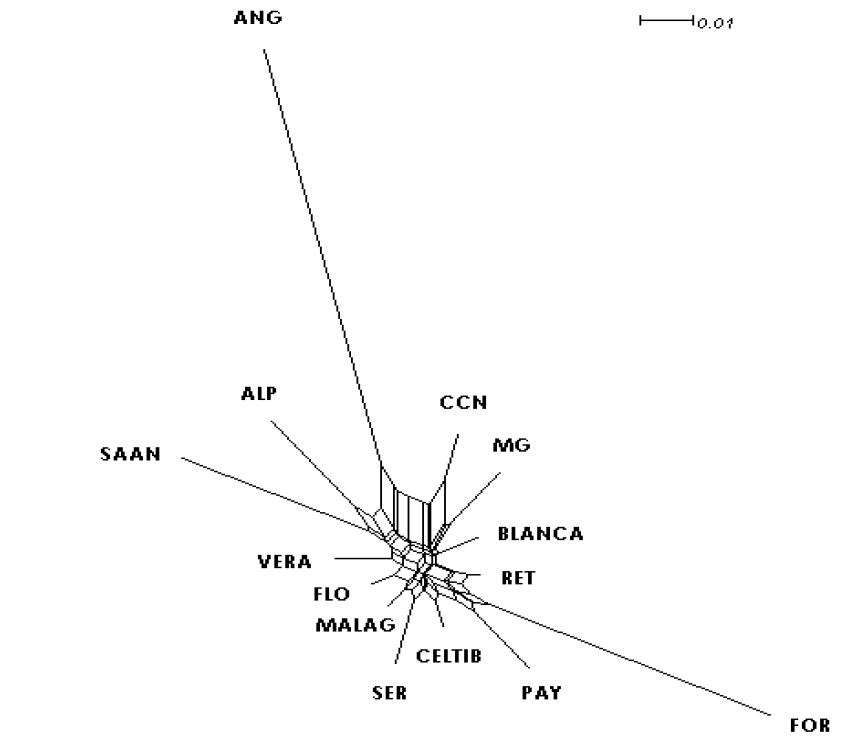
RET= Retinta, VERA= Verata, BLANCA= Blanca serrana, CELTIB= Celtibérica, MALAG= Malagueña, MG= Murciano granadina, FLO= Florida, PAY= Payoya, SER= Serrana, FOR= Formentera, SAAN= Saanen, ALP= Alpina, ANG= Anglonubia, CCN= Criolla negra.
Figura 1: Dendrograma Neighbor-Net construido con la distancia genética de Reynolds entre 14 razas caprinas
Análisis de la estructura genética en las poblaciones evaluadas
El K óptimo para la estructura genética de las poblaciones incluidas en el estudio fue de 9. La representación gráfica del resultado de la estructura genética obtenida de las 14 razas de cabras incluidas en el estudio se presenta en la Figura 2. En la cual, cada individuo está representado por una línea vertical, misma que se divide en segmentos de color que representan la fracción de pertenencia de cada individuo a cada uno de los grupos (K).
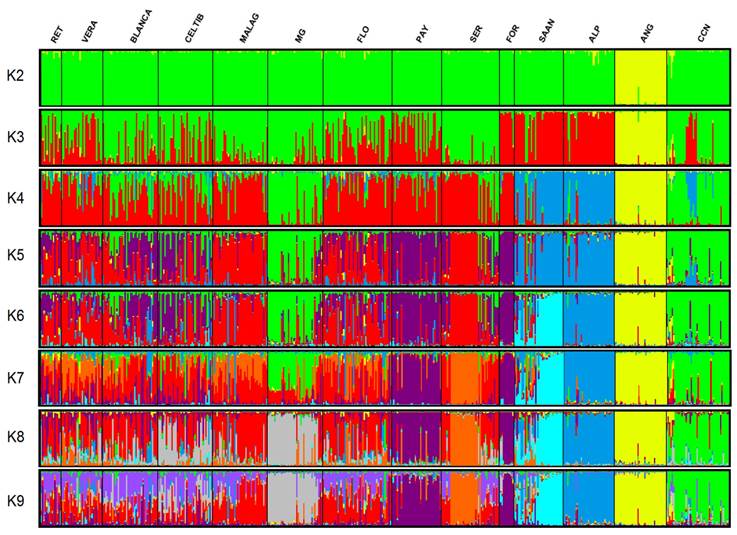
RET=Retinta, VERA= Verata, BLANCA= Blanca serrana, CELTIB= Celtibérica, MALAG= Malagueña, MG= Murciano granadina, FLO= Florida, PAY= Payoya, SER= Serrana, FOR= Formentera, SAAN= Saanen, ALP= Alpina, ANG= Anglonubia, CCN= Criolla negra.
Figura 2: Representación gráfica de la estructura genética de las 14 razas analizadas, asumiendo un número de poblaciones ancestrales K que van de 2 a 9
Discusión
Variación genética en la población Criolla Negra
Se ha recomendado que los microsatélites utilizados tengan por lo menos cuatro alelos y que el NEA sea superior a dos para ser considerados 7en estudios de diversidad y reducir el error estándar en la estimación de distancias genéticas38. En el presente estudio solo dos marcadores (MAF209 y SPS115) resultaron con 2 y 3 alelos. Respecto al NEA ETH225 y MAF209 presentaron valores menores a dos (1.41 y 1.25 respectivamente). Lo anterior nos indica que tan apropiados fueron los marcadores para la evaluación de la diversidad genética respecto al NA y NEA. El NMA proporcionó información sobre la diversidad genética de la población de CCN, a mayor número de alelos mayor diversidad y viceversa, el resultado obtenido (8.1) se considera alto si se compara con otros trabajos de caracterización como el de la cabra criolla cubana39, cabras saudíes19, cabras cachemira en China40,41 y algunas razas de cabras iraníes42. Sin embargo, tiene valores cercanos al NMA obtenido para cabras lecheras en Sudáfrica43. El CIP de los marcadores utilizados reveló un valor promedio (0.66) similar al obtenido en el trabajo de la cabra Retinta extremeña44.
Otra manera de apreciar la diversidad genética es mediante los valores de heterocigosis, ya que dependen del número y de las frecuencias relativas de los alelos45. Los valores de heterocigosis promedio obtenidos (He= 0.71 y Ho= 0.62) se muestran muy similares a los observados en la cabra Blanca Andaluza46 y Retinta Extremeña44, aun cuando esta última presenta un MNA mayor a la CCN. Los valores obtenidos para He y Ho indican un nivel importante de variabilidad genética para la CCN teniendo en cuenta el estado de reducción de la población en el que se encuentra y donde se esperaría un marcado nivel de consanguinidad. La prueba para el HWE reveló 14 marcadores con desviaciones significativas (P≤0.05), lo cual indica que en estos existe un déficit de heterocigosis. El hecho que algunos de estos marcadores se comporten homocigóticos puede deberse a acciones que van desde las condiciones de manejo, como el préstamo de sementales y el poco flujo genético en cada rebaño hasta marcadores que pudieran estar vinculados a rasgos productivos debido a selección enfocada a producción de leche y ganancias de peso sin importar relaciones de parentesco47.
Diferenciación genética entre poblaciones
Tomando en cuenta que la estimación de FIS y FIT pueden variar de 1 a -1 y que valores positivos indican una deficiencia de heterocigotos y valores negativos un exceso. Los resultados obtenidos para ambos índices (FIS=0.067 y FIT=0.121) indican que algunos de los marcadores en las razas resultaron homocigotos. Aun cuando los valores son cercanos a cero, estos indican un posible apareamiento entre parientes, resultado que concuerda con lo observado en cabras autóctonas de China48, India47, España y Portugal24. El FST indicó que el 94.2% de la variabilidad genética en las razas estudiadas se debía a diferencias entre los individuos dentro de la raza y el 5.8% a diferencias genéticas entre las razas. Este efecto se muestra de forma parecida con el coeficiente de diferenciación genética (GST= 0.073) que indicó que el 7.3% de la diversidad genética total se dividió entre las razas y por consiguiente la mayor parte de la variabilidad se encontró dentro de las poblaciones con un porcentaje del 92.7%. La diferencia obtenida entre ambos indicadores se debe a que FST refleja las propiedades de la distribución de las frecuencias alélicas entre las poblaciones, mientras que GST se define en términos de las frecuencias de la población49. Los valores obtenidos de FST y GST hacen referencia al nivel de variación genética que se ha mantenido en las razas incluidas en el estudio. Porcentajes similares a los obtenidos en el presente estudio se observaron para otras poblaciones caprinas17,42,50.
Distancia genética entre poblaciones y su representación gráfica
El dendrograma Neighbor-Net mostró que las razas caprinas españolas incluidas en el estudio (Retinta, Verata, Blanca Serrana, Celtibérica, Malagueña, Murciano-Granadina, Florida, Payoya, Serrana, Formentera), se mantienen agrupadas, este efecto se ha observado también en otros trabajos51 y se debe a la estrecha relación genética y geográfica que guardan estas razas. Las razas Saanen y Alpina también se visualizaron agrupadas hacia un extremo del dendrograma, sin embargo, es notoria la distancia genética obtenida para la raza Anglonubia. Esta relación también se pudo observar en una comparación entre estas razas y cabras brasileñas52, dicho efecto puede atribuirse a una mayor distancia genética con las razas comparadas y no precisamente a relaciones de origen o parentesco. Sin embargo, hay que tomar en cuenta que los animales de cada población han sido seleccionados también en base a sus características morfológicas. Las estimaciones obtenidas de la distancia genética de Reynolds también mostraron que la distancia más corta para la CCN se encuentra en relación con la raza MG (0.038), este resultado sugiere la posible relación genética entre ambas razas.
Análisis de la estructura genética en las poblaciones evaluadas
Con el análisis de la estructura genética se evaluó el grado de relación entre las diferentes poblaciones, detectando diferencias entre razas por medio del K óptimo (K=9). El análisis indica por un lado que la CCN no muestra signos de cruzamientos con el resto de las razas incluidas en el estudio y por otro lado se evaluó el grado de relación entre las diferentes poblaciones. La diferenciación genética de la población ANG, que se separa desde K2 y se mantiene separada en todos los supuestos debido a que presenta las distancias genéticas mayores, es similar a lo obtenido con la estructura poblacional en cabras criollas de las Américas53. Este resultado hace notar que la población ANG mantiene su estructura genética con un bajo nivel de mezcla de individuos de las otras 13 poblaciones evaluadas. En las razas españolas incluidas en el estudio, se presentó una estructura genética con más individuos entremezclados, muy similar a lo observado en un estudio de biodiversidad caprina24. Esto pudiera deberse a que son poblaciones geográficamente cercanas, lo que facilita la migración de individuos de otras poblaciones. Respecto a la CCN, ésta se observa similar a la cabra MG lo que podría confirmar el supuesto origen de la población. Sin embargo, en el K óptimo (K9) la población CCN presenta una estructura totalmente diferente a la raza MG y al resto de las poblaciones incluidas en el estudio. Este resultado sustenta lo observado en el dendrograma Neighbor-Net y sugiere que la CCN mantiene una estructura racial única y diferenciada de poblaciones que pudieron participar en su origen.
Conclusiones e implicaciones
El presente trabajo presenta los primeros resultados sobre la diversidad y estructura genética de una población de CCN. Los resultados sugieren que esta población presenta cierto grado de diversidad genética dado su nivel de polimorfismo encontrado en la población. Las distancias genéticas de la CCN con respecto a las otras razas incluidas en el estudio, indican que esta población se encuentra claramente diferenciada de las anteriores, motivo por el cual deberá considerarse como una raza de cabras mexicana. La reducida distancia obtenida entre MG y CCN sugiere que ambas poblaciones mantienen un ancestro en común, muy seguramente la raza Granadina. Se puede concluir que la cabra Criolla Negra presenta una estructura racial definida y diferenciada de las razas que pudieron darle origen, siendo ésta la primera raza de cabras mexicanas dentro del mosaico del centro del país descrita desde el punto de vista genético.











 texto en
texto en 


