El virus de la leucosis bovina (BLV) es el agente etiológico de la leucosis bovina enzoótica (LBE), pertenece a la familia Retroviridae, subfamilia Orthoretrovirinae, género Deltaretrovirus1. Los animales infectados con el virus generalmente no presentan signos visibles; sin embargo, la exoftalmia es el signo más específico de la enfermedad, en la cual se produce la degeneración del tejido retro ocular y de las estructuras internas del ojo2. Entre el 30 al 70 % los animales infectados con el BLV pueden desarrollar linfocitosis persistente (LP), la cual se caracteriza por la proliferación de linfocitos B. Sólo entre el 0.1 al 10 % de los bovinos con más de tres años de infección, presentan la forma tumoral de la enfermedad o linfosarcoma (LS), en la cual los nódulos linfáticos aumentan de tamaño3,4. El virus está ampliamente distribuido en el mundo; en América presenta prevalencias serológicas del 83.9 % en Estados Unidos5, 10.4 % en Uruguay6, 59 % en Chile7 y 41.8 % en Costa Rica8. En Colombia, la seroprevalencia en el 2004 fue del 44 %9. Para el 2014 se tomaron muestras de 8,150 bovinos en todo el territorio colombiano, de los cuales 3,840 presentaron anticuerpos contra el BLV, lo que equivale a una seroprevalencia del 42.7 %. De los 390 hatos evaluados, 264 presentaron infección por el virus; es decir el 67.7 %10. Actualmente no existe una vacuna comercial o tratamiento para LBE, por tanto, la erradicación y control de esta enfermedad se basa en el diagnóstico temprano y en la separación de los animales infectados. Las pruebas serológicas como ensayo por inmunoabsorción ligada a enzimas (ELISA) e inmunodifusión en gel de agar (AGID) son las pruebas recomendadas por la Organización Mundial de Sanidad Animal (OIE) para el diagnóstico de la infección4. La prueba diagnóstica AGID es ampliamente usada para la detección de anticuerpos contra los antígenos p24 o gp51 que corresponden a las proteínas de superficie del virus11; de igual forma la prueba ELISA también ha sido usada para detectar anticuerpos de BLV en muestras de suero, leche y remplazadores de calostro12. La reacción en cadena de la polimerasa (PCR) es una metodología altamente sensible de diagnóstico directo para la infección con BLV13,14,15. Sin embargo, los resultados de ambas pruebas de diagnóstico (ELISA y PCR) pueden variar considerablemente de acuerdo con diferentes aspectos relacionados con las técnicas empleadas, como: las condiciones fisiológicas de los animales (estado de preñez, edad), el kit empleado para la prueba serológica, el método de extracción del ADN, entre otros13. Los análisis de sensibilidad, especificidad y concordancia evalúan el desempeño y confiabilidad de la prueba, permitiendo dirigir con racionalidad su uso y aplicación16. Estos métodos cuantitativos son importantes para demostrar la eficacia de una prueba diagnóstica. El objetivo de este trabajo fue detectar el virus de leucosis bovina a través de una prueba molecular (PCR) y una prueba serológica (ELISA) en una población de vacas Holstein de lechería especializada en el departamento de Antioquia.
Esta investigación es de tipo descriptivo y sólo se evaluó la muestra poblacional en un momento de tiempo. Se tomó muestra de sangre por punción de la vena coccígea media de 1,000 vacas Holstein entre primer y quinto parto con edades de 3 a 7 años, las cuales pertenecen a 23 hatos de lechería especializada, distribuidos en 10 municipios del Departamento de Antioquia, Colombia: Subregión Valle de Aburra (Bello, Medellín), Subregión Norte (Belmira, Don Matías, Entrerríos, Medellín, San Pedro de los Milagros y Santa Rosa de Osos), Subregión Oriente (Rionegro, La Unión, El Retiro). Las muestras se recolectaron durante los meses de febrero a junio de 2013. Las condiciones específicas de manejo, alimentación y sanidad son variables y dependen del manejo de cada hato. Para la toma de muestras se obtuvo la aprobación por parte del comité de ética de la Universidad Nacional de Colombia (CEMED-007, 14 de mayo 2012).
La toma de muestras se hizo con una aguja calibre 18 G con sistema al vacío vacutainer (VACUETTE®) y EDTA como anticoagulante; las muestras se homogenizaron por inversión y se trasladaron al laboratorio en condiciones de refrigeración para realizar la recolección de plasma y la extracción de ADN. Las muestras se llevaron a un tubo cónico de 15 ml y se centrifugaron durante 4 min, 3,000 rpm, 4 °C; se recuperó el plasma y se almacenó a -20 °C hasta su uso. Para obtener el ADN de los leucocitos se realizó la técnica salting out17 y se resuspendió en buffer TE 1X pH 8.0 (Tris HCl 1 M y EDTA 0,5 M), el cual se almacenó a 4 °C hasta el momento del análisis. La calidad y cantidad del ADN obtenido se evaluó en un espectrofotómetro (NanoDrop2000®, Massachusetts-Estados Unidos) y en gel de agarosa al 1%.
El método ELISA se realizó con un kit comercial (SVANOVIR® BLV gp51-Ab) de acuerdo con las instrucciones del fabricante. Se adicionaron 4 μl del reactivo A (control positivo) y 4 μl del reactivo B (control negativo) en los pozos para los controles. Se adicionaron 4 μl de plasma de cada muestra en los otros pozos. Se agitó la placa y se incubó a 37 °C por 1 h. Se lavó la placa y se adicionaron 100 μl del conjugado en cada pozo y se incubó a 37 °C por 1 h. Se repitió el lavado y se adicionaron 100 μl de solución sustrato, se incubó 10 min a 25 °C. Se adicionaron 50 μl de solución de parada. Se midió la densidad óptica (DO) de los controles y las muestras a 450 nm en un espectrofotómetro para microplacas (BioTek® ELx800).
La detección del genoma viral por PCR se determinó a partir del ADN de las muestras de células sanguíneas (leucocitos) de las 1,000 vacas. Se amplificó una región del gen env (gp51) viral para obtener un fragmento de 444 pb utilizando los oligonucleótidos reportados por Beier, et al18. La primera ronda de PCR se realizó en un volumen final de 25 µl con 150 ng de ADN, 3.0 µl de 10 mM de cada oligonucleótido env 5023 (5´-TCTGTG CCAAGTCTCCCAGATA-3´) y env 5608 (5´-AACAACAACCTCTGGGGAGGGT-3´), 0.4 mM de dNTPs, 1X de tampón PCR (ThermoScientific®), 3 mM MgCl2 y 1U de Taq ADN polimerasa. En la segunda reacción de PCR se utilizó como ADN molde 5 µl del producto de PCR de la primera amplificación, con las mismas concentraciones de los otros reactivos y los oligonucleótidos env 5099 (5´-CCCACAAGGGCGGCGCCGGTTT-3´) y env 5521 (5´-GCGAGGCCGGGTCCAGAGCTGG-3´), en un volumen final de 30 µl. Las reacciones para las dos PCR fueron idénticas y se realizaron en un termociclador T3 (Biometra®) con el siguiente protocolo: desnaturalización inicial fue a 94 °C durante 5 min, seguido de 40 ciclos de 94 °C por 30 seg, 60 °C por 30 seg y 72 °C por 1 min, para terminar con una extensión final a 72 °C por 5 min. El producto de la segunda reacción se verificó en un gel de agarosa al 2% en un documentador de geles (Biorad®). Como control negativo se hicieron reacciones en ausencia de ADN y como control positivo se usó inicialmente el ADN de una vaca positiva para leucosis por la técnica de ELISA; después de esto se utilizó el ADN de una de las vacas que resultó positiva para la prueba molecular. El producto de PCR definido como control positivo fue secuenciado (Macrogen®, USA) y se verificó su identidad a través del análisis de similitud nucleotídica usando el algoritmo BLAST®. Se determinó un 99 % de identidad entre nuestra secuencia y la secuencia completa del virus de la leucosis bovina (LC164085.1), así como con la secuencia parcial de la glicoproteína de superficie gp51 (KU233560.1).
De acuerdo con la DO se consideraron positivas a la presencia de anticuerpos contra la proteína del BLV (gp51), aquellas muestras que obtuvieron un valor positivo porcentual (PP) mayor o igual a 15 (de acuerdo con los criterios del fabricante). Se utilizó una tabla de frecuencias para establecer la prevalencia del BLV. Se compararon los porcentajes para establecer las diferencias entre las subregiones, municipios y hatos utilizando tablas de contingencia. Se aplicó la prueba de independencia Ji-cuadrada (X 2 ) y razón de momios (OR) a través de una regresión logística, para determinar la relación entre el diagnóstico de la infección por ELISA, el PCR y el lugar de origen de la muestra (hato, municipio o subregión). Si el resultado del OR para el municipio fue mayor a 1, hay una asociación positiva y por tanto las vacas tienen menor posibilidad de estar infectadas con el BLV. Si el resultado del OR para el municipio fue menor a 1, hay una asociación negativa y por lo tanto las vacas tienen mayor posibilidad de estar infectadas con el BLV. La significancia del OR se evaluó con Ji-cuadrada (X 2 ) (P<0.05). Se utilizó una tabla de contingencia (2x2) para establecer los parámetros de sensibilidad y especificidad. La prueba de concordancia Kappa se utilizó para determinar la eficiencia de las dos metodologías diagnósticas. Los análisis se hicieron empleando el programa estadístico EPIDAT 3.1® y en el programa SAS® 9.2 para Windows (SAS InstituteInc, Cary NC, USA) todo con un nivel de confianza de 95%.
Se obtuvieron muestras de 10 municipios dedicados a lechería especializada, distribuidos en tres subregiones del departamento de Antioquia; la Subregión Norte tenía 640 animales distribuidos en 17 hatos, la Subregión Valle de Aburrá tenía 234 animales distribuidos en tres hatos y la subregión Oriente tenía 126 animales distribuidos en tres hatos (Figura 1). La presencia o ausencia del BLV estuvo relacionada con el lugar de origen o procedencia de la muestra. Las dos metodologías de detección fueron significativas para la presencia del BLV en los diferentes hatos (X 2 =777.6420 P<0.0001), municipios (X 2 =500.5000 P<0.0001) y la subregión evaluada (X 2 =29.2811 P<0.0001).
En Colombia el primer reporte de la enfermedad fue en 1957, a partir de muestras clínicas que llegaban a centros de veterinarios19; las primeras pruebas arrojaban seroprevalencias en ganado de leche de 24.9 % para la región Andina; 14.4 % en la región Caribe y 15.3 % para el Piedemonte Llanero19. El BLV ha aumentado su prevalencia en la región Andina, en la cual se encuentra el departamento de Antioquia. La mayor prevalencia molecular para el BLV se encontró en la subregión Oriente con el 69.79 % de infección, seguido de la subregión Norte con una prevalencia del 49.19 % y de la subregión Valle de Aburrá con el 32.44 % de infección. Estos resultados fueron similares a los encontrados por la técnica serológica ELISA, en los cuales la mayor seroprevalencia se registró en la subregión Oriente (79.17 %), seguido de la subregión Norte (53.74 %) y la subregión Valle de Aburrá (46.67 %).
Con el uso de la PCR se logró detectar 47.6 % de muestras como positivas al BLV, mientras que el ELISA logró detectar un 54.6 %. La mayor prevalencia molecular se presentó en el municipio de Rionegro, seguido de los municipios de El Retiro, y Medellín. La mayor prevalencia serológica se presentó en el municipio de El Retiro, seguido de los municipios de Medellín y Rionegro (Figura 2).
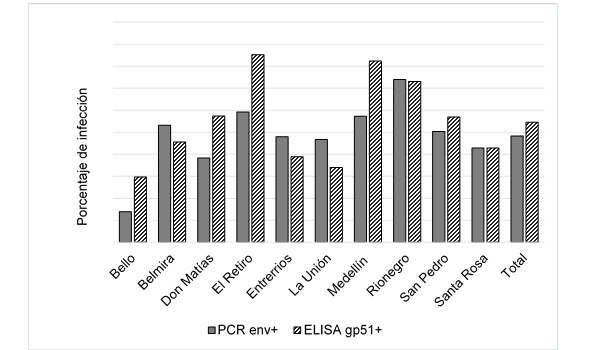
Figura 2: Porcentajes de infección al virus de leucosis bovina por las técnicas PCR y ELISA por municipio
Se determinó el OR para las técnicas de detección serológica y molecular respecto a los municipios. Con el uso de la técnica ELISA se encontró que los animales que pertenecen a los municipios de Bello (OR=2.972, P<0.0001) y Entrerríos (OR=1.205 P<0.0001) tienen una menor posibilidad de presentar la infección por el BLV; mientras que los municipios de Belmira (OR=0.784, P<0.0311), El Retiro (OR=0.617, P<0.609), La Unión (OR=0.936, P<0.0008), Medellín (OR=0.669, P<0.0001) y Rionegro (OR=0.399, P<0.0210) tienen una mayor posibilidad de presentar la infección por el BLV. Por otra parte, con el uso de la técnica PCR se encontró que los bovinos que pertenecen al municipio de Bello (OR=2.863, P<0.0001) tienen una menor probabilidad de tener la infección por el virus, mientras que los animales que pertenecen al municipio de Rionegro (OR=0.446, P<0.0018) tienen una mayor probabilidad de estar infectados con el BLV (Cuadro 1).
Cuadro 1: Razón de momios (OR) para los 10 municipios del departamento de Antioquia de acuerdo con la técnica de detección (ELISA o PCR)
| Municipio | ELISA | PCR | ||||||
|---|---|---|---|---|---|---|---|---|
| OR | CI (95%) | P> ChiSq | OR | CI (95%) | P> ChiSq | |||
| Bello | 2.972** | 3.044 | 7.455 | <0.0001 | 2.863** | 3.324 | 9.277 | <0.0001 |
| Belmira | 0.784* | 0.427 | 0.864 | 0.0311 | 0.599 | 0.567 | 1.111 | 0.2238 |
| Don Matías | 1.446 | 0.493 | 1.609 | 0.9504 | 0.896 | 0.796 | 2.648 | 0.1456 |
| El Retiro | 0.617* | 0.072 | 0.609 | 0.0040 | 0.211 | 0.285 | 1.348 | 0.2221 |
| Entrerrios | 1.205** | 1.000 | 1.992 | <0.0001 | 1.904 | 0.692 | 1.376 | 0.1646 |
| La Unión | 0.936** | 1.400 | 5.067 | 0.0008 | 2.344 | 0.566 | 1.874 | 0.9279 |
| Medellín | 0.669** | 0.151 | 0.444 | <0.0001 | 0.260 | 0.440 | 1.025 | 0.0723 |
| Rionegro | 0.399* | 0.205 | 0.644 | 0.0210 | 0.446* | 0.183 | 0.552 | 0.0018 |
| San Pedro de los Milagros | 0.851 | 0.770 | 1.334 | 0.7826 | 0.915 | 0.675 | 1.167 | 0.3693 |
| Santa Rosa de Osos | 1.196 | 0.557 | 3.143 | 0.0397 | 1.613 | 0.502 | 2.877 | 0.4501 |
CI= Intervalo de confianza, ChiSq= Jii cuadrada. Los municipios con mayor probabilidad de presentar la infección con el BLV* son aquellos que presentaron OR>1 y P<0.05. Los municipios con menor probabilidad de presentar la infección con el BLV* son aquellos que presentaron OR<1 y P<0.05 de presentar la infección con el BLV* son aquellos que presentaron OR>1 y P<0.05. Los municipios con menor probabilidad de presentar la infección con el BLV* son aquellos que presentaron OR<1 y P<0.05.
La prevalencia molecular para región centro del país fue 50.7 % (37/73); además el ganado de leche presentó el mayor número de infectados (37/73) respecto al ganado de carne (5/85)20. La prevalencia molecular de BLV para la raza Holstein en el departamento del Valle del Cauca fue del 83.3 %21, mientras que en 2012 se encontró un resultado similar con el 77.8 % de positividad en este mismo departamento22. La subregión y el municipio están altamente relacionados con la presencia del virus; otros estudios también han establecido la dependencia entre el lugar de origen y la prevalencia de infección a BLV21. Para los hatos de Antioquia, una región de lechería especializada, este estudio determinó una prevalencia molecular del 47.6 % menor a la registrada en el Valle del Cauca utilizando el mismo método diagnóstico, lo cual puede estar relacionado tanto con el lugar de origen de los animales muestreados, como con las diferencias en los sistemas de producción, ya que Antioquia cuenta con una lechería más especializada en cuanto a estructura y manejo de los animales. De acuerdo con el estudio más reciente, la seroprevalencia para Colombia es del 42.7 %, mientras que para el departamento de Antioquia el municipio de San Pedro presentó una seroprevalencia del 53.9 %10, similar a la encontrada en el presente estudio (54.6 %). La diseminación de la infección por el BLV en los hatos lecheros está relacionada con fallas en las prácticas de manejo sanitario; se ha demostrado que adecuadas prácticas iatrogénicas durante el ordeño y la vacunación (uso de una aguja por animal) son factores de protección contra la infección por el BLV10.
La técnica PCR detectó 474 vacas positivas al BLV y 526 negativas al BLV. La prueba serológica ELISA detectó 546 bovinos positivos para BLV y 454 vacas negativas al BLV. Del total de vacas evaluadas 389 fueron negativas tanto por ELISA como por PCR y 409 vacas fueron positivas por ELISA y PCR. Por otra parte, 137 vacas fueron positivas por ELISA más no por PCR y 65 vacas fueron positivas por PCR más no por ELISA. El valor de concordancia entre las pruebas fue de 0.59; este valor es considerado como moderado de acuerdo a la escala de valoración propuesta por Landis y Koch23 (Cuadro 2); sin embargo, en un estudio por conglomerados realizado en el país, se encontró una concordancia del 0.8 o excelente20, la diferencia entre ambos valores puede estar dado por el lugar de origen de las muestras, el tipo de estudio y en número de animales evaluados.
Cuadro 2: Análisis de sensibilidad, especificidad y concordancia entre la PCR y el ELISA para el diagnóstico de leucosis bovina
| Valor | IC (95%) | ||
| Sensibilidad, % | 74.91 | 71.18 | 78.64 |
| Especificidad, % | 85.68 | 82.35 | 89.01 |
| Valor predictivo positivo, % | 86.29 | 83.08 | 89.49 |
| Valor predictivo negativo, % | 73.95 | 70.11 | 77.80 |
| Índice de Youden | 0.61 | 0.56 | 0.65 |
| Concordancia: kappa | 0.59 | 0.54 | 0.64 |
| Acuerdo observado | 0.79 | ||
| Acuerdo esperado | 0.49 | ||
La concordancia muestra que ambas técnicas de detección (PCR y ELISA) coinciden en 78 % de los resultados obtenidos, es decir, que ambas técnicas detectan un número similar de animales positivos y negativos para la infección por BLV. La sensibilidad de la PCR con relación al ELISA (estándar de oro, de acuerdo con la OIE) fue 74.91 % y presentó una especificidad de 85.68 %. Otros estudios han reportado sensibilidades del 62.7 %24, 84.3 %20 y del 97 %25 para diagnóstico del BLV por estas dos metodologías (ELISA y PCR). Los resultados muestran que el valor predictivo positivo es mayor que el resultado obtenido por sensibilidad, es decir que se tiene el 86.29 % de las vacas positivas por PCR realmente tienen el BLV integrado como provirus en los linfocitos B.
Aunque la PCR deja de detectar el 25.09 % (137/546) de los bovinos infectados respecto al ELISA, esto puede ser por la baja carga proviral integrada en los linfocitos B de los bovinos infectados. Sin embargo, se ha demostrado que la amplificación y detección de secuencias ADN o ARN de BLV mediante PCR constituye una metodología altamente sensible de diagnóstico directo para la infección con BLV13,14,15; este método se basa en secuencias cebadoras del gen env que codifica la proteína gp51, este gen está altamente conservado y tanto el gen como el antígeno están generalmente presentes durante las diferentes fases de la infección26.
Las técnicas de detección PCR y ELISA presentan algunas desventajas que pueden afectar la veracidad de los resultados obtenidos. En el caso del ELISA se pueden encontrar animales con baja cantidad de inmunoglobulinas debido a que la célula blanco del virus son los linfocitos B. En otros casos la infección puede estar dada por un serotipo adaptado contra el cual no se generen anticuerpos27, o cuyos antígenos no sean detectados por la técnica debido a mutaciones en el epítope25. La técnica de detección PCR puede estar afectada por sustancias inhibidoras durante la reacción, por la cantidad de linfocitos periféricos que resultan infectados por el virus, o cuando se desarrolla una infección restringida a órganos linfoides28. Sin embargo, la prueba de detección ELISA es reconocida por la Organización Mundial de Sanidad Animal (OIE) para el diagnóstico del BLV4 debido a su simplicidad y sus ventajas prácticas y económicas25, por otra parte la PCR permite detectar animales infectados con el BLV antes que desarrollen una respuesta inmunológica, al igual que en animales que con baja carga proviral o con bajos títulos de anticuerpos.
El lugar de origen de la muestra (hato, municipio o subregión) está altamente relacionado con la presencia o ausencia del virus; lo que pude estar relacionado con diferentes prácticas de manejo y factores ambientales. La subregión Oriente del departamento de Antioquia presentó la prevalencia molecular y serológica más alta. A nivel de municipio las mayores prevalencias de la enfermedad se encuentran en Belmira, El Retiro, La Unión, Medellín y Rionegro; además los animales que pertenecen a estos municipios tienen una mayor probabilidad de presentar la infección por BLV.
La concordancia entre las pruebas de detección serológica y molecular evaluadas fue moderada, por tanto aunque la especificidad y sensibilidad de la técnica molecular PCR son inferiores respecto al ELISA, ambas técnicas coindicen en un 78 % de los resultados. Ambas técnicas de detección pueden ser aplicadas en los hatos lecheros como herramientas importantes para conocer el estado sanitario de la enfermedad. Por otra parte, los resultados de este trabajo demuestran que más del 50 % de los animales evaluados presentaron títulos contra el BLV y el provirus integrado en su genoma; este resultado presenta una visión global del nivel de infección por BLV en los hatos de lechería especializada del departamento de Antioquia.











 nueva página del texto (beta)
nueva página del texto (beta)



