Una de las causas de grandes pérdidas económicas en la industria avícola son las enfermedades reproductivas y respiratorias de las aves, las cuales causan aumento en los costos por el tratamiento y mortalidad1. La bacteria Gram negativa de Gallibacterium anatis causa salpingitis y peritonitis en gallinas ponedoras, lo cual disminuye la producción de huevo2, ocasionando problemas en la avicultura moderna por presentar lesiones como atrofia folicular, ooforitis, septicemia, folículos deformes, peritonitis, agrandamiento de los riñones, enteritis, congestión, así como edema del tracto respiratorio3,4,5. Mirle et al1 establecieron que Gallibacterium también ocasiona alteración en el tamaño del huevo, variación en la formación del cascarón y bajas significativas en la producción del 2 al 4 %, ocasionando grandes pérdidas económicas y aumento en los costos de producción por tratamientos, por lo que representa un gran reto sanitario y económico para la industria avícola mexicana. La compleja patogenia de G. anatis ha sido objeto de revisiones6. Es poco lo que se sabe hoy en día de la patogénesis y son pocos los factores de virulencia que se han identificado, como la toxina RTX7, las metaloproteasas con capacidad de degradar la IgG aviar8, y la fimbria F17-like9. Así mismo, se ha demostrado una incidencia del 48 al 61 % y una alta prevalencia con el 95 de significancia, la cual es afectada por los niveles de bioseguridad3,10.
Anteriormente G. anatis era considerado un agente secundario1, debido a que este patógeno se encuentra en la flora normal de pollos sanos, por lo que causa intriga en cuanto a su patogenicidad primaria, que probablemente se encuentre influenciada por factores adicionales que causan la inmunosupresión de las aves, permitiendo que el agente oportunista de G. anatis actúe negativamente sobre el huésped3,11,12, sugiriendo que es una bacteria oportunista que puede convertirse en el agente primario para el desarrollo de la enfermedad1,3,5.
Aunado a esto, es importante conocer los factores de patogenia y virulencia para mejorar, entender y prevenir los efectos negativos de G. anatis2; como también, la aparición de nuevas enfermedades, la modificación de la virulencia y la medicación desmedida, las cuales han contribuido a la resistencia y susceptibilidad antimicrobiana a los antibióticos, complicando la enfermedad; observando aumento de resistencia antimicrobiana entre varios miembros de la familia Pasteurellaceaes13,14.
El diagnóstico oportuno de esta enfermedad, podría prevenir algunos inconvenientes. Para ello, se debe integrar el trabajo de campo considerando la observación de signos clínicos, los resultados obtenidos a partir de las muestras enviadas al laboratorio, la identificación del agente y el diagnóstico diferencial con otras enfermedades. De igual manera un tratamiento eficaz con antibióticos específicos permitiría mantener altos niveles de salud animal. Sin embargo, en México, los productores de aves, suelen fracasar en el tratamiento con antibióticos utilizados contra la infección de G. anatis15, debido a que los organismos pertenecientes a la familia de las Pasteurellaceae muestran resistencia recurrente debido al uso desmedido de antibióticos no específicos13. Es por esto, que el objetivo del presente trabajo fue determinar la susceptibilidad y resistencia a diferentes antibióticos, incluyendo los más utilizados para el tratamiento de G. anatis.
Al iniciar la investigación, fue necesario aislar la bacteria a partir de muestras de tejidos. Los aislamientos, se obtuvieron de 189 granjas avícolas establecidas en el estado de Sonora, las cuales están integradas por múltiples casetas, durante el periodo 2007-201116. Se seleccionaron 30 granjas de postura comercial de forma dirigida por presentar sintomatología de tipo respiratorio y reproductivo como referencia, donde se obtuvieron muestras de 20 aves. El procedimiento de necropsia se realizó de acuerdo a lo establecido en el manual de necropsias y toma de muestras de las aves17,18. Se buscaron lesiones de tipo reproductivo y respiratorio, tomándose muestras de diversos tejidos como los cornetes, tráquea, pulmones, hendidura palatina, hígado, bazo, riñones, folículos y peritoneo. La superficie del tejido muestreado se cauterizó con una espátula caliente antes de introducir el hisopo estéril con el objeto de prevenir cualquier tipo de contaminación, inoculándose posteriormente en cajas Petri de plástico estériles desechables, con agar sangre (BD Bioxon), con sangre estéril desfibrinada de bovino al 5% y agar Mac Conkey (BD Bioxon), incubándose a 37 °C de 18 a 24 h en una incubadora microbiológica (1535 Shel Lab). Posteriormente, se realizó la identificación bacteriana de los aislamientos mediante pruebas convencionales de cultivo de agar sangre de bovino al 5%, identificando características morfológicas y de hemólisis19. Las bacterias se purificaron y se seleccionaron morfológicamente en base a lo descrito por Christensen et al19, quienes mencionan que las colonias deben tener un diámetro de 1 a 2 mm, ser circulares, convexas y grisáceas traslúcidas. Las bacterias se colorearon con tinción de Gram, identificándose como cocobacilos Gram negativos, de acuerdo a lo establecido por Rimler et al20, en un microscopio Olympus CX41.
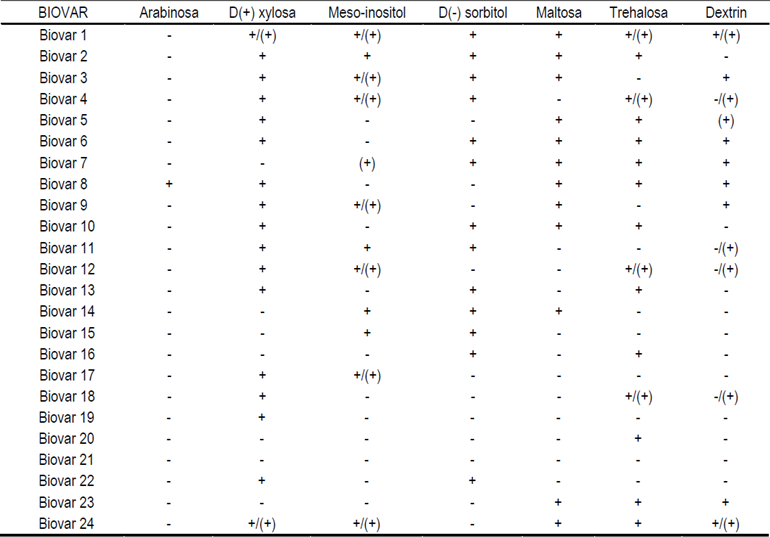
Así mismo, la identificación bioquímica se realizó de cultivos de menos de 24 h de vida, por medio del sistema comercial para cocobacilos Gram negativos (NC33) de MicroScan Renok™, según especificaciones del fabricante en el procedimiento en un equipo AS-4 con el software LABPRO MICROSCAN; las placas inoculadas (NC33) se incubaron a 37 °C durante 24 h en una incubadora 1535 Shel Lab. La interpretación fue visual; cada pozo se evaluó por su cambio de color específico, indicando así la presencia de una reacción metabólica basándose en la tabla de MicroScan RenokTM Gram negativos21, debido a que la base de datos del MicroScan no poseía referencias para G. anatis. Para complementar las pruebas y poder biotipificar las bacterias, se realizaron bioquímicas de cuatro carbohidratos más (glucosa, maltosa, trehalosa y D-xilosa) (BD Bioxon). Todas las bioquímicas se corrieron dos veces para detectar cualquier error efectuado durante el procedimiento. Así mismo, se ejecutó la prueba para buscar la presencia de la enzima oxidasa, con tiras reactivas (Bactident® Oxidase). La tipificación de los aislamientos se basaron en investigaciones anteriores, a partir de la fermentación de carbohidratos de (+) L-arabinosa, (+) D-xilosa, m-inositol, (-) D-sorbitol, maltosa, trehalosa y dextrina, diferenciando entre 24 biovares registrados19,22 (Cuadro 1).
Para complementar la identificación, se consideró identificar a G. anatis por la reacción en cadena de la polimerasa (PCR), y para esto fue necesario primero la extracción de ADN bacteriano genómico, proceso realizado mediante las instrucciones de QIAamp DNA Mini Kit (51306, QIAGEN), según especificaciones del proveedor. Para la cuantificación del ADN se utilizó un espectrofotómetro (Jenway, GENOVA) con celdas de cuarzo, a una absorbancia de 260 y 280 nm. Para la confirmación molecular de los aislamientos bacterianos mediante la amplificación por PCR de los segmentos específicos de los genes de G. anatis. Las secuencias de oligonucleótidos se diseñaron en base a las 99 secuencias de 16S RNAr del Gen Bank23. Se seleccionó el oligonucleótido 1133fgal (5’-TATTCTTTGTTACCARCGG), el cual se prevé que es específico para Gallibacterium y no tiene la capacidad de amplificar el ADN de otros miembros de la familia Pasteurellaceae, dentro de las condiciones del PCR elegido donde se espera un amplicón de 790 pb. Para la amplificación inversa se eligió el oligonucleótidos del gen 23S rRNA con secuencia 114r (5’-GGTTTCCCCATTCGG) reportados por Lane24 y Bojesen et al23, esperando un amplicón de 1080 pb. Para esto se utilizó un termociclador (Mastercycler® personal Eppendorf) en punto final. La reacción de PCR se llevó a la concentración final de 25 µl con 2.5 µl Mg 10X Buffer, 0.85 µl MgCl2 (25 mM), 0.5 µl DNTP’s mix (2.5 mM), Primer F 0.25 µl y Primer R 0.25 µl (50 µM), 0.25 µl Taq (5U/µl), 18.5 µl H2O libre de RNasas (Todos los reactivos del laboratorio InvitrogenTM), y 2 µl DNA de cada una de las muestras, obteniendo 25 µl de volumen total. Las condiciones de la PCR fueron las siguientes: la desnaturalización de 95 °C durante 4 min, seguido de 35 ciclos de 95 °C por 30 seg cada uno, alineamientos de 54 °C durante 1 min y 2 min a 72 °C, seguido por la extensión final de 72 °C durante 10 min. El amplicón esperado se amplificó en gel agarosa al 1.6% teñido con bromuro de etidio23, y posteriormente visualizados en un transiluminador UV (Macro Vue UV-25, Hoefer).
Para finalizar, el análisis de resistencia antimicrobiana se realizó mediante el método de Kirby-Bauer, descrito por el Laboratorio Internacional de Referencia: National Committe for Clinical Laboratory Standards (NCCLS)25. Se realizaron cultivos de los aislamientos de G. anatis en agar sangre de bovino al 5%, utilizando sensidiscos de papel que contenían una distribución homogénea de antibiótico, para posteriormente incubar a 37 °C por 18 a 24 h26.
Los antibióticos evaluados fueron los aminoglucósidos (gentamicina, estreptomicina, neomicina, kamicina, neo-kanamicina); betalactámicos (ampicilina, penicilina); fluoroquinolona (norfloxacina, enrofloxacina); cefalosporina (ceftiofur, cefalexina); fenicoles (florfenicol, tianfenicol); lincosamidas (lincoespectin, lincomicina); tetraciclinas (oxitetraciclina); macrólidos (tilosina); fosfonatos (fosfomicina) y sulfonamidas (sulfametoxazol). La concentración de los sensidiscos fue de 100 µg en los nitrofuranos, 5 µg en las fluoroquinolonas y 30 µg en todas las demás; todo esto es fundamentado en la Farmacopea Americana del Compendio de Normas o Estándares y Técnicas de Laboratorio. La interpretación de la susceptibilidad antimicrobiana se estableció en la medición del halo de inhibición, comparándolo con la concentración mínima inhibitoria (CIM)27.
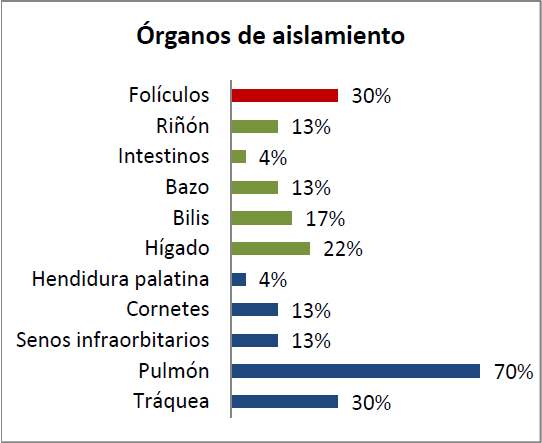
Se lograron aislar 23 cepas bacterianas de G. anatis, de las cuales la cepa de referencia y cuatro cepas más, fueron donadas de estudios anteriores. A pesar que se hicieron 600 muestreos, no se pudo aislar de todos los casos, posiblemente debido a que las aves se encuentran inmunizadas contra este microorganismo; sin embargo, el muestreo fue considerado exitoso, debido a que hubo un brote en el 2010-2011, lo que permitió que del 100 % de las bacterias aisladas, el 34 % correspondiera a G. anatis (Figura 1), ya que en otras circunstancias hubiera resultado complicado. El 70 % de las bacterias se asilaron de pulmón, 30 % en tráquea y 30 % en folículos (Figura 2), lo que indica que este microorganismo causa patologías respiratorias y reproductivas. Las bacterias se identificaron morfológicamente en agar sangre de bovino al 5%, apoyados en la técnica y morfología descrita por Christensen et al19.
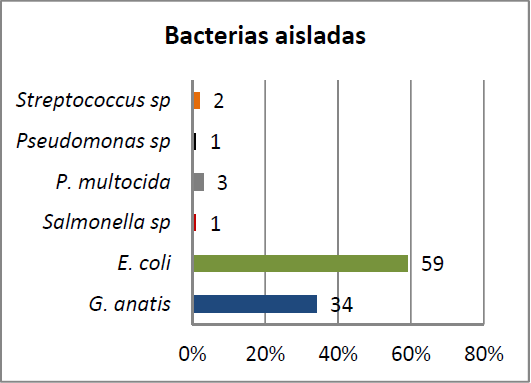
Figura 1 Porcentaje de bacterias aisladas en aves del sur de Sonora, durante en el período 2010 - 2011
En la identificación bioquímica se identificaron los aislamientos como G. anatis19,22,28, realizando repetición de las pruebas con la finalidad de disminuir errores.
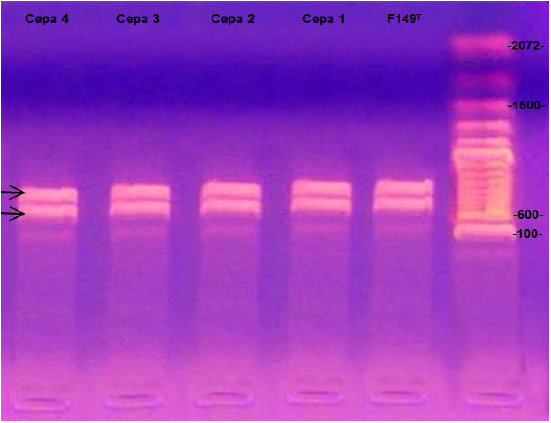
En la identificación molecular por PCR, se obtuvo una media de 20.5 µg/ml y una relación de pureza de 1.8 en la extracción de ADN bacteriano; con esto fue posible continuar con el proceso, donde los productos de amplificación de las bacterias aisladas obtuvieron 790 pb y un segundo amplicón de 1080 pb, donde están ubicados los segmentos específicos de G. anatis que codifican para los genes 16S rRNA y 23S rRNA, según lo establecido por Bojesen et al23, confirmando como positivas a las 23 cepas aisladas (Figura 3).
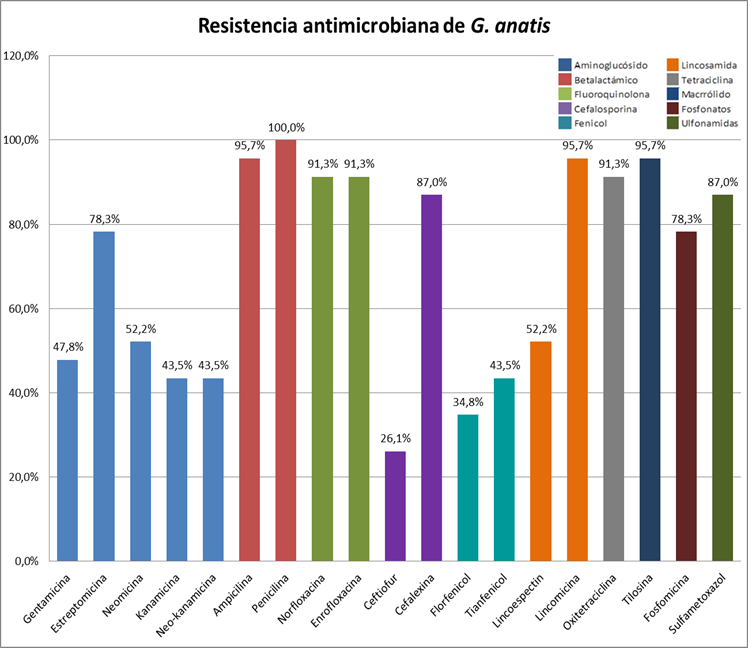
En el análisis general de la resistencia antimicrobiana de los 23 aislamientos, los resultados revelaron que las bacterias presentaban mayor resistencia a la penicilina con el 100 % y la ampicilina con el 95.7 %, ambos del grupo correspondientes a los betalactámicos. Lincomicina con el 95.7 % y también tilosina con el 95.7 % (Cuadro 2) (Figura 4). G. anatis reflejó más susceptibilidad a ceftiofur, con el 73.9 %, seguido de los fenicoles, donde se encuentra al florfenicol con el 65.2 % y al tianfenicol con el 56.5 %. En los antibiogramas realizados a cada una de las bacterias aisladas, todos los aislamientos, a excepción de dos, mostraron una resistencia mayor al 50 % de los antibióticos, demostrando, que la mayoría de los aislamientos de G. anatis en el estado de Sonora han creado resistencia marcada.
Los 23 aislamientos de G. anatis se obtuvieron de aves con patologías respiratorias y reproductivas; se identificaron macroscópicamente19, también se identificaron bioquímicamente. Esto se confirmó genotípicamente por la amplificación, mediante la PCR de los segmentos específicos que codifican para los genes 16S rRNA y 23S rRNA, estipulados en diversos métodos19,23,29. Se debió haber tenido un marcador molecular más específico para determinar los pares de bases con mayor exactitud y descartar del 1030 a 1080 propuesto por Bojesen et al23. La PCR puede ser útil para el diagnóstico de rutina de Gallibacterium. Si bien, el uso de los métodos moleculares es limitado debido a los altos costos de la PCR (3 veces más caro que una bacteriología), también ahorra tiempo sin tener que recurrir a las pruebas fenotípicas, las cuales suelen llevar días antes de obtener un resultado.
Cuadro 2 Susceptibilidad y resistencia de bacterias de Gallibacterium anatis aisladas en el Sur de Sonora, México

*Ilegibilidad de origen
La patogenia de G. anatis está poco definida, por lo que existen pocos estudios que expliquen la farmacocinética de este microorganismo, lo que ha inclinado al productor a medicar desmedidamente esta enfermedad, ocasionando que la bacteria desarrolle una resistencia marcada, como se ha demostrado30,31, al comparar los fenotipos de resistencia con los informes de otros taxones de Pasteurellaceae, encontrándose una alta multirresistencia de las tetraciclinas y sulfametoxazol del 92 y 97 % en los aislamientos de campo de Gallibacterium. Confirmando así los resultados obtenidos en el presente estudio, donde la mayoría de los aislamientos presentaron resistencia marcada con las tetraciclinas y los betalactámicos.
En este estudio, los aislamientos resultaron altamente resistentes, indicando la dificultad para el control de esta infección; la medicación no se debería de aplicar sin algún análisis de susceptibilidad antimicrobiano previo, para poder prescribir correctamente los antibióticos, considerando también la naturaleza del proceso infeccioso, la historia clínica, la vía de administración, la dosis, la duración del tratamiento, el mecanismo de acción del antibiótico y su toxicidad27. Sin embargo, aunque la resistencia parezca ser muy común entre los aislamientos de G. anatis, los que no son de campo, muestran menor resistencia a algunos antibióticos30.
Se puede concluir que los resultados de este trabajo muestran que la presencia de Gallibacterium anatis está frecuentemente asociada a problemas respiratorios y reproductivos en las poblaciones de gallinas de postura comercial en el sur de Sonora, afectando la producción. Los aislamientos de G. anatis mostraron una resistencia marcada a distintos antibióticos, probablemente por la medicación desmedida ante la presencia de microorganismos, lo que sugiere implementar un programa eficiente de control para esta bacteria.











 nueva página del texto (beta)
nueva página del texto (beta)