Introducción
México se encuentra entre los cinco primeros países en producción y exportación de miel a nivel mundial. La calidad de la miel se establece a través de sus diferentes propiedades como el sabor, color y aroma1,2, mismas que son producto de las especies vegetales de la zona y que le confiere su origen botánico. Actualmente existen algunos esfuerzos por comercializar la miel clasificada, siendo la Península de Yucatán la región que más ha avanzado en este rubro3.
La Península de Yucatán aporta el 30 % de la producción nacional de miel y aproximadamente el 90 % se destina al mercado internacional4,5. Estas mieles poseen características fisicoquímicas y sensoriales específicas debido a que provienen de una gran diversidad de especies nativas propias de la región. Hasta la fecha se han registrado aproximadamente 2,400 especies vegetales, de las cuales 600 son melíferas y 30 de ellas consideradas de gran importancia apícola6,7,8.
A pesar de la diversidad de especies melíferas existentes, cerca del 90 % de la producción anual de miel en la Península de Yucatán, proviene de dos principales flujos de néctar; floración de tajonal (Viguiera dentata) en los meses de diciembre a febrero y floración de t´sit´silche´ (Gymnopodium floribundum) durante marzo a mayo. Ambas especies florecen en el período de más baja precipitación pluvial. En el período de junio a noviembre (período de mayor precipitación pluvial) florecen una alta proporción de leguminosas y enredaderas; sin embargo, solamente se cosecha un 8 % del total anual9.
El método empleado más comúnmente, para distinguir las especies vegetales que dieron origen a una miel, es mediante el análisis palinológico, fundamentado en la morfología particular de cada polen presente en la miel10-14; sin embargo, existen en la actualidad diferentes metodologías que complementan el análisis melisopalinológico.
El contenido de polen en las mieles es muy bajo (niveles trazas) pero varía dependiendo de la riqueza floral de cada zona y del aporte de néctar y polen de cada especie vegetal, por lo cual, algunas especies se encuentran sub- o sobre-representadas en el perfil palinológico y se reconoce la necesidad de establecer otras metodologías menos dependientes de las trazas de polen en la miel, como la proteómica15, la espectroscopia óptica y sensores electrónicos16 o espectrometría de fluorescencia para estimar parámetros físico-químicos y contenido floral17, entre otros.
Una opción para identificar las especies vegetales presentes en la miel, son los métodos analíticos basados en el ADN; adecuados para la normalización como base para la armonización en la Unión Europea y otros países18. En este sentido, la definición del origen botánico de las mieles con fines de proporcionar un valor agregado al producto, establece la necesidad de contar con un sistema de detección de secuencias nucleotídicas homólogas, empleando oligonucleótidos genéricos derivados de genes metabólicos que, si bien son afines a todas las especies, también tienen patrones de amplificación muy particulares entre familias de plantas.
Este estudio se centró en el desarrollo y validación de sistemas de amplificación de oligonucleótidos genéricos por PCR cuantitativo (qPCR) empleando SYBR-Green y análisis de las curvas de disociación, para la caracterización de especies de plantas de importancia apícola en la península de Yucatán presentes en la miel a través del polen, como una herramienta para identificar el origen botánico de las mieles.
Material y métodos
Muestras de miel. Diez panales con al menos el 80 % de operculado se colectaron del apiario del Campo Experimental Mocochá, Yucatán, ubicado en km 1.5 carretera Mocochá-ex Hacienda Carolina, a 21º 07’ LN y 89º 26’ LO, a 10 msnm, durante los diferentes ciclos de floración que se presentan en la región. Los panales se llevaron al laboratorio para extraer individualmente la miel utilizando un extractor de acero inoxidable, el cual se lavó y secó con agua y alcohol al 70% para evitar contaminaciones entre muestras. Las muestras se filtraron y almacenaron a temperatura ambiente en frascos de polietileno tereftalato (PET) para su posterior envío al laboratorio de Cultivo de Tejidos Vegetales e Ingeniería Genética, en el Campo Experimental Bajío del INIFAP. Se obtuvieron cinco muestras de miel (M-1, M-3, M-4, M-7 y M-8) del ciclo apícola 2013 y cinco (M-11, M-12, M-13, M-14 y M-15) del ciclo 2014. Para tener de referencia el origen floral de las mieles, se realizó el análisis palinológico de cada muestra.
Análisis melisopalinológico. Las mieles se acetolizaron de acuerdo al método reportado por Louveaux19. Los gránulos de polen acetolizados se montaron en portaobjetos con gelatina glicerinada y se observaron sus características estructurales como forma, tamaño, aberturas y elementos estructurales utilizando un microscopio Motic BA 410 provisto de un analizador de imágenes. Los gránulos de polen se clasificaron e identificaron de acuerdo a las referencias publicadas por Palacios et al20.
Material Vegetal. Se recolectó en los alrededores del apiario del Campo Experimental Mocochá, hojas de cinco especies de importancia apícola; Viguiera dentata (tajonal) (florece diciembre a febrero), Gymonopodium floribundum (t´sit´silche´) (florece febrero a abril), Piscidia piscipula (ja´bin) (florece marzo a abril), Acacia angustissima (xáax x) (florece junio a agosto) y Mimosa bahamensis (sak káatsim) (florece julio a noviembre), para su análisis molecular y utilizarlo como referencia de cada especie.
Extracción de ADN. La extracción de ADN de plantas se realizó empleando el kit comercial DNeasy Plant Mini (Qiagen®), siguiendo la metodología descrita por el proveedor. La extracción de ADN de miel se realizó empleando 50 g de miel por cada muestra distribuyéndose en 4 tubos de 50 ml (NALGEN®), se agregaron aproximadamente 30 ml de agua doble destilada-estéril, y se incubó en baño María a 40 ºC durante 15 min. Posteriormente cada tubo se agitó por inversión y se centrifugó a 10,000 xg por 15 min, hasta obtener un sedimento (trazas de polen). A partir de este punto se siguió el protocolo de extracción de ADN de trazas de polen en miel publicado por la Oficina Federal de Protección al Consumidor y Seguridad Alimentaria de la Unión Europea (2011), con modificación en el laboratorio de Cultivo de Tejidos Vegetales e Ingeniería Genética del Campo Experimental Bajío del INIFAP. La concentración de ADN osciló entre 40 y 200 ng/L, dependiendo del contenido de polen por muestra de miel.
Iniciadores. Se utilizaron siete iniciadores de secuencias consenso publicadas previamente por Laube et al18 para establecer los patrones de amplificación de especies vegetales, y posteriormente identificar estos patrones de amplificación y sus curvas de disociación en las muestras de miel: 1) secuencia no codificante de DNA cloroplástico tRNA-Leu de cloroplasto trnL (UAA) intrón consenso de 19 especies incluyendo algas, briofitas, pteridofitas, gimnospermas y angiospermas18,21 (Plant 1), 2) secuencia anidada de Plant 1 (Plant nest)18, 3) Actina de canola y girasol (Act)18, 4) Profilina de girasol (Helli-all)18, 5) Lipasa de canola (Brass-lip)18, 6) Alcohol deshidrogenasa de sorgo, arroz y centeno (Adh1)18 y 7) 3-hidroxi metilglutaril CoA de algodón, chile, tomate, papa y tabaco (Hmg2)18.
Amplificación qPCR. La amplificación se realizó empleando el sistema SYBR Green a través de la siguiente mezcla de reacción: buffer Fast Master mix SYBR Green (Applied Biosystems No. Cat. 4385612) 1X, iniciadores derivados de los marcadores genéricos (150 nM) y ADN (100 ng). Las condiciones de amplificación requeridas para el equipo ABI PRISM 7000 (Applied Biosystems) fueron 1 ciclo a 95 °C por 20 seg (pre-desnaturalización), seguido de 40 ciclos a 95 °C durante 3 seg (desnaturalización) y 60 °C por 30 seg (alineación y extensión). Al finalizar la amplificación se realizó un análisis de las curvas de disociación (curva de temperatura de fusión (Tm)) para verificar la presencia de productos específicos e inespecíficos según la temperatura de fusión que posean22,23. Cada muestra de ADN de planta y de miel se amplificó por triplicado para establecer la consistencia del patrón de amplificación.
Como control interno para verificar concentraciones homogéneas se utilizó la sonda comercial TaqMan 18s (human18s rRNA No. Cat. 4319413E Applied Biosystems) utilizando la mezcla TaqMan Universal PCR Master Mix II with UNG (No. Cat. 4440038 Applied Biosystems). El control negativo consistió de los elementos de reacción sin ADN molde.
Resultados
Las curvas de amplificación del control interno (18S) validaron la calidad la concentración de ADN estándar para el desarrollo de las reacciones subsecuentes con los iniciadores de genes metabólicos genéricos. La concentración establecida fue de 50 ng/reacción, presentando amplificación entre los ciclos 17 y 20, teniendo menor diferencia de separación entre cada una de las réplicas, incrementando la confiabilidad de homogeneidad de una reacción a otra (Figura 1).
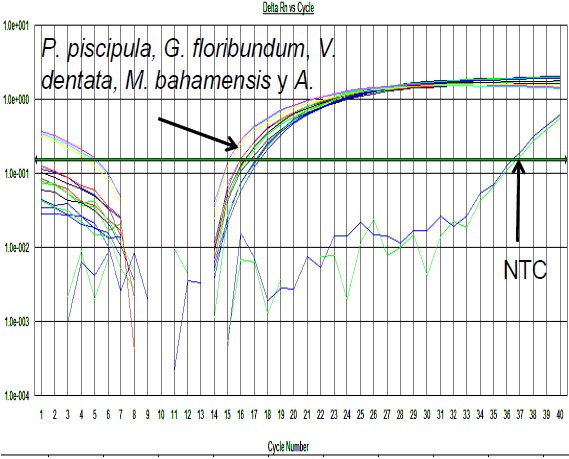
Figura 1 Curvas de amplificación qPCR del gen constitutivo 18S (control interno) de ADN de plantas (Piscidia piscipula, Gymnopodium floribundum, Viguiera dentata, Mimosa bahamensis y Acacia angustissima) (Software 7000 SDS v 1.2.3, Applied Biosystems)
Amplificación de genes genéricos en especies vegetales melíferas
El iniciador Plant 1 mostró evidencia de amplificación en todas las muestras de ADN de plantas, no así para el control negativo sin ADN molde (Figura 2A). La amplificación con los iniciadores Plant nest también mostró amplificación en todas las especies; sin embargo, se observó un diferencial en los ciclos de amplificación entre las diferentes especies, es decir, la secuencia de iniciadores de Plant nest demostró tener mayor similitud en las especies M. bahamensis, G. floribundum y A. angustissima que en P. piscipula y V. dentata (Figura 2B).
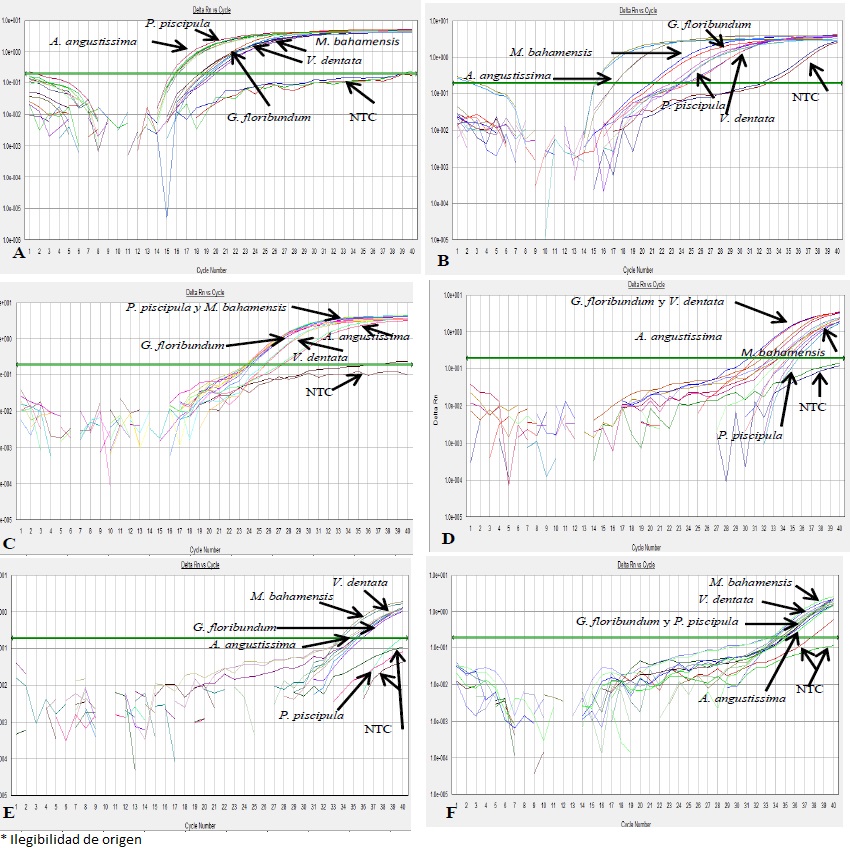
Figura 2 Curvas de amplificación qPCR de ADN de plantas (V. dentata, G. floribundum, P. piscipula, A. angustissima y M. bahamensis) con el sistema SYBR Green. Iniciadores A) Plant 1; B) Plant nest; C) Actina; D) Brass-lip; E) HMG; F) Helli-all. NTC control negativo sin ADN (Software 7000 SDS v 1.2.3, Applied Biosystems)
Los iniciadores del gen Actina, Bras-lip y Helli-all evidenciaron amplificación en todas las muestras de ADN de plantas en un rango de ciclos diferenciales (Figura 2C, 2D y 2F). Con los iniciadores Hmg2 se observó una amplificación para las muestras de plantas M. bahamensis, V. dentata, G. floribundum y A. angustissima, mientras que P. piscipula no mostró amplificación (Figura 2E). Finalmente, no se observó evidencia de amplificación con los iniciadores Adh1 de alcohol deshidrogenasa en ninguna de las especies de planta bajo estudio.
Análisis de curvas de disociación de los productos de amplificación en especies florales
En las curvas de disociación derivadas de la amplificación con iniciadores Plant 1 se observó un producto específico para cada una de las especies, respecto a la derivada de fluorescencia (el pico máximo de la curva correspondiente a la temperatura de fusión (Tm) del producto) con una variación en las áreas bajo la curva asociado al número de copias del gen en cada una de las especies (Figura 3A). Al igual que Plant 1, cuando se empleó el iniciador Plant nest, la temperatura y el valor de la fluorescencia variaron entre las especies; observándose un producto específico en las curvas de disociación para las cinco especies de plantas (Figura 3C). De esto se confirma que Plant nest reduce el espectro de similitud entre las especies, haciendo más precisa la detección. Sin embargo, es de notar que aun cuando hay amplificación en todas las especies, el control negativo (sin ADN molde) también presentó amplificación, lo que establece un fenómeno de amplificación inespecífica derivado posiblemente de formación de dímeros entre los iniciadores eliminando la posibilidad de emplear al iniciador Plant nest.

Figura 3 Comparación del patrón de curvas de disociación producto de la amplificación con iniciadores de genes genéricos en plantas A: Cloroplasto trnL (UAA) intrón (Plant 1); C: Cloroplasto trnL (UAA) intrón (Plant nest); E: Actina (Act); G: Lipasa (Bras lip); I: 3-hidroxi metilglutaril CoA (Hmg2); K: Profilina (Helli all) y mieles B: Cloroplasto trnL (UAA) intrón (Plant 1); D: Cloroplasto trnL (UAA) intrón (Plant nest); F: Actina (Act); H: Lipasa (Bras lip); J: 3-hidroxi metilglutaril CoA (Hmg2); L: Profilina (Helli all) (Software 7000 SDS v 1.2.3, Applied Biosystems)
Los resultados de la amplificación empleando los iniciadores Act del gen actina, muestran que las curvas de disociación muestran un solo producto en todas las especies con variantes en la temperatura y fluorescencia, eliminando así las posibilidades de amplificación cruzada (Figura 3E).
En la curvas de disociación para el iniciador Brass-lip, las especies que obtuvieron un producto específico fueron M. bahamensis y A. angustissima (Figura 3G). Las muestras G. floribundum, V. dentata y P. piscipula presentaron dos o más productos inespecíficos, esto derivado de la unión no específica del iniciador a las secuencias en los genomas de estas especies de plantas.
La curva de disociación obtenida en la amplificación con los iniciadores Hmg2 se observó un producto específico para A. angustissima y G. floribundum (Figura 3I). Se observó que con este iniciador los valores de fluorescencia son comparativamente más bajos que con los otros genes, haciendo que el área bajo la curva sea menor, esto puede ser debido a que el número de copias del gen Hmg2 es considerablemente menor en las especies de plantas estudiadas aun cuando hay especificidad en sus secuencias.
Finalmente, en las curvas de disociación obtenidas al amplificar con los iniciadores Helli-all, se observó un producto específico para la especie G. floribundum y V. dentata (Figura 3K). Las muestras de M. bahamensis, P. piscipula y A. angustissima presentaron un perfil de desnaturalización diferente (valores de temperatura de fusión distintas) mostrando productos de amplificación inespecíficos.
Análisis de curvas de disociación de los productos de amplificación en ADN de miel
Una vez realizado el análisis de las curvas de disociación de las especies melíferas, se analizaron y compararon las curvas de disociación derivadas de los productos de amplificación de ADN de muestras de miel (datos no mostrados).
Nuevamente y al igual que ocurrió en la detección en las especies de planta, no se observó evidencia de amplificación con los iniciadores Adh1 en ninguna muestra de miel. Una vez más se confirmó que las especies de gramíneas (pastos y cereales), no son parte de la composición taxonómica de las muestras de miel bajo estudio. Con el iniciador Plant 1 del gen cloroplasto trnL (UAA) intrón, se observó amplificación para todas las muestras de ADN de mieles, sin embargo, no se obtuvo un único producto para ninguna muestra de miel de acuerdo al análisis de las curvas de disociación (Figura 3B).
El iniciador Plant nest del gen de cloroplasto trnL (UAA) intrón, también mostró amplificación para todas las mieles, asimismo, este iniciador conservó el patrón de curvas de disociación de la amplificación de ADN de todas las plantas coincidiendo con las mieles; sin embargo, el control negativo (sin ADN molde) también mostró amplificación, evidenciando así la formación de dímeros entre iniciadores y eliminando la posibilidad de hacer una detección confiable con estos iniciadores (Figura 3D).
Cuando se emplearon los iniciadores Act del gen actina se observó amplificación para todas las muestras de miel; sin embargo, en el análisis individual de las curvas de disociación, la muestra de miel M-1 mostró un producto específico a 76.9 ºC con una fluorescencia 0.11, con la probable presencia de la especie V. dentata, mientras que la muestra M-3 obtuvo un producto específico a 73.3 ºC con una fluorescencia de 0.13 con la oportunidad de presencia de M. bahamensis (Figura 3E). Esto se confirmó con el análisis palinológico, el cual indicó la presencia de V. dentata y M. bahamensis en las muestras M-1 y M-3, respectivamente.
El iniciador Brass lip que amplifica el gen lipasa mostró amplificación para las mieles M-3, M-4, M-8 y M-11; sin embargo, en el análisis de las curvas de disociación las muestras M-3 y M-8 fueron las muestras que presentaron un solo producto específico a 70.2 ºC y con una fluorescencia de 0.5. Esto indicó la presencia de M. bahamensis coincidiendo en el análisis palinológico encontrando un 45.3 % de gránulos de dicha especie en la muestra (Figura 3H). Es de hacer notar que la curva de disociación que acota la amplificación de ADN de A. angustissima no fue obtenida para ninguna de las mieles, no obstante que mediante el análisis palinológico, esta especie sí se encontró en M-3 (22.2 %) y M-4 (58 %).
Cuando se empleó el iniciador Hmg2 del gen 3-Hidroximetilglutaril CoA se observó amplificación en todas las muestras de miel excepto en M1 y M8. La curva de disociación indica que las muestras M-3 y M-4 presentaron un producto específico similar en características a la curva de disociación (temperatura/ fluorescencia) de la amplificación de ADN de G. floribundum, sugiriendo que estas dos muestras de miel contienen la especie; coincidiendo con la fecha de colecta de la miel, la floración de esta especie y el análisis palinológico (Figura 3J). Es de resaltar que con el iniciador Hmg2, las muestras de miel M-14 y M-15 muestran presencia de un producto específico a una temperatura mayor y a una fluorescencia también superior a la obtenida en las muestras M-3 y M-4; las características de estas curvas de disociación no tienen precedente en la caracterización de ninguna de las especies melíferas analizadas con este iniciador en el presente trabajo,
La amplificación con los iniciadores Helli-all que codifican al gen profilina se obtuvo para las muestras de miel M-3, M-4, M-7, M-11, M-12, M-13, M-15. La curva de disociación mostró presencia de un producto específico para la miel M-3 similar al patrón obtenido de la curva de la especie G. floribundum (Figura 3L). Este resultado coincide y ratifica al obtenido con el iniciador Hmg2 donde se sugiere la presencia de esta misma especie (Figuras 3J). Por su parte la muestra de miel M-13 mostró una curva de disociación semejante al patrón de la curva de disociación de la especie V. dentata (Figura 3L).
Discusión
La identificación del origen floral de las mieles producidas en México por medio de la utilización de iniciadores provenientes de secuencias consenso de genes metabólicos genéricos y el análisis con curvas de disociación establecen una nueva posibilidad de apoyo a la melisopalinología en una primera etapa de detección cualitativa.
El fluoróforo SYBR® Green suele adherirse a cualquier producto amplificado sea éste el producto blanco o no-blanco, y todas estas señales se suman produciendo una sola curva de amplificación, por lo que el patrón de amplificación no puede ser empleado directamente para determinar especificidad. Para establecer esta especificidad es conveniente realizar un análisis curvas de disociación. En una curva de disociación la fluorescencia decrece de forma gradual a medida que aumenta la temperatura23. Mediante el análisis de las curvas de la temperatura de disociación (curvas de fusión) es posible identificar productos específicos, cuya forma se relaciona con el contenido de GC (guanina y citosina), tamaño de los amplicones y la secuencia de los mismos22.
De los siete pares de iniciadores provenientes de secuencias de genes genéricos, fue interesante observar los diferentes patrones de amplificación aun cuando estas secuencias son altamente conservadas en múltiples organismos de familias y reinos divergentes. En un primer análisis se establece que el gen Cloroplasto trnL (UAA) intrón, que conformó la base para el diseño de los iniciadores Plant 1 y Plant nest, amplifican en las muestras de ADN de las cinco especies de plantas bajo estudio; sin embargo, este resultado no concuerda cuando las muestras de miel son sometidas a escrutinio de sus curvas de disociación, al mostrar inespecificidad de la secuencia blanco con las contenidas en las muestras de ADN extraídas de miel. Una posible explicación es que el polen al ser una célula germinal masculina tiene muy bajas probabilidades de contener plastidios (células de herencia materna) limitando así la identificación de secuencias cloroplásticas en ADN proveniente de las muestras de miel24. Lo anterior contrasta con lo publicado por Laube et al18, quienes afirman que esta secuencia multicopia en cloroplasto fue adecuada para la detección de bajos niveles de ADN en plantas, siendo aplicable para la detección de ADN de plantas en la miel.
Durante el análisis con el iniciador Act del gen actina, se encontró la presencia de V. dentata en la miel M-1 y M. bahamensis en la miel M-3. La especie V. dentata se produce entre el mes de diciembre y enero coincidiendo con la fecha de colecta (24/01/2013) de la muestra M-1 y con el análisis palinológico, mediante el cual se encontró un porcentaje del 7.5 % de la especie. El porcentaje de polen de esta especie encontrado en la miel se considera como “polen minoritario importante” de acuerdo a la Comisión Internacional Botánica Apícola19; sin embargo, el hecho que el iniciador Act del gen actina lo haya detectado es muy positivo, ya que podría identificarse dicha especie en muy bajos porcentajes difíciles de detectar por el método tradicional. La miel M-3 de la cual se detectaron dos especies M. bahamensis (Act y Brass-lip) y G. floribundum (Hmg2 y Helli-all) se clasificó como monofloral de la especie M. bahamensis, ya que los gránulos se encontraron en un porcentaje del 65 % considerado como “polen predominante” por el análisis melisopalinológico, coincidiendo con la fecha de floración de la especie y la de colecta de la miel. De manera similar, Brass lip confirmó la presencia de M. bahamensis en la muestra M-8 con temperaturas de fusión similares a las obtenidas para la especie; pero con un área bajo la curva inferior a M-3 indicando contenidos bajos de esta especie.
Los iniciadores Hmg2 y Helli-all confirmaron la presencia de G. floribundum en la muestra M-3, corroborando no solo la presencia de la especie en la muestra de miel, sino que además se reportan dos genes metabólicos genéricos que son consistentes en los resultados, proporcionando con esto iniciadores adicionales de verificación. Es interesante notar que si bien estos dos genes provienen de familias distantes (Asteraceae en el caso del iniciador Helli-all y Solanaceae, en el caso de Hmg2), es muy posible que haya una alta similaridad de secuencias entre los genes profilina (Helli-all) y 3-hidroxi metilglutaril CoA (Hmg2) con sus contrapartes homólogas en G. floribundum. En este sentido de acuerdo con Huang et al25) una de las profilinas que conforman parte de la familia de profilinas de Arabidopsis (PRF4) presentó mayor similitud con otras especies que con las otras tres profilinas de Arabidopsis (PRF1, PRF2, PRF3). A nivel aminoácido PRF4 fue 78.6 a 82.4 % idéntica a profilinas de polen-específico de monocotiledóneas como Zea mays (maíz), Phleum pratense (hierba timotea), Betula verrucosa (abedul blanco), Nicotiana tabacum (tabaco) y Triticum aestivum (trigo).
La muestras de miel M-14 y M-15 amplificaron el gen 3-hidroxi metil glutaril CoA (Hmg2); sin embargo, se trata de un producto específico que no se observó en la amplificación de ninguna de las especies estudiadas por tener temperatura muy superior a la obtenida para las otras especies en el análisis de las curvas de disociación; además es importante resaltar que estas muestras fueron colectadas al final del periodo de producción de las principales especies florales de importancia apícola, por lo que se considera que la similitud de estos iniciadores está asociada a otras especies secundarias encontradas en el mismo ciclo, como Mimosa pudica, Acacia gaumeri, Bursera simaruba, Pithecellobium albicans, Senna pallida, Guazuma ulmifolia, Lysiloma latisiliquum y Leucaena leucocephala, entre otras.
En general, las especies que mostraron mayor frecuencia en las muestras de miel fueron V. dentata (tajonal), G. floribundum (t´sit´silche´) y M. bahamensis (sak káatzim), estas tres especies tienen presencia floral por más tiempo a lo largo del año; mientras que A. angustissima (xáax x) y P. piscipula (ja´bin) no presentaron evidencia de presencia en ninguna muestra de miel, debido probablemente a que estas especies florecen por periodos muy cortos en la temporada de floración.
De acuerdo con nuestros resultados fue posible establecer el patrón de amplificación de las cinco especies bajo estudio de acuerdo a la derivada de la fluorescencia en cada producto de amplificación, y con ello comparar el patrón obtenido para cada una de las mieles estudiadas.
En este sentido se obtuvo evidencia de la presencia de tres de las especies de mayor importancia en el ciclo apícola. De esto observamos que los genes profilina, lipasa, 3-hidroxi metilglutaril CoA y en mayor medida actina, están conformados de secuencias útiles para identificar patrones de amplificación en cada especie y compararlas con las obtenidas en mieles para una aproximación de la posible presencia de una o más especies.
Se encontró que, aun cuando las muestras de miel se cosecharon dentro de los periodos de floración de las cinco especies estudiadas, no se detectó la presencia de dichas especies vegetales en las muestras de miel M-7, M-11 y M-12, confirmando dichos resultados con los análisis palinológicos, donde se observaron diferentes especies de Asteraceae, Malvaceae y Fabaceae principalmente. Por otra parte, aún cuando las muestras M-14 y M-15 no indican la presencia de alguna de las cinco especies bajo estudio, demostraron tener un patrón de amplificación diferente y posiblemente relacionado con especies no incluidas en nuestro análisis, concordando con los análisis palinológicos, ya que en dichas muestras de miel se detectaron especies como Lysiloma bahamensis (tzalam), Mimosa pudica (xmuuts´) y Acacia gaumeri (box káatsim), entre las mas importantes.
La especie V. dentata fue identificada por los genes actina y proflina, M. bahamensis por los genes actina y lipasa; y G. floribundum por los genes Hmg2 y profilina. Es importante mencionar que la especie A. angustissima se detectó por palinología en las mieles M-4, M-8, y M-12 en muy bajos porcentajes (6.2, 2.0 y 0.5 % respectivamente) lo cual probablemente estuvo debajo del límite de detección de los marcadores utilizados.
Finalmente, para la caracterización de las especies endémicas que no fueron detectadas por los iniciadores diseñados de secuencias consenso de genes metabólicos generales, se emplearán otros genes con características de amplia similitud en función metabólica a través de especies, que pudieran establecer el patrón de amplificación e identificación de Piscidia piscipula (ja´bin) y Acacia angustissima (xáax x). Asimismo, el diseño de nuevos iniciadores para la amplificación de genes que no amplificaron para ninguna especie o muestra de miel como el caso de Adh1, podrá establecer una estrategia para la identificación de especies importantes de la Península de Yucatán.
Conclusiones e implicaciones
Los resultados de amplificación y posterior comparación de curvas de disociación de plantas y muestras de miel, establecen que un solo gen puede detectar más de una especie (actina y profilina), asimismo, dos genes diferentes en función pueden detectar la misma especie en una misma muestra (actina y lipasa para M. bahamensis y Hmg y profilina para G. floribundum), confirmando y reforzando con ello los resultados de detección. Por otro lado, secuencias de genes ligados entre si, por su actividad metabólica en pared celular (actina y profilina), identificaron independientemente la misma especie (V. dentata) en muestras de miel de cosechas diferentes. De esta manera se puede establecer que este sistema basado en detección por amplificación de ADN empleando secuencias consenso de genes genéricos, constituye un nuevo protocolo de apoyo a la melisopalinología para identificar contenido de especies.











 texto en
texto en 


