El agente causal de la tuberculosis está conformado por el complejo M. tuberculosis, capaz de sobrevivir y crecer dentro de los macrófagos, un ambiente hostil por la limitación de nutrientes y acidificación1-3. Su adaptación se efectúa a través de mecanismos que le permiten detectar su entorno, regulando los genes que se expresan o reprimen durante su crecimiento y reproducción4, que favorecen su persistencia en el hospedero5.
Las células pueden responder a estímulos externos al iniciar un proceso que se da a partir de la unión de un efector a un receptor unido a la membrana. Este es un mecanismo de transducción, en el que los factores estresantes desencadenan una respuesta a través de la expresión de los genes, que preparan a las bacterias durante el agotamiento de los recursos6,7. Hecho que se puede observar in vitro en la fase exponencial y son decisivos para la adaptación a las condiciones que se presenten en la fase estacionaria y sobrevivir8. Lo anterior, implica que la célula acumule protones del medio para mantener la homeostasis del pH en el citoplasma9, polifosfatos para la conversión en fuentes de energía10. En M. tuberculosis y M. smegmatis, además se acumula el glutamato11 y la asparagina12 que se relaciona con la inducción del aletargamiento de M. tuberculosis13.
Los bacilos tuberculosos responden a situaciones de estrés por agotamiento de nutrientes, pH ácido, especies reactivas de oxígeno y nitrógeno causado por su crecimiento en los macrófagos7,8,14. Para responder a estos estímulos, las micobacterias secretan enzimas como ESAT-6 y CFP-102,15, que en las células del hospedero, actúan en los procesos de necrosis, tales como la lisis del epitelio alveolar y membranas de los macrófagos, induce la fragmentación del ADN y la permeabilidad de la membrana de las mitocondrias15. Entre las funciones metabólicas de las micobacterias, se propone que regulan la transferencia de ADN16.
M. smegmatis mc2155, mutante de la cepa silvestre M. smegmatis, es 10 a 100 mil veces más eficiente transformada con plásmidos, utilizada en el análisis de la función de las micobacterias17. Por su rápido crecimiento se ha propuesto como modelo para entender los cambios que promueven la adaptación18 y la persistencia de las micobacterias patógenas19. Comparte 12 de 19 genes de virulencia con M. tuberculosis20, entre los que se encuentran los genes esat-6 y cfp-10, que codifican para las proteínas correspondientes secretadas por el sistema ESX-1 (por sus siglas en inglés: Secretion Ejectosoma BOX)21. Converse y Cox22 observaron el efecto de los medios de cultivo en el sistema de secreción ESX-1, por lo que proponen que este sistema puede ser sensible a uno o más nutrientes del medio de cultivo.
Durante el proceso de adaptación, los genes relacionados con redes metabólicas específicas se expresan como una respuesta al estrés ocasionado por la privación de nutrientes23. La adaptación de las micobacterias cultivada se puede observar después de la fase estacionaria, al cambiarse a un medio de cultivo fresco reinician su actividad. Por lo que en el presente estudio se evalúan las condiciones de cultivo limitantes de nutrientes para el crecimiento de M. smegmatis y su relación con la expresión del gen esat-6.
M. smegmatis mc2155, se creció rutinariamente a 37 ºC en Middlebrook 7H11. Las alícuotas iniciales de los medios de cultivo testigos y probados, se obtuvieron por inóculo de una colonia de M. smegmatis en 10 ml de medio líquido Middlebrook 7H9 y se creció por 24 h hasta media fase logarítmica (0.8-1.0 DO a 600 nm).
Los medios de cultivo utilizados como control para comparar el comportamiento metabólico de los bacilos y expresión del gen esat-6 fueron 7H9 y Sauton sin zinc, suplementados ambos con 0.5 y 6 % de glicerol respectivamente y 0.05 % de Tween 80, por ser medios en los que expresa el gen esat-622. La inoculación inicial se efectuó con 100 μl del cultivo inicial en frascos de 225 ml con 150 ml de medio líquido, se incubaron a 37 ºC, en agitación a 250 rpm.
Todos los medios de cultivo probados para la limitación de nutrientes se derivan del medio mínimo HdB, se ajustó el pH 7, se adicionó, 0.2 % de glicerol, 0.05 % de Tween 8024, excepto para provocar el agotamiento de C (HdB<C) al que se limitó el glicerol a 0.08 % (v/v). Para los experimentos en los que se requirió el agotamiento de nitrógeno (HdB<N), se usó (NH4)2SO4 a una concentración 100 veces menor (0.15 mM) que el usado para el medio mínimo HdB. Para los experimentos limitados 100 veces menos en Pi (HdB<Pi), se adicionó K2HPO4 a una concentración final de 0.089 mM (0.0155 g L-1) y NaH2PO4 a una concentración de 0.0708 mM (0.085 g L-1), para reemplazar la pérdida de capacidad amortiguadora se agregó ácido propanosulfónico 3-(Nmorfolino) (MOPS) 50 mM24. La fase de crecimiento y estacionaria de todos los cultivos se monitoreó a 600 nm DO, hasta las 144 h y se registró el pH final.
El fragmento predicho del gen esat-6 se obtuvo por amplificación del ADN de M. smegmatis a partir de los iniciadores: F-5’ACAGGTATGGAATTTCGCCG-3’, R-5’-CAGGCAAA CATTCCCGTGA-3’, los cuales se diseñaron con el programa Oligo TM Software DNA Star Program. Para llevar a cabo la identificación mediante secuenciación enzimática, el producto amplificado se purificó mediante un protocolo a base de columnas de silica (QIAquick Gel Extraction Kit, QIAGEN®), para ello, el fragmento amplificado se visualizó en el gel de agarosa al 1.2 % a través de un transiluminador de luz UV y se cortó mediante un bisturí estéril, llevando a cabo el protocolo mencionado. La calidad y la cantidad de ADN purificado se observó mediante un gel de agarosa y se procedió a enviar esta muestra amplificada para su secuenciación. El proceso de secuenciación se realizó en el Laboratorio Nacional de Genómica para la Biodiversidad (LANGEBIO) del Centro de Investigación y Estudios Avanzados (CINVESTAV), Unidad Irapuato, Guanajuato, México, del Instituto Politécnico Nacional (IPN), basado por el método ddNTPs25, utilizando un secuenciador 3730 XL DNA, (Applied Biosystems, Foster City, CA) y el kit Big Dye Terminator 3.1 (Applied Biosystems, Foster City, CA).
Para la extracción de ARN el cultivo de M. smegmatis se adaptó en los medios de cultivo correspondientes hasta el inicio de la fase estacionaria (96 h). En esta fase se cosechó el 30 % de las células, se lavaron con PBS y se colocaron en 150 ml de medio de cultivo fresco con las mismas condiciones nutritivas, se incubaron por 14 h. El ARN se extrajo mediante el uso de TRIzol (Invitrogen®). Se observó por electroforesis en geles de agarosa (1.2 %) con MOPS X (200 mM de MOPS, 50 mM de acetato de sodio, y 10 mM de EDTA, pH 7) y 3.15 % de formaldehído. La electroforesis se corrió en una solución amortiguadora TAE (40/1 mM pH 8.0).
La hibridación Northern se efectuó a partir de 8 a 12 ng del ARN extraído, transferido por capilaridad descendente en membranas de nailon, en citrato de sodio y cloruro de sodio 20X. El ARN se fijó con luz ultravioleta durante 5 min. La sonda se elaboró a partir del fragmento del gen esat-6 amplificado por PCR y marcada con biotina acoplada a fosfatasa alcalina (Gene Images Alkphos Direct Labelling and Detection System-GE Health Care®). La hibridación se efectuó durante toda la noche y se reveló por autoradiografía (Hyperfilm-MP autoradiography, Amersham Pharmacia Bioscience®)26.
Los datos obtenidos del crecimiento y pH, se obtuvieron de seis repeticiones y se aplicó la prueba t de student, para expresar los resultados de la media, el error estándar de la media y las correspondientes comparaciones entre ellas de todos los cultivos de M. smegmatis27.
En los medios de cultivo probados, se observó a M. smegmatis con crecimiento más bajo en los medios HdB<N, HdB<C (0.659 y 1.697) respecto al medio de cultivo HdB mínimo y HdB<Pi (2.170 y 1.982) (P<0.05). Además, M. smegmatis en HdB<Pi tiene un crecimiento similar al cultivo en HdB mínimo (P>0.05) (Figura 1). Lo que es indicativo de un crecimiento continuo a pesar de las condiciones de estrés por la disminución de 100 veces Pi. Con excepción del cultivo en HdB<Pi, en los demás medios de cultivo probados y los usados como controles, M. smegmatis reanuda su crecimiento al cambiarlo al medio de cultivo fresco correspondiente, después de 144 h de cultivo.
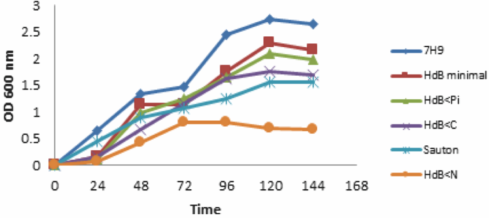
Figura 1 M. smegmatis crecido a 144 h en los medios de cultivo usados para el experimento HdB<N (0.659±0.12), Sauton (1.559±0.03), HdB<C (1.697±0.07), HdB<Pi (1.982±0.11) HdB mínimo (2.170±0.12) y 7H9 (2.638±0.12).
En el medio de cultivo HdB<Pi se observó una relación entre el crecimiento de M. smegmatis, el pH y la pérdida de resistencia alcohol-ácido. Es decir, a mayor número de horas de cultivo (144 h), se disminuyó el pH (5.4 ± 0.16), aunado a la incapacidad de recuperar su actividad metabólica cuando se cambia el cultivo a medio fresco. Esto se observó por los cúmulos de células teñidas con el color característico de la técnica Ziehl-Neelsen desaparecen partir de las 100 h hasta teñirse todas las células de color azul (Figuras 2a y (2b).
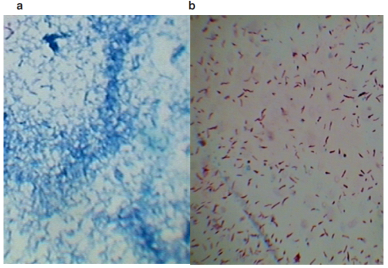
Figura 2 M. smegmatis a) Pérdida de resistencia alcohol-ácido en cultivo HdB<Pi a 96 horas. b) mismo cultivo cambiado a medio fresco 1 hora después, en el que se observa la recuperación de la capacidad de respuesta al alcohol-ácido.
Con excepción del medio HdB<Pi, las micobacterias en los medios de cultivo restringidos en nutrientes tienen un menor crecimiento hasta la fase estacionaria. Además, las bacterias crecidas en HdB<Pi pierden paulatinamente su capacidad de resistencia al alcohol-ácido a partir de las 96 h, para no volver a recuperarla después de 144 h de cultivo, cuando las bacterias se cambiaron a medio fresco. Por lo que las 96 h se tomaron en cuenta para hacer los cambios de M. smegmatis a medios de cultivo fresco de todas las pruebas, extraer el ARN total 12 ± 1 h después (Figura 3A) y llevar a cabo la hibridación Northern. Esta prueba revela que M. smegmatis cultivado en HdB<C, HdB<N, y HdB medio mínimo, no expresa el gen esat-6. Sin embargo, el cultivo limitado 100 veces en Pi (Figura 3B carril 7) si se observó la expresión, de manera similar a los medios de cultivo usados como control (7H9 y Sauton carriles 1 y 3 de la Figura 3B).
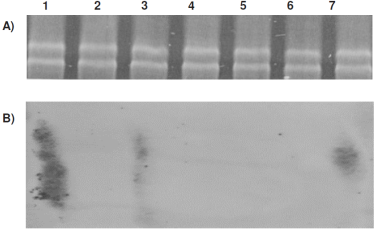
Figura 3 M. smegmatis A) ARN total de la bacteria en los medios de cultivo: 1) 7H9, 2) HdB medio mínimo, 3) Sauton sin Zinc, 4) variable no controlada 5) HdB<N, 6) HdB<C, 7) HdB<Pi. B) Mismos carriles con los resultados de la hibridación Northern para el gen esat-6.
La sonda elaborada a partir del fragmento de esat-6, se secuenció y se remitió al GeneBank con el número SeqID KR363260. La comparación homológica consultada en la base de datos corresponde al gen esat-6, y se localiza en el sitio del 87,387 a 87,604 pb de la secuencia del genoma completo de M. smegmatis cepa MC2155 (ID: gb|CP000480.1).
La capacidad que poseen las micobacterias para acumular las fuentes de energía, les permiten persistir in vivo durante la fase de infección28. En este mismo sentido, las micobacterias in vitro logran adaptarse al agotamiento de los nutrientes y sobrevivir por más de 650 días24, debido a la acumulación de energía de los compuestos almacenados y de la degradación de proteínas innecesarias y ARNm bacteriano6. Esto puede provocar una situación emergente, en la cual las micobacterias responden al ambiente a través de los genes que regulan los cambios adecuados a las condiciones impuestas. De esta manera, el modelo del presente estudio permite observar la expresión del gen esat-6 de M. smegmatis en HdB<Pi.
La adaptación de M. smegmatis hasta por 144 h es evidente en todos los medios de cultivo, excepto para las micobacterias crecidas por en HdB<Pi (Figura 1). Por lo cual se plantea la inestabilidad de la bacteria al cultivo, por su crecimiento continuo con respecto al cultivo en HdB mínimo (P>0.05) y su metabolismo inactivo después de 144 h al cambiar las micobacterias a un medio de cultivo fresco, en donde se observa la pérdida de la capacidad alcohol-ácido (Figura 2: a y b), resultados similares a los de Rifat et al29 al restringir Pi en medio 7H9 en el crecimiento de M. tuberculosis.
Esto hace suponer bajo las condiciones propuestas, la existencia del estado de emergencia, en el cual M. smegmatis puede tener diferentes formas de adaptación, donde se comprometen diversas rutas de transducción mediante los cuales las micobacterias detectan y dan una respuesta al medio. Estas rutas, pueden estar relacionados con la alarmona ppGpp que desencadena la respuesta estricta. Un mecanismo indispensable para la adaptación de las bacterias durante la transición de la fase exponencial a la fase estacionaria30-32. Al respecto, en E. coli, la carencia de aminoácidos, carbono, o Pi, se acompaña por el incremento de los niveles de ppGpp y se observan acumulaciones de polifosfatos33,34.
Bajo las condiciones limitantes de Pi y un crecimiento sostenido muy cercano al HdB medio mínimo (Figura 1), M. smegmatis aparentemente termina los recursos externos y las reservas de energía interna. Para evitar este desgaste de las reservas de energía se ha propuesto la respuesta estricta, que se caracteriza por la regulación de ppGpp, que incrementa la ciclopropanación35, la inhibición y síntesis de los ácidos grasos y los fosfolípidos36. Aunque la propuesta de la respuesta estricta está relacionada con la posibilidad de la estabilización del metabolismo para la adaptación de la micobacteria a las condiciones de estrés, es decir, bajo condiciones de abundancia, los polifosfatos pueden servir como donadores de ADP, GDP, y otras reacciones en las que se involucra la enzima Rel que actúa en la síntesis e hidrólisis de ppGppp5,37. Pero cuando no hay suficientes reservas de Pi y las reservas de energía son mínimas, es probable que la respuesta estricta falle para que M. smegmatis se adapte, y su estado metabólico no le permita responder de inmediato a los cambios favorables para reanudar su crecimiento, como lo sucedido en el presente estudio.
Un incremento en los niveles de ppGpp y polifosfatos induce la transcripción de 150 genes involucrados en la disminución del crecimiento y el metabolismo38. Por el contrario, lo encontrado en la presente investigación respecto a la limitación de Pi en HdB, es que se mantiene el crecimiento, similar al del medio HdB mínimo en el que no se restringe el Pi (P<0.05). Por otra parte, la expresión de gen esat-6 en HdB<Pi que se dio a nivel de los ARNm (Figura 3B), que es comparable a 7H9 y Sauton, aunque la proteína codificada por este gen, se secreta sólo en el medio Sauton22, lo que falta por determinar es si bajo las condiciones de cultivo HdB<Pi se secreta la proteína ESAT-6. Esta proteína, también se ha propuesto como una ATPasa39, que modifica las propiedades del compartimiento del fagosoma donde reside M. tuberculosis, como lo evidenciado por la retención aberrante o adquisición defectuosa de un rango de proteínas del hospedero, entre las que se incluyen las Rab GTPasas40,41 y la protón-ATPasa de la vacuola42.
En M. smegmatis y M. tuberculosis, los polifosfatos regulan la síntesis de ppGpp a través del control de la transcripción de relA vía la ruta mprA-sigE-relA37,43. Existen otros sistemas de los dos componentes que pueden contribuir a la regulación de relA. Por lo que se plantea como perspectivas, estudiar las relaciones entre los reguladores de la expresión de los genes que dependen de Pi. Entre ellos, está SenX3-RegX3 y su relación con los genes que regulan el sistema ESX-1 y las proteínas de secreción en M. smegmatis44. Pang et al45, confirman que en M. tuberculosis, esat-6 no es regulado por MprAB bajo crecimiento en 7H9 normal. Por lo que se debe entender si esat-6 de M. smegmatis es regulado por este sistema en condiciones limitantes de Pi. Al respecto, los estudios de Rifat et al29, enfocados a la restricción de Pi, demuestran que no se expresa esat-6 en M. tuberculosis, cuando restringieron el Pi en medio 7H9 con L-glutamato como fuente de nitrógeno orgánico; sin embargo, en este modelo se limita el Pi y la fuente de nitrógeno es inorgánico. Pang et al45 también indican que al menos indirectamente hay una regulación del ESX-1 por PhoPR. Por lo que se propone investigar las relaciones entre la limitación de Pi y PhoPR, bajo el modelo propuesto en esta investigación.
En conclusión, M. smegmatis se adapta a HdB medio mínimo, HdB<C y HdB<N y reanuda su actividad metabólica en medio fresco, pero no expresa el gen esat-6. En cambio el medio de cultivo HdB<Pi, M. smegmatis presenta una fase de crecimiento exponencial continuo, estadísticamente similar al de HdB medio mínimo (P<0.05), manifiesta un estado metabólico en el cual pierde su capacidad ácido-resistente en la fase estacionaria y expresa el gen esat-6 después del cambio del medio de cultivo a 96 h al medio de cultivo fresco correspondiente. Por lo anterior se propone al modelo de investigación para la expresión génica en condiciones de estrés por nutrientes.











 texto en
texto en 


