Introducción
Actualmente, las pruebas de ADN son herramientas disponibles para los criadores de ganado y todos aquellos interesados en llevar a cabo el monitoreo de la calidad y salud genética de los animales del hato1,2. En bovinos, se han descrito pruebas de ADN para identificar portadores de al menos 40 desórdenes genéticos1,3.
En el caso de la raza Charolais, diferentes Asociaciones en el mundo reportan oficialmente el fenotipo doble músculo (DM). Esta es una condición genética que es producto de mutaciones disruptivas en el gen de la miostatina (MSTN)4. Además del fenotipo DM, otros desórdenes genéticos reportados para la raza Charolais son la Citrulinemia bovina (ASS), la Enfermedad de Almacenamiento del Glucógeno tipo V (EAG-V), y la Deficiencia de la Uridina 5 Monofosfato Sintasa (DUMPS); todos estos desórdenes son recesivos y son causados por mutaciones en genes específicos5.
En el caso de las pruebas de ADN para predecir la calidad de la carne y la productividad, las que a la fecha han alcanzado mayor difusión son las de rasgos sensoriales de la carne6, específicamente suavidad y contenido de grasa intramuscular (marmoleo). El marcador TG5 del gen tiroglobulina, es una transición C/T localizada en el sitio consenso de unión de la RNA polimerasa III, 537 pb río arriba del inicio del primer exón del gen de tiroglobulina (NW_001493192.1.g.290170C>T). El polimorfismo de este marcador está definido por los alelos 2 y 3, mientras que el alelo 2 muestra la secuencia GATC, y el alelo 3 tiene una secuencia GATT, el cual se ha asociado a mayor marmoleo. Aunque ya hay estudios en los que se ha validado su asociación a marmoleo6, hay reportes en los que no se ha encontrado efecto significativo sobre la característica7. Para suavidad o terneza, dos marcadores localizados en el gen de la micro-calpaína 1 (CAPN1-316 y CAPN1-4751) y dos ubicados en el gen de la calpastatina (CAST), son a la fecha los más estudiados y su efecto sobre la suavidad ha sido validado6. El marcador CAPN1-316, es una transversión G/C en la posición 5709 del exón 9 del gen CAPN1, mientras que el marcador CAPN1-4751 es una transición de C/T localizada en la posición 6545 entre el intrón 17 y el exón 18 del mismo gen6. En ambos marcadores el alelo C ha sido reportado y validado como favorable para la suavidad de la carne6. El marcador UoG CAST es una transversión G/C que se encuentra en la posición 282 entre el exón 5 y 6 y el CAST-T1 es una transición G/A localizada la región 3’ no traducible del gen. Los alelos C y A, respectivamente se han identificado como favorables para la suavidad de la carne en bovinos.
En este trabajo se tipificaron poblaciones de ganado Charolais de registro, utilizando un panel de marcadores asociados con enfermedades genéticas y con rasgos sensoriales de la carne, con el fin de determinar sus frecuencias alélicas y conocer el potencial genético-molecular de las poblaciones estudiadas.
Materiales y métodos
Utilizando el estuche comercial Promega Wizard, se realizó la extracción de ADN a muestras de sangre de 493 animales provenientes de cinco ranchos de registro de ganado Charolais. Dos de ellos localizados en Sonora, región noroeste de México, SoR1 (n=83) y SoR2 (n=74), y tres en la región noreste del estado de Nuevo León, NLR1 (n=79), NLR2 (n=100) y NLR3 (n=157). Con excepción de 21 muestras de machos, todas las muestras fueron de hembras.
Las muestras se tipificaron por discriminación alélica con los marcadores CAPN1-316 (transversión G/C), CAPN1-4751 (transición de C/T), CAST-T1 (transición A/G) y alelo Q204X (transición C/T), utilizando los iniciadores y sondas específicos para cada SNP (Cuadro 1), las cuales se sintetizaron en la compañía Applied Biosystems. Todos los ensayos se realizaron en el equipo ABI Prism 7000 Sequence Detection System, utilizando las siguientes condiciones: un ciclo de 2 min a 50 °C y 10 min a 95 °C, seguido de 40 ciclos de dos pasos 15 s a 92 °C y 1 min a 60 °C. Se utilizaron 250 ng ADN, 12.5 μl of Taqman PCR master mix (Applied Biosystems), y 0.625 ml de la mezcla de sondas e iniciadores (Assay SNP mix). La llamada de los genotipos para cada marcador se llevó a cabo utilizando el software ABI Prism 7000 Sequence Detection System.
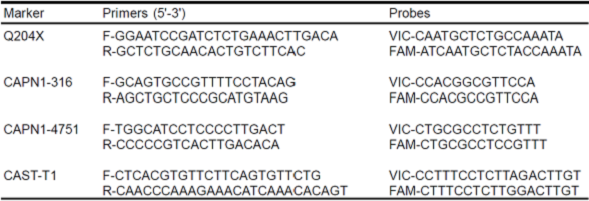
Cuadro 1 Iniciadores y sondas específicos para los marcadores Q204X, CAPN1-316, CAPN1-4751 y CAST-T1.
Adicionalmente, las muestras se tipificaron con cuatro marcadores mediante ensayos de PCR-RFLP; para estos, las secuencias de los iniciadores y las enzimas de restricción específicas para detectar el SNP se tomaron de la literatura y se describen a continuación. La PCR para el marcador TG5 se realizó utilizando los iniciadores TG5U2 5´ggg gat gac tac gag tat gac tg 3´ y TG5D1 5´gtg aaa atc ttg tgg agg ctg ta3´ que generan un fragmento de 545pb8, la transición C/T se identificó después de digerir el fragmento con la enzima Mbo I, el alelo 2 produce fragmentos de 17 pb, 73 pb, 177 pb y 278 pb mientras que el alelo 3 se caracteriza por el patrón de bandas de 73 pb, 194 pb y 278 pb. Para el marcador asociado a la EAG-V, se usaron los iniciadores reportados F-5´-CCA GGA AGA CCC TCA TTC CA-3 y R-5´-AGG GAA ACA CAC ACA CAG-3´5. La transición C/T en el codón 489 del gen de la miofosforilasa fue detectada con la enzima Sty I, donde los portadores homocigotos TT fueron identificados por un patrón de bandas de 133 pb y 119 pb y los no afectados CC una banda de 252 pb. Los marcadores asociados a DUMPS y ASS se tipificaron utilizando los iniciadores DUMPS-F 5´-GCA AAT GGC TGA AGA ACA TTC TG-3´: DUMPS_R 5´-GCT TCT AAC TGA ACT CCT CGA GT-3´y ASS-F 5´-GTG TTC ATT GAG GAC ATC-3, ASS-R 5´-CCG TGA GAC ACA TAC TTG-3, respectivamente5.
La transición (C/T) en el gen de uridina monofosfato sintasa asociado a la DUMPS, se detectó con la enzima Ava I, donde los homocigotos normales muestran un patrón de bandas de 23, 36 y 19 pb y los homocigotos recesivos de 89 y 19 pb. Mientras que para la ASS la transición C/T del codón 86 de la proteína del gen ASS se detectó con la enzima Ava II, donde un producto sin digerir identifica a los portadores mutados y un patrón de bandas de 118 y 80 pb a los portadores no afectados5.
Todas las amplificaciones se realizaron en un volumen de 12.5 μl utilizando 50 ng de ADN, 2 mM de MgCl2, 0.25 μM de cada iniciador y 0.125 U de GoTaq DNA polimerasa. Para las amplificaciones se utilizó un perfil de temperaturas tipo “touchdown” que consistió en incubación a 95ºC/10’, 5 ciclos de 95°C/45", 65°C/45” (disminuyendo 2 °C cada ciclo) y 72°C/45”; posteriormente 25 ciclos a 95°C/45”, 60°C/45” y 72°C/45”, finalmente a 72ºC/10’. La amplificación se confirmó por electroforesis en un gel de agarosa al 1.5% teñido con Syber Gold, posteriormente se visualizó en el fotodocumentador Kodak Gel Logic 112.
Las digestiones enzimáticas se realizaron utilizando 10 μl del producto de PCR y 2.5 U de enzima de restricción específica para cada polimorfismo; los patrones de bandeo de la digestión se analizaron por electroforesis en geles de agarosa Nusieve al 4.5%, la cual se preparó siguiendo las instrucciones de la casa comercial (Karlan Research Products Corporation, Phoenix, AZ 85070).
Posteriormente, se estimaron las frecuencias genotípicas y alélicas de los ocho marcadores analizados utilizando el programa Cervus 3.09. Se analizó el equilibrio de Hardy-Weinberg (HWE) y se evaluó la diferenciación genética entre las poblaciones de estudio para los cuatro marcadores asociados a calidad de la carne, utilizando el programa Genepop 4.210. En el análisis de diferenciación genética, la hipótesis nula probada fue Ho= la distribución alélica es idéntica a través de las poblaciones. Para poblaciones, la prueba se realiza automáticamente por medio de tablas de contingencia por pares de poblaciones, y se lleva a cabo una estimación no sesgada del valor de P prueba exacta de Fisher como la describen Raymond y Rousset10. Para ilustrar gráficamente la diferenciación por segregación alélica en estos marcadores se realizó un análisis de correspondencia utilizando el programa SAS ver. 9.0 (SAS Institute Inc., Cary, NC, USA).
Resultados
Marcadores asociados a desórdenes genéticos
En las poblaciones estudiadas, no se encontraron portadores de los alelos asociados a ASS y DUMPS. Sin embargo, se encontraron animales portadores para las variantes génicas asociadas a la EAG- V y al doble músculo (Cuadro 2). Para el alelo Q204X asociado al doble músculo, se encontraron en la población total 28 hembras portadoras (5.87 %) y entre hatos las frecuencias de portadoras variaron entre 0 a 14.4 %.
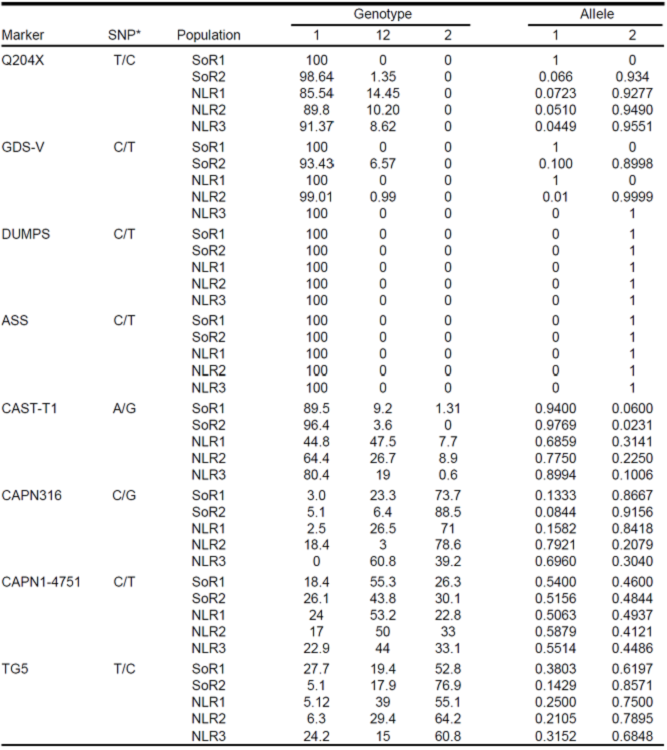
Cuadro 2 Frecuencias alélicas y genotípicas de los cuatro marcadores asociados a enfermedades genéticas y calidad de carne en ganado Charolais de registro (%).
El análisis por región permitió observar que los hatos ubicados en la región noreste de México, tienen mayores frecuencias del alelo Q204X (8.6 a 14.4 %) respecto a las localizadas en el noroeste, que mostraron una máxima frecuencia de 1 %. Este resultado podría explicarse por el origen del material genético con el que se mejoran estos hatos, ya que es común que en el noreste y en particular en los hatos analizados, la fuente de material genético sea europeo (principalmente semen Francés), y la frecuencia de sementales portadores reportadas para este alelo en algunos estudios es hasta del 27 %11,12. Para apoyar este resultado, con la información de pedigrí de las hembras portadoras identificadas en el estudio, se lograron formar seis familias de medias hermanas cuyos padres son de origen francés. De estos últimos se logró obtener una muestra biológica y la prueba de ADN confirmó que estos son portadores del alelo Q204X.
En el estudio también se encontraron cinco hembras y un macho portadores del marcador asociado a la EAG-V (Figura 1). En este caso la mayor frecuencia se registró en un hato del noroeste (6.49 %), y sólo una portadora en un hato del noreste (1 %). La verificación del pedigrí de dos de las vacas portadoras permite suponer que es posible que el alelo haya sido transmitido por vía paterna, de un semental proveniente de Estados Unidos no incluido en este estudio.
Marcadores asociados a calidad de carne
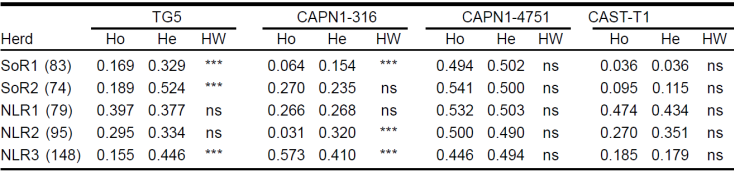
En el Cuadro 2 se muestran las frecuencias alélicas y genotípicas de los cuatro marcadores asociados a calidad de la carne. El análisis de diferenciación génica pareado entre las poblaciones y con los cuatro loci mostró que hay diferencias altamente significativas dentro de las dos poblaciones del noroeste (P<0.0001) y entre estas dos poblaciones y las tres del noreste (P<0.001). Adicionalmente entre poblaciones, la población NLR3 fue significativamente diferente de las otras dos poblaciones del noreste (NLR1 y NLR2). El análisis por loci demostró que existe diferente comportamiento para los cuatro marcadores, siendo las diferencias más significativas en el marcador TG5 entre las poblaciones del noroeste y estas dos poblaciones y la NLR3. En el marcador 4751 no se observaron diferencias significativas entre las cuatro poblaciones. A nivel de segregación alélica la Figura 2 muestra una fuerte influencia de los alelos del marcador CAPN1-316 y CAST-T1 en la diferenciación de los hatos del noreste contra los del noroeste, e indica una menor variabilidad entre los hatos de esta última región para los loci analizados.
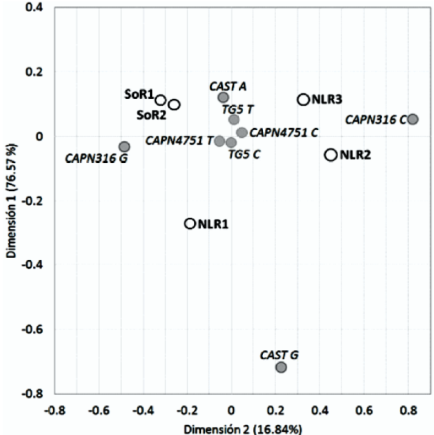
Figura 2 Gráfico factorial de correspondencia de segregación alélica de marcadores asociados a calidad de la carne en hatos del noroeste y noreste de México.
En el Cuadro 3 se muestran las heterocigosidades esperadas y observadas; sólo en dos marcadores se observó desviación al equilibrio Hardy-Weinberg. La desviación al EHW fue significativa para el marcador TG5 en las dos poblaciones del noroeste y la población NLR3 del noreste, mientras que en el marcador CAPN316 lo fue en la población SoR1 del noroeste y dos poblaciones del noreste (NLR2 y NLR3). En todos los casos la desviación fue debida a un déficit de heterocigotos.
Discusión
Enfermedades genéticas
A nivel mundial, las Asociaciones Ganaderas reconocen oficialmente, en las diferentes razas de bovinos, desórdenes genéticos que se reportan en el registro del animal. A través de esta práctica, se obtiene importante información que la Asociación Ganadera puede hacer disponible a los compradores de animales y de semen, lo que hace posible su exclusión como reproductor o manejo adecuado dentro del hato, mientras que para la Asociación es una forma de controlar la diseminación y lograr la eventual eliminación de los portadores.
Se ha descrito que el fenotipo doble músculo muestra diferencias en la frecuencia entre razas; esto se puede deber a que durante muchas generaciones los rasgos que se relacionan a este fenotipo tienen intensidades de selección que dependen de los objetivos productivos del hato o población donde se presenta4. De acuerdo a diferentes reportes, la presencia del doble músculo en la raza Charolais se ha promovido principalmente para obtener portadores en cruzas terminales12,13,14. En México, productores de la raza Charolais reportan que han optado por limitar e incluso eliminar su presencia en el hato, debido a los problemas de distocia. Para lograrlo, ellos se han basado en la inspección visual de los animales buscando las características del fenotipo DM.
En este estudio se encontraron frecuencias alélicas de 0 a 14 % y todos los animales fueron portadores heterocigotos. La presencia del alelo en las poblaciones de Charolais analizadas puede ser consecuencia de que los animales heterocigotos o portadores del alelo son difíciles de diferenciar de los portadores normales, ya que los portadores heterocigotos no muestran el marcado desarrollo muscular que es característico de los animales DM15. En situaciones como ésta, las pruebas de ADN representan una herramienta valiosa, ya que esta información dará al productor la oportunidad de decidir y manejar, de acuerdo al objetivo del hato, las ventajas y desventajas productivas y reproductivas asociadas a la presencia de esta condición genética.
En el estudio también se encontraron portadores del marcador asociado a la enfermedad de almacenamiento del glucógeno tipo V. Esta es una enfermedad muscular inducida por una mutación puntual en el gen de la enzima glucógeno fosforilasa, lo que causa intolerancia al ejercicio, mialgia y mioglobinuria recurrente. Son pocos los reportes sobre la prevalencia de este desorden recesivo en ganado bovino, y en ganado Charolais la mayoría de los reportes son casos de portadores16. Jolly et al17 reportaron la presencia de una familia portadora en hatos de ganado Charolais en Nueva Zelanda e identificaron que la presencia del alelo recesivo pudo deberse a importaciones de animales de Inglaterra o América. En México, este es el primer reporte de portadores del marcador asociado a la enfermedad de almacenamiento del glucógeno tipo V en ganado Charolais mexicano; el caso más relevante de este resultado, es la identificación de un portador macho, el cual a la fecha tiene al menos 38 crías registradas en las bases de datos de la Asociación Charolais HerdBook de Mexico (34 Machos y 4 hembras), y a las cuales a la fecha no se les ha realizado la prueba de ADN para identificar el alelo recesivo. Aun cuando las implicaciones productivas de la enfermedad de almacenamiento del glucógeno tipo V para animales portadores no han sido reportadas, el efecto de la presencia de portadores en los hatos es potencialmente dañina.
Marcadores asociados a calidad de carne
En México no se cuenta con amplia información sobre la calidad de la carne de bovinos, esto en parte se debe a que aún no están completamente definidos los criterios para su clasificación18. Sin embargo, dado que la aplicación de la biotecnología ha alcanzado a la industria de la carne y el uso del diagnóstico molecular de genes asociados a su calidad están disponibles para los ganaderos, es de interés dar el primer paso para determinar las frecuencias alélicas de estos marcadores en ganado de registro, ya que de acuerdo a la cadena productiva que prevalece en México, cualquier estrategia de mejoramiento genético empieza en las razas puras y para rasgos complejos como la calidad de la carne, se esperaría que el impacto de este mejoramiento se refleje en el ganado de sacrificio.
Los marcadores de calidad de la carne analizados en este estudio están incluidos en las pruebas comerciales para suavidad y marmoleo de la carne, por lo que se ha reportado el efecto favorable de cada variante alélica6.
De acuerdo a las frecuencias alélicas y genotípicas obtenidas en las poblaciones analizadas, todos los hatos muestran frecuencias de altas a moderadas del alelo reportado como favorable para los marcadores CAST-T1 (alelo A) y CAPN1-4751 (alelo C), mientras que para los marcadores CAPN316 y TG5 el alelo reportado como desfavorable fue el de mayor frecuencia (de 91.5 a 69.1 %, respectivamente).
En un estudio realizado en ganado Charolais francés, Allais et al11 reportaron frecuencias alélicas similares a las encontradas en este trabajo para los marcadores CAST-T1 (alta frecuencia) y CAPN316 (baja frecuencia). Se ha reportado que los dos marcadores del gen CAPN1 explican hasta el 25 % de variación genética en la suavidad de la carne19 y hasta el 18 % de su variabilidad fenotípica6; sin embargo otros autores han encontrado que este efecto es dependiente del fondo genético y de las poblaciones donde se evalúa20.
Recientemente, dos de los cuatro marcadores analizados en este trabajo han sido estudiados en toretes de ganado Charolais Mexicano21 encontrándose que el marcador CAPN1-4751 tiene efecto significativo sobre el área del músculo Longissimus dorsi (REA) y grasa intramuscular (IMF) medidos por ultrasonografía; mientras que el marcador TG5 mostró una tendencia de asociación al grado de rendimiento (YG), por lo que estudios para confirmar esta asociación podrían llevarse a cabo con las iniciativas por parte del Consejo Nacional de los Recursos Genéticos (CONARGEN) para medir rasgos de calidad de la carne basándose en ultrasonografía. El único trabajo de asociación con variables fenotípicas con los marcadores de calidad de la carne reportado a la fecha en México7, apoya la asociación favorable de los marcadores 4751 y TG5 a la fuerza de corte y marmoleo; sin embargo, dado el tamaño de muestra estudiado, no se podría considerar como un estudio de validación que permita cuantificar el efecto de los genotipos de cada marcador sobre estos rasgos. Las frecuencias encontradas para los cuatro marcadores de calidad de la carne en las poblaciones Charolais estudiadas justifican la implementación de estrategias de manejo, tendientes a aumentar su frecuencia, así como para diseñar estudios para evaluar su efecto fenotípico del manejo asistido por marcadores.
Aunque se encontró diferenciación genética en las poblaciones estudiadas y desviación al EHW para dos loci, es improbable que este resultado pueda explicarse por la selección de los rasgos de calidad de la carne (suavidad y marmoleo), ya que estos rasgos no son considerados en las estrategias de mejoramiento genético. Esta diferenciación por regiones (poblaciones del noroeste (P<0.0001) y del noreste (P<0.001) podría explicarse por el origen del material genético con el que se manejan los hatos. De acuerdo con los propietarios de los hatos del noroeste, su ganado es Charolais americano y la influencia de material genético europeo es casi nula, contrario a los tres hatos del noreste que tienen una alta influencia de material genético proveniente principalmente de Francia. Sifuentes-Rincón et al22 en un estudio para evaluar la variación genética molecular en tres poblaciones mexicanas de ganado Charolais utilizando marcadores microsatélites, encontraron diferenciación genética en la población estudiada, la cual fue seleccionada con base al material genético con el que se mejoraban las poblaciones, por lo que concluyen que la estructura encontrada es consecuencia de este manejo.
Como se expuso, los cuatro marcadores de calidad utilizados en este estudio, han sido validados en EU como predictores de la calidad y marmoleo de la carne. Existen actualmente compañías comerciales que ofertan pruebas de ADN para estos y muchos otros marcadores de calidad y productividad en ganado bovino, lo que ha abierto la posibilidad para que ganaderos o Asociaciones ganaderas las incluyan como herramienta para apoyar los programas de mejoramiento genético, evitando la selección de individuos con genotipos no favorables; sin embargo, es muy importante hacer estudios de asociación y validación de los marcadores en las poblaciones locales, a fin de precisar y cuantificar sus efectos en todos los rasgos de interés.
Conclusiones e implicaciones
De acuerdo a los resultados, la salud genética para la población estudiada es adecuada para los desórdenes recesivos UMP y ASS ya que no se detectaron portadores. Se recomienda el monitoreo de la enfermedad de almacenamiento de glucógeno donde se encontraron algunos portadores; este monitoreo es importante sobre todo en los casos de importación de semen. Respecto al alelo Q204X del gen MSTN, sus frecuencias varían entre hatos y podrían relacionarse con el origen del material genético que se utiliza en ellos; es muy importante que los criadores de esta raza reconozcan las ventajas y desventajas de la presencia de este alelo en los hatos y utilicen la prueba de ADN para aumentar o eliminar su presencia en sus poblaciones. Las poblaciones estudiadas mostraron diferenciación genética significativa cuando se evaluó con los loci asociados a rasgos de calidad y productividad. Las frecuencias encontradas en la población de estudio de los alelos previamente reportados y en algunos casos validados en la literatura como favorables para los diferentes rasgos de calidad, justifican la implementación de estrategias para confirmar su utilidad como herramienta para complementar los programas de mejoramiento genético de la raza Charolais en México.











 texto em
texto em