Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ciencias pecuarias
versión On-line ISSN 2448-6698versión impresa ISSN 2007-1124
Rev. mex. de cienc. pecuarias vol.6 no.3 Mérida jul./sep. 2015
Notas de investigación
Ensayo de PCR multiplex para la detección en leche del género Salmonella, la subespecie I y el serotipo Typhimurium
A multiplex PCR assay for detection of genus Salmonella, subspecies I, and serotype Typhimurium in milk
Ma. Soledad Vázquez-Garcidueñasª, Silvia Cristina Meléndez-Cejaª, Gerardo Vázquez-Marrufob
a División de Estudios de Posgrado, Facultad de Ciencias Médicas y Biológicas "Dr. Ignacio Chávez". Morelia, Michoacán, México.
b Centro Multidisciplinario de Estudios en Biotecnología, Facultad de Medicina Veterinaria y Zootecnia. Universidad Michoacana de San Nicolás de Hidalgo. Km 9.5 carretera Morelia-Zinapécuaro, Col. La Palma, 58893 Tarímbaro. Michoacán, México. Tel, and fax: +52 (443) 2 9 5 80 29. Correspondencia al último autor. gvazquezmarrufo@yahoo.com.mx.
Recibido el 16 de julio de 2014.
Aceptado el 25 de noviembre de 2014.
Resumen
Debido a la importancia social y económica de las enfermedades gastrointestinales causadas por alimentos contaminados con Salmonella entérica, es necesario contar con sistemas de detección rápidos, sensibles y específicos para la detección oportuna de dicho patógeno. En este trabajo se presenta un protocolo de PCR de punto final para la identificación de S. entérica en leche, que utiliza los pares de iniciadores STM3098-f2/STM3098-r2, STM4057-f/STM4057-r y STM4497-f/STM4497-r previamente validados como específicos del género Salmonella, de la subespecie I y del serotipo Typhimurium, respectivamente. El protocolo presentado permite la detección de 1 UFC/25 ml de leche contaminada artificialmente, con un periodo de pre-enriquecimiento de 12 h, en ensayos de PCR simple y multiplex con los tres pares de iniciadores. El protocolo diseñado puede ser aplicado en sistemas de vigilancia sanitaria para la detección de S. enterica en leche.
Palabras clave: Salmonella enterica, Leche, PCR multiplex.
Abstract
Gastrointestinal diseases caused by foodstuffs contaminated with Salmonella enterica implicate important social and economic issues, making it necessary to have fast, sensitive, and specific systems for opportune detection of the pathogen. An endpoint PCR protocol for identification of S. enterica in milk is presented using primer pairs previously validated as specific to the genus Salmonella (STM3098-f2/STM3098-r2), subspecies enterica (subspecies I) (STM4057-f/STM4057-r), and serotype Typhimurium (STM4497-f/ STM4497-r). This protocol allowed for detection of 1 CFU/25 mL of spiked milk after a 12 h pre-enrichment period by means of simple and multiplex PCR assay with the three used primer pairs. The designed protocol can be applied in sanitary survey programs for detection of S. enterica in milk.
Key words: Salmonella enterica, Milk, Multiplex PCR.
Las enfermedades transmitidas por alimentos representan una de las causas más importantes de morbilidad y mortalidad, tanto en países desarrollados como en aquéllos en vías de desarrollo. Se estima que 1.8 millones de personas mueren anualmente a causa de enfermedades diarreicas y una gran proporción de estos casos se puede atribuir a la contaminación de alimentos(1). Entre los principales agentes patógenos causantes de enfermedades diarreicas que se transmiten a través de alimentos se encuentra Salmonella enterica(2), incluidos la leche y los derivados lácteos(3,4). La Organización Mundial de la Salud estima que decenas de millones de casos de salmonelosis no tifoidea ocurren en el mundo anualmente, resultando en más de cien mil muertes(5). La detección oportuna de S. enterica en alimentos lácteos previene el surgimiento de brotes y permite mantener un control adecuado en las cadenas de producción(6).
Además de los métodos microbiológicos estándar para la detección de S. enterica en alimentos, se ha diseñado una gran diversidad de ensayos basados en la PCR tanto de punto final como de tiempo real(7,8,9), algunos de los cuales ya se encuentran disponibles como kits comerciales(7,10). Aunque los ensayos de PCR en tiempo real para la detección de S. enterica en alimentos tienen gran sensibilidad, rapidez y especificidad(7,8), los altos costos de implementación y el nivel de capacitación requerido limitan el acceso a dichas técnicas por las instituciones públicas encargadas de la inocuidad de alimentos y vigilancia sanitaria, manteniendo a los ensayos de PCR de punto final como una opción para la detección de S. enterica en alimentos en países en vías de desarrollo como es el caso de México.
En este trabajo se diseñó un protocolo para la detección de S. enterica en leche, con una sensibilidad 1 unidad formadora de colonia (UFC)/25 ml después de un periodo de pre-enriquecimiento de 12 h, con los iniciadores STM3098-f2r2, STM4057-fr and STM4497-fr previamente validados como específicos de género, subespecie y serotipo Typhimurium, respectivamente. Sin embargo, no s e ha evaluado la utilidad de dichos iniciadores para la detección de S. enterica en alimentos y particularmente en leche.
Se utilizaron los iniciadores STM3098-f2 (5'-TTTGGCGGCGCAGGCGATTC-3') y STM3098-r2 (5' -GCCTCCGCCTCATCAATCCG-3'), específicos del género Salmonella; STM4057-f (5'-GGTGGCCTCGATGATTCCCG-3') y STM4057-r (5' -CCCACTTGTAGCGAGCGCCG-3'), específicos de la subespecie enterica (subespecie I); STM4497-f (5' -AACAACGGCTCCGGTAATGA-3') y STM4497-r3 (5'-TGACAAACTCTTGATTCTGA-3'), específicos del serotipo Typhimurium, descritos por Kim et al(11,12). En todos los ensayos se utilizó como testigo positivo de amplificación el ADN de la cepa de Salmonella enterica Serotipo Typhimurium (ATCC14028) y como testigo negativo el ADN de la cepa de Escherichia coli (ATCC11229).
Para los ensayos de inoculación artificial se emplearon tres tipos de leche comercial pasteurizada, a las cuales se les hicieron pruebas para la detección de UFC de enterobacterias, generando resultados negativos. Las muestras se conservaron a 4 °C hasta su análisis. El tiempo entre la adquisición de las muestras de leche y el análisis fue menor a 48 h.
La inoculación con S. enterica de las muestras de leche se realizó de acuerdo a Day et al(13), inoculando 25 ml de leche con diluciones seriadas desde 108 hasta 1 UFC. El proceso de pre-enriquecimiento para cada cantidad de UFC probada y el tiempo de incubación analizado se realizó de acuerdo a Oliveira et al(14), empleando 25 ml de leche y 225 ml de agua peptonada (AP) al 1% a pH regulado.
La extracción y purificación de ADN para todas las muestras de leche se realizó como se describe a continuación. Se tomaron 500 μl de leche y se le adicionaron 500 μl de regulador de lisis (Tris-HCl 100 mM a pH 8.0, SDS 2%, NaCl 100 mM y EDTA 50 mM) agitando vigorosamente por 10 min. Enseguida se añadieron 500 μl de fenol - cloroformo (1:1 v/v) y se homogenizó durante 5 min en vórtex. Se centrifugó por 10 min a 9,300 xg y se rescató la fase acuosa a la cual se le agregó el mismo volumen de isopropanol frío, incubando por 10 min a -20 °C. Se centrifugó a 9,300 xg durante 10 min, se desechó el sobrenadante y la pastilla recuperada se lavó con 250 μl de etanol al 70%, dejando secar a temperatura ambiente y resuspendiendo en 20 μl de agua desionizada esterilizada.
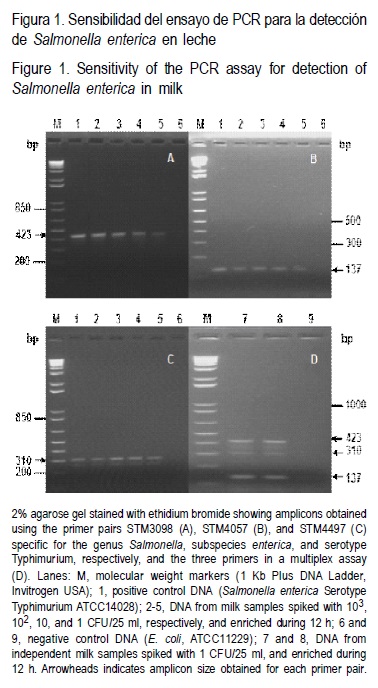
Después de diversos ensayos de optimización, la concentración de los reactivos utilizados en los ensayos de PCR fueron: buffer Tris-HCl pH 8.5 10 mM, MgCl2 1.50 mM, 0.4 μM de cada iniciador, 0.2 mM de cada dNTP, 25 ng de ADN y 0.5 U de Taq DNA polimerasa (Invitrogen, USA), en un volumen final de 25 μl. El programa de amplificación empleado tanto para los ensayos de PCR simple como multiplex consistió de un ciclo inicial de 3 min a 94 °C, seguido de 30 ciclos a 94 °C durante 45 seg, 65 °C por 30 seg, 72 °C por 30 seg y un paso final de 3 min a 72 °C(12). Tanto el ADN de alto peso molecular obtenido como los productos de amplificación generados se visualizaron mediante electroforesis en geles de agarosa al 2% y teñidos con bromuro de etidio.
Después de probar tiempos de enriquecimiento de 2 a 24 h, con intervalos de 2 h y 103, 102, 10 y 1 UFC/25 ml de leche, se observó que es factible obtener productos de amplificación con los tres pares de iniciadores empleados, en leche contaminada artificialmente hasta con 1 UFC/ 25 ml y un tiempo de pre-enriquecimiento de 12 h (Figuras 1, A,B,C). En estas mismas muestras y usando el mismo programa de amplificación, se generó un ensayo multiplex con los tres iniciadores (Figura 1, D). En este ensayo de PCR multiplex se obtuvo amplificación positiva en leche contaminada artificialmente en una concentración de 1 UFC/25 ml con un tiempo de pre-enriquecimiento de 12 h (Figura 1, D). Tanto los ensayos de PCR simple como multiplex fueron reproducibles, generando los mismos resultados de la Figura 1 en cinco ensayos independientes para cada tipo de iniciador y en el ensayo multiplex.
Otro par de iniciadores también denominados STM4497 distintos a los empleados aquí, pero dirigidos al mismo gen, han sido empleados en ensayos de PCR multiplex para la detección de S. Typhimurium en muestras de agua, carne de pollo y de res, con una sensibilidad de 100 UFC/100 ml, <60 y <50 UFC/mg, respectivamente(15). Tanto los iniciadores empleados por Shanmugasundaram et al(15) como los de Kim et al(11,12) usados en este trabajo, reafirman la especificidad de la región genética STM4497 como específica del serotipo Typhimurium, y por lo tanto, su utilidad para la detección específica de dicho serotipo en muestras de alimentos. Los tres pares de iniciadores pueden emplearse de manera independiente o en un ensayo multiplex, si interesa saber si el serotipo contaminante es Typhimirum, ya que dicho serotipo es el que más comunmente se encuentra contaminando leche y productos lácteos(3).
La sensibilidad obtenida por el ensayo aquí diseñado en leche contaminada artificialmente es superior a la reportada por Shanmugasundaram et al(15) en agua y carne. La sensibilidad reportada para otros ensayos de PCR simple o multiplex para determinar la presencia de S. enterica en leche contaminada artificialmente es 1-10 UFC/ml empleando iniciadores dirigidos al gen invA(16).
Previamente se ha reportado que la incubación directa de S. enterica en leche durante 12 h puede detectar 102 UFC/ml empleando iniciadores dirigidos al gen de la subunidad 16S de rRNA(17). También se ha notificado un protocolo de detección sin enriquecimiento, en el cual es posible detectar hasta 5 bacterias/ml en muestras de leche empleando iniciadores dirigidos al gen hilA(18). La sensibilidad de 1-10 UFC/ml se ha obtenido con un pre-enriquecimiento 18 h en caldo de soya y triptona (16). Las 12 h de pre-enriquecimiento en AP al 1% aseguran la detección hasta de 1 UFC/25 ml de leche, lo cual está por arriba de los límites de detección reportados previamente en ensayos de PCR de punto final.
En otro trabajo se ha reportado el uso de centrifugación para recuperar el pellet bacteriano y un tratamiento con EDTA y TritónX-100 para eliminar grasas y proteínas que inhiben la PCR(18), lo cual no fue necesario en el presente trabajo.
En conclusión, el protocolo aquí reportado es superior en sensibilidad, especificidad y rapidez a otros protocolos de PCR de punto final para la detección de S. enterica en leche. Adicionalmente, es de esperar que la sencillez del protocolo reportado represente una ventaja adicional para su implementación en sistemas de vigilancia sanitaria por parte de agencias de salud pública de países de escasos recursos.
AGRADECIMIENTOS
Se agradece al CONACYT por la beca de maestría otorgada a Silvia Cristina Meléndez-Ceja (No. de becario 253389). El presente trabajo fue financiado por el proyecto SALUD-2009-01-115172 del Fondo sectorial de investigación en salud y seguridad social SSA/ IMSS/ISSSTE-CONACYT, CONVOCATORIA 2009.
LITERATURA CITADA
1. Stein C, Kuchenmüller T, Hendrickx S, Prüss-Üstün A, Wolfson L, Engels D, Schlundt J. The global burden of disease assessments-WHO is responsible? PLoS Negl Trop Dis 2007;1(3):e161. [ Links ]
2. Newell DG, Koopmans M, Verhoef L, Duizer E, Aidara-Kane A, Sprong H, et al. Food-borne diseases — The challenges of 20 years ago still persist while new ones continue to emerge. Int J Food Microbiol 2010;139:S3-S15. [ Links ]
3. Oliver SP, Boor KJ, Murphy SC, Murinda SE. Food safety hazards associated with consumption or raw milk. Foodborne Pathog Dis 2009;6:793-806. [ Links ]
4. Langer AJ, Ayers T, Grass J, Lynch M, Angulo FJ, Mahon BE. Nonpasteurized dairy products, disease outbreaks, and state laws—United States, 1993-2006. Emerg Infect Dis 2012;18:385-391. [ Links ]
5. WHO (2013) http://www.who.int/mediacentre/factsheets/fs139/en/index.html. Accessed: 1st Dec, 2013.
6. Papademas P, Bintsis T. Food safety management systems (FSMS) in the dairy industry: A review. Int J Dairy Technol 2010;63:489-503. [ Links ]
7. Maurer JJ. Rapid detection and limitations of molecular techniques. Annu Rev Food Sci Technol 2011;2:259-279. [ Links ]
8. Logue CM, Nolan LK. Emerging bacterial food-borne pathogens and methods of detection. In: Simpson BK, et al, editors. Food Biochemistry and Food Processing, 2nd ed., New York: John Wiley & Sons, Inc; 2011:833- 857. [ Links ]
9. Riyaz-Ul-Hassan S, Verma V and Qazi GN. Real-time PCR-based rapid and culture-independent detection of Salmonella in dairy milk - addressing some core issues. Lett Appl Microbiol 2013;56:275-282. [ Links ]
10. Glynn B, Lahiff S, Wernecke M, Barry T, Smith TJ, Maher M. Current and emerging molecular diagnostic technologies applicable to bacterial food safety. Int J Dairy Technol 2006;59:126-39. [ Links ]
11. Kim HJ, Park SH, Kim HY. Comparison of Salmonella enterica serovar Typhimurium LT2 and non-LT2 Salmonella genomic sequences, and genotyping of Salmonellae by using PCR. Appl Environ Microbiol 2006;72:6142-6151. [ Links ]
12. Kim HJ, Park SH, Lee TH, Nahm BH, Chung YH, Seo KH, Kim HY. Identification of Salmonella enterica serovar Typhimurium using specific PCR primers obtained by comparative genomics in Salmonella serovars. J Food Prot 2006;69:1653-1661. [ Links ]
13. Day JB, Basavanna U, Sharma SK. Development of a cell culture method to isolate and enrich Salmonella enterica serotype Enteritidis from shell eggs for subsequent detection by Real-Time PCR. Appl Environ Microbiol 2009;75:5321-5327. [ Links ]
14. Oliveira SD, Rodenbusch CR, Cé MC, Rocha SLS, Canal CW. Evaluation of selective and non-selective enrichment PCR procedures for Salmonella detection. Lett Appl Microbiol 2003;36:217-221. [ Links ]
15. Shanmugasundaram M, Radhika M, Murali HS, Batra, HV. Detection of Salmonella enterica serovar Typhimurium by selective amplification of fliC, fljB, iroB, invA, rfbJ, STM2755, STM4497 genes by polymerase chain reaction in a monoplex and multiplex format. World J Microbiol Biotechnol 2009;25:1385-1394. [ Links ]
16. Balakrishna, K, Murali HS, Batra HV. A novel multiplex polymerase chain reaction for simultaneous detection of Yersinia enterocolitica, Staphylococcus aureus, Aeromonas and Salmonella from chicken meat and milk samples. J Food Safety 2010;30:263-275. [ Links ]
17. Aslam M, Hogan J, Smith KL. Development of a PCR-based assay to detect shiga toxin-producing Escherichia coli, Listeria monocytogenes, and Salmonella in milk. Food Microbiol 2003;20:345-350. [ Links ]
18. Marathe SA, Chowdhury R, Bhattacharya R, Nagarajan AG, Chakravortty D. Direct detection of Salmonella without pre-enrichment in milk, ice-cream and fruit juice by PCR against hilA gene. Food Control 2012;23:559-563. [ Links ]














