Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ciencias pecuarias
versión On-line ISSN 2448-6698versión impresa ISSN 2007-1124
Rev. mex. de cienc. pecuarias vol.4 no.4 Mérida oct./dic. 2013
Artículos
Virus del síndrome reproductor y respiratorio porcino (PRRSV) en granjas porcinas tecnificadas del Estado de México
Porcine reproductive and respiratory syndrome virus (PRRSV) in technified pig farms from the State of México
Sandra Maricruz López Heydecka, Gabriel Gerardo Huitrón Bravoa, Salvador Lagunas Bernabéb, Edgardo Soriano Vargasb, Arturo Cabrera Torresc, Fernando de la Cruz Valdésc
a Centro de Investigación en Ciencias Médicas de la Universidad Autónoma del Estado de México. Jesús Carranza No. 205 Col. Universidad 50130, Toluca, Méx., México. Teléfono y fax: 52(722) 219 4122, 219 3675. heydeck@hotmail.com. Correspondencia al primer autor.
b Centro de Investigación y Estudios Avanzados en Salud Animal. Facultad de Medicina Veterinaria y Zootecnia. Universidad Autónoma del Estado de México. México.
c Comité de Fomento y Protección Pecuaria del Estado de México. México.
Recibido el 15 de noviembre de 2012.
Aceptado el 21 de febrero de 2013.
Resumen
Se realizó un estudio transversal por conglomerados en una etapa, de abril a septiembre de 2011, en cerdos de más de 11 semanas de edad, de 11 granjas, para analizar la situación de PRRSV en granjas tecnificadas del Estado de México. Se colectaron muestras de suero (n=220) para ELISA y sangre (n=80) para transcripción en reversa y reacción en cadena de la polimerasa en tiempo real en un solo paso (RTqPCR) para PRRSV, e hisopos nasales (n= 425) para el aislamiento de posibles bacterias patógenas de la enfermedad del complejo respiratorio porcino (CRP). También se aplicó un cuestionario. ELISA mostró una seroprevalencia verdadera de 30.67 %, siendo mayor la probabilidad de ser seropositivos en granjas con antecedentes y calendario de vacunación y de ubicación urbana-semiurbana; los cerdos fueron seroreactivos en 7/11 granjas (63.63 %). RTqPCR mostró viremia en 2/80 (2.5 %) cerdos analizados, de 2/11 granjas (18.18 %); una cerda infectada con PRRSV norteamericano que mostró signos clínicos de la enfermedad y una cerda infectada con PRRSV europeo que no mostró ningún signo clínico evidente de infección. Dos granjas no mostraron cerdos seropositivos o virémicos, no tienen antecedentes ni calendario de vacunación y están ubicadas en una zona semirural. En todas las granjas se aisló Staphyloccus aureus y Streptococcus suis; en algunas granjas y con menos frecuencia Actinobacillus spp (4/11) y Pasteurella spp (3/11). Es el primer reporte en México de infección por PRRSV europeo.
Palabras clave: PRRSV, PCR, ELISA, Enfermedad del complejo respiratorio del cerdo.
Abstract
A cluster cross sectional study in a single stage was carried out from April to September 2011 on pigs over 11 wk old from 11 farms, to analyze the viral porcine reproductive and respiratory syndrome (PRRSV) status of technified farms of the State of Mexico. Sera samples (n=220) for ELISA, blood samples (n=80) for one step real-time quantitative reverse transcriptase-PCR (RTqPCR), nasal swabs (n=425) for bacterial isolation of possible porcine respiratory disease complex syndrome (PRDC) pathogens were collected. Additionally information about individuals and farm condition was gathered for further analysis. ELISA shows a 30.67 % PRRSV true seroprevalence been more as likely to be seropositives in farms with PRRSV background and vaccination schedule and of urban-semiurban location; pigs were seroreactive for PRRSV in 7/11 farms (63.63 %). Viremia was found through RTqPCR in 2/80 (2.5 %) of the total of pigs sampled for it, and was in 2/11 farms (18.18 %). We found a sow infected with a European PRRSV that did not show any evident clinical signs of infection, and a sow with a North American PRRSV who displayed clinical signs of the disease. Two farms didn't show seropositive or viremic pigs; these two farms had no PRRSV background or PRRSV vaccination schedule and are located in a semirural area. Staphyloccus aureus and Streptococcus suis where isolated from all the farms and Actinobacillus spp (4/11) or Pasteurella spp (3/11) where found less frequently in some farms. This is the first report in México of a European type PRRSV infection.
Key words: PRRSV, PCR, ELISA, Bacterial isolation, Porcine respiratory disease complex.
INTRODUCCIÓN
El PRRS es una enfermedad endémica en la mayoría de los países de producción porcina(1). En México se detectó por primera vez en 1992(2). Está más frecuentemente asociada a granjas tecnificadas que a granjas de traspatio(3). La primoinfección de la granja puede ocasionar pérdidas significativas y posteriormente reemerger como brote agudo(4,5). Ocasiona pérdidas de aproximadamente 10 % de la producción anual de lechones(2). El virus de PRRS (PRRSV) es un Arterivirus; es pequeño y envuelto, con ARN linear de una cadena, de sentido positivo, de 15 kb, con nueve marcos de lectura abiertos conocidos(6-9). Se clasifican en linajes: europeo (tipo I) y norteamericano (tipo II)(7,8,10); hay una gran diversidad genética entre estos y de cepas norteamericanas (NA), con diferentes propiedades antigénicas, que ocasionan sintomatología similar(6,10); pudiendo haber una rápida variación o recombinación genética(11,12).
La vía de entrada es oro-nasal o genital, llega a nódulos linfáticos regionales y se distribuye sistémicamente libre o en los monocitos circulantes, ocasionando leucopenia. Se replica en diferentes órganos y tejidos, pero infecta principalmente a macrófagos alveolares, células dendríticas y monocitos(13). Ocasiona fiebre, disnea, enrojecimiento de la piel, edema en los párpados, conjuntivitis, depresión, anorexia, diarrea mediana, inadecuada ganancia de peso; disminuye la calidad del semen, aumenta los índices de repeticiones de celo, abortos (a finales de la gestación), momificaciones, nacimientos prematuros, mortalidad en recién nacidos, nacimiento de lechones débiles, y mortalidad de lactantes(14,15,16); aumenta la frecuencia de problemas respiratorios de cerdos en desarrollo solo, asociado con patógenos bacterianos, o incrementando la frecuencia de otras enfermedades respiratorias(2,4,5). El PRRSV es una de muchas posibles etiologías de la enfermedad del complejo respiratorio porcino (CRP)(17). Es posible se presente de manera asintomática o tan solo con una sintomatología general indistinguible de otras enfermedades del aparato reproductor o respiratorio. De acuerdo al tipo de virus de PRRS y a la característica respuesta inmunológica individual, podría destruirlo, desarrollar un proceso crónico con diseminación prolongada del virus, inducir una inmunidad de corto plazo con posible nuevo desarrollo clínico de la enfermedad(4,5), o ser portador con baja viremia y títulos bajos en tejidos limitado a algunos tejidos linfoides, de difícil diagnóstico(4,5,6); generándose cerdos en diferentes estados de infección en cambio constante, de lo cual depende la estabilidad de la granja(4,5). La viremia varía según la edad y así el tiempo de transmisión del virus(15), siendo de una a dos semanas en adultos y de 10 a 12 semanas a incluso meses en cerdos jóvenes(4,5,12), eliminándose por saliva, orina, semen, secreciones mamarias, trasplacental (a partir de la implantación) y quizá excremento(1,7).
Existe la posibilidad de la activación, recombinación (con cepas de campo) y transmisión de virus de vacunas atenuadas(10,12). En granjas endémicas con cerdos asintomáticos, el diagnostico se basa en la combinación de un método serológico como ELISA y una técnica que determine la presencia del virus, su proteína o su ARN y su o sus linajes norteamericano o europeo, para determinar circulación y presencia del virus(1,6); el aislamiento viral, es un proceso largo (7 a 14 días), las muestras son difíciles de obtener y manejar con algunas cepas de campo difíciles de aislar; el RT PCR es más rápido, detecta infecciones en más muestras y en tiempos más tardíos de infección, el RTqPCR es más sensible, cuantificable, hay menor manejo de muestra que permite mayor rapidez, menor error y un diagnóstico más preciso, incluso para múltiples agentes en un solo tubo(6).
Se ha reportado la presencia de PRRSV en algunos estados de México(14,16,18), sin embargo, no hay reportes de referencia acerca de la situación por infección de PRRSV antecedente o actual en el estado de México, excepto ELISA de sueros de cerdos de traspatio de dos municipalidades. Por lo tanto el objetivo de este trabajo fue determinar la condición de PRRSV en granjas tecnificadas del estado de México por ELISA y RTqPCR en un solo paso (determinando linaje), análisis bacteriológico de posibles bacterias del CRP presentes en muestras de hisopos nasales y recopilación de información relacionada a PRRSV y CRP mediante un cuestionario.
MATERIALES Y MÉTODOS
Muestreo
Se realizó un estudio transversal por conglomerados, en una etapa, en cerdos, de abril a septiembre de 2011, sujeto a disponibilidad, cooperación participativa y presupuesto. Las 33 granjas tecnificadas del estado de México tenían un estimado de 77,431 cerdos (N); se realizó una selección al azar por lotería, de 11 granjas que tenían un estimado de 19,353 cerdos. Se determinó el tamaño de muestra considerando un 95% de intervalo de confianza (valor Z), seroprevalencia del 30% (p) o viremia del 3% (p)(14), precisión de error de 0.10 (d), efecto de diseño (D) 2 por conveniencia; se dividió entre 11 granjas para tamaño de muestra para cada conglomerado.

Por conveniencia se decidió para cada granja, un muestreo al azar por lotería de 20 cerdos para la circulación viral mediante ELISA y ocho cerdos para la presencia viral mediante RTqPCR, tomados de las etapas productivas de lactación, gestación, engorda y reemplazos (5 y 2 para cada prueba respectivamente). Se tomaron 40 hisopos nasales por granja para aislamiento bacteriológico, procesando por conglomerado de muestras de hisopos de 10 cerdos de cada etapa productiva de la granja. Los animales no fueron vacunados al menos un mes antes de ser muestreados. El estudio transversal para la seroprevalencia verdadera (P)(20) de PRRSV consideró una sensibilidad (Se) del 97.6 % y especificidad (Sp) del 98.6 % para ELISA(21) y aunque se conoce una sensibilidad (Se) y especificidad (Sp) del 100 % para RTqPCR(21,22,23), al ser menos de 100 muestras por granja la prevalencia absoluta (Pa) sólo se considera como proporción:
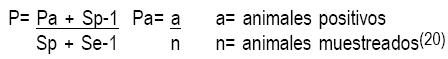
Se aplicó un cuestionario sujeto a participación cooperativa. Para la asociación de variables a PRRSV se realizaron estudios de casos-testigo, en tablas de contingencia de 2x2, para el 95% de confianza con razón de probabilidades OR (odds ratio), intervalo de confianza del 95%, valores P de "ji" cuadrada de asociación sin corrección y con corrección de Yates y de Prueba exacta de Fisher, utilizando el software EPIDAT versión 3.1(24,25).
ELISA
Los muestreos para ELISA y RTqPCR fueron realizados de manera independiente, sin embargo algunos cerdos resultaron muestreados para ambas pruebas.
Para el análisis serológico con ELISA se colectaron muestras de sangre (n=220: 20 animales/ granja) por el personal calificado del Comité de Fomento y Protección Pecuaria del Estado de México (CFPPEM) de las 11 granjas, de abril 11 a septiembre 19 del 2011, de cerdos reproductores y reemplazos de más de 11 semanas de edad. Las muestras se tomaron asépticamente por venopunción de la vena cava anterior en cerdos jóvenes o de la vena yugular externa en cerdos mayores, con agujas 21g x 1½" (0.8 x 38 mm) y tubos de 7 ml estériles; las muestras se conservaron alrededor de una hora a temperatura ambiente para después ser transportadas al laboratorio en cajas aislantes a 4 °C. La sangre se centrifugó 5 min a 2,500 rpm, se colectó el suero y se almacenó a 4 °C hasta su análisis. La serología se realizó con el kit para prueba de unión enzimática (ELISA), IDEXX kit HerdChek PRRS 2XR Ab test (Holland), No. de catálogo 99-09418, diseñado para detectar anticuerpos IgG anti-PRRSV en suero de cerdos y antígenos de células normales del hospedero (NHC), siguiendo las instrucciones del fabricante. La presencia o ausencia de anticuerpo anti-PRRSV, se determina calculando el índice S/P para cada muestra, considerando positivo para anticuerpos anti-PRRSV, valores de índice de S/P mayores o iguales a 0.40 y negativos para anticuerpos anti-PRRSV, valores de índice de S/P de menos de 0.40, cálculos realizados con el software xCHECK (USA) version 3.3 de IDEXX.
RTqPCR
En el muestreo para RTqPCR (n= 80), se procuraron ocho muestras por granja (G); algunas muestras no fueron óptimas, resultando siete muestras en G3 y G5, seis muestras en G1 y cuatro muestras en G11. Las muestras fueron de sangre completa tomadas asépticamente de abril 12 a julio 11 de 2011 por el CFPPEM, con el mismo criterio, utilizando tubos de 4 ml y agujas de 21G x 1.5" (0.8 x 33 mm), estériles. Las muestras se guardaron y transportaron en cajas aislantes manteniéndolas a 4 °C por no más de 17 h para su análisis. La extracción de ARN se realizó en el sistema automatizado del MagNa Pure LC 2.0 Roche (Japan), con el kit de extracción MagNA Pure LC RNA Isolation Kit - High Performance, Roche 3542394001 (Mannheim, Germany), siguiendo las instrucciones del fabricante para la lisis externa de 200 ul de muestra (150 ul de sangre, 50 ul PBS1X o solución salina fisiológica de NaCl2, 3-4 ul Xeno RNA testigo). La pureza y concentración del ARN se evaluó en un NanoPhotometer, Implen (Munich, Germany). El RTqPCR en un solo paso se realizó en un Fast 7500 termocycler, Applied Byosistems (Singapore, USA patented 5475.610, etc.), siguiendo el protocolo estándar para presencia/ausencia en el Software Versión 1.4, para analizar las mezclas de reacción con el kit TaqMan® NA and EU PRRSV Reagents, Ambion/Applied Biosystems 4405547 (USA), que diferencia los tipos de PRRSV NA y EU, y el kit TaqMan® NA and EU PRRSV and XenoTM RNA Controls Ambion/Applied Biosystems 4405548 (USA), siguiendo las instrucciones del fabricante para el RTqPCR en un solo paso, en una reacción de 25 ul, con no más de 100 ng de templete de ARN (60 a 80 ng) muestra en 8 ul; cada corrida tuvo sus reacciones de testigo negativo (blanco) y testigo positivo (NAPRRSV, EUPRRSV, Xeno RNA testigo, 20 copias/ul cada uno). Las sondas de hidrólisis TaqMan indicaron a sus correspondientes muestras positivas, detectándolas en sus canales correspondientes de 520 nm para PRRSV NA con FAM TM dye detector; 550 nm para PRRSV EU con VIC TM dye detector para Cal Fluor Orange 560 dye; 670 nm para Xeno RNA Control con Cy TM 5 dye detector para Quasar 670 dye; con ROX dye como referencia pasiva (610 nm).
Cultivo bacteriológico de muestras de hisopos nasales
Para los aislamientos bacteriológicos el personal de CFPPM colectó hisopos nasales (n= 425) con el mismo criterio, de abril 11 a agosto 1° de 2011. Fueron cuarenta y un conglomerados de muestras (10 hisopos cada uno) de las cuatro etapas productivas de cada granja; una de las granjas (G3) sólo tuvo un conglomerado de muestras (40 hisopos) para las cuatro etapas. Las muestras se guardaron a 4 °C para su transporte. Los cultivos bacterianos y todo el trabajo bacteriológico desde su inicio, cultivo e identificación de bacterias se realizó siguiendo los procedimientos estándares para identificar bacterias del CRP(26). Cada conglomerado de muestras fue sembrado en agar McConkey y en agar sangre (10% sangre ovina) para ser incubados durante la noche a 37 °C. También fueron sembradas en agar sangre (10% sangre ovina) con una línea central de colonias nodrizas de Staphylococcus epidermidis e incubadas durante la noche en ambiente micro anaeróbico a 37 °C. Después de su cultivo, las colonias se sometieron a las pruebas bioquímicas de rutina para caracterizar las bacterias.
Se aplicó un cuestionario a los encargados de las granjas, de agosto a octubre de 2011.
Análisis previos mostraron a las granjas seronegativas a Aujesky. Todas las granjas a excepción de una (F1) vacunan contra parvovirus porcino, leptospirosis y erisipela porcina.
RESULTADOS
Los resultados por ELISA revelaron una prevalencia verdadera de 30.67 % en suero por anticuerpos anti-PRRSV en la población estudiada (30.90 % prevalencia absoluta) (Cuadro 1); 7 de 11 granjas fueron seropositivas en el Norte y Noreste del estado (Figura 1) en un rango de proporciones por granja de 15 a 100 % (0.3090 promedio, 0.30 mediana), una con el 15 % y valores de 0 % en cuatro granjas.
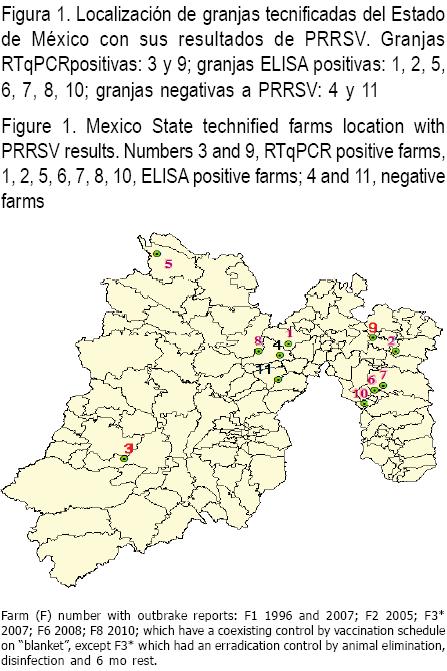
Los resultados por RTqPCR mostraron la presencia del virus de PRRS en sangre en una proporción de 2/80 (2.5 %) de los cerdos estudiados. Dos de las 11 granjas estudiadas tuvieron una cerda virémica cada una; la granja 3 (F3) tuvo una cerda infectada con PRRSV europeo; la granja 9 (G9) tuvo una cerda infectada con el PRRSV norteamericano, con una proporción aproximada de virémicos del 13.39 % en estas granjas positivas, las cuales fueron seronegativas.
Una proporción del 81.81 % (9/11) de las granjas estuvieron afectadas por PRRSV. De éstas 4/11 (36.36 %) tenían antecedentes de brote de PRRSV y su control mediante calendario de vacunación establecido contra PRRSV (PRRSVav) y sólo mostraron anticuerpos anti-PRRSV; 3/11 (27.27 %) granjas no tenían antecedentes de brote de PRRSV ni calendario de vacunación contra PRRSV (NoPRRSVav) y sólo mostraron anticuerpos anti-PRRSV; en 2/ 11 (18.18 %) granjas NoPRRSVav, sólo se detectó ARN viral. Únicamente dos granjas (18.18 %, F4 y F11), NoPRRSVav, se mostraron libres de PRRSV al no detectarse anticuerpos o ARN viral (Cuadro 1).
En todas las granjas se aislaron bacterias potencialmente patógenas. La más frecuentemente aislada fue Staphylococcus aureus en una proporción del 70.73 % (29/41) de los conglomerados de muestras, seguida de algunos microorganismos bacterianos primarios causantes de CRP: 58.53 % Streptococcus suis, 17.07 % Actinobacillus spp (como posibilidad de A. pleuroneumoniae y Haemophilus parasuis), 9.75 % Pasteurella spp (como posibilidad de P. multocida; incluyendo la granja con la cerda con PRRSVEU); las cuales se presentaron en diferentes combinaciones en los conglomerados de muestras de la granja, nunca faltando Staphylococcus aureus y Streptococcus suis (staph-strepto) en las granjas (Cuadro 1).
Durante el presente estudio ninguna de las granjas mencionó algún brote de PRRS, alguna de sus manifestaciones clínicas o problemas respiratorios de consideración. En todas las granjas se aplica el criterio de un bajo uso de la monta natural como un conocido factor de riesgo(16,18).
Las granjas con serologías positiva a PRRSV tuvieron viremias negativas, lo cual es considerado exposición con pregunta abierta a infección. De estas granjas las granjas F1 (9/20), F2 (6/20), F6 (20/20) y F8 (8/20) tienen antecedentes de brote de PRRSV y han decidido controlarlo coexistiendo con el virus mediante calendarios de vacunación contra PRRSV en "sábana", por lo que esta exposición es bajo protección (Cuadro 1). F10 (14/20), F5 (8/20) y F7 (3/20) dijeron no tener antecedentes de PRRSV y no vacunar contra éste, lo que indica una población potencialmente infectada en alto riesgo.
Las únicas dos granjas con viremia positiva declarada por la detección de ARN viral en la sangre de una cerda cada una, F3 y F9, tuvieron serologías negativas; (Cuadro 1) ninguna vacuna contra PRRSV y tampoco contra bacterias del CRP, además de staph-strepto sólo en F3 se aisló Pasteurella spp. F3 está ubicada al suroeste del estado (Figura 1) y es la única granja con antecedente de erradicación de PRRSV (2007); hasta ahora repuebla con cerdos libres de PRRSV procedentes de Sonora, sin tener brotes de PRRSV hasta la fecha, manteniendo buenos índices; la cerda virémica fue RTqPCR positiva a PRRSV europeo (PRRSVEU) y ELISA seronegativa, lo que es considerado infección aguda; fue comprada en diciembre de 2010, cuarentenada e inseminada en julio 27 de 2011 y para agosto continuaba preñada sin mostrar ningún signo o síntoma; esta población es considerada altamente expuesta a infección con un virus que no se mostró altamente patógeno. F9 está ubicada en el noreste (Figura 1), dijo no tener antecedentes de brote de PRRSV en ese lugar que rentaban desde 3 años antes y desconocían si lo hubo antes en ese lugar, pero en su sucursal donde producen el alimento sí tenían antecedentes de brote de PRRSV; frecuentemente insemina con semen de Tlaxcala, Hgo., tiene aceptables instalaciones y manejo de la granja y poco manejo de sus registros; de manera general en la granja sólo habían percibido una ligera y constante tos en maternidad; la cerda virémica fue RTqPCR positiva a PRRSV norteamericano (PRRSVNA), (Cuadro 1) no tuvo ELISA pero todos los resultados de ELISA de la granja fueron negativos, por lo que muy probablemente fue una infección aguda que la cerda mostró activa con una repetición de celo seguido de un aborto y posteriormente dos repeticiones de celo, por lo que fue enviada a rastro; esta granja fue considerada altamente expuesta a infección y brote.
Sólo dos granjas se mostraron libres de PRRSV con serologías y viremias negativas, F11 y F4, ambas ubicadas en un área semi rural, sin antecedentes de PRRSV ni calendarios de vacunación contra éste (Cuadro 1, Figura 1). F11 nunca introduce cerdos de origen externo y es la granja con menor uso de semen de origen externo (cuatro veces al mes) y tan sólo de un origen.
Se encontró una relación entre la seropositividad a PRRSV y ubicación urbana-semi urbana, siendo 12.65 veces la probabilidad (OR 13.65) de ser seropositivos en granjas de ubicación urbana-semi urbana que en granjas de ubicación rural-semi rural, IC 6.74 a 27.65 y X2 0.001. También se encontró una relación de seropositivos a PRRSV con granjas con antecedentes de PRRSV, que desde entonces han controlado con calendario de vacunación a PRRSV (PRRSVav), siendo de 4.35 veces (OR 5.34) la probabilidad de ser ELISA seropositivos siendo de granjas PRRSVav que siendo de granjas sin antecedentes de PRRSV (o antecedente erradicado o dudoso), las cuales no tienen calendario de vacunación a PRRSV (NoPRRSVav), IC 2.88 a 9.90 y X2 Pearson P<0.001. Para asociar otras variables de exposición, a falta de información en varias granjas se consideró como más conveniente tomar datos únicamente de granjas sin antecedentes (o erradicado o dudoso), las cuales no presentaban calendario de vacunación (a excepción de F4 por tener información incompleta), y se asociaron con la positividad a PRRSV por una o ambas pruebas de ELISA y RTqPCR como variable de respuesta, las posibles variables de exposición y que mostraron asociación significativa fueron: Introducción de uno (semental) o más animales externos o inseminación ≥50 % con semen de origen externo, OR 4.05, IC 1.14 a 14.31, X2 Pearson P<0.020 (Fisher ex. un. 0.01), al estratificarlo, únicamente de animales externos OR 6.12, IC 1.62-23.11, introducción de animales externos e inseminación ≥50 % con semen de origen externo OR 5.62, IC 1.33-23.77, únicamente inseminación ≥50 % con semen de origen externo OR 0.48, IC 0.04-4.94; agua ≥20 % de bordos (acceso animales trashumantes) y pipas o únicamente ≥20 % pipas, OR 0.38 IC 0.150.92, no mostró riesgo, X2 0.029 (Yates corr 0.049) mostró ligera asociación, IC al estratificarlo sólo mostró riesgo a bordos (acceso animales trashumantes) y pipas OR 1.20.
Los resultados individuales de los cerdos (n= 266) relacionados con los resultados de las granjas muestran a un 26.32 % de la población estudiada serológica o viralmente afectada, 56.39 % en algún nivel de riesgo y al 17.29 % libre (Cuadro 2, Figura 2) lo que se explica como: 0.75 % con viremia declarada y 19.17 % en alto riesgo por no estar en un calendario de vacunación contra PRRSV, de las granjas F3 y F9; en contacto con PRRSV sin la protección de un calendario de vacunación contra PRRSV un 9.4 % y en riesgo medio 15.79 %, de F10, F5 y en menor grado F7; en contacto con PRRSV estando con calendario de vacunación contra PRRSV 16.17 % y en bajo riesgo 21.43 % F1, F2, F6 y F8; una población libre de PRRSV del 17.29 % de F4 y F11 (Figura 2).
DISCUSIÓN
A nuestro conocimiento, este es el primer reporte a cerca de la circulación viral de PRRSV (30.67 % seroprevalencia verdadera) y detección viral (2/80) en el estado de México, por ELISA y RTqPCR, con el primer reporte de una infección por PRRSVEU en México. La mayoría de las granjas (81.81 %; 9/11), se mostraron positivas a PRRSV pero no hubo signos evidentes de infección en las granjas, a pesar de bacterias aisladas posibles causantes de CRP. Los posibles tipos de PRRSV presentes no se manifestaron como un brote agudo, sino como una enfermedad controlada subclínica y endémica, que puede notarse principalmente en repeticiones de celo que podrían deberse a reabsorción embriónica (notado por F2 utilizando ultrasonido), en abortos esporádicos y en algunos problemas respiratorios ligeros.
La presencia de PRRSV, el aislamiento bacteriológico de posible etiología de CRP y los signos de estas enfermedades estuvieron influenciados por las instalaciones de la granja, control sanitario, supervisión médica, programas de medicina preventiva y el tipo o tiempo de uso de calendarios de vacunación, como factores clave(27). En las granjas PRRSVav, los calendarios de vacunación contra PRRSV (además de otros factores clave), disminuyó el número de cerdos virémicos, signos, síntomas, tiempo de viremia y tiempo de diseminación viral, muy probablemente detectamos anticuerpos contra virus de campo antecedente, virus vacunal activo y algunos virus de campo nuevos(7,10,12), sabiendo que hasta ahora las vacunas del mercado no garantizan una protección satisfactoria, su eficiencia decae frente a tipos heterólogos y de la posibilidad de una prevalencia de virus vacunal del 10 % y sobre el 33 %(10), confirmación sólo posible mediante la secuenciación del ARN viral de PRRSV y estudios realizando alineaciones y dendogramas (filograma, dendograma radial) en las cepas de PRRSV mexicanas para diferenciar el virus de campo residente, virus vacunal o la introducción de un nuevo virus, no siendo aún probada la relación entre la secuenciación del virus y las características del virus principalmente en cuanto a patogenicidad(28).
La presencia de anticuerpos anti-PRRSV fue más probable en granjas con PRRSVav y en un área urbana-semi urbana que al estratificar tiene mayor asociación a semi urbana como se observa en F6 (20/20) y urbana en F1 (9/20). En F10 (14/20), granja en área urbana, sin calendario de vacunación contra CRP, NoPRRSVav, es interesante notar que a pesar de una alta proporción de seropositivos con anticuerpos anti-PRRSV no se detectó ARN de PRRSV, ni tampoco se observaron evidencias de problemas reproductivos o respiratorios persistentes asociados a PRRSV o CRP, lo cual probablemente podría explicarse por una alta calidad de las instalaciones de la granja, el uso de probióticos (ambos de los cuales no se tienen en las otras granjas), el manejo de la granja(27), y quizá a una baja patogenicidad del virus, concordando con otras en las asociaciones más altas de introducción de un semental de origen externo y ubicación urbana.
En la aplicación de cuestionarios, la mayoría de las granjas cooperó positivamente pero no todas completamente, y los registros de las granjas son llevados manualmente a diario por etapas productivas, pero en la mayoría toma demasiado tiempo para ser descargados individualmente, anualmente y electrónicamente; esto limitó los datos para más análisis estadísticos y el número de granjas, no siendo suficiente para una regresión logística múltiple.
La infección por PRRSV puede confundirse o estar en asociación con otras enfermedades como son influenza suina, enfermedad de Aujesky, fiebre porcina clásica, parvovirus porcino, encefalomiocarditis, clamidiosis o micoplasmosis(2). Un ejemplo de este tipo de infección pudo pasar en F5 (8/20), en donde los cerdos pudieron tener contacto con un virus de PRRSV que aparentemente controlaron mediante diferentes factores individuales y generales, y la infección confundirse o estar en asociación con otras enfermedades.
Por la influencia positiva de factores generales, la cerda positive a PRRSVNA (F9) fue eliminada por el manejo general debido a síntomas de repetición de celo, aborto y dos repeticiones de celo (quizá reabsorciones); F11 (granja PRRSV negativa) nunca introduce cerdos de origen externo (factor asociado a PRRSV) y es la que menos usa semen de origen externo. Presentaciones de fallas en factores generales parecen influenciar la posible presencia de PRRSV, siendo una posibilidad de deficiencias en una buena calidad de cuidado en recursos externos para agua en F5 (8/20 PRRSV seropositivos, NoPRRSVav, agua de bordo de acceso de animales de granja trashumantes y de pozo, significativo), de alimento en G9 (RTqPCR positiva a PRRSVNA), de semen de origen externo en algunas granjas pero principalmente por su uso en alta frecuencia en F5 y F9 (RTqPCR positiva a PRRSVNA); la posibilidad al introducir animales de origen externo con problemas reproductivos (altamente significativo) como se ve en F1 (9/20 PRRSV seropositivos, PRRSVav), F3 (RTqPCR EUPRRSVEU positiva), F5 (8/20 PRRSV seropositivos, NoPRRSVav) y F10 (14/20 seropositivos, NoPRRSVav); y por un calendario de vacunación mal aplicado para Actinobacillus y Pasteurella en F1, o necesario y no incluido en F2 (Cuadro 1).
No fue determinado el origen de las cepas de PRRSV detectadas por RTqPCR, pero algunas posibilidades podrían surgir de los registros de las granjas, por ejemplo la F9 (con la cerda PRRSVNA) utiliza semen de Tlaxcala, Hgo.; F1 (9/20 seropositivos, PRRSVav) introdujo 20 cerdas de reemplazo de Tlaxcala, que tuvieron problemas de fertilidad y repeticiones de celo y utiliza semen del estado de Sonora; F3 (antecedentes de erradicación de PRRSV) trajo de Sonora la cerda diagnosticada con PRRSVEU.
La granja F7 con pocos seropositivos (15%; 3/ 20; NoPRRSVav) podría no tener una importante exposición a PRRSV, ya que de acuerdo a Brown et al(8) el Idexx HerdChek PRRS 2XR enzyme-linked immunoasorbent assay (ELISA) está basado en la proteina N codificada por ORF7, y con preocupación se han observado resultados positivos en granjas seronegativas.
Diferencias en el tipo de detección de cada prueba (RTqPCR para ARN viral en etapas tempranas de infección y más en aguda, ELISA para anticuerpos en etapas mas tardías). En cuanto a posibilidades económicas de realización de cada prueba, RTqPCR es caro limitando el tamaño de muestra, edad y etapa productiva de los cerdos (más probabilidad de carga viral en gestantes en el último tercio y más en lechones de 9 a 16 semanas o menos, reciente contacto con placentas o intracamada; más posibilidad de formación de anticuerpos de 12 a 22 semanas)(21), portadores con baja carga viral de distribución limitada de tejidos, disminución de animales virémicos por los calendarios de vacunación, políticas de eliminación de cerdas con repeticiones de celo y quizá una baja patogenicidad del virus circulante, podrían explicar las diferencias en resultados entre ambos métodos de diagnóstico utilizados (+/-), mencionándose poca concordancia entre las pruebas y suficiente sensibilidad para RTqPCR en muestras de sangre en trabajos similares(16,21,29,30).
Para CRP, Done y White(27) consideran que a través de las décadas los signos y síntomas respiratorios han permanecido similares, pero las etiologías pueden diferir según cambios en el sistema productivo, el desarrollo de vacunas eficientes y la ocurrencia pasada y presente de nuevas enfermedades del sistema respiratorio. Esto podría explicar la baja frecuencia de problemas respiratorios en estas granjas gracias a sus instalaciones y medidas sanitarias de control y el aislamiento dominante de Staphylococcus aureus (patógeno posible)(31) y Streptococcus suis en todas las granjas y sobre Pasteurella spp, y Actinobacillus spp, que si en un futuro se asilaran de problemas respiratorios o lesiones neumónicas, deberían realizarse estudios de patogenicidad como posibles etiologías bacterianas de CRP(32) y consecuentemente implicarse o no en calendarios de vacunación.
De acuerdo a lo observado en este estudio, mejorar el diagnóstico y control de PRRSV sería: comparando con el estudio de Duinhof et al(21), un mejor diagnóstico de PRRSV por granja, requeriría 12 muestras individuales de sangre con EDTA de cerdos de 9 a 16 semanas de edad o más jóvenes para RTqPCR, y 12 muestras de sangre sin EDTA, para suero, de cerdos de 12 a 22 semanas de edad para ELISA. En este estudio, por economía y ampliación de área (más granjas) fueron 8 para RTqPCR y las mismas 12 para ELISA, de las edades disponibles de las etapas productivas; para RTqPCR incluir muestras de sangre e incluso de semen(18), placenta o hisopado de tonsilas de animales sospechosos como sería un verraco y su semen(29) pajillas de semen, una cerda con reabsorción fetal o repetidora en el último cuarto de gestación, o un conglomerado de muestras de sangre, hisopado de tonsila o placenta de cuatro lechones de una misma camada; los cerdos virémicos detectados aislarlos o eliminarlos, procurando antes encaminar muestras de estos para obtener el PRRSV y su secuenciación; granjas NoPRRSVav, sin signos de brote, ELISA negativas-1/8-12 RTqPCR positivas o ELISA positivas-RTqPCR negativas, probar evitar el riesgo de introducir virus vacunal e iniciar un programa de control de vacunación con virus inactivado; en toda granja comprar animales y semen certificados libres de PRRSV o hacer pruebas rutinarias de RTqPCR y ELISA antes de introducirlos; ubicar o reubicar las granjas en áreas rurales o semi rurales; revisar e implementar condiciones de instalaciones, manejo general en la granja y medidas estratégicas de prevención(30) como para densidad, temperatura y ventilación adecuados, reducir o evitar mezcla de camadas, sanidad general, de agua y alimento, calendarios de vacunación necesarios; usar ultrasonido para detectar reabsorciones fetales; mayor atención en los registros para abortos, repeticiones de celo, reabsorciones fetales, mortalidad de lechones, mortalidad de recién nacidos y retrasos en el crecimiento y en su pronta relación con los registros de la cerda, recursos de semen y la progenie derivada, para poder tomar medidas de control temprano.
AGRADECIMIENTOS
Esta investigación fue realizada y publicada gracias a la autorización del "Comité de Fomento y Protección Pecuaria del Estado de México" (CFPPEM) bajo un servicio de CFPPEM y la Universidad Autónoma del Estado de México (UAEMéx) financiado por Grupo Produce en el estado de México. Agradecemos las observaciones, confianza y cooperación otorgada por profesionistas y personas en general, de las granjas involucradas.
LITERATURA CITADA
1. Arias M, Barceló J, Muñoz A, Sánchez-Vizcaíno JM. Síndrome Respiratorio Reproductivo Porcino. Sánchez-Vizcaíno JM editor. Curso digital de enfermedades infecciosas porcinas [en línea]. http://www.sanidadanimal.info/cursos/curso/9/9-prrs.htm. Consultado Nov 15, 2011. [ Links ]
2. Morrilla A, González-Vega D, Diosdado F, Estrada E. Seroepidemiology of PRRS in México. 4th International Symposium on Emerging and Re-emerging Pig Diseases. Rome. 2003. [ Links ]
3. Cruz MC, Mogollón JD, Rincón MA, Peña NB, Ruiz S, Lora AM. Prevalencia serológica del síndrome reproductivo y respiratorio porcino (PRRS) en cerdos de explotaciones extensivas de Colombia. Rev Med Vet Zoot 2006;53:33-41. [ Links ]
4. Benfield D A, Collins JE, Dee SA, Halbur PG, Joo HS, Lager KM, et al. Porcine reproductive and respiratory syndrome. In: Straw B, DÁllairre S, Mengeling WL, Taylor DJ, editors. Diseases of swine. 8th ed. Ames (Iowa), Iowa State University Press; 1999:201-232. [ Links ]
5. Callen A. La problemática del control del PRRS en granjas de reproducción. 2006. [en línea]. http://www.porcicultura.com/porcicultura/home/articulos_int.asp?cve_art=388. Consultado Nov 15, 2011. [ Links ]
6. Kleiboeker SB, Schommer SK, Lee S-M, Watkins S, Chittick W, Polson D. Simultaneous detection of North American and European porcine reproductive and respiratory syndrome virus using real-time quantitative reverse transcriptase-PCR. Brief communications. J Vet Diag Invest 2005;17:165-170. [ Links ]
7. Flores-Mendoza L, Hernández J. Vacunas contra el virus del síndrome reproductivo y respiratorio porcino (PRRSV): escribiendo una historia. Vet Méx 2010;41:139-159. [ Links ]
8. Brown E, Lawson S, Welbon C, Gnanandarajah J, Li J, Murtaugh MP, et al. Antibody response to porcine reproductive and respiratory syndrome virus (PRRSV) Nonstructural proteins and implications for diagnostic detection and differentiation of PRRSV Types I and II. Clin Vaccine Immunol 2009;16:628-635. [ Links ]
9. Zhoua Z, Nia J, Caoa Z, Hana X, Xiaa Y, Zia Z, et al. The epidemic status and genetic diversity of 14 highly pathogenic porcine reproductive and respiratory syndrome virus (HP-PRRSV) isolates from China in 2009. Vet Microbiol 2011; 150:257-269. [ Links ]
10. Shi M, Lam TT-Y, Hon Ch-Ch, Murtaugh MP, Davies PR, Hui RK-H, et al. Phylogeny-based evolutionary, demographical, and geographical dissection of north american type 2 porcine reproductive and respiratory syndrome viruses. J Virol 2010;84:8700-8711. [ Links ]
11. Goldberg TL, Lowe JF, Milburne SM, Firkins LD. Quasispecies variation of porcine reproductive and respiratory syndrome virus during natural infection. J Virol 2003;317:197-213. [ Links ]
12. Thanawongnuwecha R, Suradhatb S. Progress in porcine respiratory and reproductive syndrome virus biology and control. Virus Res 2010;154(1-2):133-140. [ Links ]
13. Flores-Mendoza L, Silva-Campa E, Reséndiz M, Osorio FA, Hernández J. Porcine reproductive and respiratory syndrome virus infects mature porcine dendritic cells and up-regulates interleukin-10 production. Clin Vaccine Immunol 2008;15:720-725. [ Links ]
14. Sierra N, Ramírez R, Mota D. Aislamiento del virus de PRRS en México: Estudio clínico, serológico y virológico. Arch Med 2000;32(1):1-9. [ Links ]
15. Xiao S, Jia J, Mo D, Wang Q, Qin L, He Z, et al. Understanding PRRSV infection in porcine lung based on genome-wide transcriptome response identified by deep sequencing. PLoS One 2010;5(6): e11377. [ Links ]
16. Rovelo-Celorio A, Alzina-López A, Rodríguez-Buenfil JC, Segura-Correa JC, Villegas-Pérez S. Prevalencia y factores de riesgo asociados con el virus del síndrome reproductivo y respiratorio porcino en sementales de granjas porcinas en el sureste de México. Revista Científica 2010;2(1):17-23. [ Links ]
17. Hansen MS, Pors SE, Jensen HE, Bille-Hansen V, Bisgaard M, Flachs EM, et al. An investigation of the pathology and pathogens associated with porcine respiratory disease complex in Denmark. J Comp Path 2010;143:120-131. [ Links ]
18. Jordán-Cravioto A, Segura-Correa JC, Alzina-López A, Rodríguez-Buenfill JC, Villegas-Pérez S. Prevalence and risk factors associated with the PRRS virus in semen of boars in pig farms of Yucatan. Trop Subtrop Agroecosys 2010;12(1):1-6. [ Links ]
19. Segura JC, Honhold N. Muestreo en múltiples etapas. En: Métodos de muestreo para la producción y la salud animal. Ediciones de la Universidad Autónoma de Yucatán. México. 2000. [ Links ]
20. Martin SW, Meek AH, Willeberg P. Veterinary epidemiology, principles, and methods. Ames, Iowa, USA: Iowa State University Press; 1987. [ Links ]
21. Duinhof T F, van Schaik G, van Esch EJB, Wellenberg GJ. Detection of PRRSV circulation in herds without clinical signs of PRRS: Comparison of five age groups to assess the preferred age group and sample size. Vet Microbiol 2011;150:180-184. [ Links ]
22. Wellenberg GJ. Review: diagnostic methods for the detection of porcine reproductive and respiratory syndrome virus (PRRSV) infections. Tijdschr. Diergeneeskd 2006;131(16):566-572. [ Links ]
23. van Maanen C, von Bannisseht-Wysmuller TE, van Esch EJB, Wellenberg GJ. Comparision and validation of selected conventional and real-time PCR methods for the detection and differentiation of European and American-type PRRSV in field samples. Proc ESVV Congress Lisbon, 2006. [ Links ]
24. Gerstman BB. 17: Case-control studies (odds ratios). C:/ date/StatPrimer/case-control.doc [on line]. http://www.sjsu.edu/faculty/gerstman/StatPrimer/case-control.pdf. De StatPrimer (Version 6.4): http://www.sjsu.edu/faculty/gerstman/StatPrimer/. Accessed Nov 15, 2011. [ Links ]
25. EPIDAT programa para análisis epidemiológico de datos tabulados (programa para computadora) versión 3.1. Dirección Xeneral de Saúde Pública, Cosellería de Sanidade-Xunta de Galicia, Área de Análisis de Salud y Sistemas de Información Sanitaria Organización Panamericana de la Salud (OPS/OMS), 2006. [ Links ]
26. Quinn PJ, Markey BK, Leonard FC, Fitz Patrick ES, Fanning S, Hartigan PJ. Veterinary microbiology and microbial disease. 2nd ed. Ames: Wiley-Blackwell; 2011. [ Links ]
27. Done S, White M. Porcine respiratory disease and complexes: the story to date. In Practice 2003;25:410-417. [ Links ]
28. Batista L, Murtaugh M, Pijoan C. Variación genética de PRRSV. Anaporc 2003;23(236):52-60. [ Links ]
29. Rovira A, Clement T, Christopher-Hennings J, Thompson B, Engle M, Reicks D, et al. Evaluation of the sensitivity of reverse-transcription polymerase chain reaction to detect porcine reproductive and respiratory syndrome virus on individual and pooled samples from boars. J Vet Diagn Invest 2007;19:502-509. [ Links ]
30. Wasilk A, Callahan JD, Christopher-Hennings J, Gay TA, Fang Y, Dammen M, et al. Detection of U.S. Lelystad, and European-Like porcine reproductive and respiratory syndrome viruses and relative quantization in boar semen and serum samples by real-time PCR. J Clin Microbiol 2004;42(10):4453-4461. [ Links ]
31. Atanasova K, van Gucht S, Barbé F, Duchateau L, van Reeth K. Lipoteichoic acid from Staphylococcus aureus exacerbates respiratory disease in porcine respiratory coronavirus-infected pigs. Vet J 2011;88(2):210-215. [ Links ]
32. Palzer A, Ritzmann M, Majzoub M, Wolf G, Hermanns W, Heinritzi K. Frequency of occurrence of pneumonia associated agents and their correlation with clinical and pathological-anatomical findings in pigs. Berl Munch Tierarztl Wochenschr 2007;120(11-12):483-489. [ Links ]














