Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista mexicana de ciencias pecuarias
On-line version ISSN 2448-6698Print version ISSN 2007-1124
Rev. mex. de cienc. pecuarias vol.4 n.4 Mérida Oct./Dec. 2013
Artículos
Diversidad genética y estructura de poblaciones de pavos domésticos mexicanos*
Genetic diversity and population structure of Mexican domesticated turkeys
Rigoberto López-Zavalaa, Horacio Cano-Camachob, Omar Chassin-Noriac, Ken Oyamad, Gerardo Vázquez-Marrufob, María Guadalupe Zavala-Páramob
a Facultad de Medicina Veterinaria y Zootecnia, Universidad Autónoma de Tamaulipas. México.
b Centro Multidisciplinario de Estudios en Biotecnología, Facultad de Medicina Veterinaria y Zootecnia, Universidad Michoacana de San Nicolás de Hidalgo. Km. 9.5 Carretera Morelia-Zinapécuaro, Posta Veterinaria, 58000, Tarímbaro, Morelia, Mich. México. Tel. y Fax: (443) 295 80 29. gzavpar@hotmail.com gzavalap@umich.mx. Correspondencia al último autor.
c Facultad de Biología, Universidad Michoacana de San Nicolás de Hidalgo. México.
d Centro de Investigaciones en Ecosistemas. Universidad Nacional Autónoma de México. México.
Recibido el 13 de febrero de 2012.
Aceptado el 19 de julio de 2012.
Resumen
En este estudio se analizaron niveles de diversidad y estructura genética de 144 guajolotes domesticados de poblaciones de traspatio de las cinco regiones fisiográficas de Michoacán y 16 guajolotes de dos poblaciones silvestres del centro de México utilizados como grupo externo de referencia, mediante siete loci de microsatélites. En guajolotes domesticados el promedio de heterocigosidad fue moderado (0.535 ± 0.285) comparado con silvestres (0.701 ± 0.068). Los guajolotes domesticados mostraron diferenciación entre poblaciones (FST= 0.122) a través de los loci. Los porcentajes de diferenciación estimados por Análisis de Varianza Molecular fueron bajos entre poblaciones (14 % domesticados y 6 % silvestres) y altos dentro de las poblaciones (86 % domesticados y 94 % silvestres). Los análisis de similitud mostraron la formación de un grupo con guajolotes silvestres y dos grupos basales con domesticados; un grupo basal incluyó tres subgrupos con poblaciones de las regiones Bajío, Faja Volcánica Transversal y Balsas, y un segundo grupo basal incluyó dos subgrupos con poblaciones de las regiones Sierra y Costa. El análisis de estructura genética mostró tres grupos para guajolotes domesticados y uno para silvestres. Una prueba de Mantel mostró aislamiento por patrón de distancia (r=0.84, P<0.05) entre poblaciones domesticadas. Los grupos genéticos de guajolotes domesticados fueron congruentes con los datos morfométricos previamente obtenidos de la misma población; un grupo correspondió a poblaciones de Bajío y Balsas (morfometría intermedia), el segundo grupo incluyó a la población de Faja Volcánica Transmexicana (grandes y pesados), y el tercer grupo correspondió a poblaciones de Sierra y Costa (ligeros y pequeños).
Palabras clave: Diversidad genética, Distancia genética, Meleagris gallopavo, Microsatélite.
Abstract
In this study, the diversity level and genetic structure of 144 domesticated backyard turkeys from domesticated populations in five physiographic regions of Michoacán and 16 turkeys from two wild populations in Central México, which were used as a reference outgroup, were analyzed through seven microsatellite loci. The average of the observed heterozygosity within the domesticated turkeys was moderate (0.535 ± 0.285) compared with wild turkeys (0.701 ± 0.068). Across the loci, the domesticated turkeys showed differentiation between the populations (FST= 0.122). The differentiation percentages estimated by Analysis of Molecular Variance were low between the populations (14 % domesticated and 6 % wild) and high within populations (86 % domesticated and 94 % wild). Similarity analyses showed a single cluster of wild turkeys and two basal clusters of domesticated turkeys; one basal cluster included three subgroups with populations from the Bajío, Faja Volcánica Transmexicana and Balsas regions, and a second cluster contained two subgroups with populations from the Sierra and Costa regions. Structure software analyses showed three groups of domesticated turkeys and one group of wild turkeys. A Mantel test showed an isolation by distance pattern (r=0.84, P<0.05) between the domesticated populations. Genetic clusters for the domesticated turkeys were consistent with previously obtained morphometric data from the same population; one cluster grouped populations from the Bajío and Balsas regions (intermediate morphometry), the population from Faja Volcánica Transmexicana formed a second cluster (big and heavy) and the third cluster corresponded to populations from the Sierra and Costa regions (small and light).
Key words: Genetic diversity, Genetic distance, Meleagris gallopavo, Microsatellite.
INTRODUCCIÓN
El pavo domesticado (Meleagris gallopavo gallopavo) se ha criado en comunidades suburbanas y rurales de México desde su domesticación entre los años 200 y 700 AC, por culturas prehispánicas(1). Estas poblaciones de pavos han sido sujetas a una variedad de presiones de selección; las aves se encuentran en pueblos y zonas suburbanas de México bajo un sistema de producción que se caracteriza por tener pobres prácticas sanitarias y dietas desbalanceadas, factores que se adaptan a diferentes condiciones ecológicas(2,3). Hasta ahora, las variedades o poblaciones de pavos en México no se han caracterizado debido a que no hay registro de sus orígenes. Recientemente, se realizaron estudios que incluyen la caracterización de los sistemas de producción del pavo doméstico en México(2,4,5,6), la importancia del pavo dentro de las actividades económicas de la población suburbana y rural del país(4,5,6), y la caracterización morfométrica(5,6) y diversidad genética de algunas poblaciones de pavos(7).
Estudios previos de las poblaciones domesticadas de pavos del estado de Michoacán han mostrado algunas características morfométricas, tales como la presencia de plumajes cafés raros (Buffy) o grises (Slate), que se encuentran registrados en los Estados Unidos de América como poco comunes y en peligro de extinción, y variaciones en el peso corporal y tamaño, lo que sugiere la existencia de diversidad dentro de las poblaciones domesticadas debido a la baja selección en la crianza(3,6,8). El estado de Michoacán tiene un amplio espectro de climas y topografía, en donde se proponen cinco regiones fisiográficas basadas en su clima y vegetación específicos: Bajío, Faja Volcánica Transmexicana, Balsas, Sierra y Costa, y cada una de estas regiones tiene vegetación característica(3,9). Esta diversidad regional parece haber tenido impacto sobre las características morfométricas de las poblaciones de pavo domesticado, las cuales tienen sistemas de producción mayoritariamente tradicionales o carentes de tecnología (94.4 %), que utilizan pocas prácticas de manejo: dietas balanceadas en pollos de pavo de hasta dos meses de edad; poco o ningún programa de vacunación; y no utilizan variedades mejoradas. Estos factores indican que el tipo de genética del pavo es nativa y constituye un verdadero fondo genético. En aquellas regiones de clima seco, tropical o subtropical (Balsas, Costa y parte de la Sierra), 70 % de las unidades de producción no están provistas de albergues o corrales y las aves pasan la noche en árboles secos o vivos; el otro 30 % tiene albergues precarios construidos con tablones de madera de segunda mano o cartón, con techos de cartón o, en ocasiones, de teja y sin comederos permanentes(3,6). Un estudio genético previo, usando ADN polimórfico amplificado al azar (RAPD), reveló una mayor diversidad genética en algunas poblaciones de pavo domesticado de Michoacán que en poblaciones del estado de Puebla, sugiriendo que los pavos de Michoacán son descendientes de una población históricamente grande(7). Esta observación inicial debe ser reevaluada por medio del uso de mejores marcadores genéticos para estudiar la estructura de la población de pavos agregando individuos de otras regiones de Michoacán. Las poblaciones de pavos domesticados mexicanos representan un reservorio de variabilidad genética y fenotípica que debe ser explorado; la información obtenida en este sentido será útil para fines de conservación y manejo adecuado de sistemas de producción tradicional. El objetivo del presente estudio fue evaluar la diversidad de cinco poblaciones de pavo domesticado mexicano de cinco regiones fisiográficas del centro de México (Michoacán) usando marcadores microsatélites.
MATERIALES Y MÉTODOS
Colecta de muestras
Se usaron cinco poblaciones de pavo domesticado (M. g. gallopavo) originario del centro de México y dos poblaciones silvestres (M. g. mexicana) incluidas como grupo externo.
Se colectó un total de 144 muestras de sangre de hembras y machos de las cinco poblaciones domesticadas, de 10 a 18 meses de edad, criadas en un sistema de producción no tecnificado o tradicional, y que provenían de diferentes localidades en cinco regiones del centro de México: Bajío (n=31); Faja Volcánica Transmexicana (n=32); Balsas (n=28); Sierra (n=31); y Costa (n=22) (estado de Michoacán) (Figura 1). Se utilizó un muestreo estratificado simple y cualitativo, con las capas constituidas por cada región. Los sitios de colección fueron seleccionados al azar en cada región y 15 a 20 sitios fueron asignados a cada región; las unidades de muestreo fueron casas y localidades en donde los pavos se encontraban en los patios o en lugares cercanos a las casas.
Además, se colectaron siete muestras de sangre de pavos silvestres de poblaciones relicto de Mezquitic y Huejuquilla, Jalisco y Valparaíso, Zacatecas (en lo sucesivo Centro Occidente); y nueve muestras de sangre de pavos silvestres de Canatlán, Durango (Figura 1). Para cada muestra se tomaron 0.2 ml de sangre de la vena braquial y se colocaron en un tubo vial con 0.5 ml de solución amortiguadora de lisis para preservación(10).
Extracción de ADN, marcadores de microsatélites y PCR
Se realizó extracción de ADN mediante el método libre de fenol descrito por Fitz-Simmons(11). Los ensayos de PCR se llevaron a cabo con oligonucleótidos para siete loci de secuencias simples repetidas (SSR), descritos previamente(12,13,14) (Cuadro 1). La amplificación se realizó en un volumen total de 25 μl; cada mezcla de reacción contenía 20-30 ng de ADN, 10 pmol de cada iniciador, 1X de amortiguador para PCR sin MgCl2 (Invitrogen), 200 μM de cada dNTP, 0.75-1.25 mM MgCl2, 0.3 U Platinum Taq ADN polimerasa (Invitrogen) y H2O destilada, desionizada, estéril. Las condiciones de la PCR fueron como sigue: desnaturalización a 95 °C durante 5 min seguido por 35 ciclos a 95 °C durante 2 min, 53-58 °C por 2 min y 72 °C durante 2 min, con un periodo de extensión final a 72 °C durante 8-10 min (2700 Applied Biosystems). El análisis de detección se realizó en un AB 3100 Avant Genetic Analyzer (Applied Biosystems). El tamaño del producto se determinó en relación al tamaño del marcador molecular interno Genescan-350ROX (Applied Biosystems). El tamaño de los alelos se determinó usando el software Peak Scanner versión 1.0 (Applied Biosystems).
Análisis estadístico
La riqueza alélica (por el método de rarefacción), diversidad genética, diferenciación por medio de Fis, Fst y Fit y la significancia de los valores de Fst (permutando genotipos 2,500 veces a través de cada par de poblaciones) con corrección secuencial de Bonferroni (para α=0.05) se calcularon usando el software FSTAT(15). El análisis de población se realizó con el software GeneAIEx versión 6.0(16) para estimar la variación genética (He de acuerdo a Nei 1987 EQ 8.1(17)), el equilibrio de Hardy-Weinberg (HWE por sus siglas en inglés) y el análisis de varianza molecular (AMOVA por sus siglas en inglés). El análisis AMOVA se realizó en forma separada en las poblaciones silvestres y domesticadas. La distancia genética se estimó usando el método de Reynolds(18). El análisis de aislamiento por distancia entre las poblaciones domesticadas se realizó mediante la prueba de Mantel, usando el software Arlequin versión 3.5, utilizando la matriz de la distancia genética y la matriz de la distancia geográfica entre individuos(19).
Análisis de comparación
La similitud entre las poblaciones fue computarizada mediante el uso del software PHYLIP versión 3.3(20) y Treeview versión 1(21) para la edición del árbol filogenético, usando las muestras del pavo silvestre como grupo externo. Con las distancias genéticas de Reynolds, se construyó un dendograma mediante el método de Neighbor-Joining que resume las relaciones genéticas entre las poblaciones domesticadas y silvestres(18). Además, los grupos fueron identificados usando el método Bayesiano implementado en el software Structure versión 2.2(22) con 10,000 periodos eliminados y 20,000 iteraciones.
RESULTADOS
Diversidad genética
Un total de 65 alelos diferentes se detectaron de las cinco poblaciones domesticadas (144 individuos) y las dos poblaciones silvestres (16 individuos) para los siete loci estudiados, los cuales fueron polimórficos. Se calculó la riqueza alélica y la diversidad genética para cada locus (Cuadro 2). Tres loci mostraron una alta riqueza alélica y, aunque dos presentaron baja riqueza alélica, el promedio de los siete loci fue alto (6.66). Sin embargo, a pesar de que la diversidad genética de tres loci fue alta, el promedio de los siete loci fue moderado (0.560).
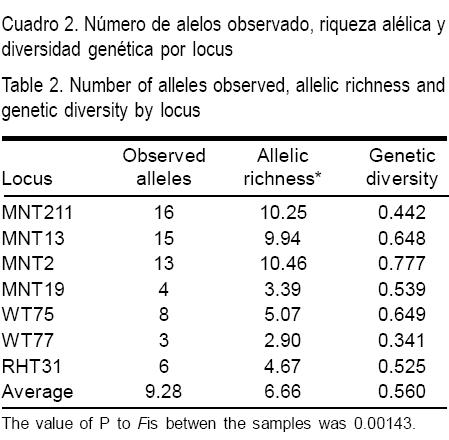
Seis de los siete loci registraron más alelos que el número previamente reportado(12,13,14) (Cuadro 3). El número promedio de alelos por locus fue de 9.28, oscilando entre 3 a 16 alelos (WT77-2 y MNT211, respectivamente). El promedio de la heterocigosidad observada para los loci, a través de las poblaciones domesticadas fue de 0.533 (0.433 a 0.613), y la heterocigosidad esperada fue de 0.606 (0.527 a 0.723). Para los loci a través de las poblaciones silvestres, el promedio de la heterocigocidad observada fue de 0.701 (0.381 a 1.000), y el de la heterocigosidad esperada fue de 0.663 (0.453 a 0.795).
El promedio del coeficiente de endogamia (Fis) a través de los loci dentro de las poblaciones domesticadas fue de 0.109 y las poblaciones silvestres -0.600 (Cuadro 3). La diferenciación genética (Fst) a través de los loci en poblaciones domesticadas fue de 0.122 y la población silvestre tuvo el valor más bajo, 0.060. El promedio del coeficiente de endogamia (Fit) a través de los loci para las poblaciones domesticadas (0.213) concordó con un déficit en individuos heterozigotos dentro y entre las poblaciones (0.533). En contraste, los valores negativos de Fis (-0.600) y Fit (-0.001) a través de los loci en las poblaciones silvestres fueron indicadores de un exceso de individuos heterocigotos dentro y entre poblaciones.
Se observó un promedio de 4.56 y 4.42 alelos por locus en poblaciones domesticadas y silvestres, respectivamente, en un rango de 3.85 en poblaciones de la región de la Costa a 5.28 en la población de la región del Balsas (Cuadro 4). En general, el valor de Ho fue menor que el de He en poblaciones domesticadas, con excepción de la población de la región del Balsas, donde el valor fue más alto (0.668 ± 0.130) (Cuadro 3). El promedio del coeficiente de endogamia (Fis) fue de 0.023 para las cinco poblaciones domesticadas y -0.266 para las dos poblaciones silvestres (Cuadro 4).
Equilibrio de Hardy-Weinberg
A través de los loci, la población silvestre del Centro Occidente de México no mostró loci que se desviaran del equilibrio de Hardy-Weinberg (HWE) (P<0.05) (Cuadro 3). En cambio, MNT19 se desvió de HWE en las cinco poblaciones domesticadas, mientras que WT75 no se desvió en ninguna población. En promedio, a través de las poblaciones domesticadas, 2.71 de los siete loci se desviaron de HWE. Las poblaciones con la mayoría de los loci que mostraron desviación fueron de las regiones de la Faja Volcánica Transmexicana, Bajío y Sierra, mientras que los loci con menor desviación se observaron en las poblaciones del Balsas y Costa, y un locus en la población silvestre de Durango (Cuadro 4).
Alelos privados
En promedio, entre las 160 aves domesticadas y silvestres, hubo 9.28 alelos por locus a través de los siete loci, dando un total de 65 alelos (Cuadro 3). De estos 65 alelos, se detectaron 41 alelos privados (63 %) distribuidos a través de los siete loci y las siete poblaciones. Los alelos privados dentro de las poblaciones silvestres sólo correspondieron a cuatro de los siete loci y fueron aproximadamente 18.46 % del número total de alelos (Cuadros 3, 4). La cantidad de algunos alelos privados entre las poblaciones domesticadas fue especialmente alta; por ejemplo, el número de alelos en el locus MNT211 fue de aproximadamente 10.76 y 9.23 % del número total de alelos de todos los loci en las poblaciones domesticadas y silvestres, respectivamente (Cuadro 4). El número de alelos privados dentro de las poblaciones domesticadas fue bajo para la región de la Costa (aproximadamente 3.07 %) y alto para la región de la Sierra (aproximadamente 10.76 %).
Diferenciación genética
Los valores pareados de Fst para la diferenciación entre las poblaciones domesticadas tuvieron un rango de 0.044 entre las poblaciones del Bajío y Balsas, a 0.148 entre las poblaciones de las regiones de la Faja Volcánica Transmexicana y la Costa, y a 0.06 entre las poblaciones silvestres (Cuadro 5). El valor pareado de Fst entre el total de las poblaciones domesticadas y silvestres fue de 0.167 (P<0.05) y este valor entre poblaciones individuales tuvo un rango de 0.109 a 0.163.
Para establecer las relaciones genéticas entre poblaciones, se midió la distancia genética(17). La mayor distancia genética se observó entre las poblaciones domesticadas y silvestres, con un rango de 0.306 a 0.210 (Cuadro 5). Se encontró una distancia genética significativa entre las poblaciones domesticadas de las regiones de la Faja Volcánica Transmexicana y de la Costa (0.260), mientras que las poblaciones del Bajío y Balsas fueron menos distantes (0.091) (Cuadro 5 y Figura 1). Los valores pareados de Fst entre las regiones del Bajío y Balsas (0.044) y entre la Faja Volcánica Transmexicana y la Costa (0.06) concordaron con su número efectivo de migrantes (5.493 y 1.434, respectivamente) (Cuadro 6).
El AMOVA produjo valores de distribución de diversidad genética de 14 y 86 % entre y dentro de las poblaciones domesticadas, respectivamente, y de 6 y 94 % entre y dentro de las poblaciones silvestres, respectivamente (Cuadro 7).

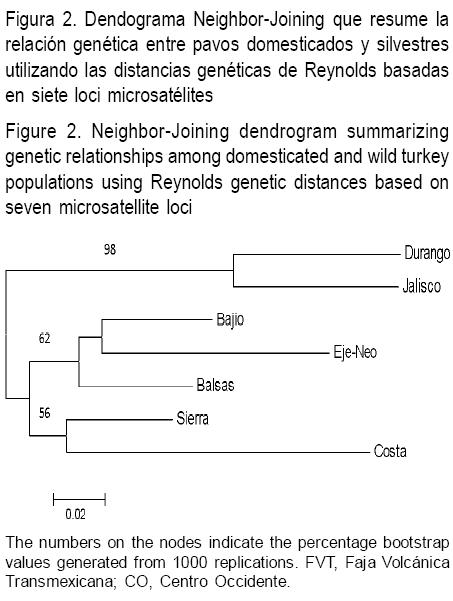
El dendograma Neighbor-Joining para las siete poblaciones analizadas mostró dos agrupamientos principales: un grupo que incluyó a las poblaciones domesticadas y un segundo grupo que incluyó a las poblaciones silvestres (Figura 2). Adicionalmente, las poblaciones de pavos domesticados fueron posteriormente agrupadas en dos subgrupos basales: uno incluyó a las poblaciones de la región del Balsas como basal de dos subgrupos con las poblaciones del Bajío y la Faja Volcánica Transmexicana, y un segundo agrupamiento que incluyó los dos subgrupos con las poblaciones de las regiones de la Sierra y Costa.
Estructura de la población
El análisis de las siete poblaciones de pavos mostró cuatro grupos genéticos basados en la estrategia descrita por Pritchard et al(22) sin una asignación a priori para una población originaria, por medio de un valor modal de DK, una cantidad basada en la tasa de cambio de segundo orden en relación a K de la función de verosimilitud (ΔK=4)(23). Tres de estos grupos correspondieron a poblaciones domesticadas y tuvieron bajos niveles de solapamiento entre ellos, mientras que el cuarto grupo incluyó a las poblaciones silvestres sin solapamiento con las poblaciones domesticadas (no mostrado). En el caso de las poblaciones domesticadas, un agrupamiento incluyó a las poblaciones de las regiones del Bajío y Balsas, un segundo agrupamiento incluyó a las poblaciones de la Faja Volcánica Transmexicana, y un tercer agrupamiento correspondió a las poblaciones de las regiones de la Sierra y Costa. De acuerdo al resultado de la prueba de Mantel, en el cual existe una correlación positiva si 0<r<1, el índice de correlación obtenido, r=0.84 (P<0.05), fue positivo, y por lo tanto las poblaciones domesticadas mostraron un aislamiento por patrón de distancia.
DISCUSIÓN
Diversidad genética
Como grupo, las poblaciones domesticadas mostraron un alto nivel de diversidad. La riqueza alélica del pavo fue alta (6.66) en comparación con las razas de patos de China (con un rango de 3.45 a 4.31)(24). Aunque la diversidad genética por locus no fue alta (0.560), la heterocigosidad pareció ser más alta en promedio en las cinco poblaciones domesticadas de pavos nativos estudiados en el presente trabajo (0.535 ± 0.285 en 144 individuos), que la heterocigosidad previamente reportada en las cinco variedades de pavos domesticados de Estados Unidos de América (0.09 en 94 individuos)(25). Los valores de heterocigosidad de este estudio también parecen altos en comparación con los valores reportados en pollos; 0.499 en 37 individuos de 37 razas(26) y 0.460 en 1,970 individuos(27). Adicionalmente, el nivel de heterocigosidad fue similar al nivel observado en poblaciones de pollos nativos en Turquía (con un rango de 0.380 ± 0.065 a 0.508 ± 0.037)(28), pollos nativos de Corea (0.630)(29), pollos japoneses nativos y comerciales (0.471 a 0.472)(30) y poblaciones de pollos de bosque y sabana de Ghana (0.568)(31).
Los valores de Fis a través de los loci de las poblaciones domesticadas y silvestres mostraron las diferencias esperadas, las cuales fueron mayores para pavos domesticados y menores para pavos silvestres. Los valores de Fst sugirieron flujo genético moderado en poblaciones domesticadas y silvestres. Los valores de Fst a través de los loci indicaron que aproximadamente el 12 y 6 % de la variación genética total en pavos domesticados y silvestres, respectivamente, fue explicada por las diferencias en las poblaciones, y 88 y 94 % correspondieron a diferencias dentro de las poblaciones, respectivamente.
En general, los valores de Fis de las poblaciones domesticadas y silvestres fueron consistentes con los valores correspondientes de heterocigosidad observados. El valor medio de Fis=0.109 en poblaciones domesticadas fue menor que el valor observado en poblaciones de pollos nativos domesticados de Turquía (Fis=0.301 ± 0.05)(28) y fue mayor que el valor en razas de pollos domesticados (0.09)(26) (-0.004 a 0.089)(27), sugiriendo que las poblaciones domesticadas estudiadas tienen mayor deficiencia de heterocigotos.
Equilibrio Hardy-Weinberg
Se esperan desviaciones de HWE si las poblaciones individuales están estructuradas en subpoblaciones que están aisladas una de la otra, o si la endogamia ha ocurrido en las poblaciones en general. El alto nivel de desviación de HWE encontrado en las poblaciones domesticadas estudiadas y el sistema de reproducción tradicional o no tecnificado(3,6), indican una estructura en subpoblaciones. Este nivel de desviación de HWE también concordó con el promedio del coeficiente de endogamia (Fis) y deficiencia de heterocigosidad debido a diferenciación en subpoblaciones (Fst). Sin embargo, las poblaciones con el menor número de loci mostrando desviación de HWE fueron de las regiones del Balsas y Costa. El sistema de producción tradicional ha contribuido a la creación de subpoblaciones; sin embargo, estas subpoblaciones muestran diferentes niveles de endogamia, indicando una diferencia en la cantidad de tiempo desde la separación a partir de una gran población original.
Alelos privados
Debido a que el flujo de genes entre poblaciones puede ser estimado por la abundancia de alelos privados, las poblaciones domesticadas de las regiones de la Sierra, Balsas y Faja Volcánica Transmexicana, que tuvieron el mayor número de alelos privados (siete, cinco y cinco, respectivamente), pudieron haber sido las primeras en separarse de las otras poblaciones(27,32). Las poblaciones domesticadas de la región de la Costa mostraron sólo dos alelos privados, sugiriendo una separación reciente. Los nuevos alelos en los loci microsatélites son el producto de procesos de mutación, y el gran número de alelos privados en loci microsatélites específicos puede indicar que están situados en una región genómica altamente mutagénica o que provienen de sitios heterocigotos, los cuales tienden a mutar más frecuentemente que los sitios homocigotos(33). Las dos poblaciones silvestres mostraron un gran número de alelos privados (cuatro y ocho), de los cuales seis correspondieron a un locus. El resto de los seis alelos privados se localizaron en tres loci, lo cual puede estar relacionado con el alto nivel de heterocigosidad que se observó y la distancia geográfica de las poblaciones silvestres.
Diferenciación genética
A pesar de la deficiencia de heterocigotos observada dentro (Fis) de las poblaciones domesticadas, el valor de Fst (0.122) y los resultados de AMOVA (14 %) indicaron una divergencia moderada de la población. Esta evidencia apoya la hipótesis del origen de estos pavos en poblaciones históricamente grandes(7).
En las poblaciones silvestres muestreadas, los valores de Fis indicaron alta heterocigosidad dentro y entre poblaciones, mientras que el valor de Fst de 0.060 y un AMOVA de 6 %, sugirieron poca diferenciación de la población y más variación dentro de las poblaciones. Los valores de Fis calculados para las poblaciones domesticadas y silvestres mostraron diferencias de deficiencia de heterocigotos entre los pavos domesticados de cada región fisiográfica y los pavos silvestres de diferentes ubicaciones geográficas, indicando una diferente separación en subpoblaciones.
La distancia genética (con un rango de 0.307 a 0.210) entre las poblaciones domesticadas y silvestres se estimó ser moderada, pero refleja el hecho de que estas poblaciones están genéticamente aisladas entre sí. El nivel de aislamiento genético entre las poblaciones domesticadas de las regiones de la Faja Volcánica Transmexicana y de la Costa (0.260) concordó con la distribución geográfica. La baja distancia genética entre las poblaciones de las regiones del Bajío y Balsas (0.091) reflejan el flujo genético entre éstas.
Como se esperaba, el análisis de similitud claramente agrupó las poblaciones domesticadas y silvestres de pavos como grupos diferentes. Aún más, las poblaciones domesticadas se segregaron en dos grupos basales y subgrupos que correlacionaron con la distribución geográfica de las regiones fisiográficas (resultado de la correlación de la prueba de Mantel, r=0.84, P<0.05). Las poblaciones de las regiones de la Faja Volcánica Transmexicana y de la Costa, con poco intercambio de individuos entre regiones (Fst=0.044 y Nm=1,434), y de las regiones del Bajío y Balsas, con importante intercambio de individuos entre regiones (Fst=0.044 y Nm= 5.493), correlacionaron con la distancia geográfica entre ellas.
La inclusión de poblaciones silvestres en el presente estudio mostró la diferencia de diversidad genética (resultados de AMOVA) y su distancia genética con poblaciones domesticadas.
Estructura de la población
El análisis de la estructura genética de las poblaciones domesticadas sugirió la existencia de tres poblaciones genéticamente distintas. Estos resultados concordaron con los resultados de similitud, donde se observó la segregación en dos grupos basales, y sugirió la separación de las poblaciones de la Faja Volcánica Transmexicana como un subgrupo genético. Los resultados también concordaron con la distribución geográfica de las poblaciones en las regiones fisiográficas (resultados de la correlación de la prueba de Mantel, r=0.84, P<0.05) y con los datos morfométricos anteriormente recopilados en las mismas regiones. El estudio morfométrico reveló pesos corporales más altos y envergadura más amplia en las poblaciones de la región de la Faja Volcánica Transmexicana (el peso promedio en machos fue de 9.09 kg y envergadura de 113.75 cm), pesos corporales y envergadura intermedios en las regiones del Bajío, Balsas y Sierra (el peso promedio en machos fue de 7.86, 7.40 y 6.90 kg y envergadura de 113.50, 110.33 y 95.35 cm, respectivamente) y pesos corporales y envergadura menores en la región de la Costa ( el peso promedio de los machos fue de 7.10 kg y envergadura de 93.14 cm)(3).
CONCLUSIONES E IMPLICACIONES
El estudio de las poblaciones de pavos domesticados de cinco diferentes regiones fisiográficas del estado de Michoacán revela diversidad genética moderada dentro de las poblaciones, a excepción de la de la región de la Costa. Las poblaciones de las regiones del Bajío, Faja Volcánica Transmexicana y Sierra pudieron haber sido las primeras en separarse de otras poblaciones, y la población de la región de la Costa pudo haber provenido de una separación reciente. Las poblaciones domesticadas mostraron una estructura genética para tres grupos genéticamente diferentes que correlacionaron con su distribución geográfica, concordando con las diferencias morfométricas de las poblaciones en peso corporal y envergadura.
AGRADECIMIENTOS
Los autores agradecen el financiamiento otorgado por el Fondo Sectorial SAGARPA-CONACYT (proyecto 2004-C01-201 a MGZP), CIC-UMSNH (proyecto 14.6 a MGZP) y a PROMEP-UAT (beca otorgada a RLZ).
LITERATURA CITADA
1. Crawford RD. Introduction to Europe and diffusion of domesticated turkey from America. Arch Zoot 1992;4:307-314. [ Links ]
2. Losada H, Rivera J, Cortés J, Castillo A, González RO, Herrera J. Un análisis de sistemas de producción de guajolotes (Meleagris gallopavo) en el espacio suburbano de la delegación Xochimilco al sur de la Ciudad de México. Livest Res Rur Develop 2006;18:52.: http://www.lrrd.org/lrrd18/4/losa18052.htm. [ Links ]
3. López-Zavala R, Cano-Camacho H, Monterrubio-Rico TC, Chassin-Noria O, Aguilera-Reyes U, Zavala-Páramo MG. Morphological and productive characteristics of guajolote (Meleagris gallopavo) raised in backyard systems in Michoacán, México. Livest Res Rur Dev 2008;20:68.: http://www.lrrd.org/lrrd20/5/lope20068.htm. [ Links ]
4. Aquino RE, Arroyo LA, Torres HG, Riestra DD, Gallardo LF, López YBB. El guajolote criollo (Meleagris gallopavo L.) y la ganadería familiar en la zona centro del estado de Veracrúz. Tec Pecu Mex 2003;41(2):165-173. [ Links ]
5. Camacho-Escobar MA, Hernández-Sánchez V, Ramírez-Cancino L, Sánchez-Bernal EI, Arrollo-Ledezma J. Characterization of backyard guajolotes (Meleagris gallopavo gallopavo) in tropical zones of Mexico. Livest Res Rur Develop 2008;20:50.: http://www.lrrd.org/lrrd20/4/cama20050.htm. [ Links ]
6. López-Zavala R, Monterrubio-Rico TC, Cano-Camacho H, Chassin-Noria O, Aguilera-Reyes U, Zavala-Páramo MG. Native turkey (Meleagris gallopavo gallopavo) backyard production systems characterization in the physiographic regions of the State of Michoacán, Mexico. Tec Pecu Mex 2008;46:303-316. [ Links ]
7. Chassin-Noria O, López-Zavala R, Cano-Camacho H, Suárez-Corona E, Juárez-Caratachea A, Zavala-Páramo MG. Genetic diversity and similarity between domestic Mexican turkey Meleagris gallopavo populations based on random amplification polymorphic DNA (RAPD). Tec Pecu Mex 2005;43: 415-424. [ Links ]
8. Christman CJ, Hawes R. Rare turkey varieties. In: American Livestock Breeds Conservancy editor. Birds of feather, saving rare turkeys from extinction. North Carolina, USA: Glover Printing, Inc. Raleigh; 1999: 25-52. [ Links ]
9. Madrigal SX. Ubicación fisiográfica de la vegetación en Michoacán, México. Ciencia Nicolaita, UMSNH 1997;15:65-75. [ Links ]
10. Dutton PH. Methods for collection and preservation of simples for sea turtle genetic studies. In: Bowen BW, Witzel WN editors. Proc Intern Symp Sea Turtle Conserv Genetics. NOOA Technical Memorandum, NMFS-SETSC-396 1996:17-24. [ Links ]
11. FitzSimmons N. Male marine turtles: gene flow, philopatry and matting systems of green turtles Chelonia mydas [PhD thesis]. Australia: University of Qeensland; 1997. [ Links ]
12. Reed KM, Chávez LD, Hall MK, Knutson TP, Rowe JA, Torgersen AJ. Microsatellite loci for genetic mapping in the turkey (Meleagris gallopavo). Anim Biotech 2003;14:119-131. [ Links ]
13. Latch EK, Smith EJ, Rhodes Jr, OE. Isolation and characterization of microsatellite loci in wild and domestic turkeys (Meleagris gallopavo). Mol Ecol Not 2002;2:176-178. [ Links ]
14. Burt DW, Morrice DR, Sewalem A, Smith J, Paton IR, Smith EJ, Bently J, Hocking PM, Preliminary linkage map of the turkey (Meleagris gallopavo) based on microsatellite markers. Anim Genet 2003;34:399-409. [ Links ]
15. Goudet J. FSTAT, a program to estimate and test gene diversities and fixation indices version 2.9.3. 2001; http://www2.until.ch/popgen/softwares/fstat.htm. Accessed Feb 22, 2011. [ Links ]
16. Peakall R, Smouse PE. GENALEX 6: genetic analysis in Excel. Population genetics software for teaching and research. Mol Ecol Not 2006;6:288-295. [ Links ]
17. Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 1978;89:583-590. [ Links ]
18. Reynolds J, Weir BS, Cockerham CC. Estimation of the coancestry coefficient: basis for a short-term genetic distance. Genetics 1983;105:767-779. [ Links ]
19. Excoffier L, Lischer HEL. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Mol Ecol Res 2010;10:564-567. [ Links ]
20. Felsenstein J. Phylogeny Inference Package (PHYLIP). Version 3.5. Seattle, USA: University of Washington; 1993. [ Links ]
21. Page RD. TREEVIEW: An application to display phylogenetic trees on personal computers. Comp Applic Biosci 1996;12:357-358. [ Links ]
22. Pritchard JK, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data. Genetics 2000;155:945-959. [ Links ]
23. Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study. Mol Ecol 2005;14:2611-2620. [ Links ]
24. Hui-Fang L, Wei-Tao S, Jing-Ting S, Kuan-Wei C, Wen-Qi Z, Wei H, Wen-Juan X. Genetic diversity and population structure of 10 chinese indigenous egg-type duck breeds assessed by microsatellite polymorphism. J Genet 2010;89;65-72. [ Links ]
25. Smith J, Geng T, Long E, Pierson FW, Spoonenberg PD, Larson C, Gogal R. Molecular analysis of relatedness of five domesticated turkey strains. Biochem Genet 2005;43:35-47. [ Links ]
26. Berthouly C, Bed'Hom B, Tixier-Boichard M, Chen CF, Lee YP, Laloe D, Legros H, Verrier E, Rognon X. Using molecular markers and multivariate methods to study the genetic diversity of local European and Asian chicken breeds. Anim Genet 2008;39:121-129. [ Links ]
27. Granevitze Z, Hillel J, Chen GH, Cuc NTK, Feldman M, Eding H, Weigend S. Genetic diversity whitin chiken populations from different continents and management histories. Anim Genet 2007;38:576-583. [ Links ]
28. Kaya M, Yildiz MY. Genetic diversity among Turkish native chickens, Denizli and Gerze, estimated by microsatellite markers. Biochem Genet 2008;46:480-491. [ Links ]
29. Kong HS, Oh JD, Lee JH, Jo KJ, Sang BD, Choi CH, Kim SD, et al. Genetic variation and relationships of Korean native chickens and foreign breeds using 15 microsatellite markers. Asian Aust J Anim Sci 2006;19:1546-1550. [ Links ]
30. Tadano R, Sekino M, Nishibori M, Tsudsuki M. Microsatellite marker analysis for the genetic relationships among Japanese long-tailed chicken breeds. Poult Sci 2007;86:460-469. [ Links ]
31. Osei-Amponsah R, Kabang BB, Naazie A, Osei YD, Youssao JA, Yapi-Gnaore VC, Tixier-Boichard M, Rognon X. Genetic diversity of forest and savannah chicken populations of Ghana as estimated by microsatellite markers. J Anim Sci 2010;81:297-303. [ Links ]
32. Slatkin M, Barton NH. A comparison of three indirect methods for estimating average levels of gene flow. Evolution 1989;43:1349-1368. [ Links ]
33. Li YC, Korol AB, Fahima T, Beiles A, Nevo E. Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review. Mol Ecol 2002;11:2453-2465. [ Links ]
* Proyecto Apoyado por Fondos Sectoriales CONACYT-SAGARPA 2004. Clave de Registro: SAGARPA-2004-C01-201.














