Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ciencias pecuarias
versión On-line ISSN 2448-6698versión impresa ISSN 2007-1124
Rev. mex. de cienc. pecuarias vol.3 no.4 Mérida oct./dic. 2012
Artículos
Diversidad genética y distribución regional de cepas de Mycobacterium bovis del ganado en México
Genetic diversity and geographic distribution of Mycobacterium bovis from cattle in Mexico
Feliciano Milián Suazoa, Leticia García Casanovaa, Cecilia Romero Torresb, Germinal J. Cantó Alarcónc, José A Gutiérrez Reyesd, Susana Gallegos Sosaa, Marcela Mercado Pezzatb, Félix Mejía Estrada F.e, Aída L. Peña Cisnerosf, Ciro Estrada Chávezg, Oscar Pizano Martíneza
a CENID-Fisiología y Mejoramiento Animal, INIFAP. Km. 1 Carretera a Colón, Ajuchitlán, Qro. México. Teléfono y Fax: 419 2920036, 2920033. milian.feliciano@inifap.gob.mx. Correspondencia al primer autor.
b CENASA-SENASICA-SAGARPA, Tecámac, Estado de México.
c Universidad Autónoma de Querétaro. Querétaro, Qro.
d Dirección General de Salud Animal, SENASICA-SAGARPA, México D.F.
e INIFAP, SE Pichucalco, Pichucalco, Chiapas.
f INIFAP, CE Los Altos de Jalisco, Tepatitlán, Jal.
g CIATEJ, AC-CONACYT, Guadalajara, Jalisco.
Recibido el 2 de mayo de 2011.
Aceptado el 17 de octubre de 2011.
RESUMEN
Se obtuvieron patrones moleculares (espoligotipos) de 878 aislados de Mycobacterium bovis de ganado de diferentes regiones de México entre los años 2009 y 2010. Setenta y dos por ciento (72 %) de los espoligotipos cayeron en nueve grupos y 27 % de los aislados dentro de sólo dos espoligotipos; 149 fueron espoligotipos individuales. Los dos espoligotipos predominantes, arbitrariamente identificados como SP1 y SP2, se distribuyen en la mayor parte del territorio nacional, en especial en la zona centro de México en ganado especializado en producción de leche. A pesar de la amplia distribución geográfica de los espoligotipos de mayor frecuencia, algunos muestran cierta localización, en especial los encontrados en zonas geográficas distantes, como es el caso de Chihuahua y Baja California. Aunque pocos, algunos espoligotipos muestran patrones moleculares distintos a los mostrados por los espoligotipos de mayor frecuencia, sugiriendo fuentes de infección desconocida. La mayoría de los Estados con ganadería predominantemente lechera muestran espoligotipos comunes, lo que sugiere intercambio regional frecuente de ganado. Algunos espoligotipos son comunes en ganado para leche y ganado para carne, lo que sugiere transmisión entre estas dos poblaciones; se desconoce, sin embargo, si los animales de carne infectados provienen de explotaciones extensivas o si son de engordas ubicadas dentro de la explotación lechera. Se propone la tipificación rutinaria de aislados de M. bovis obtenidos en todos los laboratorios de diagnóstico y mejorar la captura de información epidemiológica de los casos, para hacer mejores conclusiones epidemiológicas de la distribución espacial de las cepas de este agente en el territorio nacional.
Palabras clave: Tuberculosis, Mycobacterium bovis, Epidemiología molecular, Spoligotyping, Tipificación.
ABSTRACT
The molecular fingerprints of 878 isolates of Mycobacterium bovis from cattle, mostly dairy cattle, collected from cattle between 2009 and 2010 in different regions of Mexico were obtained by spoligotyping. Seventy-two percent (72 %) of the spoligotypes fell into nine clusters, and 27 % of the isolates fell into only two spoligotypes; 149 were orphan spoligotypes. The two predominant spoligotypes, arbitrarily designated as SP1 and SP2, were found in almost all States in Mexico, especially in central Mexico, where a concentration of dairy cattle is known. In spite of the wide distribution of spoligotypes observed, some show high regional preference, especially those in geographically distant regions. Only a few spoligotypes show patterns completely different from those shown by the most frequent spoligotypes, suggesting strange sources of infection or the formation of new genetic lines derived from non-lethal mutations. Most States with predominantly high dairy cattle populations showed similar spoligotypes, suggesting exchange of animals between regions. Some spoligotypes are common to dairy and beef cattle, suggesting transmission between populations, most probably due to the movement of dairy cattle to non-dairy regions.
Key words: Tuberculosis, Mycobacterium bovis, Molecular epidemiology, Spoligotyping, Genetic typing.
INTRODUCCIÓN
La tuberculosis bovina (TBb) es causada por el bacilo M. bovis, bacteria de crecimiento lento e intracelular obligado(1), de distribución mundial y capaz de infectar a diferentes especies animales(2) y al hombre(3,4, 5). Esta enfermedad causa pérdidas económicas en la ganadería y representa un riesgo para la salud pública(6); la participación de M. bovis en humanos en México ha sido estimada en 7 %(7), mientras que en sur de los Estados Unidos de Norteamérica (USA) llega al 40 %(8). En la última década la TBb se ha convertido en una barrera no arancelaria para el comercio de ganado(9). Para reducir su impacto, la Dirección General de Sanidad Animal del Servicio Nacional de Sanidad Inocuidad y Calidad Agroalimentaria (SENASICA) ha establecido un Plan Estratégico que contempla a la investigación científica para sustentar actividades de campaña. Uno de estos proyectos está relacionado con la epidemiología molecular (EM) del agente etiológico, M. bovis(10).
Los mecanismos de persistencia y transmisión de la TBb en las poblaciones es poco entendida(11). Estudios sobre la dinámica de la enfermedad han sido tradicionalmente afectados por diversos factores que impiden la identificación de la relación epidemiológica de los casos. La movilización del ganado, la transmisión entre bovinos dentro y entre hatos, y la participación de la fauna silvestre, son factores que al igual que ocurre en otros países(12,13) en México son importantes, sin embargo, cuantificar el impacto de cada uno es complicado y costoso(14).
La EM es usada para entender mejor el comportamiento de la tuberculosis. Estos estudios se basan en la premisa de que animales infectados con cepas de patrón molecular idéntico son el resultado de transmisión reciente, mientras que aquéllos infectados por cepas de patrón único, se consideran cepas nuevas resultantes de mutaciones no letales(15). Sin embargo, es importante resaltar que patrones moleculares idénticos no necesariamente indican relación epidemiológica, y casos epidemiológicamente ligados no siempre tienen el mismo patrón molecular, lo que hace indispensable sustentar los hallazgos moleculares con información epidemiológica rutinaria.
Uno de los métodos más usados para la tipificación de cepas de M. bovis es la espoligotipificación (spoligotyping)(16)- Este método ha demostrado su utilidad en una gran cantidad de estudios con una amplia diversidad de objetivos y especies animales(17-23). En comparación con otros métodos, es fácil de realizar, requiere poca cantidad de ADN, es robusto y altamente reproducible, además de que puede ser aplicado a especímenes clínicos(16). Más que eso, spoligotyping es de utilidad en cepas de M. bovis con pocas copias del elemento de inserción IS6110, como ocurre con M. bovis obtenido del ganado(24). Así, el objetivo del presente trabajo fue identificar los patrones moleculares (espoligotipos) de cepas de M. bovis más frecuentes en las diferentes regiones de México, con el fin de apoyar las actividades de la campaña nacional contra la tuberculosis bovina, y elaborar una base de datos que apoye los programas de control y los acuerdos de comercialización interna y de exportación de ganado en México.
MATERIALES Y MÉTODOS
Aislados
Ochocientos setenta y ocho (878) aislados de M. bovis fueron obtenidos del ganado en diferentes regiones de México entre el 2009 y el 2010. En un muestreo pasivo por conveniencia y buscando la representación de cada Estado, a cada uno de los Laboratorios Regionales Acreditados para el Diagnóstico de la Tuberculosis Bovina en México se le solicitó, a través de la Dirección General de Salud Animal, el envío de un mínimo de 15 aislados de M. bovis de cada uno de los estados de los que tuvieran muestras, incluyendo la hoja de laboratorio con la información epidemiológica correspondiente. Por otro lado, buscando incrementar la muestra, y cubrir los faltantes de Estados no representados o con bajo número de aislados, en un proceso activo se obtuvieron lesiones sospechosas de tuberculosis en rastro para el cultivo y aislamiento de la bacteria: 269 aislados se obtuvieron en el muestreo pasivo y 609 en el muestreo activo.
Análisis de laboratorio
En el caso de las muestras de laboratorios, los aislados se obtuvieron por cultivo en medio de Stonebrink o Lowenstein-Jensen, y en el caso de lesiones obtenidas en el muestreo activo, los aislados se obtuvieron por cultivo en Stonebrink. Todos los aislados se analizaron genéticamente por el método de spoligotyping de acuerdo a Kamerbeek et al(16), utilizando el kit de ISOGEN (Maarssen, Países Bajos). Este método se basa en la presencia o ausencia de variantes en las regiones variables (DVR, por sus siglas en inglés) de secuencias directas repetidas (DR, por sus siglas en inglés). Las DVR se componen de una secuencia repetida de 36 pb conservada y una secuencia espaciadora variable de 35 a 41 pb. Por lo tanto, el blanco de la tipificación es la región variable(25).
Análisis molecular
Los patrones moleculares (espoligotipos) fueron convertidos a datos binarios de acuerdo a la presencia (1) o ausencia (0) de cada uno de los 43 espaciadores. Con apoyo de Excel los espoligotipos fueron organizados de acuerdo a su similitud genética. A todos aquéllos con espoligotipo idéntico se les asignó un número único, el cual se utilizó para realizar un análisis de clusters en SPSS ver. 16. Del resultado del análisis de clusters general se tomaron aquellos espoligotipos idénticos con dos o más aislados, para, por apreciación visual primero y por medio de un análisis de clusters parcial después, organizar grupos de similitud genética. Posteriormente, por medio de un análisis de frecuencia los espoligotipos se relacionaron con origen (Estado), sexo, raza y fin zootécnico de los animales afectados. Con la información relacionada al origen del animal (localidad), y con el apoyo de GoogleEarth se determinaron las coordenadas espaciales para cada uno de los aislados.
RESULTADOS
En el Cuadro 1 se muestra el estado de origen de los 878 aislados de M. bovis analizados. De un total de 26, los Estados que mayor cobertura tuvieron en términos de número de aislados fueron Jalisco (n=153), el Estado de México (n=110), Hidalgo (Tizayuca, n=106), Aguascalientes (n=78), Querétaro (n=71), Baja California (n=55), Guanajuato (n=51) y Coahuila (n=48); el resto tuvo entre 23 y 1 aislados. El 66 % (n=585) provinieron de ganado especializado en leche, el 7.5 % fueron de cruzas de otras razas con ganado especializado en leche, el 4 % de ganado especializado en carne y del 21 % no se obtuvo información (Cuadro 2).


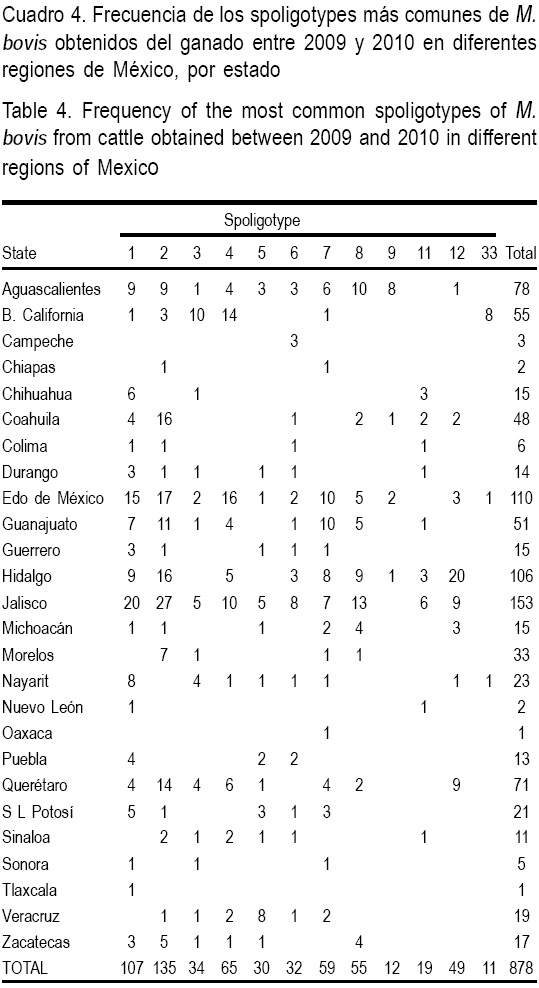
A partir de los 878 aislados se obtuvieron 194 espoligotipos, 45 con dos o más aislados y 149 con uno solo. Setenta y dos por ciento (72 %) de los espoligotipos agruparon en nueve clusters y 27 % de los aislados cayeron dentro de dos espoligotipos SP1 (n=107) y SP2 (n=135) (Cuadro 3). En este cuadro se muestran los espoligotipos que incluyeron dos o más aislados con su respectivo patrón molecular, y se puede observar que los espoligotipos SP1 y SP2 fueron por mucho los de mayor frecuencia y cobertura, ya que incluyeron al mayor número de aislados y su presencia se observó en 18 y 20 Estados, respectivamente, de un total de 26 representados. La información de este cuadro se complementa con la Figura 1, donde se muestran los grupos (A-I) de similitud genética. El análisis de clusters indica una alta similitud de espoligotipos dentro de los grupos, lo que sugiere que la mayoría de los espoligotipos son derivados de los espoligotipos de mayor frecuencia en cada uno de los grupos. Aunque los espoligotipos SP1 y SP2, están presentes en la mayoría de los Estados, su presencia es especialmente notoria en aquéllos tradicionalmente especializados en la producción de leche: Aguascalientes (n=9 y 9, respectivamente), Coahuila (n=4 y 16), Estado de México (n=15 y 17), Hidalgo (n=9 y 16), Guanajuato (n=7 y 11), Jalisco (n=20 y 27), y Querétaro (n=4 y 14). En el caso de Coahuila, la diferencia en la frecuencia entre SP1 y SP2 es notoria, el 33 % de los aislados de ese estado pertenecen al SP2.

Aunque en general se observa una gran diversidad regional de espoligotipos, en algunos de ellos se observa cierta regionalización (Cuadro 4). Por ejemplo, a pesar de que los espoligotipos SP1 y SP2 fueron los más frecuentes y los de mayor distribución en el País, en la región de Tijuana, BC tuvieron poca presencia (n=3). En esta región los espoligotipos más frecuentes fueron SP3 (n=10), SP4 (n=14) y SP33 (n=8), los cuales agrupan en el grupo B, sugiriendo parentesco de cepas. SP3 y SP33 fueron casi exclusivos de la región. En el caso de Chihuahua, sólo se observó presencia de SP1. Para Nayarit y Morelos, dos estados no considerados especializados en producción de leche, tuvieron la presencia de SP1 (n=8) y SP2 (n=7), donde sólo un aislado fue de ganado productor de carne, por lo que se presume que el resto provienen de ganado productor de leche o de sus cruzas. En Zacatecas se observó la presencia de SP1 (n=3) y SP2 (n=5), seis de estos fueron obtenidos de ganado para carne, por lo que se supone contacto estrecho entre razas en esta región. Situación similar se presentó en Guanajuato, donde siete aislados fueron de ganado para carne, los cuales comparten espoligotipo con aislados de ganado para leche. En Aguascalientes, además de SP1 y SP2 se nota la frecuencia de SP8 y SP9, donde SP8 está emparentado con SP1 y SP2, ya que están en el mismo grupo; sin embargo, SP9 pertenece a un grupo diferente. En la región de La Laguna (Coahuila y Durango), los espoligotipos de mayor frecuencia fueron SP1 y SP2; no obstante, otros espoligotipos de alta frecuencia en otras zonas lecheras del País, tales como SP4, SP7 y SP8, estuvieron ausentes. En el caso de Chihuahua, sólo dos espoligotipos predominan, SP1 y SP11, los cuales son muy diferentes entre sí genéticamente, lo que sugiere dos fuentes grandes de infección y poca entrada de animales de otras regiones.
DISCUSIÓN
A partir de 878 aislados de M. bovis obtenidos del ganado en diferentes regiones de México se generaron 197 espoligotipos, 45 con dos o más aislados y 149 con sólo uno. Los espoligotipos más frecuentes fueron SP1 y SP2, presentes en la mayoría de los estados representados, en especial en aquéllos tradicionalmente enfocados a la producción de leche. La mayoría de los espoligotipos que involucran a un solo aislado son muy parecidos a los espoligotipos de mayor frecuencia en la muestra, la diferencia en la presencia/ausencia de espaciadores en la mayoría de los casos es de sólo uno, lo que sugiere que estos son el resultado de nuevas líneas genéticas derivadas de mutaciones no letales en infecciones recientes.

La distribución de un mismo espoligotipo en diferentes regiones puede ser un indicativo de su alta movilización de ganado. De acuerdo a la frecuencia de los espoligotipos, esta movilización es más frecuente entre cuencas lecheras del centro del país y la región de La Laguna. Las cuencas del norte, Chihuahua y Baja California, parecen tener menos intercambio de animales, lo cual puede ser una consecuencia lógica de la distancia geográfica. La zona de Baja California tiene tres espoligotipos que la diferencian del resto, SP3, SP4 y SP33, los cuales están altamente emparentados, ya que sólo existe un espaciador de diferencia entre ellos, lo que sugiere poca entrada de animales de otras regiones de México. El caso de Hidalgo es muy particular, y que casi todos los aislados provienen de Tizayuca, un complejo lechero con alta densidad de población, donde se esperaría predominancia de algún es poligotipo en particular; no obstante en la base de datos, se observó la presencia de casi todos los de mayor frecuencia, si bien resalta el SP12, observado también con frecuencia en Jalisco y Querétaro, lo que sugiere intercambio frecuente de animales entre Tizayuca y estos dos estados.
Varios de los espoligotipos encontrados en aislados de ganado para leche fueron también encontrados en aislados de ganado para carne y en aislados de Estados no especializados en producción de leche. Esto fue notorio en los estados de Zacatecas, Guanajuato y Morelos, lo que sugiere transmisión entre razas. Desde luego, en el caso de otros Estados no puede descartarse que se trate de ganado para leche, pero desafortunadamente las muestras no tenían información sobre la raza del animal afectado en todos los casos para confirmar este dato.
Nuestros resultados coinciden con lo reportado con anterioridad sobre diversidad genética de espoligotipos de M. bovis por región en México(22,26,27). Los espoligotipos SP2, SP8, SP9, SP12 y SP17 ya habían sido señalados en hatos lecheros de Ciudad Juárez, Chihuahua(22); sin embargo, espoligotipos clasificados en www.mbovis.org como SB0987, SB0272, SB0985, SB0327 y SB0807 reportados en ese mismo estudio no fueron encontrados en nuestra base de datos, lo que sugiere que estos espoligotipos pueden ser particulares de esa región del País. Una ventaja de nuestro estudio en relación a trabajos previos es el alto número de aislados incluido y el número de estados representados, lo que garantiza una mayor representatividad de aislados por región y permite conclusiones más sólidas sobre la regionalización de algunos espoligotipos.
El la mayoría de los estudios de epidemiología molecular donde se involucra una cantidad indeterminada de aislados de M. bovis para determinar su distribución regional, se carece de representatividad en el muestreo. Esto es, el número de aislados analizados es más bien consecuencia de la disponibilidad de los mismos, que de un tamaño de muestra probabilístico, nuestro estudio no es la excepción. Por lo tanto, es probable que la poca o nula presencia de algunos de los espoligotipos en algunas regiones sea más bien consecuencia de la poca cantidad de aislados proporcionados, que de una ausencia real de los mismos. La sistematización en la tipificación (espoligotipificación) de todos los aislados de M. bovis obtenidos por los laboratorios regionales de diagnóstico ayudaría enormemente a determinar la posible regionalización de los espoligotipos del ganado en México.
Otra deficiencia de este tipo de estudios es la carencia de información epidemiológica disponible para cada aislado, lo que limita la validez de las conclusiones, y que se pueda explicar mejor el comportamiento del Mycobacterium en una determinada región geográfica. En el caso de México, existe un temor infundado por parte de autoridades de la Campaña de Tuberculosis de algunos Estados, de que la información se use para fines de "fiscalización", por lo que no proporcionan la información epidemiológica completa a pesar de contar con ella, lo que desde luego imposibilita el desarrollo de estudios epidemiológicos completos.
Es pertinente mencionar que spoligotyping no es un método de alto poder de discriminación, más bien se le considera un método apropiado para estudios globales para la identificación de grupos, los cuales deberán ser posteriormente analizados con un método más discriminatorio, como es el caso de VNTR (variable number tandem repeats, por sus siglas en inglés). De hecho se ha determinado por conveniencia internacional utilizar spoligotiping para la formación de grupos de aislados genéticamente relacionados y VNTR para definir cepas específicas y definir transmisión(28). En el caso de los aislados de este estudio, los análisis con VNTR están pendientes.
CONCLUSIONES E IMPLICACIONES
Se observa una gran diversidad de espoligotipos en las diferentes regiones de México, lo que puede ser consecuencia de la movilización frecuente de ganado. La mayoría de los espoligotipos de un solo aislado tienen una alta similitud con aquéllos de mayor frecuencia, por lo que se deduce que son consecuencia de mutaciones no letales que dan origen a nuevas líneas genéticas. La presencia de espoligotipos idénticos en ganado especializado en leche y ganado para carne, indica transmisión de tuberculosis de una raza de ganado a la otra, lo cual puede tener serias repercusiones epidemiológicas por razones de comercialización y de exportación de ganado. Aunque en este caso no se puede descartar que se trate de engordas familiares de ganado dentro de las explotaciones lecheras, en cuyo caso no representaría un riesgo para la ganadería de carne en su generalidad. Se concluye que una mayor representatividad de aislados de las diferentes regiones, una mayor representatividad de las diferentes razas de ganado y una mejor captura de información epidemiológica ayudaría a tener un diagnóstico de la situación de los espoligotipos de M. bovis en México más precisa. No obstante, ahora se cuenta con una base de datos importante que en mucho apoyará la toma de decisiones para el mejor control de la tuberculosis en México, y para mejorar los acuerdos de comercialización nacional e internacional de animales.
AGRADECIMIENTOS
Se agradece la participación de los Laboratorios Regionales de Diagnóstico de TB en México, que amablemente proporcionaron aislados de M. bovis para la realización de este trabajo.
LITERATURA CITADA
1. Bourne FJ. Bovine TB: The scientific evidence. Final report of the independent scientific group on cattle TB. Defra, UK. 2007. [ Links ]
2. Pollock M, Neill D. Mycobacterium bovis infection and tuberculosis in cattle. Vet J 2002;(2):115-27. [ Links ]
3. Gibson AL, Hewinson G Goodchild T, Watt B, Story A, Inwald J, Drobniewski FA. Molecular epidemiology of disease due to Mycobacterium bovis in humans in the United Kingdom. J Clin Microbiol 2004;(42):431-434. [ Links ]
4. Romero B, Aranaz A, de Juan L, Alvarez J, Bezos J, Mateos A, Gómez-Mampaso E, Domínguez L. Molecular epidemiology of multidrug-resistant Mycobacterium bovis isolates with the same profile as isolates from animals. J Clin Microbiol 2006;(449): 3405-3408. [ Links ]
5. Lari N, Rindi L, Bonanni D, Tortoli E, Garzelli C. Molecular analysis of clinical isolates of Mycobacterium bovis recovered from humans in Italy. J Clin Microbiol 2006;(4411):4218-4221. [ Links ]
6. de la Rua-Domenech R. Human Mycobacterium bovis infection in the UK: incidence, risks, control measures and review of the zoonotic aspects of bovine tuberculosis. Tuberc 2006;(86):77-109. [ Links ]
7. Pérez-Guerrero L, Milián SF, Arriaga DC, Romero TC, Escartín ChM. Epidemiología molecular de la tuberculosis bovina y humana en Querétaro, México. Rev Sal Púb Méx 2008;(50):1-6. [ Links ]
8. Rodwell TC, Moore M, Moser KS, Brodine SK, Strathdee SA.Tuberculosis from Mycobacterium bovis in binational communities, United States. Emerg Infect Dis 2008;(146):909-916. [ Links ]
9. Milian-Suazo F, Harris B, Arriaga DC, Romero TC, Stuber T, Alvarez OG, Morales LA, et al. Molecular epidemiology of Mycobacterium bovis: usefulness in international trade. Prev Vet Med 2008;(87):261-271. [ Links ]
10. Plan Estratégico de la Campaña Nacional de la Tuberculosis Bovina en México, 2008-2012. Dirección General de Salud Animal. Campaña Nacional Contra la Tuberculosis Bovina. [en línea]. http://www.senasica.gob.mx/?id=801. Consultado Abr 15, 2011. [ Links ]
11. White PC, Bohm M, Marion G, Hutchings, MR. Control of bovine tuberculosis in British livestock: there is no 'silver bullet'. TIM 2008;420-427. [ Links ]
12. Green DM, Kiss IZ, Mitchell AP, KAO RR. Estimates for local and movement-based transmission of bovine tuberculosis in British cattle. Proc Royal Soc Biol Sci 2008;(275):1001-1005. [ Links ]
13. Ramirez-Villaescusa AM, Medley GF, Mason S, Green IE. Herd and individual animal risks associated with bovine tuberculosis skin test positivity in cattle in herds in south west England. Prev Vet Med 2009;(92):188-198. [ Links ]
14. Skuce RA, Mallon TR, McCormick CM, McBride SH, Clarke G, Thompson A, Couzens C, Gordon AW, McDowell SWJ. Mycobacterium bovis genotypes in Northern Ireland: herd- level surveillance (2003 to 2008). Vet Rec 2010;(167);684-689. [ Links ]
15. Durr PA, Clifton-Hadley RS, Hewinson RG. Molecular epidemiology of bovine tuberculosis II. Applications of genotyping. Rev Sci Tech Off Int Epiz 2000;(19):689-701. [ Links ]
16. Kamerbeek J, Schouls L, Kolk A, van Agterveld M, van Soolingen D, Kuijper S, Bunschoten A, Molhuizen H, Shaw R, Goyal M, van Embden J: Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol 1997;(35):907-914. [ Links ]
17. Collins DM, Gabric DM, de Lisle GW. Typing of Mycobacterium bovis isolates from cattle and other animals in the same locality. NZ Vet J 1988;(36):45-46. [ Links ]
18. van Soolingen D, de Haas PEW, Haagsma J, Eger T, Hermans PWM, Ritacco V, Alito A, van Embden JDA. Use of various genetic markers in differentiation of Mycobacterium bovis strains from animals and humans and for studying epidemiology of bovine tuberculosis. J Clin Microbiol 1994;(32):2425-2433. [ Links ]
19. Zumarraga MJ, Martin C, Samper S, Alito A, Latini O, Bigi F, Roxo E, Cicuta ME, Errico F, Ramos MC, van Soolingen D, Romano MI, Usefulness of spoligotyping in molecular epidemiology of Mycobacterium bovis-related infections in South America. J Clin Microbiol 1999;(372):296-303. [ Links ]
20. Gutiérrez M, Samper S, Gavigan JA, García Marín JF, Martín C. Differentiation by molecular typing of Mycobacterium bovis strains causing tuberculosis in cattle and goats. J Clin Microbiol 1995;(33):2953-2956. [ Links ]
21. Aranaz A, Liébana E, Mateos A, Dominguez L, Vidal D, Domingo M, Gonzolez O, et al. Spacer oligonucleotide typing of Mycobacterium bovis strains from cattle and other animals: a tool for studying the epidemiology of tuberculosis. J Clin Microbiol 1996;(34):2734-40. [ Links ]
22. Cobos-Marín L, Montes-Vargas J, Zumarraga M, Cataldi A, Romano MI, Estrada-Garcia I, Gonzalez-y-Merchand JA. Spoligotype analysis of Mycobacterium bovis isolates from Northern México. Can J Microbiol 2005;(51):996-1000. [ Links ]
23. Parra A, Larrasa J, García A, Alonso JM, de Mendoza JH. Molecular epidemiology of bovine tuberculosis in wild animals in Spain: a first approach to risk factor analysis. Vet Microbiol 2005;(110):293-300. [ Links ]
24. Amadio A, Romano MI, Bigi F, Etchechoury I, Kubica T, Niemann S, Cataldi A, Caimi K. Identification and characterization of genomic variations between Mycobacterium bovis and M. tuberculosis H37Rv. J Clin Microbiol 2005(435):2481-2484. [ Links ]
25. Acosta-Salinas R, Estrada-Chávez C, Milián-Suazo F. Tipificación de cepas de mycobacterium bovis: revisión. Tec Pecu Méx 2009;(47):389-412. [ Links ]
26. Milián SF, Banda RV, Ramírez CC, Arriaga DC. Genotyping of Mycobacterium bovis by geographic location within México. Prev Vet Med 2002;(55):255-264. [ Links ]
27. Santillan-Flores MA, Flores J, Arriaga-Diaz C, Romero-Torres C, Suárez-Güemes F, Espitia C. Polymorphism of the PE domain of PE/PEPGRS sequences in clinical isolates of Mycobacterium bovis in Mexico. Vet Microbiol 2006;(115):364-369. [ Links ]
28. Smith NH, Kremer K, Inwald J, Dale J, Driscoll JR, Gordon SV, Van Soolingen D, Hewinson RG, Smith JM. Ecotypes of the Mycobacterium tuberculosis complex. J Theor Biol 2006;(239):220-225. [ Links ]














