Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ciencias pecuarias
versión On-line ISSN 2448-6698versión impresa ISSN 2007-1124
Rev. mex. de cienc. pecuarias vol.3 no.3 Mérida jul./sep. 2012
Artículos
Efectos genéticos para características de crecimiento de bovinos Charolais y Charbray mexicanos estimados con modelos alternativos
Genetic effects for growth traits of Mexican Charolais and Charbray cattle estimated with alternative models
Ángel Ríos Utreraa, Guillermo Martínez Velázquezb, Vicente Eliezer Vega Murilloa, Moisés Montaño Bermúdezc
a Campo Experimental La Posta, Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP), km 22.5 Carretera Veracruz-Córdoba, Paso del Toro, 94277 Veracruz, México. Tel 01 (285) 596-0111. rios.angel@inifap.gob.mx; ariosu@hotmail.com. Correspondencia al primer autor.
b Sitio experimental El Verdineño, INIFAP.
c CENID Fisiología y Mejoramiento Animal, INIFAP.
Recibido el 3 de febrero de 2011.
Aceptado el 8 de abril de 2011.
RESUMEN
Para estimar parámetros genéticos para peso al nacimiento (n=105,599), peso al destete ajustado a 205 días (n=89,111) y peso al año ajustado a 365 días (n=55,284), se analizaron los datos registrados de 1997 a 2009 por la Charolais-Charbray Herd Book de México, con seis diferentes modelos animal. El Modelo 1 incluyó el efecto genético aditivo directo. El Modelo 2 consideró el ambiente permanente de la madre, incluyéndolo como un efecto aleatorio adicional. El Modelo 3 incluyó el efecto genético aditivo directo y el efecto genético aditivo materno, asumiendo una covarianza igual a cero entre ellos. El Modelo 4 fue similar al Modelo 3, pero además incluyó el ambiente permanente de la madre. El Modelo 5 fue similar al Modelo 3, pero además incluyó la covarianza entre los efectos genéticos directo y materno. El Modelo 6 ajustó los tres efectos aleatorios más la covarianza entre efectos directos y maternos. Comparado con los demás modelos, el modelo más completo (Modelo 6) redujo sustancialmente -2[logaritmo de la verosimilitud] (P<0.05), proporcionando el mejor ajuste para las tres características. La comparación entre el Modelo 1 y el Modelo 6 para cada característica, mostró que el Modelo 1 produjo menores estimadores de la varianza genética aditiva directa, en contraste con los resultados de estudios previos. Sin embargo, los estimadores de la varianza residual obtenidos con el Modelo 1 fueron significativamente mayores que los obtenidos con el Modelo 6.
Palabras clave: Parámetros genéticos, Características de crecimiento, Bovinos carne.
ABSTRACT
Data recorded from 1 997 to 2009 by the Charolais-Charbray Herd Book of Mexico were analyzed with six different animal models to estimate genetic parameters for birth weight (n=1 05,599), weaning weight adjusted to 205 d (n=89,111 ) and yearling weight adjusted to 365 days (n=55,284). Model 1 included direct additive genetic effects of the animal. Model 2 allowed for a common environmental effect due to the dam, fitting it as an additional random effect. Model 3 included the animal's direct genetic effect, and the dam's maternal genetic effect, assuming a covariance equal to zero between them. Model 4 was the same as Model 3 but it also allowed for a common environmental effect due to the dam. Model 5 is identical to Model 3, but included a covariance between direct and maternal genetic effects. Model 6 was fitted for all three random effects plus the covariance between direct and maternal genetic effects. Compared with any of the other models, the most complete model (Model 6) substantially reduced the -2log L (P<0.05), providing the best fit for the three growth traits. Comparison between Model 1 and Model 6 across traits showed that Model 1 yielded smaller estimates of direct genetic variance, in contrast with results from previous studies. However, estimates of the residual variance for Model 1 were significantly greater than estimates for Model 6.
Key words: Genetic parameters, Growth traits, Beef cattle.
INTRODUCCIÓN
En bovinos Hereford(1), Simmental(2), Nelore(3), Angus(4), Gelbvieh(5), Charolais(6), Brahman(7) y Limousin(8) se ha encontrado que los efectos maternos influyen sobre características de crecimiento predestete. Sin embargo, la magnitud de los estimadores de parámetros genéticos para características de crecimiento en bovinos productores de carne puede variar dependiendo del grupo racial y de los efectos genéticos incluidos en el modelo estadístico(9). Se ha reportado que la exclusión de efectos maternos (genético y ambiental) del modelo estadístico resulta en la sobreestimación de la varianza genética aditiva directa del peso al destete(10). Por lo tanto, antes de realizar la evaluación genética de cualquier característica económicamente importante, se debe seleccionar el modelo más apropiado para poder desarrollar programas de mejoramiento genético efectivos y estimar valores genéticos exactos.
La prueba de razón de verosimilitudes se ha usado extensivamente para comparar el ajuste de diferentes modelos estadísticos para la estimación de parámetros genéticos en bovinos productores de carne(11-16), borregos(17-21) y cerdos(22,23,24). Con base en lo anterior, el objetivo del presente estudio fue identificar el modelo más apropiado para estimar parámetros genéticos para características de crecimiento de bovinos Charolais y Charbray criados en condiciones extensivas en diferentes regiones (Estados) de México.
MATERIALES Y MÉTODOS
Descripción de la información
Datos de pesos al nacimiento, destete y año de edad, así como de pedigrí de animales criados en diferentes regiones de México se obtuvieron de la Charolais-Charbray Herd Book de México, correspondientes al periodo de 1997 a 2009. Los registros de pesos al destete y año de edad se ajustaron a 205 ({[peso al destete - peso al nacimiento]/edad al destete} x 205 + peso al nacimiento) y 365 días ({[peso al año - peso al destete]/días entre pesajes} x 160 + peso ajustado a 205 días), de acuerdo a los Lineamientos para Programas Uniformes de Mejoramiento de Bovinos Productores de Carne(25). Los pesos ajustados a 205 y 365 días de edad deben ser calculados para becerros pesados entre 160 y 250 y entre 320 y 410 días de edad. Los registros de becerros con edades fuera de estos intervalos fueron eliminados de los análisis, pero no del pedigrí. Después de la edición, 105,599 registros de peso al nacimiento, 89,111 de peso al destete ajustado a 205 días, y 55,284 de peso ajustado a 365 días de edad se usaron en los análisis. El número de sementales con progenie en la base de datos fue 3,923, 3,791 y 3,054 para peso al nacimiento, peso al destete ajustado a 205 días y peso al año ajustado a 365 días de edad, respectivamente. El archivo de pedigrí, el cual incorporó toda la información de pedigrí disponible, fue el mismo para las tres características y consistió de 131,659 animales, incluyendo padres sin registros. El número de registros y de sementales para cada característica e información adicional de la estructura de los datos se resumen en el Cuadro 1.

Descripción de los modelos
Efectos aleatorios. Cada característica de crecimiento se analizó con seis diferentes modelos animal, como los descritos por Meyer(9), para evaluar la importancia de diferentes efectos genéticos. El Modelo 1 incluyó el efecto genético aditivo directo. El Modelo 2 incluyó el efecto del ambiente materno permanente, incluyéndolo como un efecto aleatorio adicional. El Modelo 3 incluyó el efecto genético aditivo directo y el efecto genético aditivo materno, asumiendo que estos efectos no estuvieron correlacionados. El Modelo 4 fue similar al Modelo 3, pero incluyó además el efecto ambiental permanente de la madre. El Modelo 5 fue similar al Modelo 3, pero se asumió que los efectos genéticos aditivos directos y maternos estaban correlacionados. El Modelo 6 incluyó los tres efectos aleatorios más la covarianza entre los efectos genéticos directos y maternos.
Efectos fijos. Todos los modelos animal incluyeron los mismos efectos ambientales fijos, grupo contemporáneo y edad de la madre al parto. El grupo contemporáneo comprendió hato, año, época y sexo del becerro, mientras que la edad de la madre se incluyó como una covariable lineal. El año de nacimiento comprendió de 1997 a 2009, y los meses de nacimiento se agruparon en cuatro épocas de nacimiento: 1) enero-marzo, 2) abril-junio, 3) julio-septiembre, 4) octubre-diciembre. Además, todos los modelos incluyeron los efectos genéticos fijos de proporción de genes Charolais, heterocigosis y pérdidas por recombinación como covariables. La heterosis y las pérdidas por recombinación se calcularon de acuerdo con Akbas et al(26). Dickerson(27) reportó que el cruzamiento entre animales híbridos o el retrocruzamiento rompen las combinaciones epistáticas de los genes en las razas, pero en diferente magnitud en diferentes cruzas. Además, Demeke et al(28) concluyeron que ignorar los efectos de heterosis y las pérdidas por recombinación presentes en los animales resulta en la sobreestimación de las varianzas genéticas directas y maternas, y de la heredabilidad directa de características de crecimiento tempranas en una población de bovinos Bos indicus y cruzados Bos taurus x Bos indicus.
Modelos en notación matricial. Los seis modelos animal univariados pueden describirse de la siguiente manera:

donde y es un vector de observaciones para cada una de las diferentes características, b es un vector de efectos ambientales y genéticos fijos (grupo contemporáneo, edad de la madre, proporción de genes Charolais, heterocigosis y pérdidas por recombinación), a es un vector aleatorio desconocido de efectos genéticos aditivos directos, m es un vector aleatorio desconocido de efectos genéticos aditivos maternos, pe es un vector aleatorio desconocido de efectos ambientales maternos permanentes, e es un vector aleatorio desconocido de efectos ambientales temporales, y X, Z1, Z2, y Z3 son matrices conocidas de incidencia que relacionan los registros con b, a, m, y pe, respectivamente.
Se asumió que los efectos genético aditivo directo, genético aditivo materno, ambiental permanente de la madre y residual se dis tribuyeron normalmente con media 0 y estructura de varianzas y covarianzas que dependió de las suposiciones de cada modelo. En particular, la estructura de varianzas y covarianzas para el Modelo 6 fue:
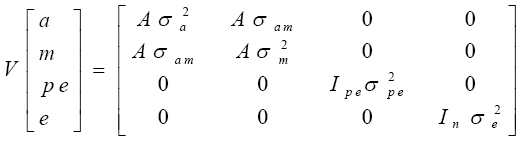
donde A es la matriz de relaciones aditivas de Wright entre todos los animales en el pedigrí, σ2a es la varianza genética aditiva directa, σ2m es la varianza genética aditiva materna, σam es la covarianza entre los efectos genéticos aditivos directo y materno, σ2pe es la varianza del ambiente materno permanente, σe2 es la varianza del ambiente temporal, e I pe e Inson matrices identidad de tamaño igual al número de madres y número de observaciones, respectivamente. Se asumió que todas las demás covarianzas fueron igual a cero.
Estimación de componentes de varianza y covarianza
Las varianzas y covarianzas se estimaron con Máxima Verosimilitud Restricta Libre de Derivadas(29), usando el programa MTDFREML(30). Se asumió que la convergencia se alcanzó cuando la varianza de menos dos veces el logaritmo de la verosimilitud en el simplex fue menor que 10-8. Después de que el programa convergió por primera vez, se realizaron varios reinicios para asegurarse de que se había alcanzado un máximo global, en lugar de un máximo local. En cada nuevo análisis, se usaron como valores iniciales los estimadores de los parámetros obtenidos en el análisis previo. Las soluciones de los efectos aleatorios se obtuvieron del último ciclo de iteración donde se alcanzó el máximo global.
Estimadores de parámetros genéticos Se obtuvieron estimadores para la varianza fenotípica (σ2p = σ2a+σ2m+σam+σ2pe+σ2e), heredabilida para efectos genéticos aditivos directos (h2a = σ2a / σ2p), heredabilidad para efectos genéticos aditivos maternos (h2m = σ2m / σ2p) , covarianza genética entre efectos directos y maternos como proporción de la varianza fenotípica (cam = σam / σ2p) , correlación entre efectos genéticos aditivos directos y maternos (rram= σam / (σ2a σ2m)½), fracción de la varianza fenotípica debida a efectos del ambiente materno permanente (c2 = σ2pe / σ2p ) , y varianza residual como proporción de la varianza fenotípica (e2 = σ2e/ σ2p). Los errores estándar para los estimadores de los parámetros genéticos fueron aproximados y fueron calculados usando la matriz de información promedio(31) y el Método Delta(32). Todas las fracciones de la varianza fenotípica y sus errores estándar fueron calculados por el programa MTDFREML, excepto la fracción de la covarianza, la cual fue calculada a mano. El error estándar para el estimador de este parámetro genético no se proporciona. Adicionalmente, se calcularon los estimadores de la heredabilidad total (h2t), usando la ecuación (ht2 =[ σ2a +0.5 σ2m+1.5 σam ]/σ2p) propuesta por Willham(33). Esta ecuación representa la regresión del genotipo (directo y materno) de un animal sobre su fenotipo.
Comparación entre modelos
La superioridad de un modelo sobre otro para explicar significativamente mejor los datos de peso al nacimiento, destete y año de edad se determinó mediante la prueba de razón de verosimilitudes. La diferencia entre los valores de la función para pares de modelos puede ser probada mediante la prueba Chi cuadrada, donde los grados de libertad son la diferencia en el número de los componentes de varianza y covarianza en los modelos(2). Dicha prueba se realizó usando la función Probchi de SAS(34).
RESULTADOS Y DISCUSIÓN
Las estadísticas descriptivas de los registros para las tres características evaluadas se muestran en el Cuadro 2. Las medias y sus desviaciones estándar para peso al nacimiento, peso al destete ajustado a 205 días, y peso al año ajustado a 365 días fueron 39 ± 5.4, 227 ± 41 y 348 ± 64 kg, respectivamente. Como se esperaba, los coeficientes de variación para los pesos al destete y al año fueron similares, pero ligeramente mayores que el coeficiente de variación para peso al nacimiento.

Los estimadores de los componentes de (co)varianza, junto con los valores de la varianza de menos dos veces el logaritmo de la verosimilitud (-2logL), y los estimadores de los parámetros genéticos obtenidos con los seis diferentes modelos animal para peso al nacimiento, peso al destete ajustado a 205 días y peso al año ajustado a 365 días, se resumen en los Cuadros 3 y 4, respectivamente.
Peso al nacimiento
Cuando el efecto del ambiente permanente (Modelo 2) o el efecto genético aditivo materno (Modelo 3) fue incluido, ocurrió una disminución sustancial (P<0.01) en -2logL (de 357,820 a 357,601 o 357,544) comparado con el Modelo 1, mostrando la influencia de estos dos efectos sobre el peso al nacimiento. Por lo tanto, se determinó que los efectos maternos fueron importantes para peso al nacimiento. El estimador de la varianza del ambiente materno permanente del Modelo 2, y el estimador de la varianza genética aditiva materna del Modelo 3 explicaron 5.3 y 5.0 % de la varianza fenotípica para peso al nacimiento, respectivamente. El estimador de la varianza genética aditiva directa del Modelo 1 fue 24 y 27 % mayor que los estimadores correspondientes de los Modelos 2 y 3, respectivamente. Consecuentemente, los estimadores de heredabilidad directa para peso al nacimiento también fueron sobreestimados con el Modelo 1, comparado con los estimadores correspondientes de los Modelos 2 y 3, los cuales proporcionaron iguales estimadores de heredabilidad directa (0.21). El estimador de la heredabilidad total obtenido con el Modelo 1 fue mayor que los estimadores de la heredabilidad total obtenidos con los Modelos 2 y 3 (0.26 vs 0.21 y 0.23).
Cuando los efectos maternos (genético aditivo y ambiental permanente) fueron incluidos simultáneamente en el Modelo 4, ocurrió una mayor reducción en -2logL (P<0.01; 357,511 vs 357,820), el estimador de la varianza genética aditiva directa (3.09 vs 4.00 kg2) y el estimador de la heredabilidad directa (0.20 vs 0.26), comparado con el Modelo 1. Se ha argumentado(35) que si existen efectos maternos, pero no son tomados en cuenta, el estimador de la varianza genética aditiva directa incluirá por lo menos parte de la varianza materna; por lo tanto, los estimadores de la heredabilidad directa disminuyen cuando se incluyen los efectos maternos. Los estimadores de la varianza genética aditiva materna y de la varianza del ambiente materno permanente obtenidos con el Modelo 4 fueron similares (0.46 y 0.42 kg2, respectivamente), pero menores que los estimadores correspondientes obtenidos con los Modelos 2 (0.81 kg2) y 3 (0.73 kg2), indicando que estos últimos estimadores de parámetros genéticos fueron sobreestimados.
Comparado con el Modelo 1, el modelo más completo (Modelo 6) sustancialmente redujo (P<0.01) -2logL (357,820 vs 357,051), proporcionando el mejor ajuste para los datos de peso al nacimiento. Esta comparación muestra que el Modelo 1 produjo un menor estimador de la varianza genética aditiva directa que el Modelo 6, en contraste con los resultados de estudios previos(9,28,36); sin embargo, el estimador de la varianza residual del Modelo 1 fue mayor que el estimador correspondiente del Modelo 6. El estimador de la heredabilidad total del Modelo 6 fue 5 2. 9 % menor que el estimador de la heredabilidad total del Modelo 1. Esta considerable reducción se debió a la alta magnitud absoluta (-0.78; Cuadro 4) del estimador de la correlación genética entre efectos directos y maternos obtenidos con el Modelo 6.
El estimador de la heredabilidad directa para peso al nacimiento fue del 36 % con el mejor modelo. Meyer(37) obtuvo un menor estimador (0.21) para bovinos Charolais en Australia. Trabajando con la misma raza productora de carne, Grotheer et al(38) y Phocas y Laloë(39) reportaron similares estimadores de heredabilidad directa para peso al nacimiento (0.38, 0.33). En contraste con el presente estimador, se han reportado mayores estimadores de heredabilidad directa (0.54, 0.55, 0.45) para bovinos Charolais criados en Brasil(40), Canadá(41) y Suiza(42). El estimador de heredabilidad para efectos maternos obtenido con el mejor modelo (0.13) es comparable con, y está dentro del intervalo de estimadores (0.11 a 0.18) reportados por otros(39-43) para bovinos Charolais. El estimador de la correlación genética entre efectos genéticos directos y maternos para peso al nacimiento fue -0.78, el cual es mayor que los estimadores obtenidos en otros países para la raza Charolais(38,39,41,42). Ríos-Utrera(44) reportó que los estimadores de la correlación genética entre efectos directos y maternos para peso al nacimiento variaron de -1.05 a 0.99. Altas correlaciones genéticas entre efectos directos y maternos implican un progreso genético lento. Sin embargo, la magnitud de nuestro estimador de la correlación pudo ser el resultado de la ausencia de algunos efectos en el modelo (e.g., interacción semental x hato).
Peso al destete ajustado a 205 días
Las reducciones significativas en -2logL (P<0.01) de los Modelos 1 al 6 indican que el peso al destete ajustado a 205 días fue significativamente influenciado por efectos genéticos maternos y ambientales permanentes, como ocurrió con el peso al nacimiento. La inclusión del efecto del ambiente permanente en el Modelo 2 disminuyó 55 kg2 la varianza genética aditiva directa, en relación con el Modelo 1. De forma similar, en el Modelo 3 la adición del componente genético aditivo materno redujo 59 kg2 la varianza genética aditiva directa. Comparados con el Modelo 1, los Modelos 2 y 3 mostraron una disminución de la heredabilidad directa, de 0.23 a 0.15 y 0.14. La comparación entre los Modelos 3 (modelo animal con efectos genéticos maternos) y 4 (modelo animal con efectos genético materno y ambiental permanente) mostró que el estimador de la heredabilidad materna disminuyó de 0.08 (Modelo 3) a 0.04 (Modelo 4). La inclusión de la covarianza entre efectos genéticos directos y maternos (Modelos 5 y 6) resultó en altos estimadores (valores absolutos) de correlaciones genéticas (-0.76 y -0.81, respectivamente), y causó que las heredabilidades directa y materna aumentaran y que -2logL disminuyera dramáticamente. Este fenómeno ha sido observado en otros estudios similares con bovinos productores de carne(9,11,38,45,46) y ha sido atribuido a grandes covarianzas de muestreo o a la presencia de una covarianza madre-cría no considerada en el modelo(37). Cuando se incluyó la covarianza entre efectos directos y maternos (Modelos 5 y 6), los estimadores de heredabilidad total para peso al destete ajustado a 205 días disminuyeron, comparado con los modelos que no incluyeron dicha covarianza (Modelos 1, 2, 3 y 4). El modelo más apropiado (Modelo 6) indica que los efectos genéticos maternos fueron más importantes que los efectos ambientales permanentes.
El estimador de la heredabilidad directa para peso al destete ajustado a 205 días (0.27) fue menor que el estimador correspondiente para peso al nacimiento. Para bovinos Charolais estadounidenses y canadienses, Donoghue y Bertrand(41) estimaron heredabilidades directas de 0.25 y 0.27 para peso al destete ajustado a 205 días, las cuales son similares al presente estimador obtenido con el modelo completo. La heredabilidad materna estimada con el Modelo 6 fue de 0.15. Estimadores similares han sido reportados para bovinos Charolais estadounidenses(5), franceses(47) y neozelandeses(41). El estimador de la varianza del ambiente materno permanente para peso al destete ajustado a 205 días explicó 6 % del estimador de la varianza fenotípica, y fue mucho menor que el estimador de la heredabilidad materna.
Peso al año ajustado a 365 días
Aunque los efectos maternos para peso al año ajustado a 365 días fueron relativamente menos importantes, estos siguieron siendo evidentes después del destete (efecto residual). La comparación de los estimadores de los Modelos 1 y 2, y de los Modelos 1 y 3 indica que la adición de los efectos genético aditivo materno y ambiental permanente causó la disminución del estimador de la heredabilidad directa de 0.23 a 0.18, menos que para peso al destete ajustado a 205 días. La prueba de razón de verosimilitudes fue significativa (P<0.01) para los Modelos 1 y 2, y para los Modelos 1 y 3. La inclusión simultánea de los efectos genético aditivo materno y ambiental permanente en el Modelo 4 resultó en pequeñas reducciones de ambas fracciones de dichos efectos maternos, en relación con los Modelos 2 y 3; sin embargo, los estimadores de heredabilidad directa obtenidos con los Modelos 2, 3 y 4 fueron de la misma magnitud (0.18). Los estimadores de los parámetros genéticos fueron similares para los Modelos 5 y 6, aunque los valores de -2logL mostraron que el Modelo 6 ajustó mejor los datos (P<0.01). Con el Modelo 6 se obtuvo un estimador de heredabilidad directa de 0.30. Este estimador es similar a los valores de 0.32 y 0.33 reportados para bovinos Charolais australianos(37) y alemanes(38). La heredabilidad materna para peso al año ajustado a 365 días tuvo un valor bajo (0.12). Los efectos genéticos directos y maternos para peso al año ajustado a 365 días estuvieron fuertemente correlacionados (-0.81).
CONCLUSIONES E IMPLICACIONES
Con cada modelo, los estimadores de heredabilidad directa para peso al nacimiento fueron mayores que los estimadores correspondientes para peso al destete y al año. Los estimadores de heredabilidad materna para peso al destete fueron mayores que los estimadores correspondientes para peso al nacimiento y peso al año. Por otro lado, los efectos genéticos aditivos directos fueron más importantes que los efectos maternos (genético y ambiental permanente) para determinar las tres características evaluadas. La comparación de los diferentes modelos animal mostró que el modelo que incluyó efectos genéticos directos y maternos, su correlación, y efectos del ambiente permanente es recomendable para la estimación de diferencias esperadas en la progenie para peso al nacimiento, peso al destete ajustado a 205 días y peso al año ajustado a 365 días de bovinos Charolais y Charbray mexicanos. El progreso genético podría ser lento debido al fuerte antagonismo entre los efectos genéticos directos y maternos; sin embargo, dicho antagonismo pudo ser consecuencia de efectos adicionales presentes en los datos, tales como la interacción semental x ambiente o una covarianza ambiental negativa entre las madres y su descendencia.
LITERATURA CITADA
1. Meyer K, Carrick MJ, Donnelly BJP. Genetic parameters for growth traits of Australian beef cattle from a multibreed selection experiment. J Anim Sci 1993;71:2614-2622. [ Links ]
2. Swalve HH. Estimation of direct and maternal (co)variance components for growth traits in Australian Simmental beef cattle. J Anim Breed Genet 1993;110:241-252. [ Links ]
3. Eler JP, Van Vleck LD, Ferraz JB, Lobo RB. Estimation of variances due to direct and maternal effects for growth traits of Nelore cattle. J Anim Sci 1995;73:3253-3258. [ Links ]
4. Berweger Baschnagel M, Moll J, Künzi N. Comparison of models to estimate maternal effects for weaning weight of Swiss Angus cattle fitting a sire x herd interaction as an additional random effect. Livest Prod Sci 1999;60:203-208. [ Links ]
5. Duangjinda M, Bertrand JK, Misztal I, Druet T. Estimation of additive and nonadditive genetic variances in Hereford, Gelbvieh, and Charolais by Method R. J Anim Sci 2001;79:2997-3001. [ Links ]
6. Eriksson S, Näsholm A, Johansson K, Philipsson J. Sex-specific genetic parameters for field recorded birth weight of beef calves. In: Proc 7th World Congr Genet Appl Livest Prod. Montpellier, France [CD-ROM]. Session 02, Breeding ruminants for meat production. 2002;(02-25). [ Links ]
7. Pico BA, Neser FWC, van Wyk JB. Genetic parameters for growth traits in South African Brahman cattle. S Afr J Anim Sci 2004;34(Suppl 2):44-46. [ Links ]
8. van Niekerk M, Neser FWC. Genetic parameters for growth traits in South African Limousin cattle. S Afr J Anim Sci 2006;36(5, Suppl 1):6-9. [ Links ]
9. Meyer K. Variance components due to direct and maternal effects for growth traits of Australian beef cattle. Livest Prod Sci 1992;31:179-204. [ Links ]
10. Robinson DL. Models which might explain negative correlations between direct and maternal genetic effects. Livest Prod Sci 1996;45:111-122. [ Links ]
11. Gutiérrez JP, Canon J, Goyache F. Estimation of direct and maternal genetic parameters for preweaning traits in the Asturiana de los Valles beef cattle breed through animal and sire models. J Anim Breed Genet 1997;114:261-266. [ Links ]
12. Ferreira GB, MacNeil MD, Van Vleck LD. Variance components and breeding values for growth traits from different statistical models. J Anim Sci 1999;77:2641-2650. [ Links ]
13. Mwansa PB, Kemp RA, Crews DH, Kastelic JP, Bailey DRC, Coulter GH. Comparison of models for genetic evaluation of scrotal circumference in crossbred bulls. J Anim Sci 2000;78:275-282. [ Links ]
14. Rump JM, Koch RM, Gregory KE, Cundiff LV, Van Vleck LD. Comparison of models for estimation of genetic parameters for mature weight of Hereford cattle. J Anim Sci 2002;80:583-590. [ Links ]
15. Knackfuss FB, Razook AG, Mercadante MEZ, Cyrillo JNSG, Figueiredo LA, Tonhati H. Seleção para peso pós-desmama em um rebanho Gir. 2. Estimativas de variâncias e parâmetros genéticos dos efeitos direto e materno para características de crescimento. R Bras Zootec 2006;35(3):726-732. [ Links ]
16. Hoque MA, Arthur PF, Hiramoto K, Gilmour AR, Oikawa T. Variance components due to direct genetic, maternal genetic and permanent environmental effect for growth and feed efficiency traits in young male Japanese Black cattle. J Anim Breed Genet 2007;124:102-107. [ Links ]
17. Al-Shorepy SA, Notter DR. Genetic variation and covariation for ewe reproduction, lamb growth, and lamb scrotal circumference in a fall-lambing sheep flock. J Anim Sci 1996;74:1490-1498. [ Links ]
18. Cloete SWP, Scholtz AJ. Environmental effects, heritability estimates and genetic trends In a Western Cape Dohne Merino nucleus flock. S Afr J Anim Sci 1998;28(3/4):185-195. [ Links ]
19. Abegaz S, van Wyk JB, Olivier JJ.Model comparisons and genetic and environmental parameter estimates of growth and the Kleiber ratio in Horro sheep. S Afr J Anim Sci 2005;35(1):30-40. [ Links ]
20. Ekiz B. Estimates of maternal effects for pre- and post-weaning daily gain in Turkish Merino lambs. Turk J Vet Anim Sci 2005;29:399-407. [ Links ]
21. Rashidi A, Mokhtari MS, Safi Jahanshahi A, Mohammad Abadi MR. Genetic parameter estimates of pre-weaning growth traits in Kermani sheep. Small Rum Res 2008;74:165-171. [ Links ]
22. Ferraz JBS, Johnson RK. Animal model estimation of genetic parameters and response to selection for litter size and weight, growth, and backfat in closed seedstock populations of Large White and Landrace swine. J Anim Sci 1993;71:850-858. [ Links ]
23. Ruíz-Flores A, Johnson RK. Direct and correlated responses to two-stage selection for ovulation rate and number of fully formed pigs at birth in swine. J Anim Sci 2001;79:2286- 2297. [ Links ]
24. Wittenburg D, Guiard V, Teuscher F, Reinsch N. Comparison of statistical models to analyse the genetic effect on within-litter variance in pigs. Animal 2008;2(11):1559-1568. [ Links ]
25. BIF. Beef Improvement Federation. Guidelines for uniform beef improvement programs. 8th ed. The University of Georgia, Athens. 2002. [ Links ]
26. Akbas Y, Brotherstone S, Hill WG. Animal model estimation of non-additive genetic parameters in dairy cattle, and their effect on heritiability estimation and breeding value prediction. J Anim Breed Genet 1993;110:105-113. [ Links ]
27. Dickerson GE. Inbreeding and heterosis in animals. In: Proceedings of animal breeding and genetics symposium in honor of Dr. Jay L. Lush. Champaign, IL, Am Soc Anim Sci 1973:54-77. [ Links ]
28. Demeke S, Neser FWC, Schoeman SJ. Variance components and genetic parameters for early growth traits in a mixed population of purebred Bos indicus and crossbred cattle. Livest Prod Sci 2003;84:11-21. [ Links ]
29. Graser HU, Smith SP, Tier B. A derivative-free approach for estimating variance components in animal models by Restricted Maximum Likelihood. J Anim Sci 1987;64:1362-1370. [ Links ]
30. Boldman KG, Kriese LA, Van Vleck LD, Van Tassell CP, Kachman SD. A manual for use of MTDFREML. A set of programs to obtain estimates of variances and covariances (DRAFT). USDA, ARS, Washington, DC. 1995. [ Links ]
31. Johnson DL, Thompson R. Restricted maximum likelihood estimation of variance components for univariate animal models using sparse matrix techniques and average information. J Dairy Sci 1995;78:449-456. [ Links ]
32. Dodenhoff J, Van Vleck LD, Kachman SD, Koch RM. Parameter estimates for direct, maternal, and grandmaternal genetic effects for birth weight and weaning weight in Hereford cattle. J Anim Sci 1998;76:2521-2527. [ Links ]
33. Willham RL. The role of maternal effects in animal breeding: III. Biometrical aspects of maternal effects in animals. J Anim Sci 1972;35:1288-1293. [ Links ]
34. SAS. SAS/STAT User's Guide (Release 8.20). Cary, NC, USA. SAS Inst. Inc. 2001. [ Links ]
35. Bahreini Behzadi MR, Shahroudi FE, Van Vleck LD. Estimates of genetic parameters for growth traits in Kermani sheep. J Anim Breed Genet 2007;124:296-301. [ Links ]
36. Ferraz Filho PB, Ramos AA, Silva LOC, Souza JC, Alencar MM. Alternative animal models to estímate heritabilities and genetic correlations between direct and maternal effects of pre and post-weaning weights of Tabapua cattle. Arch Latinoam Prod Anim 2004;12(3)119-125. [ Links ]
37. Meyer K. Estimates of covariance components for growth traits of Australian Charolais cattle. Aust J Agric Res 1993;44:1501-1508. [ Links ]
38. Grotheer VV, Röhe R, Kalm E. Entwicklung einer zuchtwertschatzung für fleischrinder in Deutschland. 2. Mitteilung: Schatzung genetischer parameter.Züchtungskunde 1997;69:349-365. [ Links ]
39. Phocas F, Laloë D. Genetic parameters for birth and weaning traits in French specialized beef cattle breeds. Livest Prod Sci 2004;89:121-128. [ Links ]
40. Fernandes HD, Ferreira GBB, Rorato PRN. Tendências e parámetros genéticos para características pré-desmama em bovinos da raça Charolês criados no Rio Grande do Sul. Rev Bras Zootec 2002;31(Suppl 1):321-330. [ Links ]
41. Donoghue KA, Bertrand JK. Investigation of genotype by country interactions for growth traits for Charolais populations in Australia, Canada, New Zealand and USA. Livest Prod Sci 2004;85:129-137. [ Links ]
42. Eriksson S, Näsholm A, Johansson K, Philipsson J. Genetic parameters for calving difficulty, stillbirth, and birth weight for Hereford and Charolais at first and later parities. J Anim Sci 2004;82:375-383. [ Links ]
43. Trus D, Wilton JW. Genetic parameters for maternal traits in beef cattle. Can J Anim Sci 1988;68:119-128. [ Links ]
44. Ríos-Utrera A. Estimadores de parámetros genéticos para características de crecimiento predestete de bovinos. Revisión. Tec Pecu Mex 2008;46(1):37-67. [ Links ]
45. Haile-Mariam M, Kassa-Mersha H. Estimates of direct and maternal (co)variance components of growth traits in Boran cattle. J Anim Breed Genet 1995;112:43-52. [ Links ]
46. Domínguez-Viveros J, Rodríguez-Almeida FA, Ortega-Gutiérrez JA, Flores-Mariñelarena A. Selección de modelos, parámetros genéticos y tendencias genéticas en las evaluaciones genéticas nacionales de bovinos Brangus y Salers. Agrociencia 2009;43:107-117. [ Links ]
47. Fouilloux MN, Renand G, Laloé D. Genetic evaluation using commercial carcass data in French beef cattle. In: Proc. 7th World Congr Genet Appl Livest Prod. Montpellier, France [CD-ROM] . Session 0 2. Breeding ruminants for meat production. 2002;(02-20). [ Links ]














