Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista mexicana de ciencias pecuarias
versão On-line ISSN 2448-6698versão impressa ISSN 2007-1124
Rev. mex. de cienc. pecuarias vol.3 no.2 Mérida Abr./Jun. 2012
Artículos
Caracterización fenotípica y molecular de poblaciones de zacate punta blanca (Digitaria californica (Benth.) Henr.)
Phenotypic and molecular characterization of populations of arizona cottontop [Digitaria californica (Benth.) Henr.]
Carlos Raúl Morales Nietoa, Alicia Melgoza Castillob, Pedro Jurado Guerraa, Martín Martínez Salvadora, Carlos Avendaño Arrazatec
a Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias. Campo Experimental Campana–Madera. Km 33.3 carretera Chihuahua–Ojinaga. México. morales.carlos@inifap.gob.mx. Correspondencia al primer autor.
b Facultad de Zootecnia y Ecología, UACH.
c Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias. C. E. Rosario Izapa, Chiapas.
Recibido el 5 de enero de 2011.
Aceptado el 10 marzo de 2011.
Resumen
Se exploró y caracterizó la diversidad fenotípica y genotípica de poblaciones del pasto punta blanca [Digitaria californica (Benth.) Henr.], en pastizales de Chihuahua. Se realizó análisis de componentes principales y conglomerados. Se observaron diferencias significativas (P<0.05) entre variables, indicando alta diversidad fenotípica y molecular entre poblaciones. Los tres primeros componentes explicaron 79 % de la variación morfológica mostrada por los 91 ecotipos. Para el CP1, las variables que mejor explicaron la variabilidad morfológica fueron densidad de tallos, rendimiento de forraje y altura de forraje, las cuales se caracterizan por agrupar variables relacionadas con producción de forraje. Al analizar la correlación del CP1 con rendimiento de forraje, se observó que a medida que la densidad de tallos se incrementó, el rendimiento de forraje se incrementó casi en la misma proporción (r= 0.94). Efecto similar ocurrió sobre rendimiento de forraje con altura de forraje (r= 0.77), altura de planta (r= 0.72) y diámetro de macollo (r= 0.72). Los marcadores moleculares presentaron 179 bandas, donde el 86.6 % presentan polimorfismo y 13.7 % monomorfismo. El número de bandas polimórficas fue de 38, 38, 38 y 40 para las combinaciones de iniciadores EcoRI–AAG + MseI–CTG, EcoRI–ACT+MseI–CTG, EcoRI–AGG + MseI–CAG y EcoRI–AAC + MseI–CAG, respectivamente. El dendograma presentó cuatro grupos. Los valores de similitud, muestran a los ecotipos PB–270 y PB–277 por un lado y PB–313 y PB–337 como los de mayor heterogeneidad genética, al presentar el valor más bajo de similitud (0.14). Se identificaron y seleccionaron los ecotipos 505 y 596 por su alto potencial forrajero de acuerdo a su variabilidad morfológica.
Palabras clave: Digitaria californica, Diversidad fenotípica, Diversidad molecular, Recursos genéticos, Variación morfológica.
Abstract
Phenotypic and genotypic diversity were explored and characterized in arizona cottontop [Digitaria californica (Benth.) Henr.] populations at Chihuahua state grasslands. Principal component and cluster analysis were applied to the data using Ward method. There were differences (P<0.05) within variables and high phenotypic and genotypic diversity were detected among populations. Three principal components explain 79 % of the variation on 91 ecotypes. For the PC1, shoots number, biomass and forage height were the variables contributed on morphological diversity. Those variables are related with forage production and regrowth. When analyzing the correlation of PC1 with forage yield, was observed that as stem density increased, forage yield was also increased at about the same proportion (r = 0.94, P<0.01). A similar effect occurred on the forage yield with forage height (r= 0.77, P<0.01), plant height (r= 0.72, P<0.01) and tiller diameter (r= 0.72, P<0.01). According with Ward method, five groups were detected. Molecular markers showed 179 bands which represents 86.3 % of the polymorphism (154 bands) and 13.7 % of the monomorphysm (25 bands). Polymorphic bands were 38, 38, 38, and 40 for EcoRI–AAG + MseI–CTG, EcoRI–ACT+MseI–CTG, EcoRI–AGG + MseI–CAG y EcoRI–AAC + MseI–CAG combination, respectively. The AFLP discriminated the 30 populations evaluated with similitud values between 0.14 and 0.55. Ecotypes PB–270 and PB–277 in one side and PB–313 and PB–337 had the highest genetic heterogeneity; they have the lowest similitude (0.14). Ecotypes with high forage potential, based on morphology and genetic variability, were identified (ecotypes 505 y 596).
Key words: Digitaria californica, Phenotypic variation, Molecular variation, Genetic resources.
INTRODUCCIÓN
La pérdida de biodiversidad es un problema presente en las tierras de pastoreo de México(1). En especies forrajeras nativas, esta pérdida adquiere mayor relevancia al desaparecer especies con características que pueden servir en programas de mejoramiento. No sólo los ecosistemas pierden su funcionalidad(2), sino que como consecuencia se reducen los servicios naturales o ambientales (ecoturismo, hábitat, agua, etc.) que se obtienen como forraje(3). Entre las especies perennes nativas del norte de México, se encuentra el pasto punta blanca [Digitaria californica (Benth.) Henr.], distribuido en pastizales amacollados, pastizales medianos y matorrales, especie apetecida por el ganado, produce altos rendimientos de forraje y responde rápidamente a la defoliación por pastoreo(4,5,6).
En la rehabilitación de pastizales es necesario utilizar especies nativas con alto rendimiento de forraje y adaptadas al medio. Un primer paso en la rehabilitación, es caracterizar y determinar la variabilidad fenotípica y genética para identificar y seleccionar ecotipos con atributos forrajeros sobresalientes(7). Las características morfológicas, citológicas o bioquímicas ligadas a los caracteres de interés, son importantes para su empleo en selección y mejoramiento genético de especies(8,9,10), y para desarrollar variedades con alta producción de forraje y calidad. Esto se logró al caracterizar la diversidad morfológica de 177 ecotipos del pasto banderita [Bouteloua curtipendula (Michx.) Torr.], utilizando descriptores como altura de forraje, altura de planta, densidad de tallos, grosor del tallo, ancho de hoja, largo de hoja, longitud de inflorescencia, ramillas por inflorescencia, espiguillas por ramillas, longitud de ramillas, raquis de inflorescencia, longitud del pedicelo y rendimiento de forraje(11). También, Peter–Schmid et al(10) evaluaron 16 características morfológicas y fisiológicas en Festuca pratensis y Lolium multiflorum. En F. pratensis hubo correlación significativa entre la diversidad de características morfológicas y localidad de sitios muestreados. Para L. multiflorum no se observó una clara separación por grupos de la variabilidad de los 38 sitios muestreados. Sin embargo, considerando que las características morfológicas son influenciadas por el ambiente, es necesario utilizar marcadores de ADN que no son afectados por el ambiente, y detectan variantes a nivel del genotipo.
El uso de marcadores moleculares como es el polimorfismo en la longitud de fragmentos amplificados (AFLP), ha sido utilizado en estudios de diversidad genética en pastos(12–15). El uso de estos marcadores, facilita una valoración rápida y eficiente de esta diversidad, comparada con otros marcadores(12,16), al generar mayor número de bandas polimórficas(17). La recolecta de poblaciones en diversas localidades es una práctica para evaluar persistencia de ciertas características deseables en plantas. Es mejor trabajar en la selección de una característica que sea heredable, en vez de múltiples características por ecotipo, porque es más fácil y rápido el proceso de mejoramiento(18). En otro estudio se concluye que la evaluación de material ex situ, en condiciones climáticas naturales y simulación de pastoreo, permite determinar ecotipos resistentes a sequia y pastoreo(19). El objetivo del presente trabajo fue explorar y caracterizar la diversidad fenotípica y genotípica de poblaciones de pasto punta blanca para producción de forraje, presentes en los pastizales del estado de Chihuahua, México.
MATERIALES Y MÉTODOS
Recolección
En 2006 se recolectaron 91 ecotipos del pasto punta blanca en diferentes ranchos ganaderos, distribuidos en todo el estado de Chihuahua, México (Cuadro 1; Figura 1). Se recolectaron cuatro plantas por ecotipo, con un diámetro de 2.5 cm, provista de raíz; la parte aérea se cortó a una altura de 15 a 20 cm y se identificó cada planta (ecotipo)(20). Los ejemplares se transportaron en hieleras, provistas de suelo comercial de jardín húmedo, y se trasplantaron en el área agronómica del Campo Experimental La Campana, del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP). Este sitio experimental cuenta con una topografía plana, clima seco templado con veranos cálidos BWk, temperatura media anual de 15 a 18 °C y precipitación promedio de 355 mm anuales. Los suelos son de origen aluvial con textura franco arenosa, pH de 6.5 y vegetación de pastizal mediano abierto(21). Posteriormente, los ecotipos se manejaron en condiciones naturales.

Caracterización fenotípica
Después del establecimiento y durante la floración del segundo año de crecimiento, se evaluaron en cada ecotipo las siguientes características: altura de forraje, desde el suelo hasta la presencia de hojas (en cm); altura de planta, desde el nivel del suelo hasta la punta de la inflorescencia (en cm); densidad de tallos; grosor de tallos, tomando al azar un tallo de la parte central (en mm); ancho de hoja, tomando una hoja al azar de la parte central (en cm); largo de hoja, tomando una hoja al azar en la parte central (en cm); longitud de inflorescencia, tomando una inflorescencia desde la base hasta la punta de la misma (en cm); diámetro del macollo, en la base al nivel del suelo (en cm); rendimiento de forraje, en materia seca. Las plantas, se cortaron a 5 a 7 cm del nivel del suelo y las muestras se pesaron y colocaron en estufa de aire forzado a 60 °C por 48 h, hasta alcanzar un peso seco constante.
Caracterización genética
Extracción de ADN y análisis AFLP: para realizar el análisis molecular, se agruparon en 30 poblaciones, las cuales representaban a todos los ecotipos muestreados. Para aislar el ADN se utilizó el método descrito por Doyle y Doyle(22). Las reacciones de AFLP se realizaron con el protocolo establecido por Vos et al (12).
Digestión: se añadieron 1.5 μl de solución amortiguadora de reacción (RL) 10X, 2 μl de templado de DNA a 50 ng/μl, 0.5 μl de enzima Eco RI (10 U/μl), 0.5 μl de enzima Mse I (10 U/μl) y se llevó la reacción hasta un volumen de 12.5 μl con agua deionizada estéril. La mezcla se centrifugó e incubó a 37 °C durante 2 h y por 15 min a 70 °C para in activar las enzimas de restricción. Se corroboró en un gel de agarosa al 1% teñido con bromuro de etidio.
Ligación de adaptadores: a la reacción de digestión se añadieron 0.3 /d de adaptador Eco RI (50 pmol), 0.3 μl de adaptador Mse I (50 pmol), 1.2 μl de ATP (10 mM, pH 7.0), 1.0 μl de solución amortiguadora de reacción (RL) 10X, 1.0 μl de T4 DNA ligasa (5 U/μl) y 6.2 μl de agua deionizada estéril, se mezcló, centrifugó e incubó por 2 h a 16 °C.
Preamplificación: se añadieron 2.5 /d de DNA digerido, ligado y diluido 1:10, 1.15 μl de Oligo Eco RI + A (50 ngμl), 1.15 μl de Oligo Mse I + A (50 ngμl), 0.5 μl de dNTPs (10 mM), 2.5 μl de solución amortiguadora de reacción de la enzima Taq DNA polimerasa, para PCR (10X), 0.65 μl de MgCl2 (50 mM), 0.2 μl de enzima Taq DNA polimerasa (5 U/μl) y 16.85 μl de agua deionizada estéril. Se mezcló, centrifugó y se puso en un termociclador a 20 ciclos a 94 °C por 30 seg, un minuto a 56 °C y un minuto a 72 °C, y mantenimiento final a 4 °C.
Amplificación selectiva: se añadieron 2.0 μl de DNA preamplificado y diluido 1:40, 4.9 μl de agua deionizada estéril, 1.1 μl de solución amortiguadora de reacción de la enzima Taq DNA polimerasa para PCR (10X), 0.3 μl de MgCl2 (50 mM), 0.5 μl de enzima Taq DNA polimerasa (5 U/μl), 1.0 μl de Oligo Mse I + 4 bases selectivas (30 ngμl), 0.2 μl de dNTPs (10 mM), 0.5 μl de Oligo Eco RI + 3 bases selectivas marcado a 700, 0.5 μl de Oligo Eco RI + 3 bases selectivas marcado a 800. Se programó el termociclador con 1 ciclo a 94 °C por 30 seg, 30 seg a 65 °C y un minuto a 72 °C. Doce ciclos en donde subsecuentemente se disminuye la temperatura de hibridación (65 °C) 0.7 °C por ciclo, mientras las otras temperaturas se mantienen igual. Seguido de 23 ciclos a 94 °C por 30 seg, 30 seg a 56 °C y un minuto a 72 °C; al final, se mantuvo la reacción a 4 °C. La electroforesis se reali zó en gel de acrilamida al 6.5 %, con urea 8M y TBE 1X (Tris 1M, ácido bórico 1M, EDTA 20 mM, pH 7.0). La separación de los fragmentos amplificados se hizo en el analizador de DNA LI–COR, cargando 0.8 μl de muestra en un pozo y utilizando el marcador de peso molecular de 50 a 700 pb. Se utilizaron oligos o primers marcados con fluorescencia a diferentes longitudes de onda (700 y 800 nm)(12).
Análisis de datos
A los datos morfológicos se les realizó un análisis de componentes principales (ACP) y un análisis de conglomerados (AC), mediante el método de Ward(23). Para obtener la grafica de dispersión se utilizó el programa PC–ORD y el MINITAB v15 y obtener el dendograma. Con el patrón de bandeo se realizó una matriz binaria de presencia y ausencia de bandas. Los datos moleculares se analizaron con el paquete estadístico NTSysPC (v 2.1). Además, se estimó la similitud genética entre los ecotipos utilizando el programa SIMQUAL, con el coeficiente Dice y como método de agrupamiento se utilizó el de Medias Aritméticas por Grupo No Ponderadas (UPGMA).
RESULTADOS Y DISCUSIÓN
Caracterización fenotípica
En general se registraron incrementos en rendimientos de forraje (g/planta), asociados a incrementos en densidad de tallo, altura de forraje, altura de planta y densidad de macollo. El análisis de componentes principales (CP) mostró que los tres primeros explican 79 % de la variación (Cuadro 2). Al obtener el coeficiente de determinación (R2) para cada una de las variables originales, respecto a los tres primeros CP, se presentaron correlaciones significativas en todas las variables evaluadas en las diversas poblaciones de pasto.
Al analizar la correlación del CP1 con las variables originales, se encontró que las variables con mayor contribución fueron: densidad de tallos (r= 0.89; P<0.01), altura de forraje (r= 0.89; P<0.01), altura de planta (r= 0.88; P<0.01) y diámetro de macollo (r= 0.78; P<0.01) (Cuadro 3). Sin embargo, cuando se analizó la correlación del CP2 con las variables originales, se encontró que las variables con mayor contribución fueron: ancho de hoja (r= 0.71; P<0.01) y grosor de tallo (r = 0.67; P<0.01). Algunas de estas correlaciones y el porcentaje de la varianza observada, coinciden de alguna manera con los obtenidos por otros trabajos(24), cuando determinaron la extensión y los patrones de distribución de la variación morfológica de 15 caracteres cuantitativos en 415 accesiones de sorgo (Sorghum bicolor (L.) Moench), al reportar que los primeros cinco CP, explicaron el 79 % de la varianza total, y que variables como altura de planta, longitud de hoja, longitud de inflorescencia, entre otras, fueron agrupadas por el CP1; sin embargo, grosor de tallo, longitud de hoja y número de hojas se agruparon en el CP2. También, estudios similares se han llevado a cabo en otros pastos del genero Lolium(25), Sorghum(26), Panicum(27), Bromus(28) y Bouteloua (29).
Al analizar la correlación del CP1 con el rendimiento de forraje, se observó que a medida que la densidad de tallos se incrementó, el rendimiento de forraje se incrementó también casi en la misma proporción (r= 0.94; P<0.01). Un efecto similar ocurrió sobre el rendimiento de forraje con altura de forraje (r= 0.77; P<0.01), altura de planta (r= 0.72; P<0.01) y diámetro de macollo (r= 0.72; P<0.01) (Cuadro 3).
La dispersión observada en la Figura 2, refleja la amplitud de la variación morfológica existente en esta especie. Al mismo tiempo, se observa que la dispersi ón no es aleatoria, ya que es posible establecer grupos con base en sus áreas de adaptación climática o entidades de origen. Con la variación encontrada, se puede establecer un banco de germoplasma en el cual se conserve y evalúe dicha variación, y permita la creación de programas de mejoramiento genético para generar ecotipos mejorados con alto potencial productivo.

Al efectuar el análisis de conglomerados jerárquicos, se obtuvieron cinco grupos basados en el método de ligamiento WARD (dendograma). A una distancia euclidiana de 10, el grupo I y II integraron a 11 y II ecotipos, respectivamente; sin embargo, el grupo III y IV agruparon 19 y 21 ecotipos, respectivamente. Además, el grupo V integró a 29 ecotipos (Figura 3). La variación morfológica observada en los ecotipos del pasto, probablemente es el resultado de la adaptación de la diversidad de factores ambientales en el área muestreada, tales como precipitación, temperatura y altitud(29).
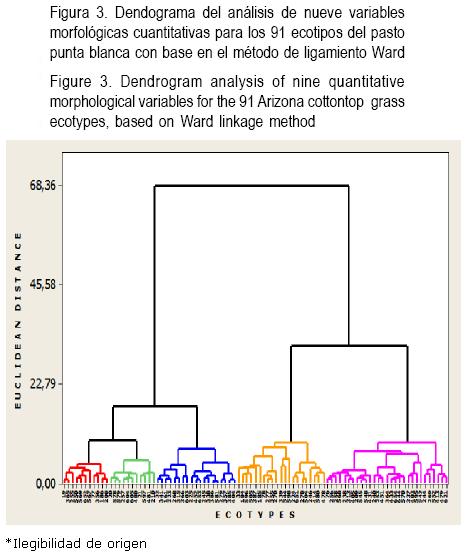
Caracterización genética
Al aplicar los cuatro pares de iniciadores AFLP, se presentaron 179 bandas totales, de las cuales el 86.3 % (154 bandas) presenta polimorfismo y el 13.7 % (25 bandas) monomorfismo (Cuadro 4). Dichos resultados son respaldados por otras investigaciones, principalmente con relación a la eficiencia del uso de esta técnica para generar gran número de polimorfismo(30).
Por otra parte, el número de bandas polimórficas fue de 38, 38, 38 y 40 para las combinaciones de iniciadores EcoRI–AAG + MseI–CTG, EcoRI–ACT + MseI–CTG, EcoRI–AGG + MseI–CAG y EcoRI–AAC+ MseI–CAG, respectivamente. Los fragmentos variaron de 38 a 40 bandas polimórficas. También, los fragmentos monomórficos en estas combinaciones fueron de 11, 5, 5 y 4 , respectivamente. El mayor porcentaje de polimorfismo (90.9 %) y bandas polimórficas (40), se obtuvo con la combinación de iniciadores EcoRI–AAC+ MseI–CAG (Cuadro 4). Además, el número total de patrones de bandas identificados (38, 38, 38 y 40), fueron muy parecidas en todas las combinaciones utilizadas, lo cual sugiere que dichas combinaciones pudieran ser empleadas para estudios de variabilidad genética en poblaciones del pasto punta blanca, utilizando marcadores de AFLP, debido a su eficiencia para detectar polimorfismos. Lo anterior refleja el potencial que representan los AFLP para realizar estudios y revelar polimorfismo en esta especie. Estos resultados concuerdan con los obtenidos en otros trabajos con ecotipos de Agrostis stolonifera, ya que obtuvieron fragmentos que variaron de 100 a 150 bandas, con 22 a 94 bandas polimórficas(31). También, en Schizachyrium scoparium reportan 854 y 653 fragmentos en tallo y semilla, pero sólo 158 bandas polimórficas, que representan 18.5 y 24.2 % para tallo y semilla y 22 a 34 bandas polimórficas(32). Lo anterior indica que existe amplia variabilidad en polimorfismo en los diferentes pastos de acuerdo a los marcadores AFLP utilizados.
La Figura 4 muestra las relaciones genéticas entre las 30 poblaciones evaluadas del pasto punta blanca, y se observa que el marcador AFLP permitió la discriminación de todas las poblaciones. Además, se encontró que los valores de similitud, obtenidos de la aplicación del coeficiente de Dice en las comparaciones pareadas de las 30 poblaciones del pasto, variaron entre 0.14 y 0.55, con un promedio global de 0. 345. Estas escalas fueron muy similares a las obtenidas en Festuca spp. (0.05 a 0.54)(33) y por Wu et al(15) en Cynodon transvaalensis (0.01 a 0.51).
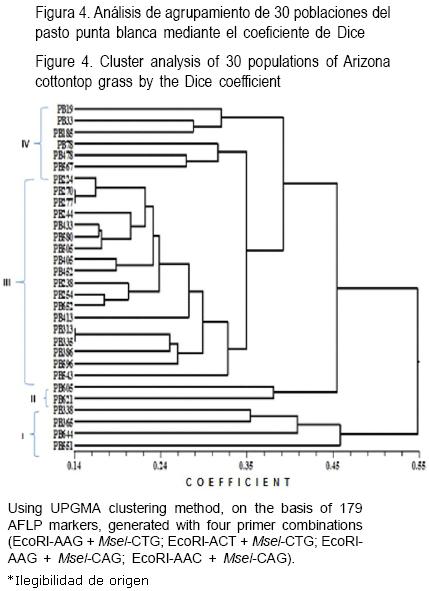
También, el dendrograma presenta cuatro grupos bien definidos (Figura 4). El grupo I incluye poblaciones recolectadas en los municipios de Buenaventura, Chihuahua y Aldama~ el grupo II incluye poblaciones que fueron recolectadas en los municipios de Ojinaga y Delicias; el Grupo III fue el más grande, ya que incluyó 18 poblaciones que se encuentran en los municipios de Janos, Sueco y Villa Ahumada; por último el grupo IV incluyo poblaciones recolectadas en los municipios de Parral, Chihuahua y Ojinaga (Cuadro 1).
Los valores de similitud obtenidos muestran a los ecotipos PB–270 y PB–277 por un lado y PB–313 y PB–337 como los de mayor heterogeneidad genética, al presentar el valor más bajo de similitud (0.14). Estas poblaciones heterogéneas con distancias genéticas bajas, son originarias de Janos y Casas Grandes por un lado y San Buenaventura, Chihuahua, respectivamente. Estas poblaciones fueron recolectadas a una altitud de 1,550 y 1, 590 m, respectivamente. Estos resultados coinciden con otros estudios respecto al uso efectivo de la técnica AFLP para estudios de diversidad(34). Además, se demostró la facilidad para realizar una valoración rápida y eficiente de la diversidad genética en estas poblaciones nativas(35).
Considerando las variables morfológicas y genéticas evaluadas, los ecotipos 505 y 596, procedentes del Rancho Los Ojos (municipio de Chihuahua) y km 111 Carretera Chihuahua–Ojinaga (municipio de Aldama) , respectivamente, fueron los más sobresalientes de acuerdo a su potencial para producción de forraje.
Los resultados demuestran que entre las poblaciones del pasto punta blanca, presentes en el estado de Chihuahua, existe amplia diversidad genética en las variables altura de forraje, densidad de tallos, grosor de tallos, tamaño de hoja, longitud de inflorescencia, diámetro del macollo y rendimiento de forraje.
CONCLUSIONES E IMPLICACIONES
El pasto punta blanca presenta amplia diversidad genética en el estado de Chihuahua, dado el alto polimorfismo detectado con la combinación de iniciadores EcoRI–AAG + Msel–CTG; EcoRI–ACT + Msel–CTG; EcoRI–AAG + Msel–CAG; EcoRI–AAC + Msel–CAG. Los ecotipos 505 y 596 son una opción para incrementar la producción animal, dado su alto potencial forrajero de acuerdo a las variables altura de forraje, densidad de tallos, grosor de tallos, tamaño de hoja, longitud de inflorescencia, diámetro del macollo y rendimiento de forraje; por lo que se dispone de riqueza genética de esta especie. Lo anterior, representa una oportunidad para iniciar programas de mejoramiento genético de estas poblaciones y establecer programas de rehabilitación de pastizales. El análisis de AFLP permitió estructurar la variabilidad genética del pasto, dado el alto polimorfismo detectado con la combinación de iniciadores EcoRI–AAC + Msel–CAG. Además, de que estos elevados niveles de variación genética, representan un recurso genético valioso para su aprovechamiento en conservación y restauración.
AGRADECIMIENTOS
Al Consejo Nacional de Ciencia y Tecnología (CONACyT) y al Fondo Mixto CONACYT–Gobierno del estado de Chihuahua por el apoyo financiero.
LITERATURA CITADA
1. Gauthier DA, Lafon A, Toombs TP, Hoth J, Wiken E. Grasslands toward a North American conservation strategy. Canadian Plains Research Center. University of Regina. Commission for Environment Cooperation. Montreal, Canadá. 2003. [ Links ]
2. Pellant M, Shaver P, Pyke DA, Herrick JE. Interpreting indicators of rangeland health. Versión 3. Tech. Ref. 1734–6. USDI, Bureau of Land Management. Denver, CO. 2005. [ Links ]
3. Báez AD, Reyes JG, Melgoza A, Royo MH, Carrillo R. Características productivas del sistema vaca–cría en el estado de Chihuahua. Téc Pecu Méx 1999;37:11–24. [ Links ]
4. Cox JR, Giner–Mendoza M, Dobrenz AK, Smith MF. Defoliation effects on resource allocation in Arizona cottontop (Digitaria californica) and Lehmann lovegrass (Eragrostis lehmanniana). Afr J Range Forestry Sci 1992;9:53–59. [ Links ]
5. Stubbendieck J, Hatch SL, Butterfield CH. North American range plants. 4th ed. University of Nebraska Press; 1992. [ Links ]
6. Ramírez G, E nríquez A, Lozano F. Valor nutricional y degradabilidad ruminal del pasto buffel y nueve pastos nativos del NE de México. Ciencia UANL 2001;4:313–321. [ Links ]
7. Morales NC, Quero AR, Pérez J, Hernández A, Le Blanc O. Caracterización morfológica de poblaciones nativas de pasto Banderita [Bouteloua curtipendula (Michx.) Torr.] en México. Agrociencia 2008;42:767–775. [ Links ]
8. Zhou Y, Kuster HK, Pettis JS, Danka RG, Gleason JM, Greenfield MD. Reaction norm variants for male calling song in populations of Achroia grisella (Lepidoptera: Pyralidae): toward a resolution of the lek paradox. Int J Org Evol 2008;62:1317–1334. [ Links ]
9. Damalas CA, Dhima VK, Eleftherohorinos GI. Morphological and physiological variation among species of the genus Echinochloa in Northern Greece. Weed Sci 2008;56:416:423. [ Links ]
10. Peter–Schmid MK, Kcolliker R, Boller B. Value of permanent grassland habitats as reservoirs of Festuca pratensis Huds. and Lolium multiflorum Lam. populations for breeding and conservation. Euphytica 2008;164:239–253. [ Links ]
11. Morales NC. Caracterización morfológica, citológica y molecular de recursos genéticos de Bouteloua cutipendula [tesis doctorado]. Montecillo, Texcoco, Estado de México: Colegio de Posgraduados; 2006. [ Links ]
12. Vos P, Hogers R, Bleeker M, Rejjans M, van de Lee T, Hornes M, Frijters A, et al. AFLP: a new technique for DNA fingerprinting. Nucleic Acids Res 1995;23:4407–4414. [ Links ]
13. Puecher DI, Robredo CG, Ríos R, Rimieri P. Genetic variability measures among Bromus catharticus Vahl. Populations and cultivars with RAPD and AFLP markers. Euphytica 2001:121(3);229–236. [ Links ]
14. Renganayaki K, Read JC, Fritz AK. Genetic diversity among Texas bluegrass genotypes (Poa arachnifera Torr.) revealed by AFLP and RAPD markers. Theor Appl Genet 2001;102:1037–1045. [ Links ]
15. Wu YQ, Taliaferro CM, Bai GH, Anderson MP. Genetic diversity of Cynodon transvaalensis Burtt–Davy and its relatedness to hexaploid C. dactylon (L.) Pers. as indicated by AFLP markers. Crop Sci 2005;45:848–853. [ Links ]
16. Kim MS, Moore PH, Zee F, Fitch M, Steiger DL, Manshardt RM, Paull RE, Drew RA, Sekioka T, Ming R. Genetic diversity of Carica papaya as revealed by AFLP markers. Genome 2002;45:503–512. [ Links ]
17. Rafalski JA, Vogel M, Morgante M, Powell W, André C, Tingey SV. Generating and using DNA markers in plants. In: Birren B, Lai E. editors. Non mammalian genomic analysis: A practical guide London: Academic Press; 1996:75–134. [ Links ]
18. Cox JR, Dobrez AK, McGuire B. Evaluation of some alkali sacaton ecotypes collected in Mexico. Applied Agric Res 1990;5:164–168. [ Links ]
19. Rivero HO. Diversidad morfológica del Bouteloua curtipendula (Michx.) Torr. y Leptochloa dubia (Kunth) Ness. en el estado de Chihuahua [tesis maestría]. Chihuahua, Chih: Universidad Autónoma de Chihuahua; 2009. [ Links ]
20. Morales NCR. Metodología para la recolecta y conservación de germoplasma de plantas forrajeras en la zonas áridas y semiáridas de México. Folleto Técnico No. 21. S.E. Campana–Madera. INIFAP–SAGARPA. 2009. [ Links ]
21. Royo MM, Lafón A. Descripción fisiográfica, diversidad vegetal y vertebrados del rancho experimental La Campana. En: Chávez A, Carrillo R. editores. Rancho Experimental La Campana 50 Años de Investigación y Trasferencia de Tecnología en Pastizales y Producción Animal. INIFAP. Chihuahua, Chih. 2008. [ Links ]
22. Doyle JJ, Doyle JL. A rapid total DNA preparation procedure for fresh plant tissue. Focus 1990;12:13–15. [ Links ]
23. Statistical Analysis System (SAS). Institute Inc. User's guide. Statistics. Version 8. Sixth edition. SAS Inc. Cary, North Carolina, USA. 1999. [ Links ]
24. Ayana A, Bekele E. Multivariate analysis of morphological variation in Sorghum [Sorghum bicolor (L.) Moench] germplasm from Ethiopia and Eritrea. Gen Res Crop Evol 1999;46:273–284. [ Links ]
25. Bennett SJ, Hayward MD, Marshall DF. Morphological differentiation in four species of the genus Lolium. Gen Res Crop Evol 2000;47:247–255. [ Links ]
26. Grenier C, Bramel PJ, Dahlberg JA, Ahmadi AE, Mahmoud M, Peterson GC, Rosenow DT, Ejeta G. Sorghums of the Sudan: analysis of regional diversity and distribution. Gen Res Crop Evol 2004;51:489–500. [ Links ]
27. Casler MD. Ecotypic variation among switchgrass populations from the Northern USA. Crop Sci 2005;45:388–398. [ Links ]
28. Ferdinandez YSN, Coulman BE. Genetic relationships among smooth bromegrass cultivars of different ecotypes detected by AFLP markers. Crop Sci 2004;44:241–247. [ Links ]
29. Morales NC, Quero AR, Melgoza A, Martínez M, Jurado P. Diversidad forrajera del pasto banderita [Bouteloua curtipendula (Michx.) Torr.], en poblaciones de zonas áridas y semiáridas de México. Téc Pecu Méx 2009;47(3):231–244. [ Links ]
30. Valdés–Infante J. Utilización de caracteres morfoagronómicos y de marcadores de ADN para el desarrollo de una metodología que contribuya al mejoramiento genético del guayabo (Psidium guajava L.) en Cuba [tesis doctorado] . La Habana, Cuba: Universidad de la Habana; 2009. [ Links ]
31. Vergara GV, Bughrara S. Genetic differentiation of tetraploid creeping bentgrass and hexaploid redtop bentgrass genotypes by AFLP and their use in turfgrass breeding. Crop Sci 2004;44:884–890. [ Links ]
32. Fu YB, Ferdinandez YSN, Phan AT, Coulman BE, Richards KW. Genetic diversity in natural populations and corresponding seed collections of little bluestem as revealed by AFLP markers. Crop Sci 2004;44:2254–2260. [ Links ]
33. Mian MA, Hopkins A, Zwonitzer J. Determination of genetic diversity in Tall Fescue with AFLP markers. Crop Sci 2002;42:944–950. [ Links ]
34. Roldán–Ruiz I, Dendauw J, Van Bockstaele E, Depicker A, De Loose M. AFLP markers reveal high polymorphism rates in ryegrass (Lolium spp.). Mol Breed 2000;6:125–134. [ Links ]
35. Hammer K. A paradigm shift in the discipline of plant genetic resourses. Gen Res Crop Evol 2003;43:337–341. [ Links ]














