Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ciencias pecuarias
versión On-line ISSN 2448-6698versión impresa ISSN 2007-1124
Rev. mex. de cienc. pecuarias vol.2 no.4 Mérida oct./dic. 2011
Notas de investigación
Inclusión del coeficiente de consanguinidad en los modelos de evaluación genética de bovinos Jersey y Suizo Americano en México
Inclusion of inbreeding coefficients into genetic evaluation models for Jersey and Brown Swiss cattle in Mexico
Agustín Ruíz-Floresa, Carlos Alberto García-Munguíaa, Rafael Núñez-Domíngueza, Rodolfo Ramírez-Valverdea, Rufino López-Ordaza, José Guadalupe García-Muñiza
a Posgrado en Producción Animal, Departamento de Zootecnia, Universidad Autónoma Chapingo, Carretera México-Texcoco km 38.5. Chapingo, Estado de México. 56230. arf@correo.chapingo.mx. Correspondencia al primer autor.
Recibido el 18 de octubre de 2010.
Aceptado el 9 de marzo de 2011.
RESUMEN
Se estudió el efecto de incluir el coeficiente de consanguinidad del animal (F) en los modelos para la evaluación genética de bovinos mexicanos Jersey y Suizo Americano de registro. El modelo sin el efecto de consanguinidad que incluyó el efecto fijo de hato-año-estación y los aleatorios genético directo y de ambiente permanente, se comparó con otro donde además se incluyó F como covariable. Las características analizadas en Jersey fueron las producciones por lactancia de leche (PL), grasa (PG) y proteína (PP), y los porcentajes de grasa (%G) y proteína (%P) en leche; mientras que en Suizo Americano sólo se analizó la PL. Los pedigríes incluyeron 21,026 animales Jersey y 101,861 Suizo Americano. El cambio más notorio correspondió a los valores genéticos predichos (VGP) para %P en Jersey; los VGP para las demás características fueron similares en ambas razas. Los porcentajes de coincidencia entre los 100 sementales superiores evaluados con ambos modelos variaron de 90 a 98 %. Los coeficientes de regresión de los VGP y de exactitudes obtenidos con el modelo sin el efecto de la consanguinidad sobre los del modelo con el efecto de consanguinidad fluctuaron alrededor de 1.0 en ambas razas. Los estimados de las varianzas genéticas y de h2 obtenidos con ambos modelos fueron similares para todas las características. No se considera necesaria la inclusión de F en los modelos para evaluar genéticamente las poblaciones estudiadas; sin embargo, se recomienda realizar nuevamente este estudio cuando el promedio de F sea mayor que el actual.
Palabras clave: Coeficiente de consanguinidad, Evaluación genética, Bovinos, Jersey, Suizo Americano.
ABSTRACT
The inclusion of the inbreeding coefficient (F) into the models to predict breeding values (BV) for Jersey (J) and Brown Swiss (BS) Mexican registered dairy cattle was studied. The conventional model including the fixed effect of herd-year-season, and the genetic additive and permanent environmental random effects was compared to an alternative model that additionally included F. Traits analyzed in J were the yields per lactation of milk (MY), fat (FY) and protein (PY), and the percentages of fat (%F) and protein (%P) in milk. In BS only MY was analyzed. The analyses were done using an animal model and the MTDFREML program; pedigree files included 21026 and 101861 animals in J and BS. Several criteria to compare results from the alternative models were used. The most notorious change was for the BV for %P in J; the BV for the other traits they were similar in both breeds. Coincidence among the top 100 sires from the two models ranged from 90 to 98 %. Regression coefficients of BV and their accuracies from the conventional model on the BV and their accuracies from the alternative model were around 1.0 in both breeds. The estimates of the genetic variance and h2 obtained with both models remained essentially the same for all traits in both breeds. The results suggest that the inclusion of F into the models to evaluate genetically Jersey and Brown Swiss purebred Mexican dairy cattle is not necessary. However, it is required to perform this evaluation when the average for F is higher than it is today.
Key words: Inbreeding coefficient, Genetic evaluation, Jersey, Brown Swiss.
En algunos estudios se ha encontrado que no incluir la covariable del coeficiente de consanguinidad del animal (F) en el modelo para la evaluación genética puede sesgar la predicción de los valores genéticos (VG)(1,2). Tohidi y Torshizi(3) recomiendan incluir F en el modelo para la evaluación genética de las producciones de leche y grasa en bovinos Holstein, ya que observaron que al ignorarlo, aumentaron las diferencias entre los valores genéticos predichos (VGP). Por el contrario, al evaluar el efecto de incluir los coeficientes de consanguinidad del animal y de la madre en los modelos para predecir los VG de características productivas y reproductivas en bovinos Piedmontese, se concluyó que no fue necesario incluir estos coeficientes(4). En bovinos lecheros se encontraron resultados similares(5,6).
La consanguinidad puede tener un efecto doble. Por una parte, influye directamente en los supuestos del modelo genético, por ejemplo, cuando se ignora F en la construcción de la inversa de la matriz de relaciones aditivas (A), la varianza de los VG de los animales consanguíneos se sobrestimará por la covarianza entre los VG de los padres, y se reducirá la varianza de muestreo mendeliano entre la progenie de animales consanguíneos. Por otra parte, la consanguinidad deprime el comportamiento fenotípico de los animales, y así probablemente los resultados de la evaluación(6). La consanguinidad también puede influir en la estimación de la varianza del error de predicción (VEP). Al respecto, al comparar dos métodos para estimar la VEP con un modelo animal, se observó un mayor sesgo conforme el promedio de consanguinidad aumentó (7).
En México la población de bovinos Jersey es relativamente pequeña; Larios-Sarabia et al(8) mencionan que 63 % del total posee algún grado de consanguinidad. El promedio de F en el total de animales fue 1.7, y 2.8 % en los consanguíneos. En Suizo Americano se identificaron 25,027 animales consanguíneos(9), 25 % del total; el promedio de F fue 1.5 ± 4.5 en la población completa y 5.8 ± 7.5 % en la consanguínea.
Las poblaciones mexicanas de ganado Jersey y Suizo Americano se evalúan genéticamente desde 2004(10), y el coeficiente de consanguinidad no se incluye en los modelos utilizados. El objetivo de este estudio fue evaluar el efecto de la inclusión del coeficiente de consanguinidad en los modelos para evaluar genéticamente las producciones por lactancia de leche, grasa y proteína; y porcentajes de grasa y proteína en leche de bovinos Jersey, y producción de leche por lactancia en Suizo Americano.
La información analizada de bovinos Jersey fue parte de la base de datos productivos y genealógicos de la Asociación Mexicana de Criadores de Ganado Jersey de Registro, A.C; los registros de comportamiento productivo corresponden a registros de producción de lactancias ocurridas entre 1998 y 2008 en ranchos localizados en siete estados del país. La información de Suizo Americano fue facilitada por la Asociación Mexicana de Criadores de Ganado Suizo de Registro, A. C; este ganado está ubicado en 33 ranchos, localizados en 12 Estados de México.
Las variables analizadas en Jersey fueron las producciones por lactancia ajustada a 305 d de leche (PL), grasa (PG) y proteína (PP), y los porcentajes de grasa (%G) y proteína de la leche (%P). Para Suizo Americano sólo se analizó la PL ajustada a 210 d ya equivalente maduro. Los pedigríes incluyeron 21,026 animales Jersey y 101,861 Suizos. En la edición de la información, los registros que estuvieron tres desviaciones estándar por arriba o debajo de la media fueron eliminados. Los grupos contemporáneos se formaron al combinar los efectos de hato, año y estación; la conectividad genética entre hatos fue determinada con el programa AMC(11). En el Cuadro 1 se presentan estadísticos descriptivos de los datos analizados.
Los modelos utilizados para evaluar el efecto de la inclusión del coeficiente de consanguinidad fueron: el modelo sin el efecto de consanguinidad que incluyó el efecto fijo de grupo contemporáneo, y los efectos aleatorios genético aditivo directo, de ambiente permanente de la vaca y residual; y el modelo con el efecto de la consanguinidad que adicionalmente incluyó el efecto fijo de la covariable coeficiente de consanguinidad del animal (F). La representación matricial de los modelos es:
y = Xb + Za + Wpe + e
y = X(F)b+ Za + Wpe + e
donde, y es el vector de registros de las variables analizadas; b, a, pe y e son los vectores de efectos fijos; de efectos aleatorios de valores genéticos aditivos directos, ambiente permanente de la vaca y residuales, respectivamente; y X, Z y W son matrices de incidencia que asocian los vectores correspondientes con y; X(F) es la matriz X con una columna adicional para el coeficiente de consanguinidad del animal. Suponiendo normalidad, las esperanzas y varianzas son:

donde, A es la matriz de relaciones genéticas aditivas; I es una matriz identidad; V es el número de vacas; N es el número de animales con registro, y s2a, s2pe, y s2e son las varianzas genética aditiva, de ambiente permanente, y residual. Los análisis para estimar los coeficientes de consanguinidad y las varianzas, y predecir los valores genéticos y sus exactitudes, se realizaron con el programa MTDFREML(12).
Los criterios estadísticos(13) para decidir la inclusión de F en los modelos para la evaluación genética fueron: 1) los estimados de las correlaciones producto-momento de Pearson y de rango de Spearman, entre los VGP y entre las exactitudes obtenidas con ambos modelos; 2) el porcentaje de coincidencia para los 100 sementales con los mayores VGP con los modelos en comparación; 3) los estimados de los coeficientes de regresión lineal de los VGP y sus exactitudes obtenidas con el modelo sin el efecto de consanguinidad sobre los obtenidos con el modelo que lo incluyó; 4) los estimados de las varianzas de los efectos genético aditivo directo, de ambiente permanente y residual, así como las heredabilidades obtenidas con ambos modelos. Adicionalmente, se estimaron los rangos de los VGP para las poblaciones completa y consanguínea, y el efecto de la consanguinidad en las variables estudiadas se cuantificó con el estimado del coeficiente de regresión lineal para cada una de las variables evaluadas sobre F.
En el Cuadro 2 se muestra la distribución de ambas razas por niveles de F. Las proporciones de animales consanguíneos fueron 48.6 y 29.5 %, para Jersey y Suizo. La proporción de animales con F mayor que 10 % fue 0.98 % en Jersey y 4.24 % en Suizo Los promedios de F fueron 1.4 ± 2.4 y 1.2 ± 2.2 % para el total de animales y 2.8 ± 2.9 y 5.1 ± 7.3 % para los consanguíneos en Jersey y Suizo. Las proporciones de animales consanguíneos estimadas en este estudio son menores que el 96.0 % observado en bovinos Holstein del Reino Unido en 2004(14). En otro estudio realizado en EE.UU. con bovinos Holstein, se encontró que 92.8 % de los animales fueron consanguíneos(5). En bovinos Jersey mexicanos de registro nacidos entre 2001 y 2007, se estimaron promedios de F de 1.9 ± 2.6 y 2.7 ± 2.7 para las poblaciones completa y consanguínea(8). En forma similar, en bovinos Suizo Americano de registro se obtuvieron promedios para F de 1.48 ± 4.50 y 5.80 ± 7.50 en las poblaciones completa y consanguínea(9).
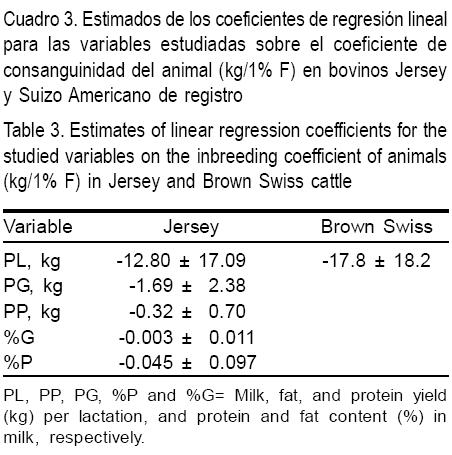
En el Cuadro 3 se muestran los estimados de los coeficientes de regresión para las variables estudiadas por cada 1.0 % de aumento en F. En Jersey hubo una disminución por lactancia de 12.8 ± 17.1 kg de leche, 1.7 ± 2.4 kg de grasa y 0.3 ± 0.7 kg de proteína; para %G y %P también hubo reducciones aunque proporcionalmente de menor magnitud. En bovinos Suizos la reducción fue de 17.8 ± 18.2 kg en PL. Los estimados de este estudio se encuentran dentro de la escala reportada en la literatura, aunque con errores estándar altos, posiblemente debido al número reducido de registros analizados y a los bajos niveles de consanguinidad en las poblaciones estudiadas. En bovinos Holstein se encontró que cada aumento de 1.0 % en F del animal se asoció con una disminución de 27 kg, 0.9 kg y 0.8 kg en las producciones a equivalente maduro en vacas de primera lactancia para leche, grasa y proteína(15). En otro estudio, Wiggans et al(5) observaron en bovinos Holstein reducciones de 29.6 kg de leche, 1.08 kg de grasa y 0.97 kg de proteína por lactancia, por cada 1 % de aumento en F. En bovinos Ayrshire, Guernsey, Holstein, Jersey y Pardo Suizo de primera lactancia, se observó que por cada 1.0 % que aumentó F, se redujeron de 14.8 a 39.5 kg y de 0.78 a 1.36 kg las producciones por lactancia de leche y grasa(16).
Los estimados de las correlaciones producto momento de Pearson y de rango de Spearman entre los VGP y entre las exactitudes fueron positivos y cercanos a la unidad, lo que significa que los VGP con ambos modelos fueron similares. Cuando F se incluyó en el modelo, el cambio más notorio fue en los VGP para %P y PG en Jersey (0.97 y 0.98), mientras que para las demás características los estimados de las correlaciones (>0.99) sugieren que los VGP cambiaron poco en ambas razas.
Los resultados del presente estudio coinciden con los obtenidos en bovinos Holstein por Tohidi y Torshizi(3), quienes estimaron coeficientes de las correlaciones de Pearson y de rango de Spearman mayores que 0.997 entre los VGP para PL y PG, lo cual sugiere que la inclusión de F en los modelos no influyó en la predicción de los VG. Estos autores atribuyeron este resultado al bajo promedio de F en la población estudiada (0.18 %) y a la alta proporción de animales no consanguíneos (91 %); también señalaron que las diferencias en los VGP promedio para PL y PG obtenidos con los modelos alternativos, incrementaron conforme F aumentó. Los resultados obtenidos en el presente estudio son similares a los observados por Varona et al (2), quienes compararon un modelo de dominancia con otro que excluyó F, los estimados de las correlaciones entre los VGP fueron superiores a 0.999.
Los porcentajes de coincidencia para las variables estudiadas variaron de 90 a 98 % en ambas razas, lo que es consistente con los valores de correlación entre los VGP. Esto implica que se esperarían cambios pequeños en la jerarquización de los VGP de los sementales sobresalientes al utilizar uno u otro modelo. Estos resultados concuerdan con los obtenidos en bovinos lecheros de Islandia(1), donde no se observó un efecto importante de la inclusión de F en la jerarquización, dentro de año, de los animales evaluados; los promedios de las correlaciones de rango dentro de año de nacimiento fueron 0.997, 0.998 y 0.996 para la primera, segunda y tercera lactancias.
Los estimados de los coeficientes de regresión para los VGP y las exactitudes obtenidas con el modelo sin el efecto de la consanguinidad sobre los del modelo que lo incluyó, variaron de 0.96 a 1.10 para VG, y entre 0.97 y 1.01 para exactitudes en Jersey; una tendencia similar fue observada en Suizo (Cuadro 4). Estos resultados eran de esperarse dada la correlación alta entre los VG predichos con ambos modelos; e indican, en ambas razas, que los valores genéticos obtenidos con un modelo pueden predecir con un alto nivel de confiabilidad los VG predichos con el otro modelo.

En el Cuadro 5 se muestran los estimados de heredabilidad (h2) y de componentes de varianza obtenidos con los modelos con y sin el efecto de consanguinidad. En bovinos Jersey los valores de los estimados de la varianza genética aditiva obtenidos con el modelo sin F fueron ligeramente menores para PL y PG, mayores para PP, e iguales para %G y %P, en comparación con los del modelo con F; los estimados de la varianza residual para todas las características fueron similares entre modelos. En Jersey los cambios en los estimados de las varianzas de ambiente permanente para PP fueron de magnitud pequeña; para PL fue menor y para PG fue mayor con el modelo sin F en comparación con los del modelo con F, mientras que para %G y %P no hubo cambios. En Jersey los cambios más notorios en los estimados de la varianza fenotípica de un modelo a otro fueron observados para PL y PG, manteniéndose muy similares para PP, %G y %P. Los estimados de h2 y la proporción de varianza de ambiente permanente sobre la varianza fenotípica se mantuvieron iguales para todas las características en ambas razas. En Suizo, los estimados de todos los componentes de varianza con ambos modelos fueron similares.
En bovinos Hereford, al comparar 12 modelos que difirieron en los efectos incluidos, se observó que los estimados de varianza y h2 fueron similares, independientemente si F fue incluido o no en el modelo(17). También en bovinos Piedmontese se encontró que las diferencias en los estimados de h2 variaron de 0.0 a 0.01, para edad a la primera inseminación, edad al primer parto, peso a los 120 días, peso al año en hembras y peso al año en machos(4).
En general, cuando se consideró la progenie de padres consanguíneos el rango para los VGP disminuyó 7.8 % en Jersey, para Suizo no hubo cambios. Los resultados para Jersey son contrarios a los observados en otras investigaciones. En un estudio realizado para evaluar el efecto de F en la predicción de los VG en bovinos Braunvieh, se observó que en la población completa, sólo 0.8 % de los VGP para PL difirieron más de 5.5 kg, y cuando se hizo la evaluación con F de los padres mayor que 10 %, 66.4 % de los VGP difirieron en más de 5.5 kg(6). Estos autores sugieren que en las evaluaciones genéticas, la progenie de padres consanguíneos pudiera tener VGP sobreestimados con relación a la población completa.
En el presente estudio, la comparación de los diferentes criterios utilizados para determinar la inclusión de F en los modelos para la evaluación genética de las diferentes características analizadas en ambas razas, sugiere que no es necesario incluir F en el modelo, probablemente debido a que los niveles de consanguinidad observados son bajos en las poblaciones de Jersey y Suizo Americano de México. A esta misma conclusión arribaron otros(17), quienes no observaron cambios en los estimados de parámetros genéticos y componentes de varianza obtenidos con los modelos con o sin F en bovinos Hereford. Conclusiones similares se obtuvieron en bovinos Piedmontese(4) y en bovinos lecheros de Islandia(1). Por el contrario, Tohidi y Torshizi(3) recomendaron incluir F en el modelo para la evaluación genética de las producciones de leche y grasa en ganado Holstein; y argumentan que ignorar F en el modelo, incrementa las diferencias entre los VGP para las variables estudiadas y que F está aumentando en la población Holstein que estudiaron.
En resumen, actualmente no es necesaria la inclusión de F en los modelos para la evaluación genética de producción de leche y sus componentes en bovinos Jersey y de producción de leche en Suizo Americano. Sin embargo, se recomienda evaluar nuevamente la inclusión de F en los modelos para la evaluación genética, cuando la consanguinidad en estas poblaciones se incremente sustancialmente.
AGRADECIMIENTOS
Se agradece a la Asociación Mexicana de Criadores de Ganado Suizo de Registro y a la Asociación Angus Mexicana A. C. por facilitar la información de sus bases de datos genealógicos y productivos para el presente estudio; al Consejo Nacional de Ciencia y Tecnología, y al Consejo Mexiquense de Ciencia y Tecnología, por el financiamiento otorgado para los estudios de maestría del segundo autor.
LITERATURA CITADA
1. Sigurdsson A, Jonmundsson JV. Inbreeding and its impact in the closed population of Icelandic dairy cattle. Acta Agric Scand 1995;45:11-16. [ Links ]
2. Varona L, Misztal I, Bertrand JK, Lawlor TJ. Effect of full sibs on additive breeding values under the dominance model for stature in United States Holsteins. J Dairy Sci 1998;81:1126-1135. [ Links ]
3. Tohidi R, Torshizi RV. Effect of inbreeding on breeding values for milk and fat yields in Iran Holstein. Proc World Congress on Genetics Applied to Livestock Production. Belo Horizonte, Brazil. 2006. [ Links ]
4. Fioretti M, Rosati A, Pieramati C, Van Vleck LD. Effect of including inbreeding coefficients for animal and dam on estimates of genetic parameters and prediction of breeding values for reproductive and growth traits of Piedmontese cattle. Livest Prod Sci 2002;74:137-145. [ Links ]
5. Wiggans GR, VanRaden PM, Zuurbier J. Calculation and use of inbreeding coefficients for genetic evaluation of United States dairy cattle. J Dairy Sci 1995;78:1584-1590. [ Links ]
6. Casanova L, Hagger C, Kuenzi N, Schneeberger M. Inbreeding in Swiss Braunvieh and its influence on breeding values predicted from a repeatability animal model. J Dairy Sci 1992;75:1119-1126. [ Links ]
7. Boichard D, Lee AJ. Approximate accuracy of genetic evaluation under a single-trait animal model. J Dairy Sci 1992;75:868-877. [ Links ]
8. Larios-Sarabia N, Ramírez-Valverde R, Núñez-Domínguez R, García-Muñiz JG, Ruíz-Flores A. Variabilidad genética en el hato bovino Jersey de registro en México y su potencial de mejoramiento. Rev Mex Cienc Pecu [En prensa] [ Links ].
9. Reséndiz HM. Niveles y tendencias de consanguinidad en bovinos Suizo Americano de registro en México. [Tesis licenciatura]. Chapingo, Méx: Universidad Autónoma Chapingo; 2007. [ Links ]
10. Núñez DR, Ramírez VR, Ruíz FA, Domínguez VJ. Resumen de evaluaciones genéticas para ganado Jersey 2004. Boletín Técnico. Universidad Autónoma Chapingo, México. 2004. [ Links ]
11. Roso VM, Schenkel FS. AMC-A computer program to assess the degree of connectedness among contemporary groups. Proc World Congress on Genetics Applied to Livestock Production, Belo Horizonte, Brazil. Poster 27-26. 2006. [ Links ]
12. Boldman KG, Kriese LA, Van Vleck LD, Van Tassell CP, Kachman SD. A manual for use of MTDFREML. A set of programs to obtain estimates of variances and covariances (DRAFT). USDA, ARS. 1995. [ Links ]
13. SAS. User s Guide: Statistics. Version 9.1.3 Edition. SAS. Institute Inc., Cary, NC, USA. 2002. [ Links ]
14. Kearney JF, Wall E, Villanueva B, Coffey MP. Inbreeding trends and application of optimized selection in the UK Holstein population. J Dairy Sci 2004;87:3503-3509. [ Links ]
15. Smith LA, Cassell BG, Pearson RE. The effects of inbreeding on the lifetime performance of dairy cattle. J Dairy Sci 1998;81:2729-2737. [ Links ]
16. Hudson GFS, Van Vleck LD. Inbreeding of artificially bred dairy cattle in the northeastern United States. J Dairy Sci 1984;67:161-170. [ Links ]
17. Ferreira G B, MacNeil MD, Van Vleck LD. Variance components and breeding values for growth traits from different statistical models. J Animal Sci 1999;77:2641-2650. [ Links ]














