Introducción
México es el centro de origen de la nochebuena (Euphorbia pulcherrima, Willd ex Klotzsch) también conocida como Pascua, Flor de navidad, Estrella de navidad o Poinsettia, es originaria de la región que va desde el norte del estado de Morelos, hasta el municipio de Taxco, Guerrero (Martínez, 2001; Steinmann, 2002). La nochebuena en México es una planta ornamental de temporada, para las fiestas de navidad, que contribuye al ingreso económico del país; anualmente se producen 25 millones de plantas terminadas, con un valor de producción de 500 millones de pesos, además genera empleos directos e indirectos (Parra-Quijano et al., 2012).
Esta especie se encuentra ampliamente distribuida por las zonas tropicales de México (Lee, 2000), desde el nivel del mar hasta los 2 000 m de altitud, alcanzando su diversidad y abundancia en los bosques tropicales caducifolios; esta especie se distribuye desde el litoral del Océano Pacifico hasta Guatemala, en todo el Golfo y centro de México (Steinmann, 2002). E. pulcherrima, es una planta arbustiva que alcanza una altura cercana a los 5 m, presenta flores femeninas simples, sin pétalos ni sépalos, rodeado por flores masculinas que están encerrados en una estructura denominada ciato, cada flor forma una glándula que atrae a los polinizadores (Ecke et al., 2004; Taylor et al., 2011; Trejo et al., 2012).
La hibridación es uno de los métodos de mejoramiento genético que aprovecha la generación F1 proveniente del cruzamiento entre dos poblaciones parentales P1 y P2 (Márquez, 1985). Por lo general, en un cruzamiento entre dos progenitores se hereda material genético de ambos; es decir, el híbrido combina caracteres de ambos progenitores (Bai y Lindhout, 2007). La hibridación ofrece ventajas únicas en las plantas de propagación vegetativa, en las que una recombinación puede fijarse fácilmente (León, 2000). Después de la hibridación es necesaria la identificación o detección de similitudes o diferencias de un cruzamiento (Watson y Eyzaguirre, 2002).
La identificación tradicional de las plantas se hace por caracterización fenotípica, procedimiento lento y limitado porque la expresión de los caracteres cuantitativos está sujeta a fuerte influencia ambiental. Los marcadores moleculares permiten identificar, clasificar y aprovechar la diversidad genética existente en los genomas de las plantas, ya que las diferencias o similitudes a nivel del ADN entre individuos se observan en forma directa, así como la expresión influenciada tanto por la relación dominante-recesiva como por interacción epistático-pleiotrópica (Gutiérrez-Díez et al., 2009; Idrees y Irshad, 2014).
Hoy en día gracias a los avances en la biología molecular los marcadores moleculares se han convertido en una herramienta para la investigación agropecuaria en el mejoramiento genético de plantas, como mapeo genético, detectar genes mutantes, mejoramiento de cultivos asistidos por marcadores, historia de la población, epidemiología, seguridad alimentaria, población de estudios, pruebas de paternidad e identificación de cultivares con ello se clasifica y analiza la variabilidad genética de un material vegetal para así aprovechar la diversidad existente en los genomas de las plantas (Hartl y Jones 2005; Azofeifa-Delgado, 2006; Gutiérrez-Díez et al., 2009).
Zane et al. (2002) indicaron que la amplificación aleatoria de ADN polimórfico (RAPD), secuencias simples repetidas (SSR) y polimorfismos en la longitud de fragmentos amplificados (AFLP) son herramientas fundamentales para estudiar la variabilidad genética. El estudio de parámetros genéticos como la caracterización molecular se puede realizar para la identificación de híbridos, para determinar las relaciones filogenéticas y la diversidad a nivel genotipo y con ello registrar nuevas variedades ornamentales de rasgos deseables sobresalientes comerciales. Por lo anterior, el objetivo de la presente investigación fue identificar híbridos F1 de E. pulcherrima mediante el uso de marcadores moleculares RAPD.
Materiales y métodos
Material vegetal
En un estudio previo Rodríguez et al. (2016) se realizaron cruzas directas donde los progenitores hembra fueron tres nochebuenas de sol (Amanecer navideño, Belén y Juan Pablo) y los progenitores macho fueron tres nochebuenas de sombra (Festival red, Ice Punch y Burgundy). De la progenie obtenida se seleccionaron como objeto de estudio 39 plantas F1 por sus características morfológicas diferentes y sobresalientes a sus progenitores macho y progenitor hembra, como color de la bráctea, forma de a bráctea y porte de la planta. Se tomaron muestras de 200 mg de hojas jóvenes de cada una de las 39 plantas, así como también se tomaron hojas de los seis progenitores, se pesaron, etiquetaron y congelaron a -20 °C hasta su extracción de ADN.
Extracción y calidad de ADN
Para la extracción de ADN se utilizó el método descrito por Andrade et al. (2005) el cual se basa en CTAB. La integridad del ADN se estimó mediante electroforesis en gel de agarosa (ultrapura GIBCO BRL) al 1%, el gel fue teñido con 1 (g ml-1 de bromuro de etidio (Invitrogen®) y posteriormente se observó en un transiluminador de luz ultravioleta con foto documentador (Syngene® GVM20). Se determinó la pureza de las muestras cuantificando la cantidad de ADN con un espectrofotómetro nanodrop 2000 (Thermo scientific®). El ADN se diluyó a una concentración de 20 ng µl-1 para ser utilizado en las reacciones de PCR.
Amplificación de fragmentos
En un estudio previo Rodríguez et al. (2016) se realizó la amplificación de ADN con 68 iniciadores de secuencia aleatoria de diez nucleótidos del kit OPA, OPB, OPC, más ocho iniciadores, de los cuales se eligieron 10 con base en la cantidad y brillo de los fragmentos RAPD amplificados (OPA-07, 5’ GAA ACG GGT G 3’; OPA-11, 5’ CAA TCG CCG T 3’; OPA-12, 5’ TCG GCG ATA G 3’; OPB-07, 5’ GGT GAC GCA G 3’; OPB-08, 5’ GTC CAC ACG G 3’; OPB-09, 5’ TGG GGG ACT C 3’; OPB-11, 5’ GTA GAC CCG T 3’; OPB-20, 5’ GGA CCC TTA C 3’; OPC-06, 5’ GAA CGG ACT C 3’; OPC-13, 5’ AAG CCT CGT C 3’. Los componentes de reacción fue la siguiente: 10 μl de dNTPs (5 μM de cada dNTP, GIBCO BRL), 2.5 μl de amortiguador PCR (10x), 1.5 μl MgCl2 (3 mM), 2 μl iniciador (10 pmol μl-1), 0.3 μl (1.5 U) de taq DNA polimerasa nativa (Invitrogen), 4 μl ADN (20 ng μl-1) ajustando a 25 μl con 4.7 μl agua desionizada esterilizada.
Se llevó a cabo la amplificación del ADN mediante marcadores RAPD los cuales se basan en la técnica de la reacción en cadena de la polimerasa (PCR). La separación de fragmentos amplificados se hizo por electroforesis en gel de agarosa (Invitrogen®) al 1.5% (w/v) teñidos con bromuro de etidio (10 μg L-1). La electroforesis se realizó a 75 V por 4 h con TAE 1X. Se realizó la documentación de los geles de la misma manera que en la calidad del ADN.
Análisis de datos
El tamaño de los fragmentos de ADN que fueron generados por los iniciadores RAPD se obtuvo utilizando el programa Gene tools Labworks 4®. Se realizaron los perfiles electroforéticos generados por los progenitores y los híbridos de cada variedad de nochebuena. Se realizó la construcción del dendrograma, para obtener el coeficiente de similitud entre progenie y progenitores, los datos se analizaron con base en la presencia (indicada por 1) o la ausencia (indicada por 0) de fragmentos amplificados con los iniciadores decámeros seleccionados. Los datos se procesaron utilizando el sistema de análisis multivariado y taxonómico numérico (NTSYSpc 2.1). Los análisis de agrupamiento se realizaron sobre la relación de matrices con el método de ligamiento promedio aritmético de grupos de pares no ponderados (siglas en inglés, UPGMA) (Avise, 1994).
Resultados y discusión
Identificación de progenie de Juan Pablo x Ice punch y Juan Pablo x Festival red
Con los iniciadores utilizados para la amplificación de ADN de las cruzas de Juan Pablo con Ice Punch y Festival Red, se obtuvieron bandas monomórficas y polimórficas, en total se obtuvieron 149 fragmentos amplificados de los cuales el 13% fueron polimórficos. El iniciador OPB-11 amplificó el mayor número de bandas de ADN con 23 fragmentos amplificados y sólo 9% fueron polimórficos (Cuadro 1), el iniciador que obtuvo mayor porcentaje de polimorfismo fue el OPB-09, con 63%. El porcentaje de polimorfismo varió de 9 hasta 63%.
Cuadro 1 Fragmentos de ADN totales, polimórficos y porcentaje de polimorfismo para la identificación de plantas híbridas similares al progenitor macho de Juan Pablo x Ice Punch mediante RAPD.
| Iniciador | Fragmentos amplificados | Fragmentos polimórficos | Polimorfismo (%) | Fragmentos marcadores (pb)/plantas identificadas por familia |
| OPA-07 | 19 | 5 | 27 | IP (1971 pb/2, 3, 4, 7 y 8), IP (1391 pb/2, 3 y 4), IP (1101/1, 2, 3, 4 y 8), IP (1080/2) y IP (1 y 2) |
| OPA-11 | 18 | 3 | 17 | IP (2135 pb/2, 3 y 5) y IP (941 pb/3) |
| OPA-12 | 16 | 3 | 19 | IP (909 pb/ 6 y 7) |
| OPB-07 | 18 | 3 | 17 | IP (1924 pb/4) |
| OPB-08 | 8 | 3 | 38 | IP (749 pb/6) (666 pb/6 y 7) |
| OPB-09 | 8 | 5 | 63 | - |
| OPB-11 | 23 | 1 | 4 | IP (914 pb/3, 4 y 5) |
| OPB-20 | 14 | 3 | 21 | IP (1267 pb/1) |
| OPC-06 | 9 | 1 | 11 | IP (1979 pb/1, 2, 3 y 4) |
| OPC-13 | 16 | 2 | 13 | - |
El tamaño de las bandas que identificaron el origen híbrido de las plantas F1 varió de 446 pb a 2526 pb. Los iniciadores que permitieron identificar el origen híbrido de las plantas F1 de la cruza Juan Pablo x Ice punch, fueron los que se muestran en el Cuadro 2, siendo el iniciador OPA-07 el que más identificó plantas provenientes de la cruza, y las plantas que pudieron identificarse como híbridos con otros iniciadores fueron las plantas 2 JP x Ip, 3 JP x Ip, 4 JP x Ip, 5 JP x Ip, 6 JP x Ip, 7 JP x Ip y 8 JP x Ip. Esta condición de similitud de bandas con el progenitor macho permitió corroborar que se obtuvo la hibridación entre el progenitor hembra Juan Pablo con el progenitor macho Ice Punch.
Cuadro 2 Fragmentos de ADN totales, polimórficos y porcentaje de polimorfismo para la identificación de plantas híbridas de nochebuena de las cruzas del progenitor belén mediante RAPD.
| Iniciador | Fragmentos amplificados | Fragmentos polimórficos | Polimorfismo (%) | Fragmentos marcadores (pb) |
| OPA-07 | 14 | 3 | 16 | - |
| OPA-11 | 16 | 4 | 25 | 2234 pb, 1894 pb, 1220 pb y 887 pb |
| OPA-12 | 9 | 2 | 22 | 1619 pb, y 536 pb |
| OPB-07 | 10 | 1 | 10 | - |
| OPB-08 | 8 | 2 | 25 | 1035 pb |
| OPB-09 | 9 | 3 | 33 | 1382 pb y 1299 pb |
| OPB-11 | 7 | 2 | 28 | 1569 pb |
| OPB-20 | 13 | 3 | 23 | - |
| OPC-06 | 13 | 5 | 38 | - |
| OPC-13 | 14 | 5 | 36 | 609 pb |
El análisis del conglomerado obtenido (Figura 1), muestra que a un nivel de 0.26 de distancia genética se forman 6 agrupaciones: 1) Juan Pablo y Festival Red, 2) Ice Punch, 3) 2 JP x Ip, 4 JP x Ip, 3 JP x Ip, 6 JP x Ip, 7 JP x Ip, 8 JP x Ip, 1 JP x Fr y 2 JP x Fr, 4) 3 JP x Fr, 5) 3 JP x Ip y 6) 1 JP x Ip. El grupo que concentró más plantas F1 fue el grupo 3. Los híbridos más similares entre sí (con un coeficiente de 0.15) son 1 JP x Fr y 2 JP x Fr estas son las que menor distancia genética presentan. En contraste, la planta que no comparte similitud genética es el progenitor macho Ice Punch con ningún progenitor ni con la progenie (Figura 1).
Lo anterior pudiera explicar por qué en el cruzamiento de Juan Pablo x Festival red se logró menos progenie (3), ya que al compartir mayor similitud genética hay menor posibilidad de que se realice la fecundación por la cercanía genética. Al respecto Jiménez-Durán y Cruz-García (2011) mencionan que el fuerte control genético que ejercen los sistemas de incompatibilidad sexual restringe grandemente la introgresión con los padres y las cruzas con otros miembros de la progenie.

Figura 1 Relación de las cruzas generadas en nochebuena, por Juan Pablo x Ice punch (8 plantas) y Juan Pablo x Festival red (3 plantas) según lo determinado por el análisis de agrupamiento UPGMA tomando como referencia 149 fragmentos RAPD.
Caso contrario el parental femenino Ice punch se encuentra en un cuadrante separado de su progenie y de los otros progenitores, mostrado una distancia más larga de separación entre ellos, debido a que Ice punch es producto de una cruza inter especifica entre otras nochebuenas y hay mayor distancia genética. La distribución obtenida en las cruzas generadas por Juan Pablo x Ice punch y Juan Pablo x Festival red mediante una distribución gráfica bidimensional, dio lugar a cuatro grupos más detallados: 1) el progenitor macho Ice punch con las cruzas de Juan Pablo x Ice punch 2, 3, 4, 6, 7 y 8; 2) el progenitor hembra Juan Pablo con la cruza 5 de Juan Pablo x Ice punch; 3) el progenitor macho Festival red con las cruzas 1 y 2 de Juan Pablo x Festival red y 4) las cruzas 1 de Juan Pablo x Ice punch y 3 de Juan Pablo x Festival red (Figura 2).

Figura2. Distribución gráfica de dos progenitores macho Ice punch y Festival red, un progenitor hembra Juan Pablo, ocho plantas generadas en la cruza de Juan Pablo x Ice punch y tres plantas generadas en la cruza de Juan Pablo x Festival red.
Los marcadores RAPD no lograron identificar a la progenie proveniente del progenitor macho Festival red, lo cual puede ser debido a que la hibridación no se efectuó, entre Juan Pablo con Festival red, a pesar de que se cuidó mucho el proceso de hibridación para que no fueran fecundadas por otro polen de otras plantas o autofecundadas, existe la posibilidad de que las plantas sean producto de autofecundación y que por eso se agruparon juntos en el grupo III, contrario con el grupo I en donde se agrupó el progenitor masculino de Ice punch con su progenie de Juan Pablo x Ice punch, la uniformidad del comportamiento de este grupo y la identificación con marcadores RAPD demuestra que se efectuó la hibridación, evidenciando la identidad genética de las seis progenies estudiadas.
Identificación de progenie de Amanecer navideño x Ice punch y Amanecer navideño x Festival red
La amplificación de iniciadores RAPDs utilizados en el estudio de las cruzas obtenidas de Amanecer navideño x Ice punch y Amanecer navideño x Festival red reveló la presencia de 146 fragmentos con un porcentaje de polimorfismo de 34% (Cuadro 3). El iniciador OPB-07 y OPB-20 fueron los que mayor amplificación de fragmentos generaron (20) con polimorfismo de 35% y 50% respectivamente, aunque el iniciador OPB-11 generó menor cantidad de fragmentos, tuvo 56% de polimorfismo, por lo que el porcentaje de polimorfismo varió desde 27 hasta 56%.
Cuadro 3 Fragmentos de ADN totales, polimórficos y porcentaje de polimorfismo para la identificación de plantas híbridas de nochebuena de las cruzas del progenitor Amanecer navideño mediante RAPD.
| Iniciador | Fragmentos amplificados | Fragmentos polimórficos | Polimorfismo (%) | Fragmentos marcadores (pb) |
| OPA-07 | 15 | 5 | 33 | IP (2179 pb/2 y 4), IP (1987 pb/2, 3, 4 y 7), IP (1531 pb/2, 3, 4 y 7), IP (855 pb/2, 4 y 5), |
| OPA-11 | 12 | 5 | 42 | IP (1507 pb/2, 3, 4, 6 y 7) |
| OPA-12 | 15 | 4 | 27 | IP (908 pb/5, 6 y 7), IP (815 pb/3, 4, 5, 6 y 7) |
| OPB-07 | 20 | 7 | 35 | IP (2455 pb/2, 3, 4, 5,6 y 7), |
| OPB-08 | 6 | 2 | 33 | IP (661 pb/1, 4, 7) |
| OPB-09 | 9 | 3 | 33 | IP (1270 pb/2, 4, 7) |
| OPB-11 | 19 | 5 | 56 | IP (1873 pb/ 2, 3, 5) |
| OPB-20 | 20 | 10 | 50 | Fr (812 pb/ 2, 3) |
| OPC-06 | 12 | 4 | 33 | - |
| OPC-13 | 18 | 5 | 28 | - |
Los fragmentos que identificaron a la progenie de las cruzas de Amanecer navideño x Ice punch y Amanecer navideño x Festival red variaron de 661 a 2455 pb. El iniciador que generó el mayor número de fragmentos que identificaron a las plantas híbridas fue el OPA-07, el porcentaje de información polimórfica de este iniciador fue de 33%. Los iniciadores que no detectaron a ningún híbrido obtenido de las cruzas fueron el OPC-06 y OPC-13 (Cuadro 3).
Las plantas que pudieron identificarse como híbridos con la mayoría de los iniciadores fueron 2 An x Ip, 3 An x Ip, 4 An x Ip, 5 An x Ip, 6 An x Ip, 7 An x Ip, 2 An x Fr y 3 An x Fr. Lo anterior sugiere que las cruzas realizadas de Amanecer navideño x Ice punch se lograron efectivamente, ya que se consiguió identificar a su progenie con respecto al progenitor macho Ice punch, a excepción de la planta 1 de esta cruza que no mostró ningún fragmento en común con el progenitor macho Ice punch.
El dendograma generado (Figura 3) a partir de los datos obtenidos con los diez iniciadores RAPDs, mostró la cercanía genética entre la planta 5 An x Ip y 6 An x Ip, así como también, la planta 3 An x Ip y 4 An x Ip. Se formaron 6 grupos a un coeficiente de distancia genética de 0.17: 1) Amanecer navideño con Ice punch; 2) 3 An x Ip, 4 An x Ip, 7 An x Ip; 3) 2 An x Ip; 4) progenitor masculino Festival red, 1 An x Ip; 5) 2 An x Fr y 3 An x Fr; 6) 5 An x Ip, 6 An x Ip y 1 An x Fr.

Figura 3 Relación de las cruzas generadas en nochebuena, por Amanecer navideño x Ice punch (7 plantas) y Amanecer navideño x Festival red (3 plantas) según lo determinado por el análisis de agrupamiento UPGMA tomando como referencia 146 fragmentos RAPD.
Las plantas más similares entre sí (con un coeficiente de 0.12) son 5 An x Ip y 6 An x Ip; así como, también la progenie 3 An x Ip y 4 An x Ip estas son las que menor distancia genética presentan de igual manera, los progenitores Amanecer navideño e Ice punch comparten similitud genética a un coeficiente de distancia de 0.15. En contraste, la planta que no comparte similitud genética con su progenie ni con otro progenitor es la planta macho Festival red (Figura 3).
El conglomerado muestra que las diez plantas F1 de las cruzas de Amanecer navideño x Ice punch y Amanecer navideño x Festival red se unieron a diferentes grupos, lo cual muestran cercanía genética entre las tres de acuerdo a lo conformado en los tres grupos formados: 1) el progenitor macho Ice Punch; 2) las cruzas de Amanecer navideño x Ice punch 2, 3, 4 y 7; y 3) el progenitor hembra Amanecer navideño, el progenitor macho Festival red con las cruzas 1, 5 y 6 Amanecer navideño x Ice punch y las cruzas 1, 2 y 3 de Amanecer navideño x Festival red (Figura 4).
Identificación de progenie de Belén x Ice punch, Belén x Burgundy y Belén x Festival red
La amplificación de iniciadores RAPDs utilizados en el estudio de las cruzas obtenidas de Belén x Ice punch, Belén x Burgundy y Belén x Festival red, reveló la presencia de 113 fragmentos con un porcentaje de polimorfismo de 26% (Cuadro 2). El iniciador OPC-13 fue el que mayor amplificación de fragmentos generó (14) con polimorfismo de 36%, pero el iniciador que dio mayor polimorfismo fue el OPC-06 con 38% y 13 fragmentos amplificados, el porcentaje de polimorfismo varió desde 10 hasta 38%.
Los iniciadores generaron fragmentos marcadores monomórficos los cuales fueron invariables en todas las plantas de estudio y estos no lograron identificar a los progenitores con la progenie híbrida, los fragmentos no mostraron diferencias incluso entre los mismos progenitores. El dendograma generado (Figura 5) a partir de los datos obtenidos con los 10 iniciadores RAPDs, mostró la formación de 11 grupos a un coeficiente de distancia genética de 0.18, los grupos se conformaron de la siguiente manera: 1) el progenitor femenino Belén con Ice punch; 2) el progenitor masculino Burgundy; 3) el progenitor masculino Festival red; 4) 2 Be x Bur; 5) 9 Be x Fr, 10 Be x Fr y 13 Be x Fr; 6) 11 Be x Fr; 7) 12 Be x Fr; 8) 1 Be x Ip, 1 Be x Bur, 4 Be x Fr, 5 Be x Fr, 2 Be x Fr, 3 Be x Fr; 9) 1 Be x Fr, 6 Be x Fr; 10) 7 Be x Fr, 8 Be x Fr; y 11) 3 Be x Bur.
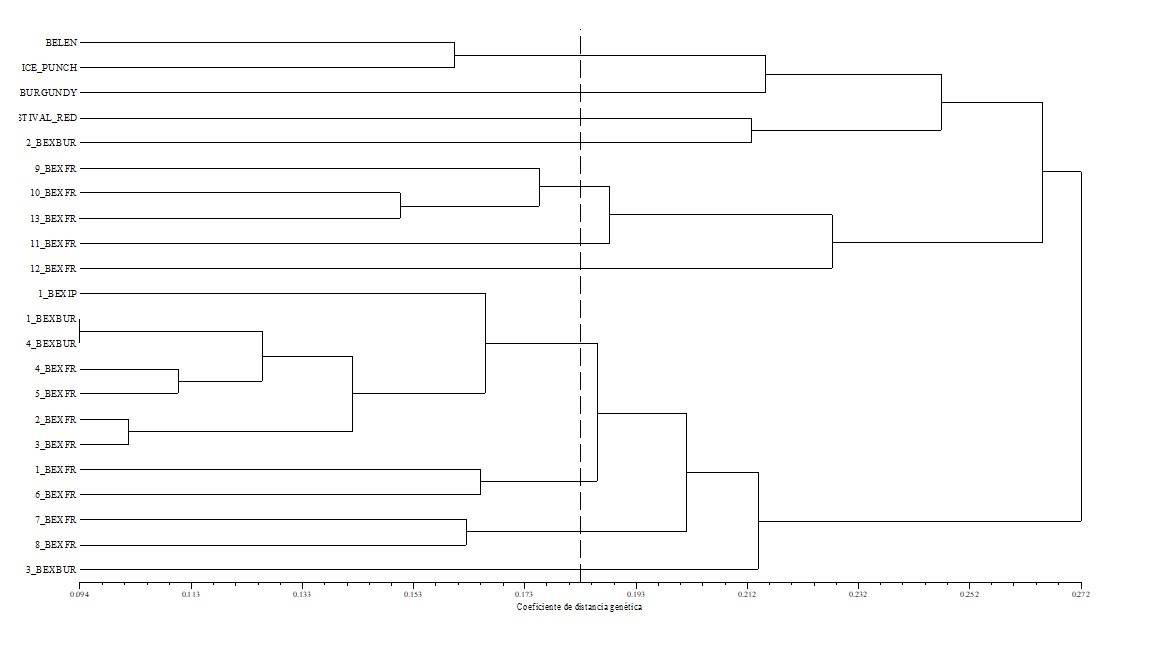
Figura 5 Agrupamiento de progenitores macho (Ice punch, Festival red y Burgundy) y la relación de las cruzas generadas con Belén, según lo determinado por el análisis de agrupamiento UPGMA tomando como referencia 113 fragmentos RAPD.
Las plantas hibridas más similares entre sí (con un coeficiente de 0.09) fueron 1 Be x Bur y 4 Be x Bur, así como también 2 Be x Fr y 3 Be x Fr, los progenitores Ice punch y Belén comparten similitud genética a un coeficiente de distancia de 0.16 (Figura 5). La distribución gráfica muestra que las 18 plantas F1 de las cruzas de Belén x Ice punch, Belén x Festival red y Belén x Burgundy son genéticamente distintas con respecto a los cuatro grupos formados: 1) el progenitor macho Ice punch; 2) el híbrido 11 de Belén x Festival red; 3) los híbridos 1, 2, 3, 4, 5, 6, 7, 8 y 9 de Belén x Festival red y 1, 3, 4 de Belén x Burgundy; y 4) las cruzas del progenitor hembra Belén con el progenitor macho Festival red y sus cruzas 1, 2, 3, 4, 5, 6, 7, 8 y 9; Belén x Ice punch, y la cruza 1, los híbridos de Belén x Burgundy 1, 4 y 5 (Figura 6).

Figura 6 Distribución gráfica de progenitor macho Belén x Ice punch 1 planta generada, Belén x Festival red 3 plantas generadas y Belén x Burgundy 14 plantas generadas en las cruzas.
Los iniciadores seleccionados para los progenitores Juan Pablo, Amanecer navideño y Belén y las plantas originadas de los cruzamientos de nochebuena de Juan Pablo x Ice punch, Juan Pablo x Festival red, Amanecer navideño x Ice punch, Amanecer navideño x Festival red, Belén x Ice punch, Belén x Festival red y Belén x Burgundy, produjeron 408 fragmentos RAPDs en total, de los cuales fueron polimórficos 109 fragmentos con 27% de polimorfismo con 10 iniciadores, estos permitieron identificar a las plantas F1 de Juan Pablo x Ice punch, Juan Pablo x Festival red, Amanecer navideño x Ice punch, Amanecer navideño x Festival red con 26 fragmentos marcadores, las cruzas con Belén no se lograron identificar con los 10 iniciadores, ya que generaron fragmentos marcadores; sin embargo, estos fragmentos marcadores no permitieron identificar a ningún hibrido, se produjeron 11 fragmentos marcadores que pudieran evaluarse con otros iniciadores.
Lo anterior muestra la capacidad de discriminación de los RAPD para la identificación de cultivares de nochebuena. Los resultados de la presente investigación coinciden con lo realizado por Jing-Tian et al. (1997) quienes identificaron cultivares de Poinsettia usando marcadores RAPD, realizaron la amplificación de 60 iniciadores y obtuvieron 69% de polimorfismo y concluyen que sus resultados indican que los RAPDs son eficientes para la identificación de cultivares de Poinsettia y para la determinación de las relaciones genéticas entre cultivares.
En otro estudio de evaluación de AFLP en Poinsettia: selección de polimorfismo, análisis e identificación de cultivares, evaluaron 81 cultivares de nochebuena mediante 41 AFLP polimórficos, de los cuales identificaron sólo 60 cultivares de nochebuena, los otros 21 no fueron identificados (Parks y Moyer, 2004). En un estudio de comparación de RAPD y AFLP como métodos de identificación genética de vid basados en el estudio de fragmentos, estudiaron 57 variedades de vid con 80 iniciadores, en su estudio de RAPD mencionan que este resultado es destacable, dado que se verifica una correspondencia entre la genealogía y la información que entrega el análisis molecular y concluyen que ambos métodos permitieron diferenciar todos los cultivares analizados (Narváez et al., 2000).
Conclusiones
Los resultados moleculares obtenidos muestran un alto nivel de identificación de híbridos F1 mediante RAPDs, se logró determinar el grado de similitud, con ello se demuestra que los RAPDs son una herramienta importante para la identificación de rasgos sobresalientes y diferencias en la variación morfológica de híbridos F1 de Nochebuena y establece la posibilidad de poder obtener y registrar el derecho de obtentor de nuevas variedades generadas de un programa de mejoramiento genético de nochebuena.
La perspectiva en cuanto a los progenitores, tanto de las variedades de sombra como las de sol, es que las tres variedades de nochebuena de sol Juan Pablo, Amanecer navideño y Belén son una opción viable como progenitoras hembra para la búsqueda y formación de híbridos novedosos. Mientras que las variedades de sombra con las que se obtuvo mayor número de híbridos fueron Ice punch y Festival red, pero los híbridos entre Belén y Festival red no se lograron identificar con los iniciadores RAPDs. En este contexto se puede establecer que los híbridos de nochebuena de Belén y Festival red, son semejantes genéticamente y para la identificación de los híbridos es conveniente aplicar e implementar un estudio posterior, con el uso de otros iniciadores o incluso otros marcadores moleculares para su identificación.











 text in
text in 



