Introducción
La utilización de técnicas moleculares para estudios relacionados con la expresión de genes diferenciales requiere la síntesis de ADN complementario de buena calidad (Aslam et al., 2017). De esta manera, la disponibilidad de un ARN de buena calidad es el primer requerimiento para el entendimiento de las investigaciones en plantas. Se han descrito diferentes métodos para el aislamiento de ARN utilizando diversos detergentes como el bromuro de cetiltrimetilamonio (CTAB) por ser económico, SDS, fenol, cloroformo, cloruro de litio y diferentes gradientes de centrifugación (Bermúdez-Guzmán et al., 2016; Silva et al., 2016). Por su parte, la preservación de la integridad de ARN durante la extracción es primordial.
La cuantificación precisa de ARNm utilizado para el perfil de expresión génica, depende de la integridad del ARN extraído. Por lo tanto, el uso de ARN de mala calidad durante la cuantificación de los niveles de ARNm puede comprometer la precisión de los resultados de la expresión génica (Bleitler et al., 2016).
La extracción de ARN de tejido de plantas de mezquite (Prosopis spp.), se dificulta debido a que es una planta rica en metabolitos secundarios, polisacáridos y polifenoles (Ghangal et al., 2009; Chang et al., 2016). La presencia de compuestos puede afectar la calidad y cantidad de los ácidos nucleicos aislados (Abdel-Latif y Osman, 2017). A su vez, se obstaculiza debido a las diferencias que existen en el contenido de metabolitos presentes en los diferentes tejidos (raíces, tallo y hojas) y etapas de desarrollo de la planta (Aslam et al., 2016). Además, existen compuestos fenólicos que pueden ser fácilmente oxidados, lo que habilita la formación de quinonas debido a sus propiedades químicas similares, afectando la calidad de la muestra y el rendimiento del ARN (Ghangal et al., 2009; da Luz et al., 2016).
Los avances en el cultivo de la fruta de la pasión se obtuvieron por mejora genética. Entonces, el mejor método para la extracción de ARN en P. edulis debe determinarse de manera que los análisis como la construcción de biblioteca complementaria de ADN (ADNc), hibridación supresiva sustractiva (SSH), RT-PCR, polimorfismo de longitud de fragmento amplificado con cDNA (cDNA AFLP) y Northern blot no están limitadas por muestras de baja calidad
Prosopis juliflora (SW), comúnmente llamado ‘mezquite dulce’, es un árbol perteneciente a la familia Fabaceae y es una planta tolerante a las sequías, salinidad, y metales pesados. Se encuentra ampliamente distribuida en regiones áridas y semiáridas. En México, es la especie más importante por su extensión y usos, ocupa casi todo el territorio mexicano, crece en climas extremos con temperaturas superiores a los 48 °C y en una variedad de suelos (Rodríguez-Sauceda et al., 2014).
Materiales y métodos
Colecta de material
El presente estudio se llevó a cabo en el Instituto de Ciencias Agrícolas (ICA-UABC) de la Universidad Autónoma de Baja California. El germoplasma de P. juliflora fue proporcionado por el banco especializado en el resguardo de semillas forestales, de árboles y arbustos, nativos o endémicos de interés comercial, operado por la Comisión Nacional Forestal en Baja California Comisión Nacional Forestal (CONAFOR). Las semillas fueron transportadas al laboratorio de Biotecnología para su desinfección con una solución de NaOCl (Clorox) al 1% por 3 min. Finalmente se realizaron cuatro lavados sucesivos con agua destilada estéril (dos minutos por lavado) para eliminar el exceso del desinfectante. Posteriormente, se pasó a la siembra y cosecha de tejidos (tallo, hoja, raíz y embrión) de las plantas de mezquite de aproximadamente dos meses de edad para la realización de las pruebas correspondientes.
Soluciones y buffers requeridos
Se evaluaron tres protocolos diferentes de extracción cada uno con ligeras modificaciones:
Buffer de extracción 1: cloruro de sodio 0.25M, Tris-HCl 0.05M (pH 7.5), EDTA 20 mM, dodecil sulfato de sodio (SDS 1%) (w/v) y polivinilpirrolidona (PVP 4%) (w/v) (Llanes et al., 2016).
Buffer de extracción 2: Buffer Fenol- Cloroformo (Ghawana et al., 2011) con modificaciones ligeras.
Buffer de extracción 3: Trizol® reagent; solución de fenol saturada con tampón citrato 0.1 M, pH 8), DNasa I, etanol, cloroformo, alcohol isoamílico e isopropanol de pureza analítica y siguiendo las consideraciones del manufacturado.
Cloroformo-alcohol isoamílico (24:1)
Fenol-cloroformo alcohol isoamílico (1:1)
Acetato de sodio 3M
Cloruro de litio
Etanol 70%
Etanol absoluto
Agua destilada libre de RNasas o DEPC
Protocolo de aislamiento de ARN total
Para los tres métodos de extracción, se colectaron muestras de los diferentes tejidos (tallo, hoja, raíz y embrión) las cuales fueron tratadas con agua destilada para su posterior análisis. Aproximadamente 100 mg de los tejidos frescos fueron pulverizados con nitrógeno líquido, el material fue colectado en tubos eppendorf de 1.5 mL y posterior se agregó 750 μL de buffer de extracción y 750 μL de cloroformo-alcohol isoamílico (24:1) y se vortexearon las muestras.
Enseguida las muestras se centrifugaron a 12.800 rpm por 2 min a temperatura ambiente 25 °C. El sobrenadante fue recuperado y transferido a tubos nuevos adicionando un volumen igual de Fenol-cloroformo alcohol isoamílico (1:1), posteriormente centrifugadas a 12 800 rpm por 2 min a temperatura ambiente 25 °C (repetir este paso hasta observar una fase clara), transferir el sobrenadante a tubos nuevos y adicionar 10% del volumen recuperado de acetato de sodio 3M y 2.5 volúmenes de etanol absoluto, mezclar e incubar a -20 °C por una h. Posterior se realizó un lavado con etanol al 70%, las muestras se centrifugaron a 12 800 rpm por 10 min a 4 °C, se realizó un último lavado de las muestras con etanol absoluto, se decantaron y dejaron secar a temperatura ambiente. Finalmente, la pastilla obtenida se resuspendió en 30 µL de agua destilada libre de RNasas para su posterior uso (Kam-Lock et al., 2007).
Estimación de la calidad y cantidad de ARN
La pureza y concentración de las muestras de ARN obtenidas de los tejidos de P. juliflora (SW) fueron evaluadas espectrofotométricamente mediante NanoDrop 2000 (Thermo Scientific) a diferentes longitudes de onda, obteniendo los radios de OD A260/280 y A260/230 nm para medida de contaminación por polifenoles-carbohidratos y proteínas, respectivamente. La integridad del ARN se evaluó por medio de electroforesis en gel de agarosa al 1%, se cargó un total de 3 µl por muestras y los geles fueron teñidos con bromuro de etidio y visualizados en un fotodocumentador de luz UV MultiDoc-It Digital Imaging System UVP (Rubio-Piña y Vázquez-Flota, 2008). Posterior se llevó a cabo un tratamiento con DNasas utilizando RQ1 RNase-Fee DNase (Promega) y siguiendo el protocolo comercial.
Síntesis de ADN complementario y RT-PCR
Los extractos obtenidos por los tres protocolos de extracción fueron evaluados por RT-PCR. La síntesis de ADNc fue sintetizada a partir de 3 µg del ARN total obtenido utilizando ImProm-IITM Reverse Transcriptase (Promega) y oligo (dT) y aplicando las instrucciones del manufacturador. La síntesis de cadena sencilla ADNc fue posteriormente utilizada para PCR para estimar los niveles de expresión del gen universal de actina en plantas utilizándose iniciadores específicos: sentido (5’CACIACTACTGCTAAACGGGAAA3’), antisentido (5’ACATCTGCTGGAAGGTGCTG3’) los cuales fueron diseños mediante el software Primer Premier v5 (PrimerBiosoft) y con base en las regiones conservadas de secuencias previamente reportadas para otras especies.
El programa de PCR implementado consistió en una desnaturalización del ADN a 94 °C por 3 min, posteriormente 35 ciclos por 30 s a 94 °C para desnaturalizar el ADN, 30 s a 60 °C para el alineamiento de los iniciadores y la extensión por 30 s a 72 °C, finalmente el programa concluye con 10 min de extensión final a 72 °C. Los productos amplificados obtenidos fueron separados en un gel de agarosa al 1%, se tiñeron con bromuro de etidio (10 mg mL-1) y se visualizaron con luz ultravioleta en un fotodocumentador.
Análisis estadístico
Para el análisis de las muestras se diseñó un modelo de análisis de varianza (Anova) completamente al azar con sus tres repeticiones y se aplicó la prueba de separación de medias de Tukey (p< 0.05) para la determinación de las diferencias entre las absorbancias obtenidas para cada uno de los tratamientos. De la misma manera, se utilizó la prueba de Duncan a (p< 0.01) para evaluar las diferencias entre los tratamientos. El análisis de los datos se realizó mediante el paquete estadístico SAS. La integridad y funcionalidad del DNA fueron evaluadas visualmente con geles de agarosa.
Resultados y discusión
Actualmente existen publicados un gran número de métodos para el aislamiento de ARN de plantas, en los cuales se ha mencionado algunas de las dificultades que existen durante el procedimiento de extracción. Generalmente, se puede obtener ARN de alta calidad de tejidos de plantas, una vez que las actividades de las RNasas son inhibidas (Wang et al., 2008). En el presente estudio, se compararon los resultados de tres diferentes protocolos con los resultados de otros protocolos que fueron diseñados para extraer ARN de tejidos vegetales con alto contenido de polisacáridos y polifenoles.
La obtención de ARN de alta calidad es la base de muchas investigaciones de biología molecular de plantas; RT-PCR, la síntesis de ADNc y el posterior análisis genético, requieren ARN con alta pureza e integridad (Liu et al., 2018). En la Figura 1, se puede observar la representación de las bandas características de las subunidades 28S y 18S del ARN ribosomal, con lo cual se confirma la purificación del ARN. A su vez, la calidad del ARN total se detectó mediante la reacción en cadena de la polimerasa. Por otro lado, en este estudio, el uso de polivinilpirrolidona (PVP) en el buffer de extracción tuvo como resultado significativo una mejora en el rendimiento y la calidad del ARN extraído comparado con la aplicación de otros métodos para tejidos de P. juliflora (SW). Wang et al. (2008), mencionan que PVP eficientemente absorbe polifenoles y remueve metabolitos secundarios de los ácidos nucleicos en plantas, así como también la mayoría de las sustancias macromoleculares.

Figura 1 Electroforesis en gel de agarosa al 1% del ARN total extraído de los tejidos (hoja, tallo, raíz y embrión) de mezquite P. juliflora (SW) de los tres protocolos evaluados. Las dos distintas bandas, 18S y 28S de ARN ribosomal muestran que el ARN está íntegro (A, C) y (B) se obtuvieron sólo muestras de ADN genómico total.
Los compuestos fenólicos presentes en las muestras de tejido se oxidan rápidamente para formar quinonas. Estas quinonas pueden unirse fácilmente con ácidos nucleicos y actuar como una barrera para el aislamiento de ARN de buena calidad (George, 2018).
El exitoso aislamiento y obtención de ARN total intacto de tejidos ricos en polisacáridos y polifenoles, es importante para prevenir la unión de contaminantes a los ácidos nucleicos (Malnoy et al., 2001). A su vez, se ha reportado que la contaminación por ADN no puede evitarse; sin embargo, la digestión de ARN con DNasas libre de RNasas es indispensable; es decir, la digestión con DNasas, es un paso común en el análisis de protocolos de ARN (Ghawana et al., 2011).
El método basado en Trizol ha sido ampliamente utilizado para el aislamiento de ARN de varios tejidos y especies. Primero intentamos usar el reactivo Trizol para aislar el ARN de los tejidos de P. juliflora. Sin embargo, el tampón Trizol no pudo eliminar eficazmente el alto contenido de metabolitos haciendo que la fase acuosa se tornara roja después de la purificación con cloroformo. Sin embargo, el protocolo 2 resultó ser favorable para la extracción de ADN de los tejidos de plantas de mezquite.
Estudios han demostrado que existen diferentes factores que afectan los perfiles de amplificación obtenidos mediante técnicas moleculares basadas en PCR, algunos de los principales factores que afectan estos perfiles de amplificación, son la calidad, pureza y cantidad del ARN extraído, lo cual tiene que ver principalmente con el co-aislamiento de polisacáridos, fenoles y metabolitos secundarios durante la extracción de ARN (Solano-Flórez et al., 2009). En este estudio, la calidad del ARN total se detectó mediante la reacción en cadena de la polimerasa. Las muestras de ARN se transcribieron de forma inversa en ADNc y el resultado se amplificó usando PCR (Liu et al., 2018). De esta manera, se demuestra que el ARN total extraído de P. juliflora mediante el protocolo 1 y 3 cumplen con los requisitos para futuras investigaciones moleculares.
Por otro lado, la electroforesis en gel de agarosa muestra que el protocolo de extracción implementado produjo ARN de alta calidad mostrado por las bandas definidas de ARN ribosomal citosólico y plasmídico (Box et al., 2011). La electroforesis observada en la Figura 2, indica que el par de cebadores usado para amplificar el fragmento del gen de actina exhibe una alta especificidad. Las curvas de amplificación demostraron que las secuencias de ARNm estaban prácticamente intactas en las muestras de ARN y que el gen de actina estaba presente en todos los tejidos en abundancia moderada; de esta manera se demuestra que el ARN total producido es de buena calidad para el análisis de RT-PCR de genes constitutivos como actina con un tamaño aproximado de 300 pb (Kam-Lock et al., 2007).
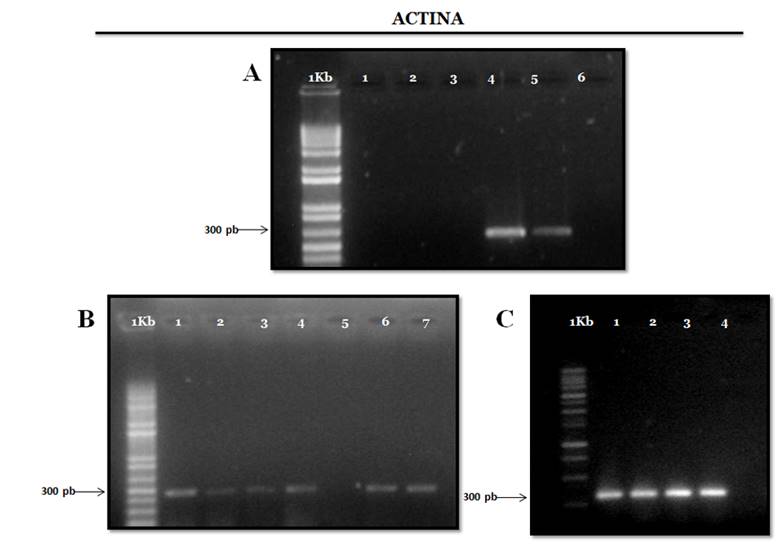
Figura 2 Electroforesis en gel de agarosa al 1% de los productos de RT-PCR del gen de actina con extractos de todos los tejidos evaluados con los tres protocolos de extracción y con iniciadores específicos para plantas (tamaño esperado 300 pb). Para los tres geles MP corresponde al marcador de peso molecular GeneRuler 1Kb Plus DNA Ladder (fermentas).
Es muy conocido el gran número de métodos originales y modificados, que en su mayoría informan modestos rendimientos y ARN parcialmente intactos, los cuales no pueden ser generalizados o aplicados a distintas especies sobre todo para extracción de ARN, donde el contenido de este ácido nucleico es relativo al momento y estado de las células a evaluar. En este estudio, los radios de absorbancias de todas las muestras analizadas, fueron mayores a 1.8, indicando una débil contaminación producida por polisacáridos y polifenoles (Cuadro 1), estos resultados son similares a los reportados por Malnoy et al. (2001) en especies de la familia Rosaceae.
Cuadro 1 Lecturas espectrofotométricas usadas para evaluar la cantidad y calidad del ARN total de tejidos de P. juliflora.
| Tejido | Protocolos | Radios de calidad OD | |
|---|---|---|---|
| A260/A280 | A260/A230 | ||
| Hoja | 1 | 2.11 | 2.19 |
| 2.2 | 2.07 | ||
| 2.26 | 2.11 | ||
| 2 | 1.85 | 1.76 | |
| 1.95 | 1.52 | ||
| 1.94 | 1.47 | ||
| 3 | 1.97 | 1.33 | |
| 1.77 | 1.59 | ||
| 1.72 | 1.17 | ||
| Tallo | 1 | 2.02 | 2.12 |
| 2 | 2.12 | ||
| 1.97 | 2 | ||
| 2 | 1.92 | 1.25 | |
| 1.91 | 1.41 | ||
| 1.95 | 2.07 | ||
| 3 | 2.02 | 1.99 | |
| 1.94 | 1.2 | ||
| 1.92 | 1.25 | ||
| Raíz | 1 | 2.11 | 2.32 |
| 2.05 | 2.1 | ||
| 2.01 | 1.9 | ||
| 2 | 2.12 | 2.09 | |
| 2.13 | 2.08 | ||
| 2.06 | 1.83 | ||
| 3 | 1.96 | 2.09 | |
| 2 | 1.9 | ||
| 2 | 1.9 | ||
| Embrión | 1 | 2.13 | 2.23 |
| 2.11 | 2.17 | ||
| 2 | 1.93 | 1.4 | |
| 1.91 | 1.19 | ||
| 3 | 2.04 | 1.94 | |
| 2.02 | 1.99 | ||
1= protocolo SDS; 2= protocolo fenol; 3= protocolo trizol (ITG).
Metodologías confiables y probadas, en ocasiones no pueden ser implementadas en todos los laboratorios, por el costo de ciertos reactivos o la dificultad para su adquisición en el laboratorio de trabajo. Aunque los resultados de los protocolos evaluados difieren, sí se obtuvieron muestras que cumplieron con las características de rendimiento y calidad, las cuales pueden ser utilizadas en la aplicación de técnicas moleculares más avanzadas.
Conclusiones
Se ha desarrollado un método rápido y eficiente para la extracción de ARN de tejidos de plantas con un alto contenido de polisacáridos y polifenoles, como es el caso de P. juliflora (SW). El ARN obtenido por este método puede ser utilizado para la detección de ARNs específicos. Asimismo, este método también puede ser aplicable para la extracción de otros tejidos en las diferentes plantas con características similares a P. juliflora (SW).
A su vez, con este documento, ofrecemos la posibilidad de un protocolo eficiente, rápido, seguro y económico para la extracción simultánea de ADN y ARN, permitiendo de esta manera, la extracción de material genético de alta calidad, asimismo, la extracción simultánea de ácidos nucleicos, puede ser una ventaja en el caso de material de origen limitado o para minimizar la variación de la muestra cuando el ADN deba compararse con el ARN de la misma fuente o condición biológica.











 texto en
texto en 


