Introducción
En México, la enfermedad conocida como secadera temprana o “damping off” se considera el principal problema fitosanitario en la fase de almácigo del cultivo de chile (Velásquez-Valle et al., 2007) y se atribuye a un complejo de hongos patógenos que incluye a Phytophthora capsici, Fusarium spp., Rhizoctonia solani, Sclerotium rolfsii y Pythium sp. (Vélazquez-Valle et al., 2013). Las plántulas pueden ser afectadas de dos formas: marchitez de la planta como consecuencia de la pudrición de sus raíces y afectación a nivel de la base del tallo (cuello); ambos síntomas originan el colapso de las plántulas (Goldberg, 1995). La incidencia de plántulas enfermas en almácigos puede llegar a ser de hasta 29% y constituyen una fuente de inóculo que los agricultores transfieren al campo durante el trasplante; las pérdidas del cultivo en almácigo, campo y cosecha pueden variar entre 10 a 100% (Montero-Tavera et al., 2013).
El uso de fungicidas químicos para el control de fitopatógenos no siempre es efectivo (Huang et al., 2012), además de causar contaminación y efectos nocivos para la salud humana y el medioambiente (Gomathi y Ambikapathy, 2011). Una alternativa para el control de enfermedades causadas por estos hongos es el uso de rizobacterias promotoras del crecimiento vegetal (PGPR, por sus siglas en inglés) (Kloepper y Schroth, 1981), las cuales no presentan efectos negativos sobre el medio ambiente (Mehetre y Kale et al., 2011).
Las PGPR pueden actuar como: a) agentes de biocontrol de enfermedades, a través de una competencia por espacio o alimento, o mediante la producción de antibióticos e inducción de sistemas de defensa de la planta (Matar et al., 2009), disminuyendo o previniendo la enfermedad (Saravanakumar et al., 2008); b) promotoras del crecimiento, a través de la producción de reguladores del crecimiento, como es el caso de la auxina ácido indol-3-acético (AIA) (Goswami et al., 2015), sintetizada por aproximadamente 80% de las rizobacterias (Ahmad et al., 2005), teniendo como efecto una mayor captación de nutrientes y promoviendo en consecuencia el crecimiento y rendimiento de los cultivos; y c) solubilizadoras de fosfato inorgánico a través de la secreción de fosfatasas y ácidos orgánicos que liberan el P mineral u orgánico no disponible, estimándose que 40% de las PGPR presentan esta capacidad (Richardson et al., 2011).
El presente estudio tuvo como objetivo caracterizar in vitro 18 aislamientos bacterianos con base en su capacidad para inhibir el crecimiento de cuatro hongos causantes de marchitez fúngica en plántulas del cultivo de chile, su capacidad para producir ácido indolacético y la solubilización de fosfato inorgánico.
Materiales y métodos
Aislamientos microbianos. Se realizó el muestreo de suelo rizosférico de maíz (Zea mays L.) establecido en un módulo agroforestal (22° 43’ 08.4” latitud norte, 101° 48’ 17.7’ longitud oeste) localizado en La Palma Pegada, municipio de Salinas, SLP, con una elevación de 2 222 msnm, donde el cultivo anterior fue chile. El aislamiento de las cepas se realizó como lo describe Kavitha et al. (2005), pero el medio de cultivo utilizado fue peptona de levadura-glucosa-agar (YPGA) (De León et al., 2008), incubando a 28 °C durante 24 h. Posteriormente se realizó la caracterización morfológica y cultural en un período de 24 h, donde se consideró el color, forma, elevación y borde de la colonia.
Por otra parte, en almácigos de plántulas de chile “guajillo” ubicados en La Dulce Grande (23° 00’ 00.1” latitud norte 102° 10’ 29.1” longitud oeste), municipio de Villa de Ramos, SLP, se realizó un muestreo de plántulas que presentaban síntomas de “damping off”. En el laboratorio del Colegio de Postgraduados, Campus San Luis Potosí, se lavaron las raíces y el cuello de las plántulas enfermas; enseguida se diseccionaron en pequeñas porciones (2 mm longitud) y se colocaron en medio de cultivo papa-dextrosa-agar (PDA, DIBICO) a 28 °C durante siete días, después de lo cual se realizó la purificación de las colonias fúngicas, obteniendo tres cepas pertenecientes al género Fusarium spp. (F). Adicionalmente, se utilizó una cepa de Rhizoctonia spp. (R) proporcionada por el Laboratorio de Edafología del Colegio de Postgraduados, Campus Montecillo. Las cepas fueron conservadas en PDA.
Determinación de antagonismo microbiano in vitro. Se realizó la reactivación de los aislamientos bacterianos y fúngicos en los medios de YPGA y PDA, respectivamente. Para evaluar la actividad antifúngica de 18 aislamientos bacterianos contra cuatro hongos patógenos, se utilizó el método de cultivo dual (Coskuntuna y Özer, 2008). Los aislados bacterianos de un día de edad fueron colocados a 2 cm de la periferia de la caja Petri, cada uno por triplicado, e incubando a 28 °C durante 24 h; pasado ese período, se colocó un disco de 5 mm de diámetro con micelio fúngico a una distancia de 4 cm de la bacteria. Los cultivos se incubaron a 28 °C, hasta que el control (sin bacteria) llenó la caja. Los resultados se expresaron como el porcentaje de inhibición correspondiente a cada una de las cepas bacterianas. El porcentaje de inhibición fue calculado de acuerdo a Mehetre y Kale (2011).
Producción de ácido indolacético (AIA). Para la activación de los aislamientos bacterianos se utilizó el medio líquido Luria-Bertani (Bric et al., 1991), modificado de acuerdo a Goswami et al. (2014), el cual fue incubado a 25 ±2 °C en agitación rotatoria (New Brunswick Scientific Edison) durante cinco días. Se tomaron 2 ml de cada aislamiento y se centrifugó a 8000 rpm; el sobrenadante de cada cultivo fue mezclado con el reactivo Salkowski (Balota et al., 1998) en una relación de 2:1 y se incubó bajo oscuridad durante 30 min, para evidenciar el desarrollo de color característico (rosado-violeta). Finalmente, la absorbancia del complejo coloreado se leyó a 530 nm en un espectrofotómetro (Bio Tek Laboratory Instrument, Synergy 2, USA). La concentración de AIA fue determinada mediante una curva estándar en un rango de 0 a 300 µg mL-1. Para cada aislamiento se evaluaron tres repeticiones, utilizando como referencia el medio de cultivo sin inóculo bacteriano.
Solubilización de fósforo inorgánico. Se inocularon 18 cepas bacterianas en frascos que contenían 20 ml de medio de cultivo líquido Pikovskaya (Pikovskaya, 1948), el cual tenía fosfato tricálcico insoluble [Ca3(PO4)2] como única fuente de fósforo, y se incubaron a 25 ±2 °C en agitación constante durante 7 días. Se tomaron 2 ml de cultivo de cada frasco y se centrifugaron a 8 000 rpm. El fósforo soluble del sobrenadante se estimó mediante el método colorimétrico azul de molibdeno (Jackson, 1967) a 480 nm. La correspondiente cantidad de fosfato soluble fue calculada a los 7 días del crecimiento del cultivo a través de una curva estándar de fosfato soluble en un rango de 0 a 300 µg mL-1 (Banerjee et al., 2010).
Supervivencia de plántulas. Esta parte de la investigación se realizó bajo condiciones de invernadero. La semilla de chile guajillo fue sembrada en una mezcla de sustratos peat moss y vermiculita (1:1) previamente esterilizada dos veces a 121° durante 1 h, con un intervalo de tres días. Siete días después de la emergencia, las plántulas fueron trasplantadas a charolas de unicel. Se dio un período de 5 días para la aclimatación de las plántulas y se procedió a realizar la inoculación con 1 mL de cultivo bacteriano conteniendo 109 células mL-1 de cada cepa bacteriana (B7, B9, B15 y B23) seleccionadas del estudio in vitro.
Las bacterias se aplicaron con una micropipeta en la base del tallo de las plántulas. Se dio un intervalo de cuatro días para realizar la inoculación del hongo patógeno (Hassan et al., 2015), el micelio fúngico provino de un cultivo de 7 días de edad y se colocó en donde empieza el desarrollo de las raíces de las plántulas. Se registró la supervivencia de las plántulas y al final se obtuvo el tratamiento con mayor número de plantas supervivientes, que fue atribuido al efecto de la bacteria antagonista inoculada frente a cada hongo fitopatógeno.
Identificación molecular de rizobacterias y hongos. Se realizó el aislamiento de ADN genómico de las rizobacterias B7, B8, B9, B14, B15 y B23 y hongos F. púrpura, F. naranja y F. amarillo a partir de cultivo bacteriano en medio agar nutritivo YPGA para rizobacterias y PDA para hongos, utilizando el protocolo de extracción CTAB y acetato de sodio (LaMontagne et al., 2002). La amplificación y secuenciación del ADN genómico se realizó en el Instituto Potosino de Investigación Científica y Tecnológica (IPICYT). Se amplificó un fragmento del gen 16S rRNA utilizando los oligonucleótidos 533F (Weisburg et al., 1991) y 1391R (Turner et al., 1999) en un termociclador Verity para PCR punto final (Applied Biosystems). Los productos obtenidos se secuenciaron con el método de didesoxinucleótidos marcados en el secuenciador 3130 Genetic Analyzer (Applied Biosystems). El porcentaje de identidad de las secuencias del fragmento del gen 16S rRNA con las cepas tipo se determinó al utilizar BLASTN de la base de datos del GenBank.
Análisis estadístico. El comportamiento de las bacterias en la inhibición del crecimiento de hongo, la producción de ácido indolacético, así como la solubilización de fósforo como reflejo de la actividad para reducir el “damping off” fueron analizados identificando diferencias (al 95% de confiabilidad.) después de identificar las fuentes de variación relevantes utilizando InfoStat (Di Rienzo, 2014).
Resultados y discusión
Aislamientos microbianos. Se aislaron en total 50 cepas de rizobacterias, de las cuales se seleccionaron 18 tomando como criterio su velocidad de crecimiento en un período de 24 h. Por otra parte, se aislaron tres cepas fúngicas provenientes de plántulas de chile, las cuales pertenecen al género Fusarium, con base en su pigmentación del micelio fúngico y del medio de cultivo donde fueron cultivados, se les asignó la nomenclatura FP, FN y FA (Fusarium púrpura, F. naranja y F. amarillo, respectivamente).
Determinación del antagonismo microbiano in vitro. Los resultados obtenidos en cultivo dual mostraron que la cepa B23 fue estadísticamente la más eficiente, al inhibir el crecimiento micelial de FP (50.02%), FN (44.05%), FA (46.67%) y Rhizoctonia spp. (R; 55.56%) (Figura 1). Estos resultados son similares a los reportados por Ríos-Velasco et al. (2016), quienes reportan un porcentaje de inhibición del crecimiento de F. oxysporum de 42 y 51.5% por efecto de B. methylotrophicus y B. amyloliquefaciens, respectivamente, así como los encontrados por Mojica-Morín et al. (2009), al confrontar F. oxisporum con las cepas de B. thuringiensis GM-23 (43.02%) y HD-121 (42.04%) y observaron porcentajes de inhibición más altos para R. solani por efecto de las cepas GM-64 (66.66%) y HD-203 (65.99%). Por su parte, Djordjevic et al. (2011), al evaluar 16 aislamientos bacterianos como antagonistas de F. oxisporum, reportan un porcentaje de inhibición más alto (70.98%) sobre el crecimiento de este hongo con 1a cepa Ab23, con resultados similares a los encontrados en el presente estudio con los aislamientos Ab9 (59.74%), Ab17 (57.7%) y Ab1 (56.6%).
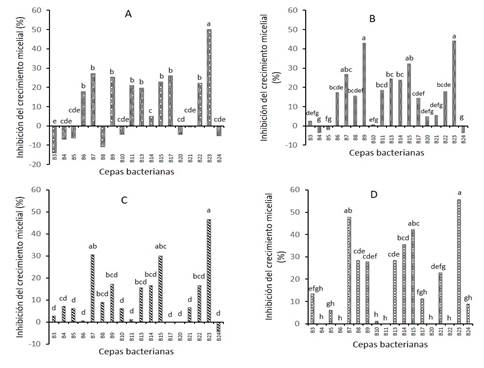
Figura 1 Efecto inhibitorio de 18 cepas bacterianas sobre el crecimiento micelial de cuatro hongos fitopatógenos. A) Fusarium púrpura, B) Fusarium naranja, C) Fusarium amarillo y D) Rhizoctonia. Los valores negativos indican estimulación del crecimiento.
En la Figura 1A se puede observar que las cepas B6; B13; B11; B22; B15; B9; B17 y B7 ejercen un efecto antagónico estadísticamente igual sobre Fusarium púrpura (FP), pero diferente al resto de las cepas bacterianas estudiadas; B23 inhibió en 50% a FP. Al contrastar las 18 cepas bacterianas con Fusarium naranja (FN) se encontró que las cepas B23 y B9 presentaron un porcentaje de inhibición de 44.05 y 42.86%, respectivamente que fue superior al resto de los tratamientos (Figura 1B).
Mientras que las cepas B7 y B15 inhibieron el crecimiento fúngico de Fusarium amarillo (FA) en 30%, y con la cepa B23 casi llega a 50% (Figura 1C). Por otra parte, se encontraron porcentajes de inhibición del crecimiento Rhizoctonia de 47.7, 42.2 y 35.5% al confrontar las cepas B7, B15 y B14 respectivamente (Figura 1D). Finalmente, las cepas B3, B4, B5, B10, B20, B24 no tuvieron ningún efecto inhibitorio en los cuatro fitopatógenos estudiados, lo cual indica que estas cepas no fueron capaces de producir una cantidad suficiente de sustancias que puedan inhibir el crecimiento de los patógenos, por lo que no se alcanza un umbral que les permita exhibir una respuesta antagónica (Figueroa-López et al., 2016.
Está bien documentado que el efecto antagónico de las rizobacterias sobre la inhibición del crecimiento de fitopatógenos es debido a la síntesis de metabolitos secundarios (Khannous et al., 2014). Trivedi et al. (2008) demostraron que metabolitos volátiles producidos por Pseudomonas corrugata juegan un papel predominante en la inhibición de los hongos F. oxysporum y A. alternata, mientras que los metabolitos difusibles solo tienen un rol secundario. Por otra parte, se ha demostrado que algunas especies de Pseudomonas producen enzimas líticas, las cuales pueden romper las membranas celulares de algunos hongos fitopatógenos; las más conocidas son: quitinasas, celulasas, proteasas y β-glucanasas 1,3 (Compant et al., 2005).
Producción de ácido indolacético (AIA). La producción de AIA es una propiedad de algunas rizobacterias para estimular y facilitar el crecimiento vegetal (Mohite, 2013). Esta auxina es un producto derivado del metabolismo del L-triptófano producido por varios microorganismos del suelo, incluyendo las PGPR (Goswami et al., 2015). En el presente trabajo, al analizar la producción de AIA de 18 aislamientos bacterianos provenientes de la rizósfera de maíz se encontró una variación de 1.3 a 11.2 µg mL-1. La producción de AIA significativamente más alta (p≤ 0.05) correspondió a B14 y las más baja a B4 (Figura 2).
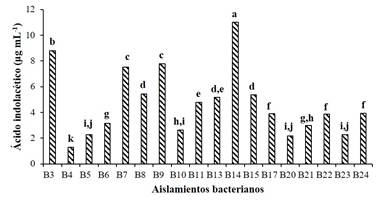
Figura 2 Producción de ácido indolacético de 18 aislamientos bacterianos provenientes de la rizósfera de maíz. Medias con letras diferentes indican diferencias significativas (Tukey 0.05).
Estos resultados concuerdan con los reportados en varias investigaciones, donde se evaluó la producción de AIA en cepas bacterianas provenientes de la rizósfera de maíz. Noh et al. (2014) mencionan una producción de 3.3 µg mL-1 de AIA. Por su parte, Josic et al. (2012) señalan que al adicionar 5mM de triptófano al medio líquido King B, la concentración de AIA se incrementó de 8.6 a 26 µg mL-1 en la cepa Q4m (Pseudomonas sp.). Se ha observado que la producción de AIA por rizobacterias varía entre diferentes especies y cepas; lo que está influenciada por las condiciones de cultivo, etapa de crecimiento y disponibilidad de sustrato (Mirza et al., 2001).
De acuerdo a la producción de AIA in vitro, las rizobacterias se pueden dividir en tres grupos: bajas (1 a 10 µg mL-1), medianas (11 a 20 µg mL-1) y altas productoras (21 a 30 µg mL-1) de AIA (Rojas et al., 2010). De acuerdo a esta clasificación, la mayoría de las cepas analizadas en el presente estudio se clasifican como bajas productoras de ácido indolacético. Sin embargo, no todas las bacterias que producen alta cantidad de AIA estimulan el crecimiento de las plantas (Noh et al., 2014). Por ejemplo, Agrobacterium tumefaciens produce hasta 78 µg mL-1 de AIA, lo cual induce el desarrollo de la enfermedad conocida como “agalla de la corona” (Lara et al., 2011). Mientras que concentraciones más bajas (1.1 a 12.1 µg mL-1) incrementaron el crecimiento de plantas de trigo in vitro (Khalid et al., 2004).
Ramírez-Malagón y Ochoa-Alejo (1996), en un estudio in vitro demostraron que el AIA tiene un efecto sobre la inducción y crecimiento de brotes adventicios del cultivo de chile, siendo su concentración la que determina el tipo de respuesta. Peña et al. (2016) reportan una producción de AIA, entre 4.0 a 24.3 µg mL-1 en cinco especies de Bacillus, la cual es suficiente para promover el crecimiento de Capsicum annuum L. Por lo anterior, en el presente estudio ocho cepas pueden considerarse promisorias para promover el crecimiento del cultivo de chile.
Solubilización de fósforo inorgánico. La cantidad de fosfato inorgánico (Pi) disuelto en el medio de cultivo varió entre los diferentes aislamientos (Figura 3). La cepa B8 fue la que solubilizó la cantidad más alta de Pi (100.4 µg mL-1), seguida de la cepa B14, con la que se registraron 80.92 µg mL-1 y luego B17 y B21 (60.67 y 56.39 µg mL-1, respectivamente). La cantidad de fosfato solubilizado por el resto de las cepas, con excepción de B10 y B9 (efecto nulo), se encontró en el rango de 2.21 a 49.12 µg mL-1. Los resultados registrados en las cepas B8, B14 y B17 son superiores a los reportados por Nautiyal (1999), quien encontró una variación de 8 a 35 µg mL-1 en cinco especies de Pseudomonas y de 8 a 17 µg mL-1 en tres especies de Bacillus.

Figura 3 Fosfato solubilizado por los aislamientos bacterianos. Columnas con lletras diferentes indican diferencias significativas (Tukey 0.05).
Se ha señalado que las bacterias solubilizan el Pi por medio de la producción de CO2, ácidos orgánicos, excreción de protones y asimilación de NH4 +. El mecanismo más importante de solubilización de fosfato de calcio es la acidificación por medio de la biosíntesis y secreción de ácidos orgánicos (Paredes y Espinosa, 2009). Tales ácidos orgánicos pueden directamente disolver el Pi como resultado del intercambio de aniones de fosfato mediante un anión ácido o bien al quelatar los iones de Fe, Al y Ca asociados con P (Omar 1998), lo que en consecuencia incrementa la biodisponibilidad de P, que puede ser aprovechado por los cultivos o la flora silvestre presente.
Supervivencia de plántulas. Al evaluar el efecto de las cepas B7, B9, B15 y B23 sobre la supervivencia de plántulas de chile inoculadas con Fusarium spp (FA, FP y FN) y Rhizotocnia sp. (R), se observa en la Figura 4 (a, b y d) el 100% de supervivencia en plántulas inoculadas con B23 y una supervivencia en las plantas control de FP, FA y R de 80, 77.8 y 83%, respectivamente (solo inoculadas con el patogéno) a los ocho días. En este mismo período, también se observó 100% de supervivencia al inocular B9 y B15 para el caso de FP (4a) y B15 para el caso de Rhizoctonia (Figura 4d). Se observa que, de 34 a 60 días existe una disminución en la supervivencia de las plántulas control en comparación con las plántulas inoculadas con rizobacterias. Por otra parte, solo las cepas B7 y B23 presentan un efecto sobre la sobrevivencia de plántulas infectadas por FA hasta los 55 días (Figura 4b), esta supervivencia coincide con lo reportado por Abdel-Monaim (2013) quien obtuvo 72% y 76.34% de supervivencia por Bacillus megaterium en plantas de frijol en dos estaciones diferentes, cuyas plantas estaban infestadas con los patógenos R. solani, F. solani, F. oxysporum y M. phaseolina causandole damping off a la planta.
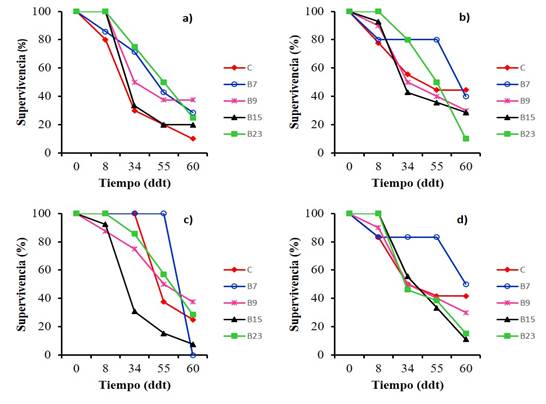
Figura 4 Efecto de cuatro bacterias antagonistas en la supervivencia de plántulas de chile “guajillo” inoculadas con diferentes hongos fitopatógenos. a) Fusarium púrpura; b) F. amarillo; c) F. naranja; y d) Rhizoctonia.
Con respecto, al hongo FN (Figura 4c), tanto en plántulas control como en las plántulas inoculadas con B7 y B23, se registró 100% de supervivencia. Esta misma tendencia se mantiene hasta los 34 días para el caso de plántulas control y las inoculadas con B7. El comportamiento de las plántulas control puede ser debido a que la cantidad de micelio fúngico no fue suficiente para que este hongo exprese virulencia en los primeros 34 días o bien a que su efecto para causar la enfermedad sea más tardío que los otros patógenos utilizados en el presente estudio.
Finalmente, para el caso de Rhizotocnia spp, la cepa B7 presentó un efecto sobre la supervivencia de las plántulas, debido a que esta fue de 80% hasta los 55 días, mientras que las plántulas control fue 41.4% (Figura 4d). Al respecto, Toppo y Tiwari (2015), reportan una variación en la supervivencia de plántulas de tomate (30 días) infectadas con Rhizoctonia la cual varió entre 93.33 a 46.67.6% al inocular Pseudomonas alcaligenes PKJ25 y Pseudomonas PKS10. En el presente estudio, a 34 días la mayor supervivencia fue de 83.3 y la menor 46.2% al inocular B7 y B23, respectivamente.
Identificación molecular de rizobacterias y hongos. Los aislados B8 y B9 se identificaron como Pseudomonas chlororaphis; B7 y B14 como Pseudomonas sp. y B15 como Pseudomonas protegens. Por su parte, B23 fue identificado como Bacillus sp. Todos presentaron una identidad 99%, con una cobertura de 100%. El género Pseudomonas está compuesto por bacterias gram negativas, que pertenecen a las γ-Proteobacteria, se encuentran distribuidas en una amplia diversidad de ambientes y son conocidas por su versatilidad metabólica en la utilización de compuestos orgánicos como fuentes de carbono y de energía (Lessie and Phibbs, 1984; Wu et al., 2011) y en la producción de diversos metabolitos secundarios (Gross y Loper, 2009). Pseudomonas es uno de los grupos bacterianos más complejos, con más de 100 especies descritas hasta el momento, en el cual hay la presencia de grupos y subgrupos.
El grupo de Pseudomonas flourescens contiene varias especies (P. brassicacearum, P. protegens, P. chlororaphis y P. fluorescence), descritas como PGPR por su habilidad para suprimir patógenos causantes de enfermedades (Raajimakers et al., 2009) y producir antibióticos o metabolitos secundarios (Gross y Loper, 2009) que las hacen muy viables para aplicaciones biotecnológicas (Saravanakumar et al., 2008). La identificación bacteriana a través del gen 16S ribosomal ha sido ampliamente utilizada; sin embargo, es bien sabido que especies bacterianas cercanas no pueden ser diferenciadas mediante el uso de este gen debido a su alta tasa de conservación (Fox et al., 1992; Kämpfer and Glaeser, 2012), por lo que se ha propuesto el empleo del análisis de secuencias multilocus mediante el uso de otros genes como marcadores moleculares: rpoB, rpoD, gyrB (Yamamoto et al., 2000; Ait Toyeb et al., 2005), permitiendo la identificación hasta el nivel de especie.
En este trabajo, al utilizar el gen 16S rRNA como único marcador molecular, permitió designar hasta nivel de género los aislados obtenidos, los cuales pertenecen a Pseudomonas y Bacillus. Miembros de estos géneros han sido reportados como PGPR, con diferentes mecanismos como producción de fitohormonas, supresión de patógenos o productoras de metabolitos secundarios de interés biotecnológico. Por otra parte, los aislamientos fúngicos FP y FN corresponden a Fusarium sp.; el primero con 93% de identidad y cobertura 84% y el segundo con 86% de identidad y 80 de cobertura. F. amarillo corresponde a un hongo no cultivado, con 94% de identidad y 33% de cobertura. Fusarium sp pertenece a la familia Nectriaceae. El género Fusarium es un grupo de hongos filamentosos ampliamente distribuidos en el suelo y plantas. Debido a su capacidad de crecer a 37 °C, son considerados oportunistas (Tapia y Amaro, 2014). Santos (2010) menciona a Fusarium sp. como uno de los principales agentes causales de la marchitez del chile que disminuye el rendimiento.
Conclusiones
La cepa B23 resultó ser la más eficiente en la inhibición del crecimiento de los cuatro hongos patógenos evaluados, Rhizoctonia (56%), F. púrpura (50%), F. amarillo (47%) y F. naranja (44%), mientras que la cepa B14 resultó ser la más sobresaliente en la producción de AIA (11.2 µg mL-1). Por otra parte, la cepa B8 mostró la más alta solubilización de fosfato (104.4 µg mL-1). Las bacterias aisladas de la rizósfera de maíz, en una parcela con cultivo previo de chile, tienen capacidad antagonista contra cuatro cepas de hongos fitopatógenos, además de que muestran actividad promotora de crecimiento (producción de AIA y solubilización de fósforo inorgánico).
Sobre la supervivencia de plántulas de chile, la cepa B7 mantuvo la mayor supervivencia de plántulas en el tiempo, mientras que las inoculadas con B15 fueron las que murieron en mayor proporción y en menor tiempo. Los resultados de este estudio sugieren que las enfermedades causadas por Fusarium o Rhizoctonia pueden ser efectivamente manejadas a través del uso de bacterias antagonistas como la B23, o la promoción del crecimiento de las plantas mediante el uso de cepas bacterianas como la B14 y la B8, disminuyendo daños al ambiente y a la salud. Además, el uso de microorganismos solubilizadores de fosfatos como inoculante en semillas y plántulas puede representar una alternativa biotecnológica para incrementar la disponibilidad de fósforo para los cultivos.











 texto em
texto em 


