Introducción
El amaranto es una planta herbácea anual predominantemente tropical que pertenece al género Amaranthus. Éste género contiene más de 70 especies de las cuales la mayoría son nativas de América y sólo 15 provienen de Europa, Asia, África o Australia. En EE. UU y México existen aproximadamente 29 especies. El género Amaranthus se distribuye ampliamente en América, mostrando gran variabilidad genética, misma que se aprecia al observar características de la planta como el tipo de inflorescencia, color de semilla, precocidad, contenido proteínico de la semilla y resistencia a plagas y enfermedades (Akaneme and Ani, 2013; Erum et al., 2012; Mwase et al., 2014). Es un cultivo fácil de establecer ya que prospera en regiones de temporal donde los cultivos básicos tienen poco éxito, se adapta a varios tipos de suelo, altitudes, temperaturas y fotoperiodos, así como a diversos requerimientos de pH y precipitación (Espitia et al., 2010).
Dentro del género se encuentran las especies A. cruentus, A. hypochondriacus y A. caudatus que son las más importantes para la producción de grano de amaranto. Las dos primeras se encuentran ampliamente distribuidas en México, conformando el centro de diversidad más importante (Espitia et al., 2010; Ruiz et al., 2013).
México es considerado un país mega-diverso por su elevado número de especies, pero también por su riqueza de endemismos (especies exclusivas de México), de ecosistemas y por la gran variabilidad genética mostrada en muchos grupos taxonómicos, resultado de la evolución o diversificación natural y cultural en el país, en su territorio se encuentran entre 4 y 8% del número de total de especies de plantas del mundo, de las cuales 51% son endémicas. Por esta razón existe una gran preocupación sobre la conservación de la flora mexicana, ya que se han detectado varias actividades y factores que la amenazan (Sosa y De-Nova, 2012).
La variabilidad genética en sentido amplio es el componente básico de la biodiversidad y se define como las variaciones heredables que ocurren en cada organismo, entre los individuos de una población y entre las poblaciones dentro de una especie. En la caracterización de una especie se estima la variabilidad existente en el genoma de la población de individuos que la conforman (Piñero, 2008).
El estudio de la diversidad genética dentro y entre las poblaciones y especies de Amaranthus es importante para planear estrategias de conservación para la continuidad de dichas especies y poblaciones, así como para satisfacer las demandas de los entornos cambiantes a través del mejoramiento genético del amaranto y esto dependerá en gran medida de la magnitud de la variabilidad disponible en su acervo genético. Por lo anterior, el objetivo de la presente investigación fue analizar la diversidad genética dentro y entre las poblaciones y especies de Amaranthus, así como comparar identidades genotípicas entre las accesiones de las siete especies mexicanas en estudio.
Material vegetal
Se estudiaron 2 especies cultivadas y 5 especies silvestres de amaranto, representadas por 13 accesiones nacionales de amaranto, previamente identificadas morfológicamente y provenientes del programa de Recursos Genéticos del INIFAP, Campo Experimental Valle de México ubicado en Coatlinchán, Texcoco, México (Cuadro 1). Las colectas y las estrategias de muestreo que se siguieron para las mismas se determinaron en base al mandato del Banco de Germoplasma y a los objetivos de la recolección. De cada accesión se evaluaron cinco individuos para el análisis de diversidad genética.
Cuadro 1 Accesiones utilizadas para el análisis de la diversidad genética en poblaciones de Amaranthus.
| Especie | Accesión | Abreviatura | Tipo de sexo |
| A. hypochondriacus | Tlax-089 | 89HYP | Monoico |
| Pue-027 | 27HYP | ||
| A. cruentus | Gto-092 | 92CRU | Monoico |
| Mor-042 | 42CRU | ||
| A. hybridus | Silv-39 | 39HYB | Monoico |
| Silv-43 | 43HYB | ||
| A. retroflexus | Silv-26 | 26RETR | Monoico |
| Silv-38 | 38RETR | ||
| A. powellii | Silv-49 | 49POW | Monoico |
| Silv-50 | 50POW | ||
| A. palmerii | Silv-27 | 27PALM | Dioico |
| Silv-35 | 35PALM | ||
| A. spinosus | Silv-47 | 47SPIN | Monoico |
Purificación del ADN
La extracción del ADN se hizo de acuerdo al protocolo de De la Cruz et al. (1997), con algunas modificaciones. Se tomaron hojas jóvenes (0.3 g) y se maceraron en nitrógeno líquido. El macerado se transfirió a un micro tubo (Eppendorf) de 1.5 mL que contenía 600 μL de amortiguador de extracción (20 mL Tris-HCL 1 M, pH 8; 20 mL EDTA 0.5 M, pH 8; 20 mL NaCl 5 M; 35 μL β-Mercaptoetanol; 40 mL dodecil sulfato de sodio 20%) y se incubó a 65 °C por 10 min en un Standard Heatblock de VWR®Scientific Products, con inversión ocasional de los tubos. Después, se adicionó 200 μL de acetato de potasio 5M, se mezcló por inversión y se incubó por 30 min a 0 °C.
Se centrifugó a 15 000x g durante 10 min a temperatura ambiente, y el sobrenadante se transfirió a otro tubo que contenía 700 μL de isopropanol frío (-20 °C). Se mezcló por inversión e incubó a -20 °C por 30 min, después se centrifugó por 5 min a 10 000x g a temperatura ambiente. Se eliminó el sobrenadante y el precipitado se disolvió en 200 μL de solución para diluir (Tris-HCl 50 mM, EDTA-Na2 10 mM, pH 8), se agitó suavemente hasta diluir bien el precipitado. Se añadió 2 μL de ARNasa A y se incubó a 37 °C por 1 h en una incubadora Felisa®.
Después se adicionó 20 μL de acetato de sodio 3M más 200 μL de isopropanol, se mezcló por inversión y se dejó precipitar a -20 °C por 2 h. Se centrifugó a 10 000x g por 5 min a temperatura ambiente. El sobrenadante se eliminó y el precipitado se lavó con 300 μL de etanol al 70%. La pastilla se secó y se disolvió en amortiguador TE (Tris-HCl 10 mM, EDTA-Na2 1 mM, pH 8) y por último se almacenó a 4 °C. La concentración del ADN se determinó usando un espectrofotómetro Genesys 10 UV Scanning® (Thermo Scientific) y la calidad del mismo se observó mediante electroforesis en un gel de agarosa al 0.8%. El ADN se utilizó en subsecuentes reacciones de PCR.
Reacción de PCR
La concentración de ADN de cada muestra se ajustó a 10 ng μL-1 y para la amplificación se usaron 10 iniciadores previamente seleccionados (Cuadro 2). Las reacciones de amplificación se realizaron en un volumen de 25 μL, que incluyó 3.2 μL de agua doble destilada estéril, 10 μL de dNTPs (500 μM), 2.5 μL de amortiguador 10X (Tris-HCl 750 mM, pH 8.8; (NH4)2S04 200 mM; Tween 20 a 1% (v/v)); 2 μL de MgCl2 (25 μM); 3 μL de iniciador a una concentración de 10 pM; 0.3 μL de enzima Taq ADN polimerasa a una concentración de 5U μL-1 y 4 μL de ADN genómico a una concentración de 10 ng μL-1. Las reacciones de PCR se realizaron en un termociclador Techne®28 TC-512. Las condiciones de reacción fueron: un paso de desnaturalización inicial a 94 °C por 3 min, 30 ciclos con desnaturalización a 94 °C por 1 min, alineamiento a 50 °C por 1 min, y extensión a 72 °C por 2 min y finalmente un ciclo de extensión final a 72 °C por 10 min.
Cuadro 2 Lista de iniciadores, su secuencia, número de productos amplificados y porcentaje de polimorfismo detectados.
| Iniciador ISSR | Secuencia | Productos amplificados | Polimorfismo (%) |
| A3 | 5’-CTCCTCCTCCTCCTCCTC- 3’ | 17 | 100 |
| A8 | 5’-AGAGAGAGAGAGAGAGT- 3’ | 21 | 100 |
| A10 | 5’-GAGAGAGAGAGAGAGA- 3’ | 17 | 94.1 |
| P2 | 5’-CTGAGAGAGAGAGAGAGAG- 3’ | 16 | 93.7 |
| P3 | 5’-AGAGAGAGAGAGAGAGTG- 3’ | 13 | 100 |
| IAH-01 | 5’-ACACACACACACA- 3’ | 16 | 100 |
| IAH-02 | 5’-GTGTGTGTGTGTGG- 3’ | 16 | 100 |
| ISSR 01 | 5’-AGGAGGAGGAGGAGGAGG- 3’ | 12 | 91.6 |
| ISSR 04 | 5’-GAGAGAGAGAGAGAGAGAC- 3’ | 6 | 100 |
| (AG) 8YA | 5’-AGAGAGAGAGAGAGAGYA- 3’ | 20 | 100 |
Los fragmentos amplificados se separaron por electroforesis en geles de agarosa a una concentración de 1.2% (p/v) con amortiguador TAE (40 mM Tris-acetato, pH 7.6; 1 mM Na2 EDTA), por 1 h a 120 V. Para el corrimiento de las muestras se utilizaron 12 μL del producto de la reacción de PCR agregando 1 μL de amortiguador de carga (0.1% azul de bromofenol, 0.1% xylen cianol, 30% de Ficoll y 1% de SDS). Como referencia se utilizó el marcador 1kb ADN ladder plus (GeneRuler). Los geles se tiñeron con bromuro de etidio (0.5 mg mL-1) por 5 min y el exceso de colorante se eliminó enjuagando en agua 5 min y se documentaron con ayuda del equipo Kodak® High Perfomance Ultraviolet Transilluminator EDAS-290, bajo luz UV.
Análisis de los datos
Los materiales se compararon en base a similitudes y diferencias en los patrones de bandeo, asignando un valor de 1 a la presencia de una banda y de 0 a la ausencia de la misma, asumiendo que bandas de igual peso molecular son idénticas entre poblaciones y especies, construyéndose una Matriz Básica de Datos (MBD). Con el uso del programa POPGENE 32 (Yeh et al., 1999), se estimaron los parámetros siguientes: porcentaje de loci polimórficos para las especies y las accesiones consideradas como poblaciones, el coeficiente de diversidad genética de Nei, el índice de información de Shannon, el parámetro GST o coeficiente de diferenciación genética entre especies y entre poblaciones, y el Nm o número de migrantes efectivos, así como la matriz de distancias e identidades genéticas de Nei (1973).
Además, con el software NTSYS-pc versión 2.2 (Rohlf, 2000) se visualizó el dendrograma generado en el programa POPGENE 32. Por último, se cuantificó la variación presente entre las especies y poblaciones mediante el análisis molecular de variancia (AMOVA) usando el programa GenAlEx 6.5 (Peakall and Smouse, 2006) con 999 permutaciones.
Resultados
Se estudiaron 154 loci generados utilizando diez iniciadores tipo ISSR para amplificar el ADN de cinco individuos de cada accesión. En las Figuras 1A y 1B, se muestra un ejemplo de los patrones obtenidos usando el iniciador P2. El promedio de bandas generadas por iniciador fue de 15.4, con un rango de 6 a 21. El porcentaje de polimorfismo promedio para los iniciadores fue de 97.9%, con un mínimo de 91.6% y un máximo de 100% (Cuadro 2).
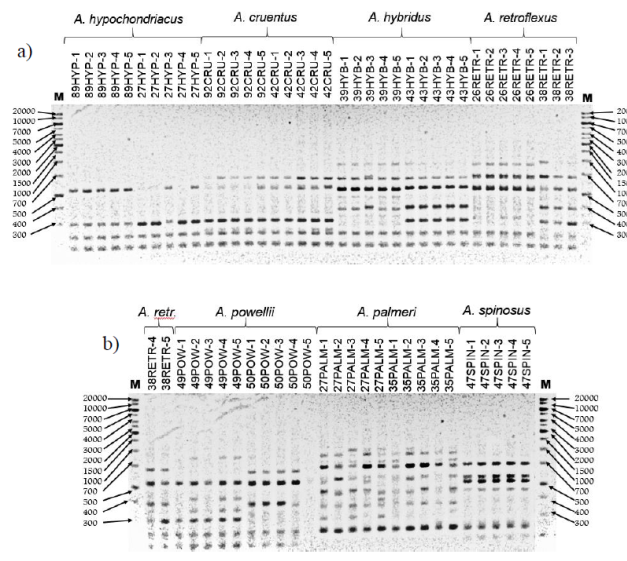
Figura 1 a)patrones de bandas obtenidos con el iniciador P2 para una muestra de cinco individuos de las poblaciones de A. hypochondriacus, A. cruentus, A. hybridus y A. retroflexus. b) patrones de bandeo obtenidos con el iniciador P2 para una muestra de cinco individuos de las poblaciones de A. retroflexus, A. powellii, A. palmerii y A. spinosus. M= marcador de peso molecular, pb.
Diversidad genética en 13 poblaciones de amaranto
En la Figura 2, se muestra el dendrograma de identidad donde se establecen las relaciones genéticas entre trece poblaciones de amaranto correspondientes a siete especies y utilizando el coeficiente de Nei. A un coeficiente de identidad de 0.718 se observa la formación de dos grupos.

Figura 2 Dendrograma de relaciones genéticas entre poblaciones y especies de amaranto, construido usando el coeficiente de identidad de Nei.
El grupo I se dividió en cuatro subgrupos. El primer sub-grupo incluyó a las especies cultivadas A. hypochondriacus y A. cruentus, donde los materiales más idénticos fueron los que pertenecen a la especie de A. cruentus (92CRU, 42 CRU), seguido de los materiales de A. hypochondriacus (89HYP, 27HYP). Los materiales más distantes dentro de este sub-grupo fueron A. hypochondriacus (89HYP) y A. cruentus (42CRU). El segundo sub-grupo se formó con accesiones de A. hybridus y A. retroflexus. El tercer sub-grupo incluyó a materiales de A. powellii (49POW) y A. retroflexus (38RETR), mientras que el cuarto sub-grupo solo integró a un material de A. powellii (50POW).
El grupo II incluyó a amarantos dioicos de A. palmerii y el material monoico con presencia de espinas de A. spinosus, donde las accesiones más similares son las que pertenecen a A. palmerii (27PALM y 35PALM), mientras que los más distantes fueron 27PALM de A. palmerii y 47SPIN de A. spinosus.
Los resultados anteriores sugieren que los amarantos cultivados A. hypochondriacus y A. cruentus están muy relacionados entre sí y luego medianamente relacionados con los amarantos silvestres A. hybridus, A. retroflexus y A. powellii, en el orden indicado, y menos relacionados con las especies A. palmerii y A. spinosus.
En lo referente a los amarantos silvestres se observó que las especies genéticamente más relacionadas fueron A. hybridus con A. retroflexus y A. powellii, y A. palmerii con A. spinosus. Entre éstas, las menos relacionadas fueron A. palmeriii y A. spinosus.
En el Cuadro 3, se muestran los índices de diversidad calculados dentro de 13 poblaciones de amaranto. Se observa que la mayoría de las poblaciones tienen porcentajes de loci polimórficos bajos, con un promedio de 13.18% y un rango que va de 3.25% para la accesión 26RETR, perteneciente a la especie A. retroflexus a 30.52% de la accesión 27PALM perteneciente a la especie A. palmerii.
Cuadro 3 Índices de diversidad genética dentro de 13 poblaciones del género Amaranthus.
| Especie | Accesión | NA | NE | H | I | NLP | LP (%) | |
| A. hypochondriacus | 89HYP |
|
1.1039 | 1.093 | 0.0487 | 0.0687 | 16 | 10.39 |
| σ | 0.3061 | 0.278 | 0.1443 | 0.2029 | ||||
| 27HYP |
|
1.1364 | 1.0997 | 0.0549 | 0.0799 | 21 | 13.64 | |
| σ | 0.3443 | 0.2737 | 0.1453 | 0.2076 | ||||
| A. cruentus | 92CRU |
|
1.1234 | 1.1144 | 0.0586 | 0.0822 | 19 | 12.34 |
| σ | 0.3299 | 0.3116 | 0.1586 | 0.2214 | ||||
| 42CRU |
|
1.0649 | 1.051 | 0.0274 | 0.0393 | 10 | 6.49 | |
| σ | 0.2472 | 0.207 | 0.1084 | 0.1536 | ||||
| A. hybridus | 39HYB |
|
1.0455 | 1.0334 | 0.0181 | 0.0263 | 7 | 4.55 |
| σ | 0.209 | 0.168 | 0.0881 | 0.1253 | ||||
| 43HYB |
|
1.0584 | 1.0446 | 0.024 | 0.0346 | 9 | 5.84 | |
| σ | 0.2353 | 0.1952 | 0.1013 | 0.1438 | ||||
| A. retroflexus | 26RETR |
|
1.0325 | 1.0192 | 0.0111 | 0.0168 | 5 | 3.25 |
| σ | 0.1778 | 0.1213 | 0.0658 | 0.0961 | ||||
| 38RETR |
|
1.1623 | 1.0964 | 0.0584 | 0.0877 | 25 | 16.23 | |
| σ | 0.37 | 0.2385 | 0.1382 | 0.2046 | ||||
| A. powellii | 49POW |
|
1.1104 | 1.0811 | 0.0451 | 0.0656 | 17 | 11.04 |
| σ | 0.3144 | 0.2464 | 0.1328 | 0.1906 | ||||
| 50POW |
|
1.1948 | 1.1573 | 0.085 | 0.1216 | 30 | 19.48 | |
| σ | 0.3973 | 0.3358 | 0.1774 | 0.2517 | ||||
| A. palmerii | 27PALM |
|
1.3052 | 1.2253 | 0.1245 | 0.1808 | 47 | 30.52 |
| σ | 0.462 | 0.3727 | 0.1977 | 0.2819 | ||||
| 35PALM |
|
1.2987 | 1.2014 | 0.1132 | 0.1667 | 46 | 29.87 | |
| σ | 0.4592 | 0.3536 | 0.1877 | 0.2686 | ||||
| A. spinosus | 47SPIN |
|
1.0779 | 1.0511 | 0.0287 | 0.0425 | 12 | 7.79 |
| σ | 0.2689 | 0.2001 | 0.1062 | 0.153 |
NA= número de alelos por locus; NE= número efectivo de alelos por locus; H= diversidad genética de Nei’s; I= índice de Shannon; NLP= número de loci polimórficos; LP (%)= porcentaje de loci polimórficos.
Los índices de Shannon (I) y diversidad genética de Nei (H) (Cuadro 3) fueron también bajos para las poblaciones. Los índices de Shannon tuvieron un promedio de 0.077, con rango de 0.0168 para la accesión 26RETR de A. retroflexus y 0.1808 para la accesión 27PALM de A. palmerii, mientras que el índice de diversidad de Nei (H) mostró un promedio de 0.054, con un rango de 0.011 para la accesión 26RETR de A. retroflexus y de 0.124 para la accesión 27PALM de A. palmerii.
Para el número de alelos por locus (NA) y el número efectivo de alelos por locus (NE) el rango de valores entre las poblaciones estuvo entre 1.032 a 1.305 y 1.019 a 1.225, respectivamente. El análisis de diversidad genética entre las trece poblaciones mostró que el porcentaje de loci polimórficos (LP %) fue de 99.35%. El coeficiente de diferenciación genética entre las poblaciones fue alto (GST= 0.82), e indica que aproximadamente 82% de la variación detectada puede atribuirse a diferencias genéticas entre las poblaciones. El resto (18%) representa diversidad genética dentro de las poblaciones. Con base al coeficiente de diferenciación genética total entre las poblaciones (GST) el nivel de flujo génico estimado (Nm) fue de 0.11. Esto indica que existe menos de un individuo migrante por generación entre las poblaciones, lo que también explica el alto nivel de diferenciación detectado entre las mismas.
Diversidad genética en siete especies de amaranto
Las especies genéticamente más cercanas fueron las silvestres A. hybridus y A. retroflexus con un coeficiente de 0.93, siguiéndoles A. powellii con un coeficiente de 0.87. Las especies cultivadas estuvieron genéticamente cercanas entre sí, aunque relacionadas con las especies silvestres antes mencionadas. A. hypochondriacus se relacionó con A. cruentus a un coeficiente de identidad genética de Nei igual a 0.86. Además, las especies silvestres A. palmerii y A. spinosus, relacionadas entre sí, estuvieron muy alejadas de los amarantos cultivados A. hypochondriacus y A. cruentus, así como de los amarantos silvestres A. hybridus, A. retroflexus y A. powellii.
En lo que se refiere a los índices de diversidad genética calculados dentro de especies, en el Cuadro 4 se observa que el porcentaje de loci polimórficos fue bastante bajo para las siete especies evaluadas, con un promedio de 24.02% y un rango de valores que va de 7.79% para A. spinosus y de 44.16% para A. palmerii.
Cuadro 4 Índices de diversidad genética dentro de siete especies del género Amaranthus.
| Especie | NA | NE | H | I | NLP | LP (%) | |
| A. hypochondriacus |
|
1.1948 | 1.1254 | 0.071 | 0.1049 | 30 | 19.48 |
| Σ | 0.3973 | 0.2916 | 0.1579 | 0.2271 | |||
| A. cruentus |
|
1.1429 | 1.102 | 0.0575 | 0.0837 | 22 | 14.29 |
| Σ | 0.3511 | 0.2671 | 0.1464 | 0.2108 | |||
| A. hybridus |
|
1.1623 | 1.1139 | 0.0656 | 0.0959 | 25 | 16.23 |
| Σ | 0.37 | 0.2695 | 0.1529 | 0.2218 | |||
| A. retroflexus |
|
1.2922 | 1.1732 | 0.1023 | 0.1534 | 45 | 29.22 |
| Σ | 0.4563 | 0.3107 | 0.1738 | 0.2531 | |||
| A. powellii |
|
1.3701 | 1.2404 | 0.1409 | 0.2085 | 57 | 37.01 |
| Σ | 0.4844 | 0.3433 | 0.1937 | 0.2817 | |||
| A. palmerii |
|
1.4416 | 1.2699 | 0.1574 | 0.2348 | 68 | 44.16 |
| Σ | 0.4982 | 0.3607 | 0.1972 | 0.2842 | |||
| A. spinosus |
|
1.0779 | 1.0511 | 0.0287 | 0.0425 | 12 | 7.79 |
| Σ | 0.2689 | 0.2001 | 0.1062 | 0.153 |
NA= número de alelos por locus; NE= número efectivo de alelos por locus; H= diversidad genética de Nei; I= índice de Shannon; NLP= número de loci polimórficos; (%) LP= porcentaje de loci polimórficos.
Los índices de Shannon (I) y de diversidad genética de Nei (H) también fueron bajos (Cuadro 4), los índices de Shannon mostraron un promedio de 0.132, con un rango de valores que va de 0.042 para la especie A. spinosus y de 0.235 para la especie A. palmerii, mientras que el índice de diversidad de Nei (H) mostró un promedio de 0.089, con un rango de 0.029 y 0.157.
Para el número de alelos por locus (NA) y el número efectivo de alelos por locus (NE) el rango de valores para las especies evaluadas estuvo entre 1.078 a 1.442 y 1.051 a 1.27, respectivamente.
El análisis de la diversidad genética entre las especies indicó un porcentaje de loci polimórficos (LP %) igual a 99.35%. El coeficiente de diferenciación genética entre las especies fue alto (GST= 0.7), e indica que aproximadamente 70% de la variación genética detectada puede atribuirse a diferencias genéticas entre las especies. El resto (30%) representa diversidad genética dentro de las especies. Con base al coeficiente de diferenciación genética total entre las especies (GST) el nivel de flujo génico estimado (Nm) fue de 0.21. Ello sugiere que hay menos de un individuo migrante por generación entre las especies, lo que también explica el alto nivel de diferenciación detectado entre las mismas.
Las especies genéticamente más cercanas fueron A. hybridus y A. retroflexus con un valor de identidad de 0.927, mientras que las especies más alejadas fueron A. spinosus y A. hybridus con un valor de 0.534.
Análisis de varianza molecular (Amova)
Los resultados del análisis de varianza molecular (Amova) indicaron diferencias genéticas significativas (p≤ 0.001) entre las poblaciones estudiadas (Cuadro 5). Entre las poblaciones se detectó un valor de variabilidad genética igual al 79%, mientras que 21% restante se encuentra dentro de las poblaciones. Al considerar especies se encontró que entre las mismas se presenta 68% de la variación genética total y solo 32% está dentro de las especies.
Cuadro 5 Análisis de varianza molecular (Amova) de especies y accesiones de amaranto.
| Fuente de variación | Análisis | df | SS | MS | Est. var. | Vt (%) | PhiPT | Valor Px |
| Entre accesiones | Entre | 12 | 1177.385 | 98.115 | 18.645 | 79 | 0.792 | *** |
| Dentro | 52 | 254.4 | 4.892 | 4.892 | 21 | |||
| Entre especies | Entre | 6 | 970.485 | 161.747 | 16.661 | 68 | 0.677 | *** |
| Dentro | 58 | 461.3 | 7.953 | 7.953 | 32 | |||
| Cultivadas vs silvestres | Entre | 1 | 171.212 | 171.212 | 5.46 | 21 | 0.214 | *** |
| Dentro | 63 | 1260.572 | 20.009 | 20.009 | 79 | |||
| Entre cultivadas | Entre | 1 | 119.85 | 119.85 | 11.531 | 72 | 0.718 | *** |
| Dentro | 18 | 81.7 | 4.539 | 4.539 | 28 | |||
| Entre silvestres | Entre | 4 | 679.422 | 169.856 | 18.041 | 66 | 0.655 | *** |
| Dentro | 40 | 379.6 | 9.49 | 9.49 | 34 | |||
| Entre dioicos vs monoicos | Entre | 1 | 284.948 | 284.948 | 15.762 | 46 | 0.464 | *** |
| Dentro | 63 | 1146.836 | 18.204 | 18.204 | 54 |
df= grados de libertad; SS= suma de cuadrados; MS= suma de cuadrados medios; vt (%)= porcentaje de variación; Px= probabilidad, * = 0.05, **= 0.01, ***= 0.001.
Al comparar los materiales cultivados contra los silvestres se encontró que la variación genética presente dentro de ambos tipos de materiales es muy baja con un valor de 21%, mientras que la variación entre los mismos fue muy alta (79%), lo que indica la existencia de variación genética susceptible de utilizar en programas de mejoramiento de los amarantos cultivados.
También, se encontró que la variación genética entre las poblaciones de amaranto cultivados fue 72%, en tanto solo 28% se encuentra dentro de las mismas, con lo cual también se demuestra el alto grado de diferenciación genética presente entre A. hypochondriacus y A. cruentus.
Cuando se compararon solo los materiales silvestres se tuvo que 66% de la variación genética se encuentra entre las poblaciones y solo 34% dentro de estas. Y por último al hacer la comparación entre los materiales silvestres dioicos contra los monoicos, los resultados indicaron que la variación genética presente entre las poblaciones es de 46% y el resto (54%) está dentro de las poblaciones.
Discusión
Los resultados de los análisis de agrupamiento del presente trabajo indican que los amarantos cultivados A. hypochondriacus y A. cruentus están muy relacionados entre sí y luego medianamente relacionados con los amarantos silvestres A. hybridus, A. retroflexus y A. powellii, y menos relacionados con las especies A. palmerii y A. spinosus (Figura 2). Pal y Khoshoo (1972) proponen a A. hybridus L. y A. powellii como los posibles progenitores de los amarantos cultivados, lo que puede explicar la cercanía genética que guardan estas especies.
En los amarantos silvestres se observó que las especies genéticamente más relacionadas fueron A. hybridus con A. retroflexus y A. powellii, y A. palmerii con A. spinosus. Entre éstas, las de menor porcentaje de similitud fueron A. palmerii y A. spinosus (Figura 2).
Los porcentajes más altos de polimorfismo se encontraron en los materiales silvestres de A. powellii (37.01%), A. palmerii (44.16%) y A. retroflexus (29.22%) (Cuadro 4) debido a que posiblemente dichos materiales se han visto menos expuestos a una presión de selección ya sea por factores del ambiente bióticos o abióticos o por el hombre mediante mejoramiento genético (Maughan et al., 2011). También, el tipo de sexo o polinización de las especies pudiera estar influyendo en la variabilidad genética presente (Štefúnová, 2014) ya que la especie dioica A. palmerii presentó el valor más alto de polimorfismo en comparación con el resto de las especies, que son monoicas (Cuadros 1 y 4).
El índice de diversidad genética de Nei (H) tuvo un promedio de 0.089 dentro de las poblaciones, con un rango de 0.029 a 0.157 (Cuadro 4), lo que significa baja variabilidad genética en las especies y las poblaciones estudiadas (Nei, 1973). Esta baja variabilidad genética presente dentro de las diferentes poblaciones y especies de amaranto se puede explicar por el alto porcentaje de autopolinización que ocurre en la planta (Agong y Ayiecho, 1991). El bajo nivel de polimorfismo detectado en Amaranthus puede reflejar el alto nivel de endogamia que existe en la especie (Htet y Park, 2013).
Se han realizado trabajos de hibridación interespecíficos tanto en materiales silvestres como materiales cultivados. Al hacerse cruzamientos entre especies de Amaranthus se ha tenido dificultad para la obtención de híbridos debido a la baja tasa de sobrevivencia de los mismos. Trucco et al. (2005) obtuvieron solo 5.9% de hibridación entre A. hybridus y A. tuberculatus, mientras que Gaines et al. (2012) al hacer cruzamientos entre A. palmerii y A spinosus obtuvieron 0.01% a 0.4% de hibridación, entre A. palmerii y A. tuberculatus menos de 0.2% de hibridación y entre A. palmerii y A. hybridus menos de 0.01% de hibridación.
Para las especies cultivadas A. hypochondriacus, A. cruentus y A. caudatus, se han reportado porcentajes de cruzamiento que varían del 5 a 30%. Al respecto, Agong y Ayiecho (1991) estiman entre 10.4% a 10.9% de hibridación entre A. hypochondriacus y A. cruentus. Con este bajo porcentaje de entrecruzamientos es posible deducir la alta tasa de diferenciación entre las diferentes especies y la baja variabilidad genética presente dentro de las poblaciones (Cuadro 5), porque el flujo génico entre las especies se ve muy limitado.
El coeficiente de diferenciación genética (GST) entre las poblaciones fue de 0.82 y entre las especies de 0.7. Estos valores son altos y se asemejan a los reportados por Chan y Sun (1997) para los amarantos de grano cultivados (A. caudatus, A. cruentus y A. hypochondriacus), quienes indican un valor medio de GST igual a 0.98 a nivel de especies. Para las especies silvestres A. hybridus, A. powellii y A. quitensis, los mismos autores reportan un valor de 0.96.
El número de individuos migrantes (Nm), que da una idea del grado de flujo génico, fue de 0.112 entre las poblaciones y de 0.214 entre las especies. Dichos valores indican que en promedio existe menos de un individuo migrante por generación entre las poblaciones y entre las especies, con lo cual se confirma el bajo porcentaje de cruzamiento y el alto grado de diferenciación entre las poblaciones y las especies.
Al comparar los materiales cultivados contra los silvestres se encontró que la variación genética presente dentro de ambos tipos de materiales es muy baja con un valor 21% (Cuadro 5), mientras que la variación entre los mismos fue muy alta (79%), lo que indica la existencia de variación genética susceptible de utilizar en programas de mejoramiento de los amarantos cultivados, así como para definir estrategias de conservación del género.
Conclusiones
Los amarantos de grano cultivados están genéticamente muy relacionados entre sí y con sus posibles progenitores silvestres A. hybridus y A. powellii. Dentro de los materiales silvestres los que estuvieron más cercanos fueron A. hybridus, A. powellii y A. retroflexus, mientras que A. spinosus y A. palmerii fueron los más alejados. La mayor parte de la diversidad genética detectable se encontró entre las especies y las poblaciones, mientras que la menor parte estuvo dentro de las mismas. Los marcadores moleculares tipo ISSR fueron útiles para caracterizar accesiones y estimar la diversidad genética en poblaciones de Amaranthus.











 texto en
texto en 


