Introducción
En los últimos años el cultivo del limón mexicano se encuentra seriamente amenazado por la llegada de enfermedades que ponen en riesgo su permanencia como la actividad agrícola más importante para el estado de Colima y de gran relevancia para los estados de Michoacán, Guerrero y Oaxaca. Destacan las enfermedades de tipo fungoso como la antracnosis (Colletotrichum acutatum), se presenta durante el periodo de lluvias y afecta el rendimiento y la calidad de fruta en los meses de otoño-invierno. Virus tristeza de los cítricos (VTC), enfermedad de tipo viral de mayor importancia económica que afecta al cultivo de los cítricos a nivel mundial y el huanglongbing (HLB) considerada actualmente como la más devastadora siendo más peligrosa que el VTC.
El desarrollo de variedades tolerantes puede ayudar a enfrentar y reducir los impactos de estas enfermedades. El programa de mejoramiento genético de limón mexicano del INIFAP- Tecomán ha generado plantas híbridas interespecíficas e intergenéricas, en la búsqueda de genotipos con resistencia o mayor tolerancia a estas enfermedades y de esa forma poder conservar el cultivo de limón mexicano en el país.
Los programas de mejoramiento genético de cítricos en varias partes del mundo, están enfocados principalmente a la generación de variedades de fruta sin semilla, producción de genotipos para uso como portainjertos que integren además de buena compatibilidad con las variedades, tolerancia a patógenos y adaptación a suelos con salinidad y carbonatos de calcio (Tusa et al., 1996; Grosser et al., 2000; Ollitrault et al., 2000; Cristofani et al., 2002; Navarro et al., 2002; Grosser y Gmitter, 2005; Shi-Ping et al., 2009).
Algunos reportes indican que es posible obtener segregantes triploides (3x) al cruzar genotipos de cítricos diploides (2x) con genotipos tetraploides (4x) (Cameron y Burnett 1978; Cameron y Soost 1969; Esen y Soost, 1972; Oiyama, 1991; Starrantino y Recupero, 1982). Las evidencias más recientes de variedades de cítricos triploides que se obtuvieron por hibridación sexual interploide son las variedades de toronja (C. maxima (Burm) Merril), ‛Oro Blanco’ y ‛Melogold’ que fueron desarrolladas en California (Soost y Cameron, 1980; 1985). Al igual que la mayoría de los cítricos, el limón mexicano es diploide (2x). Al cruzar esta especie vía hibridación sexual con genotipos tetraploides (2n= 4x) que contengan características de tolerancia a problemas sanitarios antes mencionados es posible generar variedades triploides sin semillas tolerantes.
El mejoramiento convencional de cítricos tiene importantes limitaciones debido a la compleja biología reproductiva de estas especies. La mayoría de los genotipos son apomícticos y desarrollan embriones adventicios directamente de las células nucelares que limitan o excluyen el desarrollo de embriones cigóticos, (Kobayashi et al.; 1979; Soost y Roose, 1996; Ruiz, et al.; 2000, Yildis, et al., 2013). Estos contienen el mismo material genético que la planta madre, además pueden provocar que la mayoría de los cultivares poliembriónicos produzcan pocas plantas híbridas. Por otra parte, es difícil distinguir las plantas nucelares y cigóticos en una etapa temprana de desarrollo (De Lange y Vincent, 1977). Por lo que la identificación de embriones sexuales es importante y requieren de análisis como la citología, citometría de flujo, isoenzima, o análisis molecular (Tusa et al., 2002).
Según Yildiz et al. (2013) muchos estudios dirigidos a la separación de distintos tipos y variedades de cítricos y la identificación de las plantas nucelares y cigóticas, comenzaron utilizando caracterización morfológica (Furr y Reece, 1946; Cameron, 1979), espectrofotometría (Pieringer y Edwards, 1967) y técnicas cromatográficas (Albach y Redman, 1969; Stanley y Jurd, 1971; Tatum et al., 1974; Weinbaum et al., 1982). También se utilizaron varios métodos bioquímicos, incluyendo el oscurecimiento enzimático (Esen y Soost, 1974).
Ninguno de estos métodos confirmó de manera eficiente la identidad de las verdaderas plántulas nucelares (Ruiz et al., 2000; Tusa et al., 2002). Más tarde, se emplearon isoenzimas (Iglesias et al., 1974; Moore y Castle, 1988; Ashari et al., 1988; Anderson et al., 1991). Sin embargo, ya que los productos de la expresión génica fueron utilizados en estos casos, los resultados pudieron ser influenciados por el entorno o por la etapa de desarrollo de la planta y sus órganos, por lo que este método fue poco fiable para la identificación de plántulas cigóticas.
El uso de polimorfismos de ADN para la identificación de plantas híbridas es importante en programas de mejoramiento de cítricos y otras leñosas, pues con esto se acelera el proceso de detección de la progenie. Varios estudios recientes han descrito el uso de marcadores SSR como método alternativo para distinguir plántulas sexuales de nucelares en cítricos (Mullis et al., 1986; Ruiz et al., 2000; Oliveira et al., 2002; Rao et al., 2008; Shareefa et al., 2009; Yildis, et al., 2013).
En este estudio se describe la identificación de híbridos de limón mexicano basado en el análisis molecular de marcadores SSR.
Materiales y métodos
Material biológico
Para el estudio se utilizó una población de 203 plantas originadas de 13 cruzas de limón mexicano con limones italianos, citranges e híbridos somáticos entre citranges y naranjo dulce (Cuadro 1), realizadas de 2009 a 2012 en el Campo Experimental Tecomán del INIFAP.
Cuadro 1 Lista de progenies híbridas de limón mexicano utilizadas en el estudio.
| Número de cruza | Cruza | Número de muestras | Año de cruza |
| 1 | Rosenberg (2x) X Colimex (2x) | 20 | 2009 |
| 2 | Colimex (2x) X Rosenberg (2x) | 14 | 2009 |
| 3 | Mex13 (4x) X Limoneira 8a (2x) | 12 | 2010 |
| 4 | Mex13 (4x) X Rosenberg (2x) | 19 | 2010 |
| 5 | Mex13 (4x) X Eureka (2x) | 10 | 2010 |
| 6 | Rosenberg (2x) X Mex20 (4x) | 8 | 2010 |
| 7 | Rosenberg (2x) X Mex13 (4x) | 7 | 2010 |
| 8 | Mex13 (4x) X C-35 (2x) | 50 | 2011 |
| 9 | Colimex (2x) X HS11 (4x) | 21 | 2011 |
| 10 | Colimex (2x) X Limequat (2x) | 6 | 2012 |
| 11 | Colimex (2x) X C-32 (2x) | 17 | 2012 |
| 12 | Colimex (2x) X C-swingle (2x) | 8 | 2012 |
| 13 | Colimex (2x) X Yuma (2x) | 11 | 2012 |
Extracción de ADN genómico
El ADN total fue extraído a partir de hojas jóvenes de acuerdo con el método de Doyle y Doyle, 1987, con modificaciones Hernández-Nava (2013). En un tubo eppendorf se depositó 0.1 g de tejido vegetal junto con 600 µl de buffer salino y se trituró en un disruptor Retsch® MM400 a 25 frecuencias (f/s) por tres min. Posteriormente se resuspendió en buffer de extracción CTAB 2% (1.4 M NaCl, 100 mM Tris, 20 mM EDTA, pH 8) y 3 µl de β-Mercaptoetanol a 100%. Se mezcló el contenido del tubo en vortex por 10 segundos y se incubó a 55 °C por 30 min.
A continuación se agregó 400 µl de fenol: cloroformo: alcoholisoamílico en una proporción 25:24:1 respectivamente, mezclando la muestra en vortex por 10 segundos. Se centrifugó la muestra por 10 min a 14 000 rpm a 4 ºC y se colectó 500 µl de la fase acuosa evitando que las fases se mezclaran. Posteriormente se agregó 50 µl de acetato de amonio y 500 µl de isopropanol y se incubó la muestra por 10 min a temperatura de -20 ºC. A continuación se centrifugó por 10 min a 14 000 rpm a 4 ºC y se decantó el sobrenadante sin perder la pastilla que se formó. Se adicionó 1 ml de etanol a 70% y se centrifugó por 1 min a 14 000 rpm a 4 ºC. Transcurrido el tiempo, se secó la muestra a temperatura ambiente hasta la evaporación del etanol. Finalmente se resuspendió la pastilla en 50 µl de agua libre de ARNasa y se almacenó la muestra a -20 ºC. La calidad y pureza de ADN obtenido se cuantificó en Nanodrop 2000.
Amplificación porPCR
La amplificación por PCR de segmentos específicos de ADN mediante juegos de oligos reportados por Kijas et al. (1997) (Cuadro 2), se realizó en un volumen total de 20 µl utilizando 10 µl de iTaq™ Universal Probes supermix (dNTPs, iTaq ADN polimerasa, MgCl2, potenciadores, estabilizantes, colorantes y normalización ROX) y 2.5 µl de cada iniciador Forward y Reverse. El programa de amplificación consistió en un ciclo a 94 ºC durante 5 min, seguido de 32 ciclos de 94 ºC durante 1 min, 55 ºC durante 30 s y 72 ºC durante 1 min. Una extensión final de 4 min a 72 ºC. La temperatura de hibridación se redujo a 45 ºC durante 30 s para promover la amplificación del cebador TAA52.
Cuadro 2 Iniciadores de microsatélites utilizados en la detección de híbridos de limón mexicano.
| Nombre | Secuencia 5’-3’ | Secuencia 3’-5’ |
| TAA41 | AGGTCTACATTGGCATTGTC | ACATGCAGTGCTATAATGAATG |
| TAA45 | GCACCTTTTATACCTGACTCGG | TTCAGCATTTGAGTTGGTTACG |
| TAA52 | GATCTTGACTGAACTTAAAG | ATGTATTGTGTTGATAACG |
| cAGG9 | AATGCTGAAGATAATCCGCG | TGCCTTGCTCTCCACTCC |
La amplificación de los fragmentos de ADN se realizó en un termociclador Bio-Rad T100™ Thermal Cycler. Los productos usados en la amplificación de PCR, se analizaron en gel de acrilamida a 8% y se revelaron con nitrato de plata al 0.2%. Se usó un marcador de peso molecular de 1kb (invitrogen). Las condiciones de corrida de las muestra fueron 190 V y 200 mA por 90 min.
Análisis de datos
Se realizaron dendogramas a partir de la identificación de presencia (1) o ausencia (0) de bandas utilizando los cuatro iniciadores, a través del método de agrupamiento pareado no ponderado con medias aritméticas (UPGMA) y distancia Euclediana al cuadrado. Los análisis se realizaron con el uso del paquete estadístico Numerical Taxonomy and Multivariate Analysis System (NTSYS-pc 2.21) (Rohlf, 2000).
Resultados y discusión
Las plantas híbridas se determinaron por la presencia de bandas del progenitor masculino amplificadas por PCR utilizando cuatro iniciadores. En la Figura 1a y b, se muestran algunos de los híbridos identificados con los iniciadores cAGG9 y TAA45 en una de las cruzas evaluadas, (Mex13 (4x) x Rosenberg (2x)). De 203 plantas obtenidas de las diferentes cruzas realizadas el mejor iniciador logró identificar 145 (71%) plantas cigóticas. Los resultados obtenidos son similares a los repostados por algunos autores. Frost y Soost (1967) informaron de frecuencias cigóticas de 78.7% y 14% en tangerinas ‛King’ y ‛Willowleaf’ respectivamente, usando a P. trifoliata como progenitor masculino.

Figura 1 Gel de poliacrilamida mostrando el análisis con SSR de progenie obtenida de cruzas de limón mexicano. a) Iniciador cAGG9; b) iniciador TAA45 marcador 1kb (M), progenitor femenino Mex13, masculino Rosenberg.
Otros autores como Hearn (1973) encontró que la selección de naranja dulce ‛Mediterranean’ produjo 62% de plántulas cigóticas cuando se utilizó como progenitor masculino a P. trifoliata. En otros reportes (Soost et al., 1980) encontraron una frecuencia cigótica de aproximadamente 85% cuando usaron como progenitor femenino a la mandarina ‛King’ y maculino a ‛Parson Special’. Varios estudios han demostrado que la frecuencia de plántulas cigoticas no excede 15%, pero esto va a depender de la especie y en algunos casos se obtienen solamente individuos nucelares (Cameron y Soost, 1980; Hirai et al., 1986; Ashari et al., 1988; Roose y Traugh, 1988).
Varios niveles de plántulas cigoticas se han observado en otras especies y cruzas de cítricos. Algunos otros autores, entre ellos. Hwang y Yeuh (1989) reportaron frecuencias cigóticas de 42.8% y 66.7% en cruzas de mandarina ‛Tankan’ x P. trifoliata. Hwang (1991) informó que, en plántulas identificadas por morfología de la hoja, obtuvo 73% de plántulas cigóticas de limón ‛Eureka’ en cruzas con naranja trifoliada y 38% a 56% en cultivares de limón ‛Eureka’ y ‛Lisboa’ en cruzas con naranja dulce. En otros trabajos como en el de Moore y Castillo (1988), se identificaron solo 24% de plántulas cigóticas de limón ‛Volkameriana’, estos resultados fueron utilizando marcadores basados en isoenzimas.
Con el uso de marcadores SSR, Ruiz et al. (2000), clasificó 86.95% de plántulas híbridas de cruzas entre ‛Flying Dragon’ y tangor ‛Ortanique’. Sin embargo, Yildiz et al. (2013), identificaron 86 plántulas (36. 91%) nucelares y 63% cigóticas en cruzas con mandarina ‛Fremont’ como progenitor femenino y la variedad ‛Rio Red’ como progenitor masculino. Estos mimos autores señalan que cuando utilizaron a la mandarina ‛Robinson’ como el progenitor femenino, esta proporción fue de 31.09% de plántulas nucelares y 69% cigóticas.
Datos que difieren a los obtenidos en este estudio, fueron reportados por Yun et al. (2007), que obtuvieron 0% a 13.4% plántulas híbridas en cruzas donde se utilizó a las mandarinas ‛Miyagawa Wase’, ‛Okitsu Wase’ y ‛Shiranuhi’ como progenitores femeninos y mandarina ‛Ponkan’ y citrumelo ‛Swingle’ como progenitores masculinos. Otros autores como Rao et al. (2008) cruzaron diferentes cultivares de mandarinas y toronjas para obtener portainjertos alternativos a la naranja agria y con marcadores SSR identificaron 22.2% de plántulas híbridas de estas cruzas.
De acuerdo con García et al. (1999) los embriones cigóticos producidos por autopolinización son menos vigorosos y pueden no ser competitivos con los nucelares. Los porcentajes de progenie cigótica encontrado en varios híbridos de cítricos dependen de la semilla del progenitor utilizado (Spiegel-Roy et al., 1977), el origen del polen (Cameron y Soost, 1980; Soares-Filho et al., 1995) y las influencias ambientales (Khan y Roose, 1988; Moore y Castle, 1988; Roose y Traugh, 1988).
Las plantas obtenidas diploides, triploides y tetraploides de cada combinación fueron evaluadas utilizando iniciadores SSR. Los cuatro lograron identificar plantas híbridas (Cuadro 3). Con el iniciador TAA45 fue posible identificar el mayor número de híbridos (145) siendo este el mejor de todos, seguido del cAGG9 y el TAA52 con 130 y 125 híbridos respectivamente. Caso contrario a los resultados obtenidos en este estudio, se reportaron por Yildiz et al. (2013) en sus investigaciones localizaron en una población en estudio que los cebadores AG14 y TAA03 fueron más eficaces para distinguir individuos cigóticos y el cebador TAA45 no fue capaz de identificar las plántulas cigóticas.
Cuadro 3 Plantas hibridas identificadas con cuatro de los marcadores moleculares reportados por Kijas et al. (1997).
| Cruza | Iniciador cAGG9 | Iniciador TAA41 | Iniciador TAA45 | Iniciador TAA52 |
| Colimex X Yuma | 16 | 8 | 7 | 5 |
| Colimex X C-swingle | 2 | 9 | 2 | 2 |
| Colimex X híbrido somático 11 | 25 | 20 | 20 | 14 |
| Colimex X Limequat | 5 | 0 | 2 | 2 |
| Colimex X Rosenberg | 7 | 8 | 11 | 12 |
| Colimex X C-32 | 3 | 2 | 16 | 2 |
| Mex13 X Eureka | 4 | 5 | 7 | 9 |
| Mex13 X Limoneira 8a | 7 | 8 | 10 | 3 |
| Mex13 X Rosenberg | 12 | 15 | 13 | 17 |
| Mex13 X C-35 | 39 | 32 | 48 | 53 |
| Rosenberg X Colimex | 7 | 6 | 3 | 2 |
| Rosenberg X Mex13 | 1 | 1 | 0 | 1 |
| Rosenberg X Mex20 | 2 | 0 | 6 | 3 |
| Total de plantas híbridas | 130 | 114 | 145 | 125 |
Con respecto al iniciador TAA41, fue este el que menos plantas identificó (114). Resultados similares fueron encontrados por Yildiz et al. (2013), reportaron que en combinaciones donde utilizaron la mandarina ‛Fremont’ como progenitor femenino, el iniciador TAA41 fue capaz de identificar solo 47 plantas cigóticas que el resto de los cebadores SSR utilizados. Estos mismos autores reportan que, en combinaciones donde se utilizó la mandarina ‛Robinson’ como el progenitor femenino, los iniciadores AG14 y TAA03 identificaron 59 y 58 plantas cigóticas respectivamente, más que el iniciador TAA41. En contraste, los iniciadores CAT01, TAA01, TAA45 y TAA52 (cuando se utilizó a la mandarina ‛Fremont’ y ‛Robinson’ como progenitores femeninos) no fueron útiles en la determinación de la identidad de plantas cigóticas.
En las poblaciones que son derivadas de una cruza, todos los individuos que son cigóticos muestran un genotipo diferente al de la madre en cualquier locus de discriminación, esto va a ocurrir siempre y cuando el padre tenga alelos diferentes a los de la madre (Ruiz et al., 2000). La variación en el número de bandas amplificadas por diferentes iniciadores puede deberse a factores como la estructura del iniciador, la cantidad de plantilla y el número de sitios reconocidos en el genoma (Muralidharan y Wakeland, 1993).
Otros autores, entre ellos Nageswara et al. (2008) identificaron 77.8% plántulas nucelares y 22.2% plántulas cigóticas, con marcadores EST-SSR, en híbridos de introgresión de mandarina (C. reticulata Blanco) y pomelo (C. maxima Merr.). Otros autores como Mei-lian et al. (2007) reportan en cruzas de tangerina roja (C. reticulata Blanco) X naranja trifoliada (P. trifoliata L.) 111 individuos híbridos a través de rescate de embriones y verificados por marcadores SSR y 44 plantas híbridas a partir de cruzas de mandarina Satsuma (C. unshiu Marc) X naranja trifoliada (P. trifoliata L.).
De la misma manera Oliveira et al. (2002), reportan la identificación de híbridos derivados de cruzas entre tangor ‛Murcott’ [C. reticulata Blanco × C. sinensis (L.) Osb.] y naranja dulce ‛Pera’. [C. sinensis (L.) Osb.], con análisis de marcadores SSR. Siragusa et al., 2006, identificaron y evaluaron la variabilidad genética de accesiones de naranja dulce con marcadores moleculares SSR. También Aleza et al., 2012, identificaron híbridos que muestran el alelo especifico del progenitor masculino con marcadores moleculares SSR en cruzas 4x X 2x de varios cítricos. Aleza et al. (2010) utilizaron marcadores de SSR para demostrar que todas las plantas recuperadas de semillas no desarrolladas obtenidas a partir de hibridaciones sexuales 2x X 4x fueron resultado del embrión cigótico.
En los cítricos, los marcadores SSR pueden distinguir fácilmente entre las especies o variedades que surgen de la hibridación sexual (Barkley et al., 2006; Luro et al., 2008). Por el contrario, la diferenciación de genotipos derivados de mutaciones espontáneas es muy difícil, como en el caso de las clementinas (Bretó et al., 2003) o naranjas dulces (Fang y Roose, 1997). Mediante el uso de varios marcadores heterocigotos diferenciando los progenitores masculinos y femeninos, también se puede encontrar una buena probabilidad de diferenciar entre plantas cigóticas independientes.
Fang y Roose (1997) mencionan que las mutaciones en la mayoría de los casos pueden causar cambios significativos en la morfología de árbol, pero con las modificaciones que ocurren en la secuencia de ADN, son difíciles de detectar. Novelli et al. (2000, 2006) también informaron que, a pesar de considerables diferencias morfológicas, hay pocos polimorfismos RFLP, RAPD y SSR. Sobre la base de marcadores SSR, pues Cao et al. (2007) no pudieron detectar diferencias entre 16 mandarinas Satsuma y por tanto el posible origen de la mutación de estas variantes.
Las plántulas siempre tienen un valor de similitud más alto con sus progenitores femeninos, de ese modo se indica la disminución de los antecedentes genéticos, el aumento de la endogamia puede ser la causa del bajo rendimiento del árbol en diversas condiciones ambientales (Fehr, 1993).
Al realizar los dendogramas con los datos de los cuatro marcadores SSR de los individuos obtenidos con las distintas cruzas se pudo detectar que el coeficiente de similitud de la distancia euclediana varió desde 0 a 999.09 entre los 203 individuos que se analizaron. Esto se debe a la gran variabilidad generada en estos cruzamientos, ya que se combinaron los genomas de distintas especies e inclusive de géneros distintos.
Por otra parte, tanto los limones mexicanos como los limones italianos están en forma heterocigótica y por lo tanto al segregar los alelos en los gametos y luego al recombinarse en el cruzamiento, están generando nuevas combinaciones genéticas. Por su parte, los citranges están formados por el genoma de C. sinesis y P. trifoliata, también en condición altamente heterosigótica, lo que aumenta la diversidad genética. Este resultado coincide con lo reportado por Herrero et al. (1996) quienes señalan que las limas y limones muestran un alto porcentaje de heterocigosidad.
La Figura 2 muestra uno de los 18 dendogramas construido con los perfiles de SSR obtenidos en el análisis de conglomerados (UPGMA) El dendograma de la cruza Mex13 x Rosenberg se separó en cuatro grupos. El primero lo formó el progenitor femenino Mex13. En el segundo se encuentran los individuos H40, H43, H44, H48, H51, H41, H42, H55, H53, H54, H50, H52, H46, H47, H49 y el progenitor masculino Rosenberg. El tercer grupo lo formó el individuo H45. Y en el último se encuentran los indiciduos H196, H209 y H198. En esta cruza se observó que todos los individuos analizados fueron identificados como híbridos. Los individuos H40, H43, H44, H48 Y H51 son genéticamente similares a Rosenberg, lo que indica que los cuatro iniciadores lograron identificar regiones semejantes al progenitor masculino.
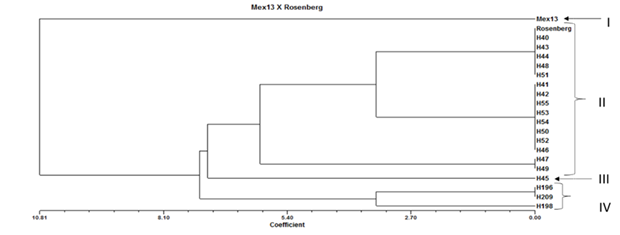
Figura 2 Dendograma generado con datos de cuatro marcadores SSR de individuos obtenidos de la cruza de ‘Mex13’ x ‘Rosenberg’ construído con el método UPGMA
El resto de los híbridos fueron identificados por tres de los iniciadores utilizados. En un estudio de germoplasma de cítricos, Shahsavar et al. (2007) reportaron que los limones (C. limon L. Burm. F.), ‛Lisboa’, ‛limón Rock’ y ‛limón forma de pera’ se separaron de otros genotipos al analizar el polimorfismo con marcadores moleculares ISSR con valor de similitud de 0.65. En este mismo trabajo estos autores reportan que las poblaciones de plantas estudiadas exhiben características morfológicas muy similares; sin embargo, los fenotipos moleculares revelaron diferencias sustanciales de similitud entre los híbridos y los progenitores.
Conclusiones
Se identificaron plantas híbridas de limón mexicano con limones italianos e híbridos somáticos entre citranges y naranjo dulce.
Los cuatro iniciadores permitieron la identificación de plantas hibridas. El mayor número de plantas hibridas se identificaron con los iniciadores TAA45 y cAGG9.
La identificación de los híbridos de la población en estudio a través del análisis de marcadores moleculares, permitirá ahorrar tiempo y reducir los costos de mantenimiento de material en campo en los programas de mejoramiento genético que implican la obtención de un gran número de plántulas derivadas de las cruzas realizadas.
Los resultados de este estudio sugieren que el análisis de marcadores moleculares puede contribuir significativamente a los programas de mejoramiento de cítricos











 text in
text in 


