Las especies del género Phytophthora son patógenos hemibiotróficos de gran importancia en cultivos agrícolas y forestales ya que causan grandes pérdidas económicas en un amplio rango de especies vegetales en el mundo (Erwin y Ribeiro, 1996; Hall y Albers, 2009; Jung et al., 2000; Roy y Grünwald, 2014). Entre las especies de este oomicete que ocasionan mayores daños o pérdidas se encuentran: Phytophthora ramorum, agente causal de la enfermedad denominada muerte repentina del roble, que afecta también a otros árboles y arbustos, P. palmivora causante de la pudrición de brotes, yemas y frutos en palmas; P. capsici patógeno que causa marchitez y pudrición de la corona y raíz en chile, cucurbitáceas y otros cultivos comerciales, P. infestans causante del tizón tardío de la papa, el tomate y otras plantas de la familia Solanaceae, P. cinnamomi que infecta a más de 3 000 especies de plantas en el mundo (Erwin y Ribeiro, 1996; Cooke et al., 2000; Drenth y Guest 2004; Shearer et al., 2004; Hardham, 2005; Dart y Chastagner, 2007; Sutton et al., 2007; Brasier, 2008; Hansen et al., 2012; Jiang y Tyler, 2012; Kroon et al., 2012; Lamour et al., 2012).
Esta última especie, es un ejemplo del impacto económico y ecológico que pueden llegar a tener algunas especies de Phytophthora en el ámbito mundial. Por ejemplo, se ha reportado que 2 284 (40%) de las 5 710 especies de plantas descritas en el suroeste de Australia son susceptibles a P. cinnamomi. Este patógeno le costó a la economía de Australia 1 600 millones de dólares en el transcurso de una década (Carter, 2004).
El número de especies descritas del género Phytophthora se ha duplicado en la última década, y posiblemente aumente de manera considerable conforme se realicen más estudios en bosques y cuerpos de agua. Por otro lado, los análisis filogenéticos multigénicos, las técnicas moleculares, la secuenciación genómica y las bases de datos con secuencias de Phytophthora han hecho posible una identificación precisa y la determinación del número real de especies descritas de este patógeno en el mundo. En la actualidad se han descrito 152 especies pertenecientes al género Phytophthorahttp://idtools.org:8080/key_server/player.jsp?keyId=48. Las páginas web que enlistan especies de Phytophthora son: http://www.phytophthoradb.org, http://www.phytophthora-id.org, http://www.qbank.eu, y http://www.boldsystems.org.
Medios de cultivo
Los medios de cultivo seleccionados para aislar, describir e identificar especies de Phytophthora son de suma importancia. Un factor que se debe considerar para elegir el medio apropiado es la especie de Phytophthora con la cual se esté trabajando (Erwin y Ribeiro, 1996). Los medios selectivos para aislar Phytophthora contienen fungicidas y antibióticos, para evitar el crecimiento de hongos y bacterias.
Los medios selectivos generalmente más utilizados son PARPH, PARP, PARPH, PVP (P-pimaricina, A-ampicilina, R-rifampicina, P-pentacloronitrobenceno, H-himexazol, V-vancomicina), 3-P (P-pimaricina, P-Penicinila, P-Polimixina B, CARP (C-cornmeal agar <harina de maíz agar>, A-ampicilina, R-rifamicina, P-pimaricina), CARPD (D-dicloran), CARP+ (+-benlato e himexazol), existiendo variaciones en las concentraciones de los antibióticos y fungicidas que se agregan (Tsao y Ocana, 1969; Jeffers y Martin, 1986; Erwin y Ribeiro, 1996; Drenth y Sendall, 2001) estos deberán añadirse después de que el medio se ha esterilizado el medio. La pimaricina es un antibiótico costoso para los laboratorios de América Latina; sin embargo, se puede substituir por Delvocid Instant (Erwin y Ribeiro, 1996), que es de bajo costo, este contiene Natamicina por lo que a este medio se le conoce como NARPH. Asimismo el himexazol que anteriormente provenía de Japón, actualmente está disponible en diferentes países. El medio selectivo se puede preparar utilizando harina de maíz agar o V8 agar. Cuando no se pueda adquirir harina de maíz esta se puede substituir por fécula de maíz (Félix-Gastélum. Comun. Pers., 2015).
Algunos ejemplos que el medio es bastante específico es el de agar centeno, que es utilizado para aislar P. infestans, P. ipomoeae y P. mirabilis, los medios 3-P y PCH (P-pimaricina, C-cloranfenicol, H-himexazol) son específicos para aislar P. cinnamomi de raíces y de suelo (Drenth y Sendall, 2001; Ivors, 2016). Los protocolos y medios (V8 agar, V8 líquido, súper V8, V8-centeno, centeno A, centeno B, harina de avena, agar zanahoria al 5%, frijol lima fresco y chícharo sacarosa), empleados actualmente se describen en el libro de Ivors (2016). En el laboratorio se pueden preparar medios de cultivo con componentes que se encuentren disponibles, se recomienda probar diferentes métodos de preparación y seleccionar los más adecuados a las necesidades individuales.
Diagnóstico directo
Cuando se lleva a cabo un diagnóstico fitopatológico se espera que el resultado se obtenga lo más rápido posible, para lo cual existen kits, pruebas de ELISA, hibridación in situ fluorescente (FISH), técnicas hiperespectrales, biosensores basados en anticuerpos, inmunoflorescencia, imágenes de florescencia, termografía, y pruebas de tiras reactivas para la detección directa de patógenos en campo que son de gran ayuda y pueden detectar Phytophthora (Miller et al., 1994; Fang y Ramasamy, 2015).
La mayoría de kits de uso en campo basados en técnicas serológicas dan una respuesta rápida in situ en pocos minutos u horas, lo cual es de gran utilidad para productores, asesores, técnicos e inspectores. Las pruebas se pueden realizar a partir de cualquier tejido de la planta que se requiere analizar y el resultado es fácil de interpretar. Si se considera necesario, los resultados pueden confirmarse posteriormente en laboratorio. Algunos de los kits disponibles en el mercado son Phytophthora late blight Pocket Diagnostic® test para la detección de P. infestans en papa, Phytophthora spp. ALERT-LF® y los ImmunoStrips que permiten identificar infecciones causadas por Phytophthora en un amplio rango de cultivos incluyendo plantas de vivero. Estos kits están diseñados para utilizarse en campo o laboratorio a partir de muestras de tejido vegetal, no requieren equipo especial para llevarlas a cabo, ya que todo el material requerido está incluido.
Aislamiento de Phytophthora
Aislamiento de Phytophthora a partir de tejido vegetal. Una adecuada colecta y selección de muestras determinan el éxito en el proceso de aislamiento de Phytophthora de tejido vegetal enfermo. Se recomienda colectar muestras de hojas, tallos, raíces, frutos, flores, etc, que tengan una zona de tejido sano frente al avance de la lesión (Figura 1). Para realizar el aislamiento se deben cortar secciones de tejido de la zona mencionada, ya que será donde el patógeno esté en crecimiento activo. Estos tejidos se pueden sembrar sin desinfestar, directamente en medio selectivo (Erwin y Ribeiro, 1996; Streito, 2002; Jung y Blaschke, 2004; Jung y Burgess, 2009; Kasuga et al., 2012).
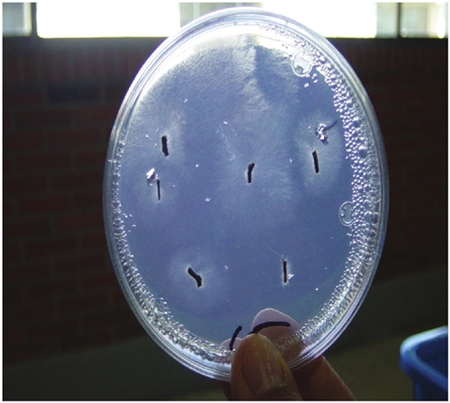
Figura 1 Aislamiento de Phytophthora a partir de raíces con pudrición en medio selectivo NARPH-harina de maíz.
No se recomienda aislar Phytophthora de tejido necrótico debido a la presencia de hongos y bacterias saprófitos. Algunos autores sugieren desinfestar superficialmente los tejidos, para lo cual, las secciones de tejido se sumergen en etanol al 70% durante 10 a 30 s, después se lavan con agua destilada estéril, posteriormente se secan con papel filtro estéril y por último se colocan en medio de cultivo selectivo (Hüberli, 2000; Davison y Tay, 2005; Ghimire et al., 2009; Moralejo et al., 2009; Tsopmbeng et al., 2012), en algunos reportes se menciona que el alcohol no deja residuos por lo que no es necesario enjuagar (Martin et al., 2012; Borines et al., 2014).
También se utiliza principalmente una solución de cloro comercial al 10% (5% hipoclorito de sodio). El proceso de desinfestación consiste en sumergir el tejido vegetal en la solución de cloro durante 30 s, enseguida enjuagar con agua destilada estéril y después secar con papel estéril, finalmente cortar las secciones de tejido antes de colocarlas en el medio selectivo (Drenth y Sendall, 2001; Ghimire et al., 2009).
El tiempo de desinfestación puede variar, lo cual depende del tipo de tejido del que se va a realizar el aislamiento. Es importante tomar en cuenta que la sensibilidad al cloro varía de una especie a otra de Phytophthora.Hong et al., (2003), han mostrado que la sensibilidad a éste es patógeno-dependiente. Algunas especies de Phytophthora, pueden ser sensibles al cloro y al etanol por lo que se debe omitir su uso (Hong et al., 2003). Cuando se desconoce si la especie es sensible a estos compuestos, se recomienda desinfestar solo algunas secciones de tejido con etanol o cloro y otros sembrarlos directamente en medio selectivo.
El tejido se debe secar preferentemente con papel filtro o toallas de papel estériles antes de sembrarse en algún medio, para evitar el crecimiento de bacterias (Erwin y Ribeiro, 1996). Las secciones de tejido sembradas en medio selectivo se deben incubar a temperatura que favorezca el crecimiento de Phytophthora (Erwin y Ribeiro, 1996; Ferguson y Jeffers, 1999; Drenth y Sendall, 2001; Martin et al., 2012).
Aislamiento de Phytophthora de agua. El agua de riego es una fuente importante de inóculo de patógenos de plantas. Los géneros Phytophthora y Pythium de la familia Pythiaceae son los más comunes, destructivos y contaminantes en sistemas de riego (Erwin y Ribeiro, 1996; Drenth y Sendall, 2001; Loyd et al., 2014; Zappia et al., 2014). Se han detectado en agua más de 30 especies de Phytophthora en el mundo, con algunas especies aún no se han realizado pruebas de patogenicidad (Zappia et al., 2014) otras que se han inoculado en diferentes hospedantes no han sido patógenas (Copes et al., 2015).
Únicamente se ha demostrado la patogenicidad de algunas como P. irrigata en azalea, chile y tomate; P. hydropathica en azalea, cineraria y tomate; P. megasperma en azalea y chile; P. nicotianae en vinca, begonia, berenjena, chile, cineraria, lupinus y tomate; P. palmivora en azalea, begonia, berenjena, chile y tomate; P. tropicalis en azalea, begonia, berenjena, chile, cineraria, lupinus, tomate y vinca (Hong et al., 2008; Loyd et al., 2014). Los reservorios o canales de agua para uso agrícola pueden ser contaminados por escurrimientos de agua de campos infestados con Phytophthora, lo que puede ocasionar la dispersión del patógeno a lugares donde no estaba presente (Erwin y Ribeiro, 1996).
El agua recirculada infestada dentro y entre viveros favorece la dispersión de propágulos de Phytophthora en forma de zoosporas, a plantas sanas y a zonas donde no se encontraba (MacDonald et al., 1994; Stewart-Wade, 2011). Por lo que, los protocolos eficaces para detectar a este oomicete en agua de riego son esenciales. Con los métodos de filtración se pueden procesar cantidades limitadas de agua comparado con la enorme cantidad de agua que existe en las represas, cuerpos de agua y sistemas de riego.
En un estudio realizado por Hong et al. (2002), obtuvieron mayores tasas de recuperación en diferentes fuentes de agua filtrando las muestras de agua a través de una membrana que retiene los propágulos, la cual posteriormente se invierte y se coloca directamente en el medio selectivo PARP-V8. Las membranas que probaron fueron de diferentes materiales y tamaños de poro observando mejores resultados con las membranas Millipore 5 y Durapore 5 (Millipore®) obteniendo mayores tasas de recuperación.
Aislamiento de Phytophthora de suelo o sustrato de siembra
Phytophthora puede dispersarse a nuevos sitios a través de la venta de diferentes sustratos de siembra o suelos infestados con propágulos (Ferguson y Jeffers, 1999; Dart et al., 2007; Dart y Chastagner, 2007). Se recomienda usar diferentes tejidos vegetales trampa con cada muestra de suelo o sustratos que se sospecha están contaminados con Phytophthora para descartar resultados negativos falsos (Jeffers y Aldwinkle, 1987; Davison y Tay, 2005b; Balci et al., 2007; Tooley y Carras, 2011). Una amplia gama de tejidos vegetales trampa se usan para recuperar Phytophthora de suelo ya que no todos son colonizados de la misma forma por las diferentes especies de Phytophthora (Erwin y Ribeiro, 1996). En un párrafo posterior se mencionan los tejidos vegetales trampa recomendados.
Para detectar Phytophthora en suelos o sustratos, se utiliza la inundación de estos con agua destilada estéril, de tal manera que los propágulos, especialmente zoosporas se liberan en el agua, nadan a la superficie e infectan el tejido vegetal usado como tejido vegetal trampa (Figura 2). Algunas proporciones utilizadas para mezclar sustrato y agua son 1:2, 2:3.5 (peso: volumen). Este método tiene varias ventajas con respecto al aislamiento de tejido, ya que puede analizar grandes cantidades de suelo, incrementando las posibilidades de detectar el patógeno cuando la concentración de inóculo es baja.

Figura 2 Hojas de Rhododrendron utilizadas como tejido vegetal trampa para detectar la presencia de Phytophthora en muestras de substrato o suelo, en cajas de Petri.
Asimismo, es más factible detectar las oosporas en especies homotálicas (Erwin y Ribeiro, 1996; Balci et al., 2007; Fichtner et al., 2007). Los tejidos vegetales trampa colonizados por zoosporas posteriormente se lavan con agua de la llave o destilada estéril para eliminar bacterias de la superficie del tejido, se secan con papel estéril y se siembran en medio selectivo (Ferguson y Jeffers, 1999; Jung et al., 2000; Balci et al., 2007; Fitchtner et al., 2007; Hwang et al., 2008; Ghimire et al., 2009). El peciolo de las hojas de Rhododendron es el tejido que se infecta primero, por lo que éste se puede cortar y sembrar en medio selectivo (Figura 3).

Figura 3 Peciolos necrosados seccionados de hojas de Rhododendron, previamente colocadas con suelo o substrato de siembra y transferidos a medio selectivo NARPH preparado con V-8 agar.
La colecta de muestras para propósitos de detección o densidad poblacional debe realizarse en las diferentes estaciones del año ya que las poblaciones pueden variar de una estación a otra (Erwin y Ribeiro, 1996). La mayoría de las especies de Phytophthora son favorecidas por alta humedad, temperaturas entre 15 y 35 °C, climas húmedos con lluvias abundantes y sustrato o suelo pobres en nutrientes. Cuando se presenten estas condiciones, es recomendable analizar muestras del sustrato o suelo para detectar al patógeno (Erwin y Ribeiro, 1996; Drenth y Sendall, 2001; Kong et al., 2009).
Se sugiere que las colectas se hagan de suelo húmedo, cerca de las raíces sanas y de las que están en crecimiento (Erwin y Ribeiro, 1996; Balci et al., 2007; Fichtner et al., 2007). Las muestras no deben exponerse a temperaturas de más de 45 °C o a temperaturas de congelamiento para mantener la viabilidad del patógeno (Drenth y Sendall, 2001). Se deben colocar en bolsas de plástico para evitar que se deshidraten y depositarlas en una hielera para su transporte al laboratorio; colocando un material aislante para evitar el contacto directo de las muestras con el hielo. Se recomienda mantenerlas húmedas, a una temperatura entre 10 y 20 °C y se deben procesar lo más pronto posible (Drenth y Sendall, 2001).
Tejidos vegetales trampa
Para que la detección de Phytophthora sea más efectiva a partir de suelo y agua se recomienda el uso de tejidos vegetales trampa. La selección del tipo de tejido es importante para la recuperación de especies de Phytophthora. Los tejidos vegetales trampa que se utilizan pueden ser de diversas plantas y de diferentes partes vegetales como: cotiledones, discos pequeños de hojas, frutos, hojas completas, pétalos, plántulas, semillas y vainas de semillas. La elección de los tejidos vegetales trampa depende de la especies de Phytophthora que se sospeche es el agente causal de la enfermedad. Las hojas de Rhododendron, Camellia, Quercus, Lupinus, acículas de Juniperus, frutos de manzana, pera y pepino son los de mayor uso mediante los cuales es posible detectar un gran número de especies de Phytophthora (Erwin y Ribeiro, 1996; Ferguson y Jeffers, 1999; Ivors et al., 2004; Balci et al., 2007; Gevens et al., 2007; Sutton et al., 2009; Reeser et al., 2011; Tooley y Carras, 2011; Themann et al., 2002; Huai et al., 2013). Se recupera un mayor número de especies de Phytophthora cuando se usan hojas enteras del tejido vegetal trampa (Ferguson y Jeffers, 1999; Ghimire et al., 2009), que cuando se utlizan discos de hojas, ya que no es recomendable porque son colonizados fácilmente por Pythium y bacterias.
Purificación de aislados contaminados por bacterias
Para realizar almacenamiento de las cepas y estudios de caracterización morfológica y genética de Phytophthora es un requisito obtener aislados libres de contaminación bacteriana. Los aislados que se obtienen en medios con antibióticos en ocasiones no están libres de bacterias. Se recomienda agregar más de un antibiótico al medio utilizado al realizar los aislamientos (Erwin y Ribeiro, 1996; Nesbitt et al., 1981), para inhibir el crecimiento de los diferentes tipos de bacterias. Por otra parte el uso de antibióticos puede inducir resistencia en las bacterias y su utilización en altas concentraciones inhibe la germinación de esporas de Phytophthora (Erwin y Ribeiro, 1996).
Algunos métodos alternativos al uso de antibióticos y de bajo costo son el método de superposición y la utilización de medio acidificado (Solache-Huacuz et al., 2010; Martin et al., 2012). El primero consiste en colocar un cubo de medio con micelio contaminado, con el crecimiento micelial hacia arriba, en una caja petri estéril vacía, sobre el cubo se coloca un triángulo de medio estéril (Figura 4).
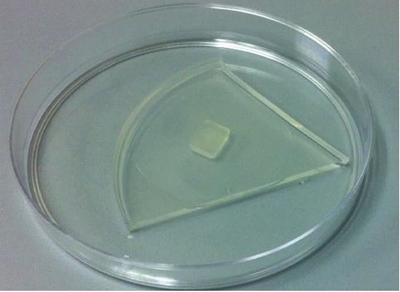
Figura 4 Purificación de un aislado de Phytophthora contaminado con bacterias, se puede realizar colocado un trozo de micelio contaminado bajo un triángulo de medio estéril.
El micelio crecerá a través del triángulo de medio hacia la superficie libre de bacterias. El segundo consiste en acidificar el medio de cultivo a pH 3, utilizando ácido tartárico al 0.14% (Koike, 2007; Solache-Huacuz et al., 2010) o PDA (papa dextrosa agar) a pH 4 con ácido láctico al 25 % (Erwin y Ribeiro, 1996). Cuando el micelio comienza a crecer se debe realizar una prueba para confirmar su pureza en medio líquido Luria Bertani (LB) en tubos de ensayo, o si se cuenta con algún otro medio que favorezca el crecimiento de bacterias también se puede utilizar. Se cortan cubos de medio con micelio, se colocan dentro de los tubos y se incuban a temperatura ambiente por 24-48 h (Erwin y Ribeiro, 1996; Solache-Huacuz et al., 2010). Antes de almacenar los aislados o realizar algún estudio es necesario llevar a cabo esta prueba, para asegurarse que están puros (Figura 5).
Identificación morfológica
La identificación de especies se realiza con el apoyo de claves tabulares (Waterhouse et al., 1963; Martin et al., 2012). Para lo cual, los cultivos a identificar deben estar puros, es decir obtenidos por medio de zoosporas o por punta de hifa. El género Phytophthora, es considerado difícil de identificar especialmente para aquellos que no tienen experiencia (Waterhouse et al., 1963), ya que las diferencias morfológicas entre algunas especies de Phytophthora son pequeñas y los caracteres morfológicos son influenciados por el ambiente, y en algunas especies como P. capsici presentan esporangios con diferentes formas. Asimismo, el creciente número de especies descritas, que algunas especies de Phytophthora no producen esporangios en los medios que se utilizan comúnmente, y la presencia natural de híbridos entre especies de Phytophthora (Man in ´t Veld et al., 2007), complica la identificación morfológica.
La producción de esporangios se puede inducir de varias maneras: a) agregando agua destilada estéril a discos o bloques de medio V-8 con micelio; b) adicionándoles una suspensión de extracto de suelo sin esterilizar; y c) agregando solución salina a los bloques (Eye et al., 1978; Schoulties et al., 1980; Fichtner et al., 2012; Almaraz-Sánchez et al., 2013). La elección del líquido que se agregará dependerá de la especie de Phytophthora que se esté estudiando. Por ejemplo, para P. cinnamomi se sugiere utilizar extracto de suelo no estéril, incubar los aislados bajo luz blanca fluorescente y realizar cambios del líquido hasta que se observen esporangios (Chen y Zentmyer, 1970; Eye et al., 1978; Ahumada et al., 2013).
También se puede utilizar agua de lluvia para inducir esporulación. Para realizar la identificación de especies actualmente existe una clave tabular publicada por Martin et al. (2012) y una clave lucida disponible en internet que incluye la mayoría de características morfológicas y especies (http://idtools.org:8080/key-server/player.jsp?keyId=48). Para la identificación de especies estas claves toman en consideración características de las esporas asexuales: tipo y forma del esporangio, dimensiones (largo y ancho), número de papilas, caducidad, longitud del pedicelo, forma de la base y algunas características del esporangióforo como tipo de ramificación, en cuanto a esporas sexuales, el tipo de anteridio, características y dimensiones del oogonio y la oospora, algunas características adicionales para la identificación de especies del género Phytophthora son la presencia o ausencia de clamidosporas, rango de temperatura a la que crecen, temperatura óptima de crecimiento, presencia o no de hinchamientos en el micelio y tipo de reproducción de la cepa (homotálica o heterotálica) (Martin et al., 2012).
Identificación molecular
Las técnicas moleculares modernas basadas en la secuenciación de regiones específicas del ADN, son herramientas de gran utilidad para la identificación de especies de Phytophthora, ya que son un complemento indispensable de la caracterización morfológica. Estas técnicas son necesarias por lo complicado que puede ser determinar las características morfológicas de aislados de Phytophthora, por las razones que se mencionaron anteriormente. Sin embargo, algunos laboratorios no cuentan con recursos para equipo y material necesario para realizar este tipo de estudios, por lo que sigue siendo una limitante en muchos países en vías de desarrollo.
El locus con mayor número de secuencias en las bases de datos que incluye todas las especies de Phytophthora descritas, es la región de los espaciadores transcritos internos (ITS). Los oligonucleótidos utilizados principalmente para la amplificación de la región ITS son ITS4 e ITS6 (White et al., 1990; Cooke et al., 2000; Martin et al., 2012). Sin embargo, las secuencias obtenidas de este locus no siempre permiten detectar variación interespecífica en especies estrechamente relacionadas filogenéticamente, por lo que es recomendable para una correcta identificación secuenciar varios loci nucleares. Otros loci comúnmente utilizados son el nuclear β-tubulina, y los mitocondriales de citocromo c oxidasa (CO1, cox2) (Bilodeau et al., 2007; Blair et al., 2008; Robideau et al., 2011). Adicionalmente, la técnica de PCR tiempo real ha proporcionado resultados que permiten detectar y cuantificar al patógeno de forma específica directamente en el hospedante (Lees et al., 2012, Engelbrecht et al., 2013).
El desarrollo de nuevas tecnologías en secuenciación masiva de ácidos nucleicos como metagenómica, permiten la detección y descubrimiento de patógenos de plantas. Las secuencias de nueva y tercera generación son tecnologías relativamente nuevas de secuenciación masiva de ácidos nucleicos muy útiles para el diagnóstico e identificación de especies de Phytophthora. Las cuales crean perfiles de grandes cantidades de secuencias permitiendo revelar todos y cada uno de los organismos presentes en la muestra (Espindola et al., 2015). Estas tecnologías están siendo cada vez de menor costo, lo que permitirá en un futuro cercano realizar identificación de especies a nivel de genomas.
Las bases de datos que se consideran confiables para la identificación de aislados de Phytophthora a través de secuencias, se pueden encontrar en los siguientes enlaces: http://www.phytophthoradb.org, http://www.phytophthora-id.org y http://www.q-bank.eu, ya que éstas cuentan con secuencias confirmadas con las que se pueden comparar mediante un análisis BLAST (Martin et al., 2012).
Conclusiones
La identificación morfológica y molecular se complementa, en particular cuando se encuentran nuevas especies. El utilizar únicamente técnicas moleculares permite encontrar un mayor número de especies; sin embargo, no es posible caracterizarlas morfológicamente lo cual es una limitante para su descripción. Asimismo, el diagnóstico oportuno e identificación correcta de especies de Phytophthora son fundamentales para prevenir la dispersión del patógeno, lo cual contribuirá a realizar un manejo adecuado de las enfermedades que ocasionan.











 texto en
texto en 



