Introducción
La guayaba (Psidium guajava, L.) es una fruta de importancia nacional, pues está integrada a la dieta del mexicano tanto como fruta fresca o sus derivados en forma de dulces regionales, mermeladas, galletas y licores, que además son exportados a los mercados latinos en el extranjero (Varela, 2010). En México se cultivan 20 899 ha de este frutal, de las cuales 30% pertenecen a huertos ubicados en el estado de Aguascalientes. De igual manera a nivel nacional se producen 302 719 t, de las cuales el 32% son producidas en dicho estado (SIAP, 2015). Estos datos de producción nacional colocan a México como el tercer productor de guayaba después de la India y Pakistán (Grattapaglia et al., 2012; Quesada et al., 2005) y a Aguascalientes como uno de los estados que más contribuyen conjuntamente con Michoacán. De la producción nacional, el 95% (288 670.85 t) se ha reportado del tipo “Media china” (SIAP, 2015).
La zona productora de Aguascalientes se ubica en el municipio de Calvillo y colinda con El Cañón de Juchipila del estado de Zacatecas. Esta zona en su conjunto se ha denominado “Calvillo-Cañones” y es una área compacta con gran importancia a nivel nacional en la producción de este frutal (Padilla et al., 2002). En dicho municipio se han reportado más de 400 huertos por parte del “Consejo Nacional Mexicano de la guayaba”, de los cuales la mayoría se encuentra en el rango de 0 a 3 hectáreas (SAGARPA, 2012).
Los genotipos considerados de origen local por los productores son compartidos en áreas vecinas de Zacatecas, conformando una región conocida como “Calvillo-Cañones” (Padilla et al., 2002; Padilla et al., 2007; Perales et al., 2005). Los frutos producidos en esta región tienen la reputación de ser los de mejor calidad a nivel nacional a pesar de que se ha observado que en campo existe variabilidad en los caracteres tales como forma de fruto, grosor del mesocarpio, color, tamaño y propiedades organolépticas que tienen influencia en el mercado nacional y recientemente internacional. No obstante, es precisamente esta calidad percibida de la guayaba de Calvillo la que ha impulsado a los productores locales a buscar una forma de impedir que frutos de otra procedencia sean etiquetados como si fuesen producidos en esta región.
Esta consideración sugiere la implementación de un método para la identificación de la “huella digital” de los frutos basándose en marcadores moleculares tales como Amplified Fragment Length Polymorphisms (AFLPs), (Randomly Amplified Polymorphic (RAPD DNAs), (Restriction Fragment Length Polymorphisms (RFLP), (Single Nucleotide Polymorphism (SNP) y (Simple Sequence Repeats (SSR). Este tipo de marcadores son utilizados para la detección de polimorfismos en la cadena de ADN, que pueden ayudar a identificar genotipos, a establecer distancias genéticas entre genotipos silvestres, mejorados, cruzas o poblaciones. Además se considera como una herramienta útil en el mejoramiento asistido (Provan et al., 1999; Chandra y Mishra, 2007; ISAAA, 2015).
En el caso específico de guayaba, se han realizado pruebas de marcadores tipo Inter Simple Sequence Repeats (SSR e ISSR), arrojando resultados confiables sobre patrones monomórficos en plantas derivadas de embriogénesis somática, de las cuales se esperaba homogeneidad genética (Liu y Yang, 2012; Rai et al., 2012). Otro uso de los marcadores SSR ha sido la evaluación de la diversidad genética en guayaba silvestre de Brasil, o la identificación de géneros Psidium, Zyzygium y Eugenia en estudios de diversidad de la familia Myrtaceae (Nogueira et al., 2012; Valdés et al., 2012a). Una nueva manera de evaluar la diversidad genética con marcadores con base en los sitios conservados de unión de retrotransposones o Inter Primer Binding Sites (iPBS) ha sido recientemente utilizada en guayaba (Mehmood et al., 2013).
Otras técnicas como RFLP combinado con Denaturing Gradient Gel Electrophoresis (DGGE) se han considerado como menos eficientes y más tardadas comparadas contra técnicas que utilizan secuencias específicas de rDNA como blanco (18S and ITS) y cpDNA (trnL intron and trnL-trnF IGS). No obstante, mediante la técnica RAPD que es aún más simple que las anteriores y con el uso de cebadores OPB17, OPG6, OPY15 y OPY18 ha sido posible obtener patrones de alto nivel de polimorfismo que han sido útiles en la discriminación de variedades cultivadas de guayaba (Tseng-Wei et al., 2007). RAPDs también ha sido útil en la discriminación de 33 genotipos de guayaba de Bangladesh diferenciados en dos principales grupos que reflejan principalmente las características morfológicas de los niveles de domesticación de las variedades presentes (Ahmed et al., 2011).
Es evidente que las condiciones ambientales no son indicadores confiables per se de la expresión genotípica (Kujal et al., 2005). Por lo tanto, el objetivo de esta investigación fue analizar el patrón de los marcadores moleculares contra el patrón de variación morfológica de las plantas analizadas en los diferentes genotipos encontrados en las huertas de Calvillo, Aguascalientes. Se espera que los resultados sean de utilidad en los programas de mejoramiento de guayaba en México.
Materiales y métodos
Material vegetal y su descripción morfológica
Muestras y fotografías de fruto y de hoja fueron colectadas durante el periodo 2012-2013 seleccionando de manera aleatoria un árbol por huerta en 140 de las 423 huertas reportadas para el área de producción de guayaba en Calvillo, Aguascalientes (SAGARPA, 2012). La morfología de fruto y de hoja fue descrita de acuerdo a las clasificaciones establecidas por la UPOV (1987) y complementados con otras descripciones (Sánchez y Peña, 2011; Sanabria, 2005). En total se utilizaron ocho descriptores morfológicos para fruto: peso, tamaño, diámetro ecuatorial, número de carpelos, forma, forma del ápice, forma de la base y forma de la cavidad. Para hoja: longitud, ancho, forma (de la hoja completa), forma del ápice y forma de la de la base. Para los datos cuantitativos se establecieron rangos de clasificación en categorías descriptivas tales como “pequeño”, “medio” y “grande”; o etiquetas de tipo ordinal de acuerdo al caso (Cuadro 1).
Extracción de ADN genómico
Las hojas colectadas fueron la fuente del ADN, extrayéndolo mediante el método de Doyle y Doyle (1990) con algunas modificaciones. El método consistió en moler 100 mg de hoja limpias con mortero y nitrógeno líquido. El polvo obtenido se colocó en un tubo para microcentrífuga que contenía 750 μL de buffer de lisis (100 mM Tris-HCl pH 8; 50 mM EDTA pH 8; 2% CTAB; 2M NaCl, 2% PVP 40; 3% β-mercaptoetanol) precalentado a 65 °C y se agitó durante tres minutos. Posteriormente se añadió un volumen de cloroformo-alcohol isoamílico (24:1), mezclando cuidadosamente durante cinco min para pasar a una centrifugación de 15 min a 13 000 rpm.
La fase acuosa se transfirió a un tubo nuevo y se añadió un volumen de cloroformo-alcohol isoamílico, repitiendo el proceso anterior. Una vez teniendo el sobrenadante en un nuevo tubo, se le agregó 5 μL de RNasa (10 mg mL-1) y se incubó durante 30 min a 37 °C. Se añadió un volumen de isopropanol frío, se incubó por 10 min a -20 °C y se centrifugó por 10 min a 13 000 rpm. Se descartó el sobrenadante y se lavó la pastilla con 500 μL de etanol al 70% agitando con vortex. Después de centrifugar los tubos a 13 000 rpm durante 10 min, se descartó el sobrenadante. Se secó el exceso de etanol mediante evaporación y se suspendió la pastilla en 50 μL de TE (10 mM Tris-HCl pH 8; 1 mM EDTA pH 8). La concentración de ADN en la muestra se determinó mediante un espectrofotómetro (UV-6100A UV/VIS, Metash, Shanghái) y las muestras se conservaron a -20 °C.
Procedimiento para efectuar las RAPD
Se utilizaron equipos con reactivos preparados de amplificación de ADN (GoTaq® Green Master Mix, PROMEGA) mediante PCR, siguiendo las indicaciones del fabricante. Después de varias pruebas para definir las condiciones óptimas de concentraciones de ADN, concentraciones de los cebadores y temperaturas de alineamiento, se definieron los parámetros para la mezcla de reacción de PCR (Cuadro 2) y para el programa del termociclador Techne TC-512 Thermal Cycler (Bibby Scientific Ltd., UK) (Cuadro 3). Se probaron un total de 22 cebadores decámeros aleatorios diseñados por Operon Technologies y producidos por IDT (Integrated DNA Technologies, USA) (Cuadro 4). Los productos de amplificación se corrieron rutinariamente en gel de 2.5% de agarosa con buffer TBE 1x durante 2:45 h aplicando 65V. El peso molecular aproximado se referenció mediante marcadores de peso molecular (50 bp, invitrogen).
*= http://www.operon.com/products/downloads/operonsrapd10mersequences.xls.
Cuadro 4 Cebadores utilizados* (operon technologies).
Elaboración de dendrogramas y su análisis
Los datos categorizados de origen molecular o morfológico (con base a ausencia/presencia, o en escala de rangos predeterminados) fueron analizados mediante el software Primer 5 (versión 5.2.8 para Windows). El método de análisis de similitud fue el de agregados (clusters) con el algoritmo de Bray-Curtis con transformación de raíz cuadrada. Con la matriz de similitud generada se construyeron los dendrogramas basándose en el método de promedio de grupo (Clarke y Gorley, 2001). El rango de correlación de Spearman de las variables consideradas en la matriz de similitud se realizó en múltiples corridas, y se reportó el mejor valor calculado tomando cinco variables a la vez por corrida para cada uno de los dendrogramas.
Resultados y discusión
Análisis de dendrograma elaborado con base a datos morfológicos
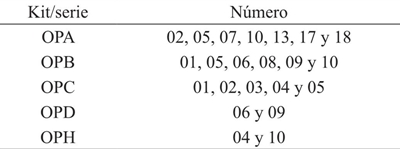
Al analizar los 16 caracteres morfológicos de hoja y fruto se encontraron especímenes de guayaba que fueron clasificados en dos o tres categorías según el descriptor. Once caracteres corresponden a fruto maduro y cinco a hoja desarrollada (Cuadro 1). El análisis del conjunto de datos morfológicos mostró un rango de similitud de 87 a 100% (Figura 1), con un rango de correlación de Spearman de r= 0.719, lo cual difiere de los resultados obtenidos con RAPDs. Sin embargo, estos resultados son comparables a los obtenidos por Muñoz (2012), quien utilizó seis descriptores para fruto y tres para hoja de 79 árboles de guayabo pertenecientes a igual número de huertos. De acuerdo con los resultados el rango de similitud de caracteres morfológicos de guayabos de Calvillo oscila de 87 a 100% con un rango de correlación de r= 0.909. De la misma manera, se tiene concordancia con los resultados de Kujal et al. (2005), ya que encontraron que los datos de RAPD obtenidos por ellos presentaron mucha mayor divergencia que los datos de morfología. Esto podría ser una evidencia de que durante el proceso evolutivo los marcadores de polimorfismos de ADN neutrales no tienen ningún efecto sobre la aptitud de los seres vivos para sobrevivir en el medio ambiente, y por lo tanto, no son sujetos a la presión de selección como ocurre con las secuencias codificantes. Los resultados de Domínguez et al. (2005), también indican que los cambios que ocurren a nivel de genoma en guayaba no siempre corresponden a los observados en el fenotipo. Gomes et al. (2010) por su parte, encontraron concordancia parcial entre los métodos de agrupamiento de marcadores RAPD y los descriptores morfológicos de guayaba. En contraparte, Feria (2008) pudo asociar tres bandas específicas de un patrón de RAPD con la acumulación de quercetina en guayaba.
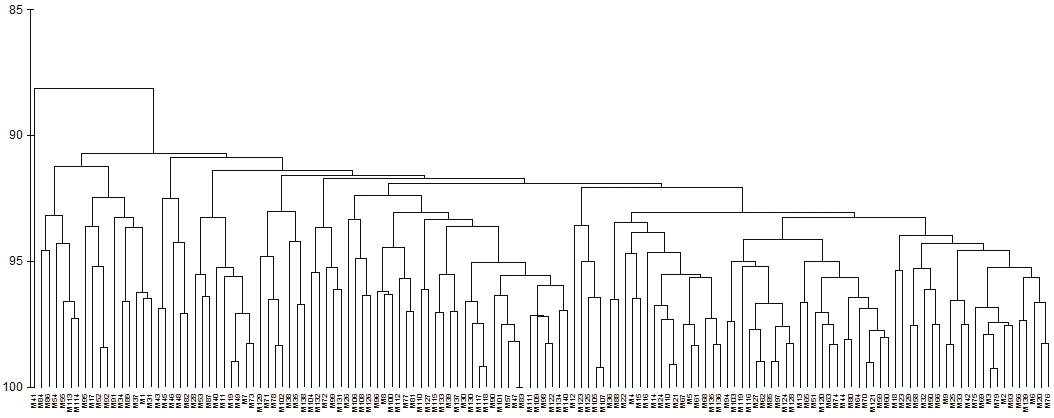
Figura 1 Análisis cladístico basado en datos morfológicos. Correlación de Spearman r= 0.719; A= frutos con base elíptica; B= genotipos con longitud y ancho de hoja medias; C= frutos redondos con diámetro ecuatorial >44 mm y grosor de mesocarpio >7 mm; D= frutos con cuatro carpelos y peso (40-70 g); E= frutos de aspecto piriforme con pulpa blanca.
Debido a que en el presente trabajo no se encontró una relación clara entre los marcadores moleculares y los descriptores de guayaba, se analizaron los caracteres morfológicos por separado, encontrándose que se generaron cinco grupos en el rango de similitud entre 91 y 94% que se describen con sus etiquetas (Figura 1).
Análisis de dendrograma elaborado con base a RAPDs
Desde las pruebas preliminares con los cebadores se encontraron bandas polimórficas en varios cebadores de la serie OPA y OPH producidas en función del genotipo (Figura 2). Las 10 bandas polimórficas fueron utilizadas para la elaboración del dendrograma en base a RAPDs. Los valores de similitud calculados para los 140 genotipos oscilaron entre 25 a 100% y un coeficiente de correlación de Spearman r= 0.794 (Figura 3), lo cual es coincidente con lo obtenido por Muñoz-Rodríguez (2012), quien encontró un rango de similitud de 30 a 100% con un rango de correlación de r= 0.927 utilizando RAPDs de solamente 26 genotipos de la zona productora de Calvillo con cebadores de las series OPA (10, 05 y 07) y OPB (01 y 09).
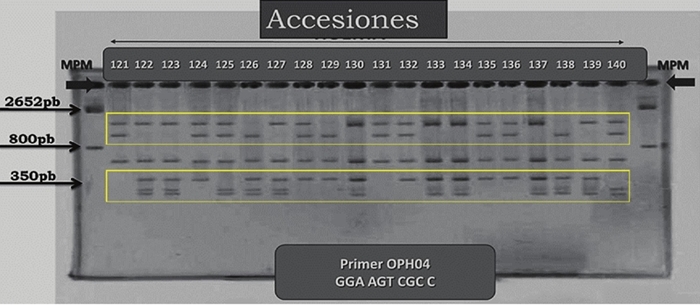
Figura 2 Ejemplo de gel que muestra la obtención de bandas polimórficas (dentro de los rectángulos amarillos) mediante RAPD en guayaba utilizando el cebador OPH-04. En la parte superior del gel se anotaron los códigos asignados a las muestras según el orden en que se fueron muestreando. MPM= marcador de peso molecular.
Figura 3 Análisis cladístico basado en RAPD realizado con 140 muestras de ADN de guayaba de Calvillo, Aguascalientes.Las etiquetas corresponden a muestras numeradas consecutivamente. Correlación de Spearman observada r= 0.794.
No es una relación clara entre la presencia de una banda en específico con las características morfológicas de las hojas o frutos de los árboles muestreados. Esto también coincide con los resultados de Muñoz (2012) aun cuando el número de muestras fue el equivalente a una quinta parte del número de muestras del presente trabajo. Los RAPDs en guayaba han sido propuestos para relacionarlos con caracteres morfológicos o nivel de domesticación. Por ejemplo, Tseng-Wei et al. (2007) fueron capaces de identificar dos grupos principales de guayaba procedentes de Taiwan utilizando dendrogramas derivados de RAPD: los genotipos comerciales y los genotipos silvestres. Este último grupo incluyó dos subgrupos que de manera general distinguían entre las guayabas de pulpa blanca y las de pulpa roja. No se pudo relacionar ninguna banda de RAPD con esos grupos.
En otro caso, con los cebadores OPA-02 y 03, Ahmed et al. (2011) observó 33.2% de bandeo polimórfico, que sirvió para determinar las distancias genéticas de 33 genotipos de guayaba en un rango pequeño (0.5253-0.6631) de índice de distancia. De acuerdo con los autores, uno de los agrupamientos encontrados está cercanamente relacionado con la morfología de los genotipos cultivados. Bajpai et al. (2008) obtuvieron dos diferentes dendrogramas de 22 genotipos de guayaba utilizando RAPD y amplificación directa de minisatélites de ADN (DAMD) con distancias genéticas de 5 a 43%. Los autores mencionaron que los genotipos de las planicies de la región del Indo-Ganges se compactan en un solo grupo, mostrando con esto que los marcadores RAPD pueden dar un patrón de identificación al menos para ese grupo.
Conclusiones
Se encontraron bandas polimórficas al utilizar la técnica de RAPD en árboles de guayaba seleccionados al azar procedentes de 140 huertas del municipio de Calvillo, Aguascalientes, México. Los marcadores de tipo RAPD obtenidos en este trabajo de investigación y anteriores, muestran la existencia de gran variabilidad a nivel molecular entre los individuos presentes en las huertas muestreadas. Estos marcadores pueden ser útiles para la selección de genotipos sobresalientes que se deseen propagar para ser utilizados en programas de mejoramiento genético asistido por marcadores moleculares.
Por su parte los agrupamientos mediante caracteres morfológicos mostraron una menor variabilidad, y no corresponden a los agrupamientos generados mediante RAPDs. No obstante, los proceso de búsqueda de marcadores moleculares asociados a características morfológicas de guayaba debe continuar, en especial para la identificación de genotipos seleccionados para el mejoramiento asistido. Alternativamente, debido a que los productores de guayaba locales que propagan vegetativamente su material están conscientes de la variabilidad de sus genotipos y reconocen a los mejores, convendría el uso de la técnica de RAPD para corroborar la uniformidad genética de los materiales que están propagando.











 texto en
texto en 





