Introducción
La producción total de jitomate en México es de 2.2 millones de toneladas en una superficie de 96 651 ha, es el primer exportador de tomates en el mundo con 1.4 millones de toneladas y de ventas externas del orden de los 2 038 millones de dólares anuales, genera 72 000 empleos directos y 10.7 millones de empleos indirectos (SIAP, 2013: Flores, 2014). Entre los factores que limitan la producción de este cultivo se encuentran los problemas fitosanitarios, como el cáncer bacteriano que es ocasionado por la bacteria Clavibacter michiganensis ssp michiganensis (Cmm), una enfermedad cuarentenada y de impacto internacional por las pérdidas económicas que ocasiona en la producción de jitomate, principalmente en invernadero. Cuando se presenta es devastadora y con gran capacidad de diseminación, Cmm puede sobrevivir en restos de plantas o en el suelo por periodos de 2 ó 3 años, lo cual dificulta su control (Fatmi y Schaad, 2002; Flores, 2014).
Cmm está distribuida en todas las zonas donde se cultiva jitomate, se han reportado epidemias en Israel, Japón, España y México, en promedio puede ocasionar pérdidas de 80 a 100% (Borboa-Flores et al., 2009; EPPO, 2010; De León et al., 2011; NAPPO, 2013). Las semillas infectadas constituyen la fuente primaria de inóculo y son responsables de la ocurrencia de brotes severos de la infección. Se requieren bajos niveles de contaminación de semilla para iniciar una epidemia, es difícil el diagnóstico oportuno debido a que cuando inicia la infección los síntomas tardan en presentarse, los primeros indicios del cáncer bacteriano generalmente aparecen entre 30 y 40 días después del trasplante, lo cual dificulta su control. Por lo anterior, es necesario contar con métodos confiables y de alta sensibilidad para realizar un diagnóstico efectivo (Chang et al., 1992; Milijašević et al., 2007; Milijasevic-Marcic et al., 2012). Por otro lado, no se han desarrollado variedades resistentes a este patógeno a pesar de los frecuentes intentos de mejoramiento (Eichenlaub y Gartemann, 2011).
Los métodos empleados para detectar a Cmm son la inmunofluorescencia y ELISA (Riley, 1987; Nemeth et al., 1991; Franken et al., 1993; Kaneshiro y Álvarez, 2001), que determinan presencia o ausencia, pero no diagnostican la virulencia. Otros métodos moleculares de detección incluyen hibridación tipo Southern empleando sondas derivadas de los genes CelA y pat-1 (Dreier et al., 1995) que codifican para una endocelulasa y una serina proteasa, respectivamente y de los plásmidos de virulencia pCM1 y pCM2 (Dreier et al., 1997); sin embargo, tales procedimientos serológicos y de hibridación tienen algunas limitantes como la baja sensibilidad y tiempos prolongados de espera de los resultados.
La detección del patógeno en plantas asintomáticas o infectadas al momento del trasplante es complicada, debido a que no se observan los síntomas, esto permite su diseminación y dificulta el control en almacigo, por ello, es necesario optimizar un método confiable y preciso de detección de Cmm, en el tejido vegetal e incluso para evitar la diseminación por el uso de semilla infectada. Las técnica de PCR es sensible y mediante el diseño de oligonucleótidos específicos combinando con procedimientos de extracción rápida del DNA (Holmes y Quigley, 1981; Dellaporta et al., 1983; Acosta-García et al., 2012) puede determinar la identidad y sugerir la virulencia de una bacteria fitopatógena (Dreier et al., 1995; Zhao et al., 2007; Rodríguez, 2013), además de evitar falsos positivos y negativos (Dreier et al., 1995; Souza-Santos et al., 1997; Pastrik y Rainey, 1999; Kaneshiro y Álvarez, 2001; Burokiene, 2006).
El objetivo de la presente investigación fue desarrollar un procedimiento de PCR para la identificación rápida y precisa de Cmm mediante el diseño de oligonucleótidos específicos anidados y la extracción del DNA por hervido para la amplificación de un fragmento del gen de virulencia pat-1 (serina proteasa) de 250 pb.
Materiales y métodos
Material biológico
La cepa de Clavibacter michiganensis ssp. michiganensis (Cmm) aislada de plantas de jitomate tipo Saladette provenientes de Zamora Michoacán, fue proporcionada por el laboratorio de fitobacteriología del Colegio de Postgraduados Campus Montecillo, Montecillo, Estado de México. La cepa Cmm fue mantenida en placas de medio selectivo YDC (Extracto de levadura, dextrosa, CaCO3 y agar) y propagada en medio líquido LB (Luria- Bertani) incubando durante 48 h a 28 oC. Las plantas de Lycopersicon esculentum Mill tipo Saladette con síntomas y asintomáticas a Cmm, fueron colectadas en campo abierto en la localidad “La Rinconada” de Zamora, Michoacán durante el ciclo primavera-verano de 2013. Para la transformación se emplearon células químicamente competentes de Escherichia coli (InvitrogenTM) TOP 10 F´. El plásmido TOPO TA 4 (InvitrogenTM) fue empleado para la clonación del producto de PCR. La enzima de restricción Eco RI fue empleada para la liberación de los productos de PCR clonados.
Extracción de DNA
Para la obtención del DNA de Cmm se empleó el método de hervido (Holmes y Quigley, 1981). Se prepararon tubos Eppendorf de 1.5 mL se colocaron 100 mg de perlas de vidrio de 450-600 μm y 150 μL de TE (Tris base, 50 mM; EDTA, 2 mM). Se transfirieron de 2-4 mg de bacterias a partir de placas sólidas con asa metálica, en tanto de material vegetal foliar con síntomas y asintomáticos fueron transferidos de 80-100 mg a cada tubo, en todos los casos se agitaron con vortex durante 2 min. Los tubos se sellaron con papel parafilm, se hirvieron durante 5 min a temperatura de 100 oC y se mezclaron por agitación durante 1 min. A cada tubo se le adicionaron 150 μL de una mezcla fenol: cloroformo (1:1, v:v), se agitó con la ayuda de un vortex por 1 min y se centrifugó a temperatura ambiente a 12 000 rpm durante 5 min. Se transfirió el sobrenadante de cada muestra a un nuevo tubo Eppendorf estéril, se adicionaron 100 μL de cloroformo, se sometió a vortex por 1 min y se centrifugó como en el paso anterior. De cada sobrenadante se empleó 1 μL como templado para la reacción de PCR. Como control, se realizó la extracción de DNApor el método de Dellapota (Dellaporta et al., 1983) recomendado para plantas. El aislamiento del DNA se verificó por electroforesis en gel de agarosa al 1.2% teñido con con GelRedTM (Biotum).
Reacción en cadena de la polimerasa (PCR)
La amplificación de un fragmento del gen pat-1 (serina proteasa) de Cmm se llevó a cabo por PCR utilizando los oligonucleótidos: CMM5F y CMM6R (Dreier et al., 1995; Cuadro 1) y como templado los DNA obtenidos por el método de hervido (Holmes y Quigley, 1981). Los componentes de la mezcla de reacción fueron: 17.4 μL de agua, 2.5 μL de reg taq 10 X, 1.5 μL de MgCl2, 1 μL de oligonucleótidos sentido y antisentido, 0.5 μL dNTPs, 0.1 μL Taq y 1.0 μL de DNA templado para un volumen total de reacción de 25 μL. Las condiciones del termociclador GeneAmp PCR System 2400 Perkin fueron: Desnaturalización 94 °C, 3 min, más 35 ciclos con las siguientes temperaturas y tiempos: 94 °C, 30 s; alineamiento 55 °C, 1 min; síntesis 72 °C, 1 min y finalmente 1 ciclo adicional a 72 °C, 5 min, en tanto para la obtención de amplicones de 250 pb se empleó una temperatura de alineamiento de 62 °C, durante 30 s y síntesis 72 °C, 30 s (Cuadro 1). Se verificó la amplificación de los productos de PCR por electroforesis en gel de agarosa al 1.2% teñido con GelRedTM (Biotum), se empleó el marcador de tamaño molecular GeneRuler 1 kb DNA Ladder (Life Technologies) y la imagen fue registrada en un transiluminador Vilber Lourmat (Marne-La-Valée Cedex, France).
Cuadro 1. Oligonucelotidos empleados en el presente trabajo.
| Iniciadores | Secuencia | Número | Tm |
|---|---|---|---|
| CMM5F | 5´GCGAATAAGCCCATATCAA3´ | 19 | 55 ºC (Dreier et al., 1995) |
| CMM6R | 5´CGTCAGGAGGTCGCTAATA3´ | 19 | 55 ºC (Dreier et al., 1995) |
| HCMA1S | 5´GCATTGCGGCGGTCGTGGTG3´ | 20 | 62 ºC (Presente estudio) |
| HCMB1As | 5´TCCGGCTACCGGCACGGATG3´ | 20 | 62 ºC (Presente estudio) |
| HCMC2S | 5´CGAAGCATTGCGGCGGTCGTG3´ | 21 | 62 ºC (Presente estudio) |
| HCMD2As | 5´TCCGGCTACCGGCACGGATGA3´ | 21 | 62 ºC (Presente estudio) |
Clonación, secuenciación y comparación
El producto de PCR fue aislado y clonado en el vector pCR4- TOPO®TA, se seleccionaron las colonias para extracción de DNA plasmídico y la digestión con la enzima Eco RI para verificar la presencia del inserto. Se realizó la purificación de DNA plasmídico de 2 clonas empleando columnas QIAprep Spin Miniprep Kit-Qiagen para obtener DNA en cantidad y calidad para secuenciar. Así, el índice A260nm/ A280nm se determinó empleando el espectofotometro Thermo ScientificTM NanoDrop 2000/200c, el cual fue cercano a 2.0. La secuencia de oligonucleótidos fue determinada empleando el secuenciador automático ABI PRISM 310 (Perkin Elmer, Norwalk, CT, USA). La comparación con la base de datos fue realizada empleando el algoritmo Blastx (Altschul et al., 1990) del Centro Nacional de Información Biotecnológica (NCBI).
Diseño de oligonucleótidos
Para el diseño de los 2 pares de oligonucleótidos anidados, temperatura de fusión (Tm) y tamaño del amplicón de 250 pb del gen pat-1 (serina proteasa) de Clavibacter michiganensis ssp michiganensis, se empleó el algoritmo OligoPerfectTM Designer de Invitrogen (Cuadro 1).
Resultados
Aislamiento del DNA de Cmm
Para la identificación de la cepa virulenta de Cmm se emplearon los métodos de extracción del DNA por hervido (Holmes y Quigley, 1981) y Dellaporta (Dellaporta et al., 1983). El resultado es mostrado en la Figura 1, en donde se observa una banda discreta y superior en tamaño a 10 000 pb correspondiente al DNA genómico de Cmm. El DNA obtenido por el método de hervido mostró considerable contaminación por RNAm (líneas 1 y 2), la cual se encuentra ausente en las muestras de DNA aislado por el método Dellaporta (líneas 3 y 4), por otro lado, el índice A260nm/ A280nm para el DNA aislado de la planta por el método de hervido fue de 1.71, en tanto para el DNA aislado por el método Dellaporta (Dellaporta et al., 1989) fue de 1.93. Adicionalmente, el DNA aislado por el método de hervido mostró una concentración de 0.595 μg μL- , en tanto la obtenida por el método Dellaporta fue de 0.117 μg μL- (Cuadro 2), sugiriendo que las muestras obtenidas por ambos métodos pueden ser empleadas como templado para realizar las reacciones de PCR.
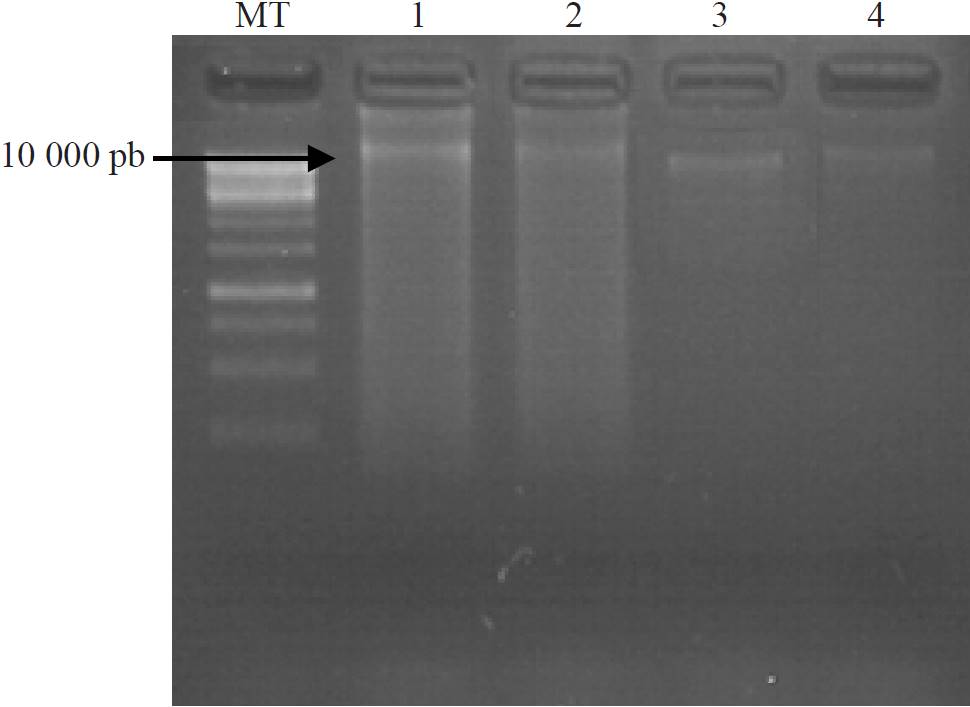
Figura 1. Extracción de DNA de Clavibacter michiganensis subespecie michiganensis (Cmm). Líneas: MT, marcador de tamaño; 1 y 2, DNA obtenido por el método de hervido; 3 y 4, DNA obtenido por el método Dellaporta.
Cuadro 2. Datos comparativos de 2 métodos de extracción de DNA genómico para Cmm y planta de jitomate.
| Método de extracción para Cmm | Tiempo | Material de partida (mg)a | Concentración de DNA (μg μL-)b | Purezad |
| Dellaporta | 19 h | 4 (0.778) | 0.125 (0.051) | 1.91 |
| Hervido | 30 min | 3 (0.408) | 0.576 (0.115) | 1.68 |
| Método de extracción para planta de jitomate | Tiempo | Material de partida (mg) | Concentración de DNA (μg/μL) | Pureza |
| Dellaporta | 19 h | 90 (14.14) | 0.117 (0.058) | 1.93 |
| Hervido | 30 min | 88 (17.79) | 0.595 (0.128) | 1.71 |
a= valor medio del material de partida para la extracción del DNA por los métodos de Dellaporta (Dellaporta et al., 1983) y de hervido (Holmes y Quigley, 1981); b= valor medio de la concentración del DNA obtenido; y c= valor medio del índice A260nm/A280nm. Las desviaciones estándar se muestran en paréntesis. Los experimentos fueron realizados por triplicado.
Amplificación del fragmento del gen pat-1 de Cmm
Para disminuir el tiempo de análisis se emplearon para la reacción de PCR como templado el DNA de Cmm obtenido por el método de hervido y los oligonucleótidos publicados CMM5F y CMM6R (Dreier et al., 1995; Cuadro 1). Los productos obtenidos fueron separados en un gel de agarosa al 1.2% teñido con GelRedTM (Figura 2), en donde se observa la amplificación de una banda superior a 500 pb (Línea 1), que pudiera corresponder al fragmento del gen pat-1 (serina proteasa), la cual posee un tamaño de 614 pb (Dreier et al., 1995). Como control negativo de la reacción de PCR (línea 2), se empleó como templado el DNA obtenido por hervido de planta de jitomate sana y los oligonuclótidos CMM5F y CMM6R.
Identificación de Clavibacter michiganensis ssp. michiganensis (Cmm)
Para identificar a la cepa Cmm patogénica, el fragmento de PCR de aproximadamente 600 pb fue clonado en el vector pCR4-TOPO®TA y transformado en células de Escherichia coli químicamente competentes. El DNA plasmídico fue purificado para la obtención de la secuencia, la cual es mostrada en la Figura 3, en donde se señalan el oligonucleótido CMM5F y el complemento del oligonucleótido CMM6R, sugiriendo que un fragmento de 608 pb del gen pat-1 (serina proteasa) de Cmm ha sido clonado. La secuencia obtenida fue comparada con la base de datos del Centro Nacional de Información en Biotecnología (National Center of Biotechnology Information NCBI) empleando el algoritmo Blastx (Altschul et al., 1990) que traduce la secuencia y la compara con las proteínas registradas, el resultado se muestra en el Cuadro 3, en donde se observan secuencias de algunas proteínas de las cepas con los respectivos números de acceso: Clavibacter michiganensis ssp michiganensis (WP_011931204.1), C. michiganensis ssp michiganensis NCPPB 382 (CAM98537.1), C. michiganensis ssp. michiganensis (AEM65933.1), C. michiganensis ssp. michiganensis (AEM65932.1), entre otras, cuyo valor E fue cercano a 0.0 con el gen pat-1 (serina proteasa) de Clavibacter michiganensis ssp michiganensis, confirmando la identificación de Cmm patogénica.

Figura 3. Secuencia de bases del fragmento de 608 pb del gen pat-1 (serina proteasa) de Cmm. Las secuencias de los oligonucleótidos CMM5F y la segunda cadena de CMM6R se muestran subrayados.
Cuadro 3. Secuencias que mostraron alineamiento significativo (NCBI) con valor E cercano a 0.0.
| Descripción | Valor E | No. de acceso |
|---|---|---|
| Serina proteasa [C. michiganensis ssp michiganensis] | 1e-126 | WP_011931204.1 |
| Serina proteasa [C. michiganensis ssp michiganensis NCPPB 382] | 4e-102 | CAM98537.1 |
| Serina proteasa [C. michiganensis ssp michiganensis] | 4e-75 | AEM65933.1 |
| Serina proteasa [C. michiganensis ssp michiganensis] | 7e-73 | AEM65932.1 |
Diseño de oligonucleótidos anidados para la producción de amplicones de 250 pb por PCR
En base a la secuencia obtenida se diseñaron 2 pares de oligonucleótidos anidados para la producción de amplicones de 250 pb, con una temperatura de alineamiento (Tm de 62 °C) los cuales fueron denominados arbitrariamente HCMA1S-HCMB1As (20-mero) y HCMC2S- HCMD2As (21-mero). Los DNA aislados por el método de hervido de plantas de jitomate con síntomas a Cmm patogénica fueron empleados como templado para la amplificación por PCR de un fragmento del gen pat-1 (serina proteasa). El resultado es mostrado en la Figura 4, en donde se observa la amplificación de una banda específica de tamaño de 250 pb con ambos pares de oligonucleótidos anidados (líneas 1 y 2). Como control positivo se realizó el PCR usando como templado el DNA de plantas de jitomate con síntomas de Cmm y los oligonucleótidos CMM5F y CMM6R, el cual generó el fragmento esperado de 608 pb. Como control negativo, se emplearon como templado el DNA aislado de plantas de jitomate asintomáticas a Cmm y el par de oligonucleótidos anidados HCMA1S-HCMB1As, permitiendo el desarrollo de un procedimiento de PCR rápido y específico, combinando la extracción del DNA por el método de hervido (Holmes y Quigley, 1981) y los oligonucleótidos anidados con Tm de 62 °C para la producción de amplicones de 250 pb del gen pat-1 (Serina proteasa) para la identificación de Cmm patogénica.

Figura 4. Amplificación por PCR de un fragmento de 250 pb del gen pat-1 de Cmm a partir de DNA de plantas con síntomas. Líneas: MT, marcador de tamaño; 1) oligonucleótidos anidados HCMA1S y HCMB1As; 2) oligonucleótidos anidados HCMC2S y HCMD2As; 3) control positivo, oligonucleótidos CMM5F y CMM6R; y 4) control negativo, DNA de planta de jitomate asintomática como templado y los oligonucleótidos anidados HCMA1S y HCMB1As.
Discusión
En el presente estudio se desarrolló un procedimiento basado en la técnica de PCR para la detección de Cmm en plantas de jitomate (Lycopersicon esculentum Mill) tipo Saladette. Para ello nosotros propusimos el combinar la extracción del DNA por el método de hervido (Holmes y Quigley, 1981) y el uso de los oligonucleótidos HCMA1S- HCMB1As (20-mero) y HCMC2S- HCMD2As (21-mero) con Tm de 62 °C para la amplificación de fragmento de 250 pb del gen pat-1 (serina proteasa) de Cmm. Con el método de extracción por hervido, el cual consume un tiempo de aproximadamente 30 min, el DNA presentó una fracción de RNA (Figura 1): sin embargo, no mostró diferencia con el método Dellaporta (Dellaporta et al., 1983) descrito para plantas, el cual requiere aproximadamente 19 h para completarse.
Debido a que en ambas muestras la banda correspondiente al DNA genómico se presenta en forma discreta de un tamaño superior a las 10 000 pb, con un índice A260nm/ A280nm promedio de 1.70 lo que indica DNA de calidad (Cuadro 2). La concentración del DNA obtenido por el método de hervido fue superior a la obtenida por el método Dellaporta, lo anterior se justifica dado que el espectofotómetro registra la concentración de los ácidos nucleicos, que en este caso incluye al RNA total (Cuadro 2). Para la amplificación por PCR del gen pat-1 (Serina proteasa) de Cmm, al emplearse el DNA aislado por hervido como templado y como iniciadores los oligonucleótidos CMM5F y CMM6R (Dreier et al., 1995), el amplicón obtenido fue una banda superior a 500 pb (Figura 2, línea 1), sugiriendo que corresponde al fragmento del gen pat-1 (serina proteasa), que ha sido reportado como un tamaño de 614 pb (Dreier et al., 1995), lo cual fue confirmado con la secuenciación del fragmento.
La nula amplificación a partir del DNA obtenido por hervido de planta de jitomate asintomática y los oligonuclótidos CMM5F y CMM6R (Figura 2, línea 2) y HCMA1S- HCMB1As (Figura 4, línea 4), confirman la especificidad de la reacción de PCR para el DNA de Cmm. La reducción del tiempo de análisis para la detección de Cmm, también es influido por el uso de oligonucleótidos anidados (HCMA1S- HCMB1As y HCMC2S-HCMD2As) con una Tm de 62 °C y tiempos de alineamiento y de síntesis de 30s para la producción de amplicones de 250 pb. Estas condiciones reducen el tiempo total del programa de PCR, al disminuir el tiempo necesario para la amplificación de 4 h que se requerían con el anterior protocolo a 3 h, empleando como templado el DNA obtenido por hervido. En la Figura 4 (líneas 1 y 2), se muestra la síntesis de fragmentos específicos de 250 pb del gen pat-1 (serina proteasa).
Conclusiones
Se describe un procedimiento rápido, sencillo y preciso basado en PCR para la detección de Cmm patogénica que consume aproximadamente 4 h a partir del arribo de la muestra comparado con el método convencional que requiere 24 h, lo que permitirá establecer en menor tiempo, estrategias para evitar la diseminación del inóculo del cáncer bacteriano en plantas de jitomate.











 texto en
texto en 



