Introducción
La especie Carica papaya L. se cree que es nativa del sur de México y los países vecinos de América Central; actualmente, se distribuye a través de las regiones tropicales y subtropicales del mundo (Fuentes y Santamaría, 2014). Aunque México es uno de los principales productores de papaya, no cuenta con variedades locales. La variedad Maradol es ampliamente utilizada, en la que se ha demeritado la calidad de forma, tamaño (Posada et al., 2010) y el color de la pulpa del fruto. La forma de fruto en papaya depende de la variedad y del tipo de flor de la cual se haya formado. Por lo general, las flores femeninas generan frutos redondos o de esféricas a ovalados, mientras que las flores hermafroditas generan frutos alargados, cilíndricos, obovoides o pirifotrmes (Magdalita y Mercado, 2003).
En varios países los frutos alargados tienen mayor aceptación comercial (Manica, 1996; Alonso et al., 2008). En América Latina hay una fuerte preferencia por la papaya de frutos grandes en los mercados domésticos (Ferraguetti, 2003). De acuerdo a Marin et al. (2003) existe preferencia por plantas de papaya hermafroditas con formato periforme y alargado, lo que está asociado a una menor cavidad ovariana y un mayor espesor de la pulpa, característica que les confiere mayor valor comercial a este tipo de frutos en el mercado. Por lo tanto, es de interés conocer el sexo de los parentales para obtener un nuevo material cultivado con una segregación propicia de plantas hermafroditas que favorezcan la presencia de estos frutos. La predicción fenotípica del sexo en papaya es difícil en etapas tempranas de desarrollo de la planta (Sánchez-Betancourt y Nuñez, 2008). El colocar tres plantas por poceta y programar un aclareo a los tres meses después del trasplante, favorece la presencia de al menos una planta hermafrodita que producirá frutos alargados, sin embargo, se incrementan los gastos por manejo del cultivo (Kanchana-udomkan et al., 2014).
Marcadores de DNA tales como SCAR, RAPD, RFLP y DAF han sido asociados al tipo sexual en plantas de papaya (Deputy et al., 2002; Urasaky et al., 2002; Lemos et al., 2002; Chaves y Nuñez, 2007; Somsri y Bussabakornkul, 2008). Por su parte el color de los frutos depende de su composición de carotenoides. En papayas Hawaianas Solo de pulpa roja, Yamamoto, (1964) mostró que los frutos rojos acumulan licopeno que representa el 63% del contenido total de carotenoides. Por su parte los frutos amarillos deben su color a una acumulación de β-caroteno y β-criptoxantina, que representa 75% del contenido total de carotenoides. , mientras que la fruta de pulpa roja contiene aproximadamente la mitad de esa cantidad. Para determinar el color de pulpa que tendrán los frutos de una determinada variedad por métodos fenotípicos hay que esperar a que la planta produzca sus primeros frutos meses después. En papaya los primeros frutos son emitidos aproximadamente 7 meses después del trasplante (Calixto, 2011).
Entre 7 a 9 meses después del trasplante, cuando se generan los primeros frutos. En relación al tamaño de fruto, se puede observar una amplia variación en el tamaño de fruto de papaya. Reportándose rangos de longitud 7-30 cm (Asudi et al., 2010), y de peso de fruto desde menos de 100 g hasta 10 kg (Jiménez et al., 2014). Sin embargo, frutos comerciales presentan un rango de peso de 0.5-2.0 kg y una longitud de fruto de 10 a 20 cm (Samson, 1986). Demey et al. (2003) y Olmos (2004) sugieren que la identificación de secuencias de ADN próximas al gen o locus de interés agronómico se puede utilizar como una herramienta para acelerar los procesos de selección tradicionales del cultivo y facilitar el trabajo del fitomejorador. Blas et al. (2010) desarrollaron un marcador molecular (CPFC1) asociado con el color de pulpa de Carica papaya L. tipo Hawaiano y Chen et al. (2007) reportaron un marcador que identifica plantas hermafroditas y femeninas en papaya Sunup y AU9.
En la presente investigación, mediante la validación de marcadores moleculares se busca determinar desde temprana edad, el tipo sexual de la planta así como el color y tamaño de frutos para favorecer la selección temprana de materiales promisorios sin tener que esperar que los materiales produzcan flores y frutos, con la inminente reducción del tiempo para generar nuevas variedades. Para ello, se emplearon marcadores moleculares para tipo sexual, color de la pulpa y ocho QTLs asociados a características morfológicas del fruto en una población segregante de papaya derivada de la cruza entre un genotipo silvestre x uno comercial (var. Maradol). A plantas jóvenes tanto de los parentales como de los materiales derivados de la cruza, se les extrajo ADN y se clasificaron en base a los resultados del análisis basado en marcadores moleculares, meses después, el agrupamiento basado en dichos marcadores, fue cotejado con el agrupamiento basado en la caracterización visual y morfológica de flores y frutos de las mismas plantas.
Materiales y métodos
Material vegetal
El experimento se desarrolló en dos periodos: octubre 2009 a mayo 2010 y septiembre 2012 a abril 2013, en el Centro de Investigación Científica de Yucatán A. C. (CICY) en Mérida Yucatán México La temperatura media anual del sitio experimental es de 26.1 °C y una precipitación de 1,012 mm (Conagua). A partir de la cruza entre papaya Criolla línea 7 (hermafrodita, pulpa amarilla-naranja) y papaya Maradol 22 (hermafrodita, pulpa naranja-rojiza), se desarrollaron 42 plántulas F1 y se realizaron autopolinizaciones de H90B, H77B y H70B (genotipos F1) para el desarrollo de 162 plántulas F2 de las cuales 83% de la población fue descartada para la evaluación por el ataque de una enfermedad viral. La presente investigación se realizó a cielo abierto en un vivero con cerca perimetral. Las plantas se manejaron bajo riego con adecuada fertilización y control de plagas y malezas siguiendo el paquete tecnológico descrito en (Cituk, 2006). Los progenitores son materiales homogéneos y estables.
Caracterización molecular
Se empleó material foliar de las plántulas F1 y F2 y se utilizó un método de extracción de ADN basado en CTAB sugerido por Doyle y Doyle (1990), con algunas modificaciones. Mediante PCR fueron amplificados los marcadores moleculares CPM1815YC2 (Chen et al, 2007) y CPFC1 (Blas et al., 2010) asociados con el carácter de sexo y color de pulpa, respectivamente; además de marcadores QTLs asociados a la morfología del fruto tales como: P3K1700CC, ctg-43C0, CPM1556C0 y P3K4311bK0 para peso, P3K6372CC y P6K969CC para longitud, CPM1550C0 para diámetro y P3K2426aK3 para forma (Blas, 2008). Los fragmentos fueron visualizados a través de electroforesis en agarosa al 5%. El tamaño de fragmento esperado fue calculado in silico con el software prfectBLAST, el cual indicó la migración aproximada del producto amplificado para los marcadores moleculares en parentales (L7 y M22) y progenie F1 y F2.
Caracterización morfológica
En toda la progenie F1 y solo en 17% de población F2 se colectaron tres frutos por planta en madurez fisiológica, esta madurez se determinó de acuerdo a Santamaría et al. (2009). Los frutos fueron transportados al laboratorio de Fisiología Molecular del CICY y se evaluaron el peso, la longitud, el diámetro y la forma y el color del fruto basado en los descriptores reportados por la UPOV (2010).
Resultados y discusión
Análisis molecular
Los marcadores moleculares ligados a caracteres de interés comercial QTL (quantitative trait loci por su siglas en inglés), constituyen una herramienta para aumentar la eficiencia de los programas de mejoramiento genético tradicionales; ya que sirven para identificar, seleccionar y mantener los genotipos que contienen la combinación de alelos deseados y para descartar aquellas que no los presenten; permitiendo una disminución en la demanda de tiempo y recursos en los estudios encaminados en la selección de genotipos elites (Valadez y Gûnter, 2000).
Marcador molecular CPM1815YC2: identificación de sexo de Carica papaya L.
El tamaño del fragmento esperado del marcador molecular CPM1815YC2 que se predijo mediante el programa bioinformático prfect BLAST (Santiago y Ramírez, 2012) fue de 167 pb. Este marcador molecular ligado al tipo sexual de Carica papaya L. amplificó una doble banda o una sola banda, dependiendo del tipo de planta analizada. El tamaño de amplicón en genotipos con doble banda corresponde a 175 y 195 pb, respectivamente, mientras que en genotipos con una sola banda fue de 190 pb (Figura 1a). Los parentales Criolla Línea 7 y Maradol 22 presentaron doble banda amplificada, ya que ambos son plantas hermafroditas. De la misma manera, se presentó doble banda en 28 genotipos F1, mientras que en los 14 individuos F1 restantes se apreció una sola banda correspondiente a plantas de sexo femenino (Figura 2a). En la progenie F2 generada por la autopolinización de H90B, H77B y H70B se presentaron 35, 40 y 39 genotipos con doble banda, y 19, 14 y 15 genotipos con una sola banda amplificada, respectivamente (Figura 3a).
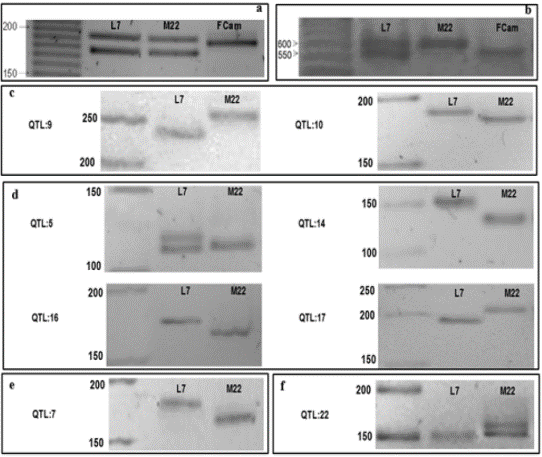
Figura 1 Electroforesis del marcador molecular de sexo CPM1815YC2 (a) y de color de pulpa CPFC1 (b) amplificado en parentales Criolla línea 7, Maradol 22 y una planta silvestre femenina y pulpa amarilla clara de Carica papaya L., y de ocho QTLs asociados a longitud (c), peso (d), diámetro (e) y forma (f) del fruto, en gel de agarosa de alta resolución al 5% teñido con bromuro de etidio. Marcador de peso molecular de 10 pb y 50 pb.
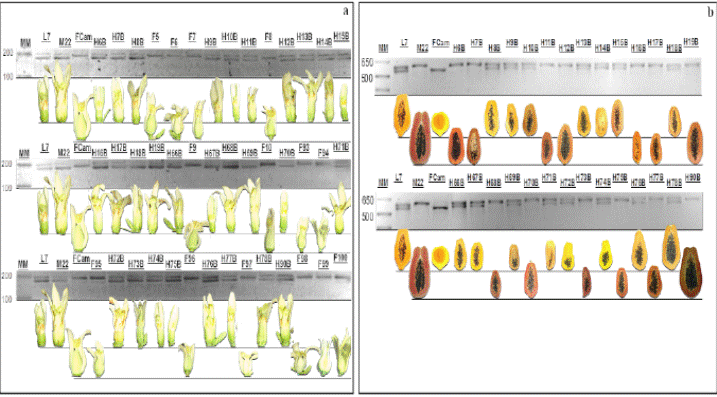
Figura 2 Electroforesis del marcador molecular de sexo CPM1815YC2 (a) y de color de pulpa CPFC1 (b) amplificado en parentales Criolla L7, Maradol 22 y progenie F1 provenientes de la cruza L7xM22 de Carica papaya L. L7: población Criolla línea 7, M22: Maradol 22, FCam: femenina silvestre, H: plantas hermafroditas en gel de agarosa al 5% teñido con bromuro de etidio. MM: 1 Kb. Se incluye la fotografía de la flor y fruto correspondiente a cada progenie F1, obtenida a los 6 y 9 meses después de haber tomado la muestra de ADN, respectivamente.

Figura 3 Electroforesis del marcador molecular de sexo CPM1815YC2 (a) y de color de pulpa CPFC1 (b) amplificado en parentales Criolla L7, Maradol 22 y progenie F2 provenientes de la autopolinización de H90B, H77B y H70B en Carica papaya L. L7: población Criolla línea 7, M22: Maradol 22, FCam: femenina silvestre, H: plantas hermafroditas en gel de agarosa al 5% teñido con bromuro de etidio. MM: 1 Kb. Se incluye la fotografía de flor y fruto correspondiente a cada progenie F2, obtenida a los 9 meses después de haber tomado la muestra de ADN.
Estos resultados coinciden con lo reportado por Chen et al. (2007) quienes probaron este marcador molecular en materiales de papaya Hawaiana e identificaron como genotipo femenino y hermafrodita a AU9 y Sunup, respectivamente con una y doble banda respectivamente. Nuestros datos arrojan que de 42 plantas en la progenie F1, 66.6% son hermafroditas y 33.3% son femeninas (relación 2:1; hermafroditas:femeninas). Dicha relación es la esperada en una segregación alélica de una planta hermafrodita autofecundada o de ambos parentales herms como lo reportan Ming et al. (2007); Niroshini, et al. (2008).
Mientras que en la progenie F2 obtenida de la autopolinización de H70B, H77B y H90B presentaron por un lado 72.2%, 74.4% y 64.8% de condición alélica hermafrodita, y 27.8%, 25.9% y 35.2% de condición alélica femenina, respectivamente. Estos resultados concuerdan de manera proporcional con lo reportado por Esquivel et al. (2008) para los cultivares HGxMR (65.5 y 34.5%, hermafroditas y femeninas, respectivamente), HGxMA (57.8 y 42.2%, hermafroditas y femeninas, respectivamente) y Maradol Roja (68.9 y 31.04%).
El tipo sexual en Carica papaya L. solo puede ser visualizado hasta iniciada la floración (a partir de los tres meses después del trasplante aproximadamente) como lo reportaron Ming et al. (2007); Niroshini, et al. (2008); Reddy et al. (2012). De manera que la determinación del sexo de las plántulas de papaya a nivel molecular antes del trasplante sería una ventaja en ahorro de tiempo, espacio y dinero para el fitomejorador, siendo un complemento de la selección tradicional de plantas aptas para la producción de frutos de acuerdo con las exigencias del mercado, o para incorporarlas al programa de selección y mejora genética de la especie (Sánchez-Betancourt y Nuñez, 2008).
Además, los frutos de papaya provenientes de flores hermafroditas, generalmente son de forma alargada, con firmeza suficiente para resistir los daños mecánicos poscosecha y de mayor demanda comercial por ocupar menor espacio por unidad de volumen al momento del empaque, lo que representa un ahorro en el flete; principalmente para mercados de exportación (Muñozcano y Martínez, 2009). La validación del marcador CPM1815YC2 para discriminar plantas femeninas de las hermafroditas en 42 genotipos F1 (provenientes de L7xM22) y 162 genotipos F2 derivados de la autopolinización de H70B, H77B y H90B es confiable ya que se logró a nivel molecular identificar y clasificar al 100% ambos tipos sexuales en plantas de papaya lo que muestra un gran potencial para su uso en la selección asistida por marcadores (SAM) en otros genotipos silvestres, criollos y comerciales.
Marcador molecular CPFC1: identificación de color de Carica papaya L.
La coloración de pulpa en papaya, además del tamaño y forma del fruto es un aspecto que determina su aceptación por el consumidor. Desde el punto de vista nutricional, la identificación y obtención dirigida a obtener materiales de Carica papaya L. con pulpa naranja-rojizo favorece la presencia de un mayor contenido de licopeno, lo que le imprime el color naranja-rojizo a la pulpa del fruto. La intensidad del color depende de la concentración de este pigmento, el cual actúa como antioxidantes en beneficio de la salud humana, al reducir el riesgo de padecer cáncer de pulmón, estómago y próstata (Giovannucci, 1999). Mientras que en genotipos con frutos de pulpa amarilla, el contenido de β-carotenos es mayor que el contenido de licopeno. Con el marcador de color de pulpa (CPFC1) se observaron tres formas alélicas en la progenie evaluada.
El parental M22 cuya característica fenotípica fue de poseer pulpa naranja-rojizo y 13 genotipos F1 presentaron una sola banda con tamaño de 600 pb, cuya condición alélica es homocigota recesiva. Mientras que el parental L7, el cual posee pulpa amarilla y 15 individuos F1 presentaron doble banda una de 600 pb y otra de 550 pb, característica alélica de genotipos heterocigotos. Además, se analizó un material silvestre (FCam) con color de pulpa amarilla claro y se observó una banda de 550 pb; es decir, su condición alélica es homocigota dominante (Figura 1b y 2b), estos datos resultaron próximos a las 568 pb calculado mediante prfectBLAST.
A nivel molecular, la proporción en la población F1 fue aproximadamente de 53.57% para genotipos con pulpa amarilla, y de 46.43% para genotipos con pulpa naranja- rojizo (relación 1:1 aprox.) (Figura 2b). Dentro de la progenie F2 evaluada se observó que el alelo recesivo de color naranja-rojizo se introgresó en un 100% para la progenie F2 provenientes del genotipo H90B, en 98.1% de los genotipos provenientes de H70B y en 92.6% de los genotipos provenientes de H77B (Figura 3b). Cabe resaltar, que en la población F2 proveniente de H70B, el genotipo H70-57 presentó condición heterocigota segregante del parental L7, por otra parte, en la población F2 provenientes de H77B, los genotipos H77-12, H77-52, y H77-54, presentaron condición heterocigota segregante del parental L7 y solamente el genotipo H77-43 presentó condición homocigota dominante semejante a la que presenta la FCam.
La condición alélica obtenida con el marcador de color CPFC1 en la progenie F1 y F2 de esta investigación coincidió con la que reportaron Blas et al. (2010), quienes indican que el material SunUp de color de pulpa roja amplifica una sola banda y tiene condición homocigota recesiva, mientras que los híbridos Rainbow y Richter presentan pulpa amarilla y tienen condición heterocigota, es decir, contienen dos alelos manifestando una amplificación de doble bandas. También mencionaron que el material Kapoho presenta color de pulpa amarilla pero posee una condición homocigota dominante. Al respecto, Rodríguez (2008) reportó que la característica de pulpa amarilla (alelo dominante) predomina sobre la pulpa naranja-rojiza, por lo que en las poblaciones silvestres de papaya rara vez aparecen frutos naranja-rojizos, debido a que este color está dado por genes recesivos.
El marcador molecular de color de pulpa (CPFC1) en genotipos F1 y F2 provenientes de la cruza L7xM22 facilitó la detección temprana de genotipos con pulpa naranja-rojizo (fenotípicamente podría ser obtenida a los 7-9 meses después del trasplante) y logró una correlación tanto molecular como morfológica en lo que se refiere a la proporción de segregación esperada. De manera que este marcador específico también puede tener un uso potencial importante en la selección asistida por marcadores (SAM) para el carácter de color de pulpa, empleado en forma temprana en plantas de vivero sin tener que esperar hasta 9 meses para visualizar el color del fruto de los genotipos usados en programas de mejoramiento genético de papaya.
Marcadores QTLs asociados a la morfología del fruto de Carica papaya L.
Los tamaños de fragmentos obtenidos in silico con valores entre 84 pb a 174 pb (datos no mostrados) fueron los esperados y aproximados a los QTLs microsatélites (SSR) amplificados por PCR, en parentales L7, M22 y progenie F1 y F2 (Figura 4), además congruentes con lo reportado por Mittal y Dubey, (2009) quienes mencionan que los fragmentos amplificados de SSR son de tamaño variante, generalmente de varias decenas hasta algunas centenas de pares de bases (pb), codominantes, polimorficos y de segregación mendeliana, atributos que pudieron constatarse en esta investigación.

Figura 4 Electroforesis de marcadores moleculares QTLs asociados a la morfología del fruto amplificados en los parentales Criolla línea 7 y Maradol 22 de Carica papaya L. y su progenie F1(a) y F2 (b). Se incluye la fotografía del fruto correspondiente a cada progenie F1 y F2, obtenida a los 9 meses después de haber tomado la muestra de ADN. MM: 50 pb.
Los ocho QTLs asociados a caracteres de morfología de fruto en parentales L7 y M22, se presentan en la Figura (1 c-f) 2 asociados a longitud (1c), 4 asociados a peso (1d), 1 asociado a diámetro (e) y 1 a forma (f) del fruto. Ninguno de los ocho QTLs ni en los parentales ni en la progenie F1 y F2, mostraron correlación molecular y fenotípica (Figura 4, Cuadro 1); es decir, no ofrecen información útil para su uso en la selección asistida por marcadores para las características del fruto de papaya tales como longitud, peso, diámetro y forma de fruto. Es posible que la falta de correlación se deba a que esos caracteres exhiben una extensa variación a lo largo de un gradiente continuo, atribuidos a la interacción de dos o más genes (poligénicos) (Obando et al., 2008). No se descarta que existan efectos pleiotrópicos sobre los QTLs asociados a la morfología del fruto de papaya, los cuales guardan estrecha relación con los genes ovate, sun y fw.2.2 presentes en el genoma de tomate y en regiones ortólogas en el genoma de papaya (Paull et al., 2008; Blas et al., 2012).
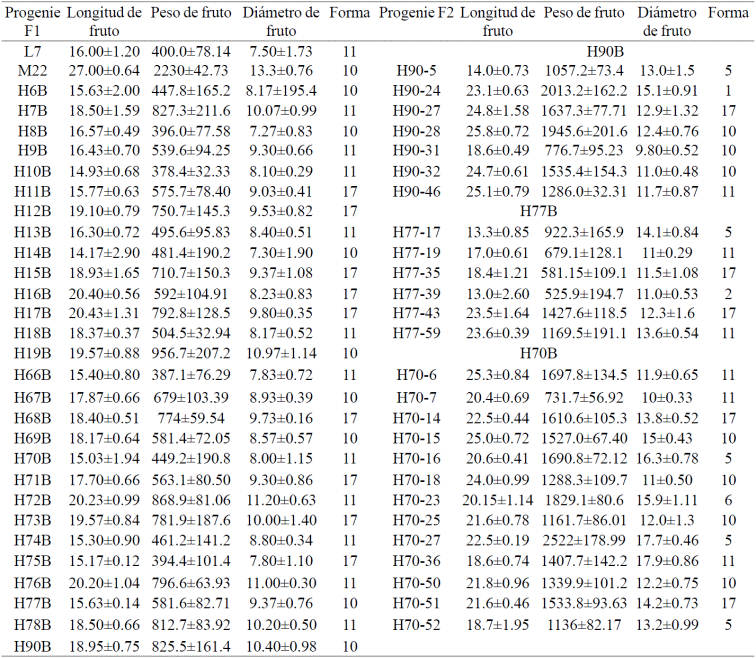
Cuadro 1 Valores medios de caracteres morfológicos asociados al tamaño y forma del fruto de progenie F1 y F2 proveniente de la cruza intraespecífica L7xM22 de Carica papaya L. Forma del fruto de Carica papaya L. según IPBGRI. 1= globular; 2= redondo; 5= oval; 6= oblongo; 10= cilíndrico; 11= periforme; y 17= ciruela.
En otras especies como tomate, se ha sugerido que factores tales como la fertilización, temperatura y humedad van a influir directamente en la expresión del gen sun que regula la elongación temprana del pericarpio del ovario, del gen ovate involucrado en la elongación del pericarpio del ovario después de la fertilización y del gen fw2.2 que regula la división celular y desarrollo del fruto. Se asume que el efecto en papaya puede ser similar, de tal manera que los QTL que estén influenciados por esos genes se verán afectados, tal es caso de algunos genotipos evaluados. O bien, estos QTLs evaluados inicialmente en papaya Hawaiana, no están asociados a los caracteres morfológicos del fruto tales como longitud, peso, diámetro y forma del fruto en híbridos de Maradol x material criollo de Carica papaya L.
Análisis morfológico
La identificación molecular del tipo sexual coincidió con el dato fenotípico registrado y evaluado tres meses después del trasplante, tanto en los parentales como en la progenie F1 y F2 de Carica papaya L. (Figuras 2a y 3a). La población F1 obtenida de la cruza intraespecífica L7xM2 y la F2 proveniente de autopolinizaciones de los genotipos H90B, H77B y H70B desarrollaron solamente plantas hermafroditas y femeninas. Para la F1 los valores fueron 28 hermafroditas y 14 femeninas; lo que representa un 67% de hermafroditismo y un 33% de feminismo. Dentro de la F2 los porcentajes fueron de 64%, 72% y 67% de hermafoditismo y 36%, 28% y 33% de feminismo, para H90B, H77B y H70B, respectivamente, quienes se mantuvieron próximos a la proporción de 2:1 esperada.
De igual forma la identificación molecular del color de fruto se contrastó con la caracterización morfológica del color de los frutos efectuada entre 7 y 9 meses después, toda vez que las plantas tanto en los parentales como en la progenie F1 y F2 correspondientes emitieron frutos (Figuras 2b y 3b). De manera similar 7 y 9 meses después de tomarse las muestras para el análisis molecular de los QTLs asociados a forma de fruto, se caracterizó la variabilidad en el largo, diámetro, peso y forma de los frutos correspondientes y se presentan en la Figura 4 y en el Cuadro 1.
Conclusiones
El marcador CPM1815YC2 logró discriminar plantas femeninas de las hermafroditas en 42 genotipos F1 y 164 genotipos F2 proveniente de la cruza L7xM22 a nivel molecular antes del trasplante. Dicha clasificación coincidió al 100% con la efectuada en forma visual en las mismas plantas 3 meses después. Lo que comprueba que este marcador es altamente confiable y tiene gran potencial para su uso en la selección asistida por marcadores (SAM) en Carica papaya L. La identificación morfológica del tipo sexual en las plantas de Carica papaya L. provenientes de la cruza L7xM22 resultó entre un 67% a 72% de plantas hermafroditas y entre un 28.4% a 35.8% de plantas femeninas en progenie F1 y progenie F2; es decir, se obtuvo una relación aproximada de 2:1. Esto corresponde a la segregación del carácter del sexo cuando la polinización artificial es entre dos individuos hermafroditas o autopolinizados como fue el caso de los parentales L7 y M22 y los genotipos H90B, H77B y H70B.
La clasificación como frutos de pulpa naranja-rojiza tanto en parentales como en la progenie F1 y F2 de la cruza L7xM22 a nivel molecular en plantas jóvenes, coincidió al 100% con el análisis morfológico efectuado en los frutos emitidos por las mismas plantas entre 7 y 9 meses después. De manera que el marcador molecular de color de pulpa (CPFC1) presenta un alto potencial para ser usado en programas de mejoramiento genético asistido por marcadores orientado a seleccionar frutos de color rojo. Sin embargo, este marcador no permite distinguir entre frutos de color de pulpa amarillo claro, de aquellos de pulpa color naranja.
Los marcadores QTLs asociados a caracteres de peso, longitud, diámetro y forma del fruto de papaya aunque si distinguieron y mostraron condición alélica en varias de las características contrastantes de los progenitores, no fueron útiles para la selección asistida por marcadores moleculares (SAM) de individuos contrastantes en esos caracteres dentro de la progenie F1 y F2 de híbridos provenientes de la cruza intra-específica L7xM22. investigaciones posteriores serán necesarias para desarrollar QTLs (u otros marcadores moleculares) mejor relacionados al tamaño y forma de frutos de papaya de híbridos entre Maradol x material criollo. Sin embargo, no se descarta que los QTLs aquí probados puedan distinguir forma de fruto en otros materiales de Carica papaya L.











 texto en
texto en 


