Introducción
Los geminivirus son un grupo diversificado y ampliamente distribuido de fitopatógenos que producen grandes pérdidas económicas alrededor del mundo. Su genoma está constituido por una o dos moléculas de ADN circular de cadena sencilla con un tamaño de 2.5 a 3 Kb (Lazarowitz, 1992). Son transmitidos por insectos del orden Homóptera (mosquitas blancas y chicharritas) a una gran variedad de plantas mono y dicotiledóneas, produciendo síntomas como mosaicos, moteados, rayados, deformaciones foliares, enanismo, amarillamientos y clorosis constituyéndose como uno de los grupos de fitopatógenos más importantes desde el punto de vista económico debido a su alta incidencia, distribución y severidad de síntomas.
Actualmente el ICTV (International Comittee on Taxonomy of Viruses) reconoce cuatro géneros (Mastrevirus, Topocuvirus, Curtovirus y Begomovirus) pertenecientes a la familia Geminiviridae (Fauquet et al, 2008) siendo los begomovirus el grupo más diversificado y de más amplia distribución a nivel global, son transmitidos por la mosquita blanca (Bemisia tabaci Genn.), su genoma puede ser mono o bipartita e infectan plantas dicotiledóneas. Los componentes A y B de los begomovirus bipartitas comparten una región común cuya longitud va de 160 a 230 pares de bases (pb) que contiene los elementos en Cis necesarios para la replicación viral (Arguello et al., 1994). El componenteAproduce las proteínas involucradas en la replicación y encapsidación del genoma viral (Gutiérrez et al., 2004) mientras que el componente B codifica dos proteínas encargadas del movimiento del virus a través de la planta (Gafni y Epel, 2002).
A inicio de los años 70 se publicó el primer reporte sobre la presencia de una nueva enfermedad que producía enchinamientos en las plantas de jitomate cultivadas en el estado de Sinaloa, México (González y Cervantes, 1973). La enfermedad se asoció con la presencia de elevadas poblaciones de mosquita blanca y fue hasta 1984 que se logró aislar y caracterizar al virus del chino del tomate (Brown y Hine, 1984), un begomovirus considerado como agente causal de dicha enfermedad.Actualmente se cree que el CdTV"está restringido a los miembros de la familia Solanaceae entre los que destacan los cultivos de jitomate y chile presentes en los estados de Chiapas, Morelos, Sinaloa y Tamaulipas mostrando una tendencia a dispersarse debido a su amplio rango de hospederos (Brown y Nelson, 1988; Torres-Pacheco et al., 1996).
En el presente escrito se reporta una nueva cepa de CdTV aislada en plantas de soya (Glycine max L.) en el estado de Sinaloa, México y cuyas diferencias a nivel de genoma con respecto a las demás cepas de CdTVreportadas en solanáceas podría favorecer su presencia en cultivos de soya.
Recolecta de muestras. Se recolectaron 12 muestras de plantas de soya en parcelas comerciales ubicadas en el municipio de Guasave, Sinaloa, México que mostraban síntomas probablemente causados por virus (enanismo, hojas enrolladas y amarillas).
Extracción de ADN. Se adoptó un protocolo de extracción de ADN genómico para todas las muestras de tejido vegetal analizadas. Este se basó en una modificación del método de Della Porta (1983) que utiliza nitrógeno líquido y el buffer de extracción "A" compuesto por Tris 100 mM - pH 8, NaCl 50 mM - pH 8, EDTA 50 mM - pH 8 y agua destilada.
Amplificación de ADN begomoviral presente en los extractos de soya. En algunas ocasiones la carga viral contenida en los extractos de ADN procedentes de plantas con posibles síntomas de virosis es reducida por lo que se implementó el uso del kit Templi phi (GE Healthcare) para incrementar la concentración de ADN viral mediante una reacción isotérmica catalizada por la ADN polimerasa del bacteriófago ¿29 la cual es capaz de producir microgramos de ADN circular de cadena sencilla a partir de picogramos.
Reacción en cadena de la polimerasa (PCR). Las amplificaciones de ADN viral se realizaron utilizando los iniciadores universales para begomovirus: pRep-DGR (Méndez et al, 2006), prCP70-Mot, prRepQGR-rev (De la Torre et al, 2006) y prC889 (Brown et al, 2001). Los productos de PCR comprenden la totalidad del componente A. La reacción de PCR se realizó utilizando como control positivo los genomas A del Pepper huastec yellow vein virus (PHYVV) y del Pepper golden mosaic virus (PepGMV) clonados en el vector pGEM-T easy (Promega Madison, WI).
Posteriormente se confirmó la presencia del componente B de CdTV-soybean utilizando los iniciadores universales: BC1-80rev (AGAYGARTATCARYTDTCNCATGA) y BV1-310for (AGGDACNGTNAAGRTYGARCGTGT) diseñados durante este trabajo y que son complementarios a secuencias del componente B de begomovirus tanto del nuevo como del viejo mundo. Para todos los juegos de oligonucleótidos, la composición de la mezcla de reacción para PCR (50 (L volumen total) fue la misma y consistió en lo siguiente: Buffer Taq DNA polimerasa 1X, MgCl2 1.5 mM, 0.2 mM de cada dNTPs, oligonucleótidos 1 μM, Taq DNA polimerasa 2.5 U, DNA 1(ig. Las condiciones para la amplificación del ADN viral fueron: desnaturalización inicial a 94 °C/2min, y 35 ciclos conformados por 94 °C/1min, 55 °C/1min, 72 °C/1min, con una extensión final de 72 °C/5 min. Los productos amplificados fueron analizados por movilidad electroforética en geles de agarosa al 1%.
Caracterización de los fragmentos amplificados y clonas obtenidas por patrón de restricción (RFLP). Se seleccionaron clonas positivas para cada combinación de oligonucleótidos utilizada y fueron analizadas mediante la técnica de RFLP con el objetivo de identificar bandas polimórficas que indicarían si se trata de una infección sencilla o mixta. Se utilizaron las enzimas de restricción MspI y Hhal en combinación con EcoRI (New England BioLabsTM). Los productos digeridos se analizaron por movilidad electroforética en geles de agarosa al 2%.
Clonación y secuenciación
Los productos de PCR obtenidos de las muestras de campo se clonaron directamente en el plásmido pGEM-T Easy (Promega, Madison, WI), según las indicaciones del proveedor. La transformación de célula calcio competente de Escherichia coli Top-10 se realizó por el método de choque térmico. Las clonas virales obtenidas fueron secuenciadas en el Laboratorio Nacional de BiotecnologíaAgrícola, Médica y Ambiental (LANBAMA) en San Luis Potosí, México. El análisis in silico de las secuencias nucleotídicas obtenidas fue realizado mediante comparaciones con las secuencias disponibles en la base de datos del NCBI, utilizando el programa BLASTN y el análisis de filogenia fue realizado mediante el método Clustal V de MegAlign (DNASTAR software, Lasergene, London).
Identificación de begomovirus por PCR
Los extractos de ADN total obtenidos a partir de plantas de soya analizados por PCR utilizando los iniciadores universales para begomovirus, prRepDGR, prCP70, prRepDGR-rev y prC889 permitieron amplificar a partir de cinco de los doce extractos de soya dos fragmentos de ADN viral traslapados de 1.8 y 1.4 Kb que representan la totalidad del componente Ade los begomovirus como se ha reportado con anterioridad (Méndez et al., 2006).
Amplificación por PCR del componente B de begomovirus
La confirmación por PCR del componente B correspondiente a cada uno de los componentes A identificados, se obtuvo de una forma eficiente empleando la combinación de iniciadores universales BC1-80rev and BV1-310for. Ésta combinación de oligonucleótidos universales son complementarios a secuencias conservadas localizadas en los componentes B de begomovirus presentes en el viejo y nuevo mundo. La longitud de los fragmentos deADN B viral amplificados fue de 1 300pb.
Análisis por RFLP
Las clonas obtenidas pertenecientes al componente A y B de begomovirus fueron digeridas con la enzima de restricción EcoRI para confirmar que contenían los fragmentos de ADN viral. Para el componente A el primer grupo de clonas positivas liberaron un fragmento de 1.8 Kb amplificado con la combinación de iniciadores prCP70 y prRepDGRrev mientras que el segundo grupo de clonas libero un fragmento de 1.4 Kb correspondiente al ADN viral que incluye la región intergénica de los begomovirus el cual fue amplificado con la combinación de iniciadores universales prRepDGR y prC889, mientras que para las clonas obtenidas a partir de los oligonucleótidos BC1-80rev y BV1-310for se confirmó las presencia de un fragmento de 1 300 pb.
Se seleccionaron ocho clonas positivas para cada combinación de oligonucleótidos utilizada y fueron analizadas mediante la técnica de RFLP. La presencia de dos patrones de restricción diferentes en las clonas obtenidas a partir de los componentes A y B sometidas al análisis por RFLP indicaron que se trataba de una infección mixta en la que participaban dos begomovirus diferentes (Figura 1). El resultado obtenido de la selección de clonas concuerda con el aumento en el reporte de infecciones mixtas causadas por begomovirus en México, cuyo ejemplo más claro es el de las infecciones mixtas causadas por PHYVV y PepGMV, los cuales producen serios daños a los cultivos de chile cuando se encuentran infectando juntos una misma planta (Rentería et al, 2011).
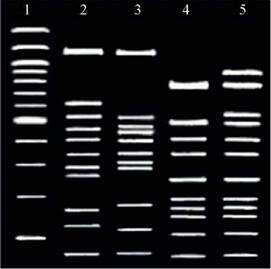
Figura 1 Patrones de restricción obtenidos por RFLP a partir de dos clonas distintas cuyos insertos tienen una longitud de 1.4 Kb del componente A de los dos begomovirus obtenidos a partir de extractos de plantas de soya utilizando la combinación de olionucleótidos prRepDGR / prC889. Los plásmidos recombinantes fueron digeridos con la enzima de restricción EcoRI en combinación con las enzimas HinfI (líneas 2 y 3) o MspI (líneas 4 y 5). Los productos de la digestión se sometieron a electroforesis en gel de agarosa al 2%. Línea 1 muestra marcador de peso molecular (100 pb DNA ladder). El plásmido que contiene el amplicón del CdTV-soybean corresponde a las líneas 2 and 4 y el correspondiente al RhGMV está representado en las líneas 3 y 5.
La alta incidencia de infecciones mixtas causadas por begomovirus en malezas y cultivos de interés económico puede influir en la aparición de nuevas cepas y variantes con cambios a nivel de nucleótidos en genes clave que pudieran producir la aparición de nuevos síntomas, cada vez más severos e inclusive ampliar su rango de huéspedes naturales (Brown et al., 2000). La importancia del análisis de secuencias mediante la técnica de RFLP radica en que nos informa sobre la presencia de dos o más genomas begomovirales infectando la misma planta con lo cual se observa que las infecciones mixtas son muy comunes en la naturaleza (Bañuelos et al., 2012) y además nos permite identificar pequeñas diferencias a nivel de nucleótidos aun entre virus pertenecientes a la misma especie mediante la presencia de una o más bandas polimórficas siempre y cuando las digestiones se realicen con enzimas que nos permitan liberar el fragmento de ADN clonado en combinación con otra enzima de restricción que reconozca sitios de corte constituidos por un máximo de cinco nucleótidos y así favorecer la aparición de más polimorfismos.
Análisis de secuencia y organización genómica
La secuencias obtenidas fueron editadas mediante el programa edit seq (DNAStar MegAlign software) y se armó el genoma completo de un primer begomovirus cuyo componente A (número de acceso NC010294) tiene una longitud de 2 604 pb mientras que la longitud de su componente B (número de acceso NC010293) es de 2 551 pb. El componenteAdel primer aislado de plantas de soya fue comparado con las secuencias presentes en la base de datos del NCBI y las que mostraron una mayor similitud a nivel de nucleótidos se analizaron con el algoritmo Clustal V de MegAlign y se obtuvo una alta identidad a nivel de secuencia nucleotídica (99%) con el componente A de Rhynchosia golden mosaic virus, su pariente más cercano. Para el segundo begomovirus se logró obtener un componente A (número de acceso DQ347945) completo cuya secuencia de 2 609 pb resultó ser 92.3% similar al componente A del CdTV-[8] y CdTV[RK], sus parientes más cercanos (Cuadro 1). Además, se logró la caracterización parcial (1 300 pb) del componente B (número de acceso EU339940) del segundo begomovirus aislado durante este trabajo.
Cuadro 1 Comparación global (%) entre los componentes A completos de los begomovirus pertenecientes a la especie CdTV y su similitud a nivel de secuencia con el componente A de CdTV-soybean.
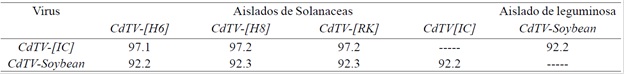
Cuadro 1. Se observa que la similitud a nivel de secuencia entre CdTV-[IC] y las variantes de CdTV aisladas de jitomate es menos 97% de similitud a nivel de secuencia nucleotídica; mientras que CdTV-soybean además de encontrarse en soya tiene una similitud de 92.3% con sus parientes más cercanos. CdTV-[H6] (número de acceso AF226665), es una variante que no es mencionada en éste reporte.
Los criterios taxonómicos establecidos por el ICTV señalan que si al comparar el componente A completo de un begomovirus con el respectivo componente A de su pariente más cercano se observa una diferencia entre 89 y 93%, el aislado analizado se considera una nueva cepa viral separada (Fauquet et al., 2008) por lo que, con base en los resultados obtenidos en el presente trabajo se reporta el aislamiento una nueva cepa correspondiente a la especie CdTV aislada de plantas de soya y cuyo nombre aceptado por el ICTV es Chino del tomato virus- Soybean (Mexico: Sinaloa: 2005), Fauquet et al., 2008.
Los dos virus aislados durante este estudio pertenecen a especies diferentes y como tales presentan iterones muy diferentes entre sí (Arguello et al., 1994) de tal forma que no se esperaría una interacción entre ellos; sin embargo, se ha observado la formación de pseudorecombinantes entre begomovirus que no comparten las mismas secuencias de iterones generando infecciones muy agresivas (Chakraborty et al., 2008) aunado a lo anterior siempre existe la posibilidad de que virus diferentes produzcan un sinergismo que potencie las afectaciones a los cultivos. Lo anterior establece la importancia de analizar más a fondo las posibles implicaciones fitosanitarias sobre la presencia de RhGMVy CdTV en cultivos de soya así como los riesgos de que estos se propaguen en nuevos hospederos.
Hasta la fecha CdTV había sido identificado previamente infectando únicamente plantas de la familia Solanaceae en forma natural. En el presente trabajo se reporta el primer aislamiento de CdTV-Soybean a partir de una leguminosa por lo que se enfatiza la necesidad de monitorear la incidencia de CdTV en cultivos de soya que permitan evitar una epidemia causada por este begomovirus.
Conclusiones
Se caracterizaron a nivel de secuencia dos begomovirus presentes en plantas de soya colectadas en el estado de Sinaloa, México. El primer aislado resultó ser una variante genéticamente muy cercana a RhGMV, reportado con anterioridad en la misma zona geográfica. Sin embargo, un segundo begomovirus aislado resultó ser una nueva cepa de la especie CdTV aislada por primera vez de una leguminosa ya que hasta la fecha su presencia estaba restringida a Solanaceas. Esto abre nuevas perspectivas con respecto a estos begomovirus las cuales nos permitirán conocer sobre su capacidad para infectar plantas de la familia Solanaceae.











 texto em
texto em 


