Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ciencias agrícolas
versión impresa ISSN 2007-0934
Rev. Mex. Cienc. Agríc vol.4 no.7 Texcoco sep./nov. 2013
Artículos
Diversidad genética de aislados de Rhizoctonia solani (Kuhn) de chile en México*
Genetic diversity of Rhizoctonia solani isolates (Kuhn) from pepper in Mexico
Víctor Montero-Tavera1, Brenda Zulema Guerrero-Aguilar1, José Luis Anaya-López1, Talina Olivia Martínez-Martínez1, Guevara-Olvera Lorenzo2 y Mario Martín González-Chavira1§
1 Instituto Nacional de Investigaciones Forestales Agrícolas y Pecuarias (INIFAP). Carretera Celaya-San Miguel de Allende, km 6.5 Celaya, Guanajuato, México. C. P. 38110. Tel: (461) 6115323 Ext. 104. §Autor de correspondencia: gonzalez.mario@inifap.gob.mx.
2 Departamento de Ingeniería Bioquímica. Instituto Tecnológico de Celaya. Ave. Tecnológico y A. García-Cubas, S/N, Col. FOVISSSTE. Celaya, Guanajuato, México.
* Recibido: noviembre de 2012
Aceptado: julio de 2013
Resumen
Una de las principales limitantes para la producción de chile son los hongos patógenos causantes de la enfermedad conocida como "marchitez del chile" o "secadera". Esta enfermedad puede ser devastadora cuando las condiciones climáticas son favorables para el patógeno. A pesar de que se han intentado diferentes medios de control (químicos y culturales) ninguno ha tenido éxito. Una alternativa para su control es producir germoplasma resistente, sin embargo para poder establecer un programa de mejoramiento efectivo es necesario conocer la distribución y diversidad genética de los patógenos involucrados, particularmente de Rhizoctonia solani, que por su ubicuidad representa un peligro potencial en todas las zonas productoras. Por ello el objetivo fue caracterizar a R. solani en las zona Centro Norte de México y determinar su diversidad genética. Para cumplir con este objetivo se consideraron los estados de Chihuahua, Durango, Zacatecas, San Luis Potosí, Colima, Querétaro y Guanajuato donde en 2009 se colectaron plantas adultas de Chile con síntomas de marchitez, se aisló al hongo y se encontró una incidencia del 33%, encontrándose tanto en tallo como en raíz. Las células miceliales fueron multinucleadas, características de las cepas patogénicas. Las pruebas de anastomosis demostaron la presencia en México de los grupos GA4, GA-2.1, GA-IIB, GA-2IV, GA7, GA11, GA12 y GA13. La diversidad genética de este hongo fue muy alta, de tal manera que las relaciones demostradas por la construcción de dendrogramas no muestran tendencias homogéneas pues los principales grupos formados contienen elementos de todos los estados.
Palabras clave: Rhizoctonia solani, AFLP, variabilidad genética, grupos de anastomosis.
Abstract
One of the major constraints for the production of pepper are pathogenic fungi causing diseases known as "pepper blight" or "damping off". This disease can be devastating when weather conditions are favorable for the pathogen. Although different means of control (chemical and cultural) have benn used but none has been successful. An alternative to control is to produce resistant germplasm, however in order to establish an effective breeding program is necessary to know the distribution and genetic diversity of the pathogens involved, particularly Rhizoctonia solani, which by its ubiquity represents a potential danger in all producing areas. Thus, the objective was to characterize R. solani in North Central area from Mexico and determine its genetic diversity. To achieve with this goal are considered the states of Chihuahua, Durango, Zacatecas, San Luis Potosi, Colima, Queretaro and Guanajuato where in 2009 were collected adult plants of pepper with pepper blight symptoms, the fungus was isolated and found an incidence of 33%, finding it in both stem and root. Mycelial cells were multinucleated, a characteristic from pathogenic strains. The anastomosis testing showed that in Mexico are present the groups GA4, GA-2. 1, GA-IIB, GA-2IV, GA7, GA11, GA12 and GA13. The genetic diversity of this fungus was very high, so that the relationships demonstrated by the construction of dendrogram show no homogeneous trends so as the main groups formed contain elements of all states.
Keywords: Rhizoctonia solani, AFLP, genetic variability, anastomosis groups.
Introducción
El chile es uno de los cultivos de mayor importancia económica en México, en el año 2012 su producción generó más de $13 000 millones de pesos. Los principales estados productores son Zacatecas, Chihuahua, Sinaloa, San Luis Potosí, Durango, Veracruz, Jalisco, Guanajuato, Chiapas y Sonora (SIAP, 2012).
En México, la producción de chile se afecta por diversos factores, entre los que destacan las enfermedades ocasionadas por hongos, bacterias, virus y nemátodos (Guigón-López y González-González, 2001). Sin embargo, las enfermedades fungosas han sido la principal causa de pérdidas económicas (Sneh et al, 1996; Krechel et al, 2002). Los patógenos de mayor incidencia en chile son Phytopthora spp., Fusarium spp., Rhizoctonia solani y Pythium spp. (Silva-Rojas et al, 2009), los cuales están asociados al síndrome de la tristeza o marchitez del chile, enfermedad que puede causar la muerte prematura de las plantas y ocasionar pérdidas en la producción entre 10% y 60%, aunque en el Bajío y Puebla se han reportado pérdidas totales (Pérez-Moreno et al, 2003).
La capacidad de infección de Rhizoctonia solani Kühn (teleomorfo Thanatephorus cucumeris) está determinada por las condiciones de temperatura y humedad (González-Hernández, 2002), y es uno de los hongos fitopatógeno de mayor incidencia en el cultivo del chile (Velásquez y Victoriano, 2007), aunque también puede infectar a un extenso grupo de plantas de diferentes especies de importancia económica (Gour, 2012) en las que produce lesiones oscuras en raíces y semillas, pudrición de tallos y de las partes de la planta que están en contacto con el suelo (González-García, 2008).
La clasificación de R. solani ha sido compleja, por lo que se han propuesto diversos criterios. Algunos de los más utilizados son la determinación de los grupos de anastomosis (GA) al que pertenecen los aislados, y el grado de interacción entre las hifas. En relación a los grupos de anastomosis R. solani se ha clasificado con base en la compatibilidad de la fusión entre hifas. De esta manera, las hifas de aislamientos de GA iguales se fusionan, lo que sugiere la existencia de compatibilidad vegetativa, mientras que las de GA diferentes no presentan interacción (Boidin, 1998; González-García et al., 2006).
De acuerdo a este criterio, R. solani se ha clasificado en 14 GA, del GA1 al GA13, que se anastomosan sólo con individuos del mismo grupo, y el GA-BI, que incluye aislados capaces de fusionarse entre ellos y con otros grupos (González-García et al., 2006). A su vez los grupos GA 1, GA2, GA4, GA6 y GA9 se dividen en subgrupos con base en caracteres morfológicos, patológicos, bioquímicos y moleculares (Liu y Sinclair, 1993; González-García et al., 2006). Otra clasificación, basada en los cambios citológicos en la zona de anastomosis, propone las categorías adicionales C0, C1, C2 y C3 para describir el grado de interacción entre las hifas en las reacciones de anastomosis (MacNish et al., 1993; Cubeta y Vilgalys, 1997). Algunos otros criterios para una clasificación más precisa de R. solani se basan en las secuencias de ADN.
La mayoría de las técnicas moleculares disponibles para establecer la diversidad genética se basan en la detección y tipificación de polimorfismo genómico en varios niveles. La diversidad genética, y la clasificación taxónomica de R. solani se ha establecido por múltiples técnicas moleculares como microsatélites amplificados al azar (RAMS-fingerprinting), polimorfismo en la longitud de fragmentos de restricción (RFLP) de la región del espaciador transcrito interno (ITS), secuencias de ADN ribosómico nuclear (rDNA) y beta-tubulina (Ceresini et al., 2007), región del espaciador transcrito interno 5.8S (ITS-5.8S rDNA) (Çebi and Ózkoc, 2013), repetidos de intersecuencias simples (ISSR) (Mirmajlessi et al., 2012) y polimorfismo en la longitud de fragmentos amplificados (AFLP), basado en un sistema de marcadores dominantes que permiten el análisis simultáneo de un gran número de marcadores en el genoma, es altamente reproducible, tiene un costo moderado y se ha usado ampliamente para analizar diversidad y huellas genéticas en varias especies de hongos (Bensch and Akesson, 2005). Los AFLP se han usado para identificar diversidad genética entre poblaciones de R. solani asociadas a papa (Fiers et al., 2011), Vitis vinifera (Meza-Moller et al., 2011); frijol (López-Olmos et al., 2005); y tomate (Taheri, 2011). Por ello el objetivo de este estudio fue identificar los grupos de anastomosis de R solani involucrados en la marchitez del chile en las principales regiones productoras de México y establecer la diversidad y relaciones genéticas entre aislados genéticamente puros de Capsicum annuum L.
Materiales y métodos
Aislamiento de Rhizoctonia solani
En 2008 se realizó una colecta de plantas de chile (Capsicum annuum L.) en 40 municipios de los estados de Guanajuato, Querétaro, San Luis Potosí, Zacatecas, Durango, Chihuahua y Colima. Se seleccionaron parcelas que estuvieran separadas al menos 5 km entre sí, y se colectaron al azar de 3 a 5 plantas completas con síntomas característicos de marchitez.
Las raíces y tallos de las plantas con síntomas de marchitez se lavaron con agua corriente y se cortaron en fragmentos de 1 cm2. El tejido se desinfectó durante 1 min en hipoclorito de sodio al 3% y se sembró en medio agar papa dextrosa (PDA) acidificado (1 300 μM de ácido láctico por litro de medio). La incubación se realizó entre 19 y 25 °C durante 2 días. Los aislamientos se identificaron de acuerdo a sus características morfológicas (Sneh et al, 1991), y se transfirieron nuevamente en PDA acidificado con la finalidad de obtener cultivos puros para la extracción de puntas de hifa. Los aislados se identificaron por parcela de colecta y por la estructura de la planta donde fue aislado (tallo y raíz).
Cultivos puros
Los cultivos puros de R. solani se obtuvieron de la siembra de puntas de hifa. En cajas Petri esterilizadas se colocó un portaobjetos sobre el que se agregaron 6.5 ml de medio agar-agua al 2%. Una vez que solidificó el medio, se colocaron fragmentos de 5 mm de micelio del aislado de R. solani creciendo sobre PDA, y se incubó a temperatura ambiente (19-25 °C) durante 24 h. El nuevo micelio se separó de la caja y bajo el microscopio se seleccionó una hifa aislada, a la cual se le cortó la punta con un alfiler entomológico. Las puntas seleccionadas se transfirieron a placas con PDA acidificado y se incubaron a 25°C durante 24 h. De la misma forma, se obtuvieron puntas de hifa jóvenes y se cuantificaron los núcleos de las células vegetativas hifales a través del método de tinción con safranina O (Bandoni, 1979).
Reacciones de compatibilidad
Los grupos de anastomosis de los aislados de R. solani se determinaron mediante confrontaciones con cepas representativas de grupos de anastomosis internacionales (AGsI). En cajas Petri se colocaron portaobjetos y se adicionaron 6.5 ml de agar agua 2%. Sobre este medio se colocaron fragmentos de 5 mm del micelio de los aislados obtenidos en campo y de las cepas representativas de grupos de anastomosis a una distancia de 2 cm, también se hicieron confrontaciones entre las hifas de aislados obtenidos en campo. Cada prueba se hizo por duplicado y se incubaron a temperatura ambiente (19-25 °C). Para observar las reacciones de compatibilidad, se sacaron los portaobjetos de las placas y se hicieron tinciones con Azul de Tripano con Lactofenol al 0.1% y se examinaron bajo microscopio las reacciones de compatibilidad. Para la clasificación de la reacción de anastomosis se usaron las categorías propuestas por MacNish et al. (1993).
Extracción de ADN
El ADN genómico total fue extraído de los cultivos genéticamente puros siguiendo la metodología para hongos filamentosos propuesta por Raeder y Broda (1985) con modificaciones menores. Se colectó y liofilizó el micelio de R. solani obtenido de la inoculación de 50 mL de caldo papa dextrosa (PDB, Difco) con cinco o seis cubos de 0.25 cm2 de micelio creciendo sobre medio Agar Papa Dextrosa (PDA, Difco) durante 48-72 hrs a 28-25 °C. El micelio se molió y maceró con buffer de extracción (200 mM tris/HCl pH 8.5, 250 mM NaCl, 25 mM EDTA, 0.5% SDS).
Análisis AFLP
Se llevaron a cabo análisis de Polimorfismo en la Longitud de Fragmentos de Restricción siguiendo el protocolo de Vos et al. (1995) con ligeras modificaciones. ElADN genómico se digirió con 5U de EcoRI y MseI (Roche®) a 37°C durante 3h. Las muestras digeridas se incubaron a 70 °C durante 15 min para desactivar las enzimas de restricción. Los adaptadores EcoRI (5 pmol) y MseI (50 pmol) se ligaron a los fragmentos de ADN cortados en amortiguador de ligación (1X T4ADN ligasa) y 1U de T4 ADN ligasa y se incubaron a 37 °C toda la noche. La amplificación selectiva se hizo usando seis combinaciones de iniciadores (E-ACA + M-GTA; E-ACG + M-GTA; E-ACA + M-GGT; E-AGA + M-GGT; E-AAC + M-CAG; EACG + M-CAG). La preamplificación del ADN ligado y diluido 10-1 se llevó a cabo con iniciadores complementarios a los adaptadore s EcoRI y MseI usando un nucleótido selectivo de adenina y citosina, respectivamente, en un termociclador (PX2 Thermo Electron Corporation, Milford, MA, USA.) usando los siguientes parámetros: 20 ciclos a 94 °C durante 30 s, 56 °C durante 30 s and 72 °C durante 60 s.
La segunda amplificación se llevó a cabo con seis combinaciones de iniciadores de EcoRI (700 y 800 nm) y MseI usando tres nucleótidos selectivos. La electroforesis se llevó a cabo en un secuenciador LiCor IR2 equipado con dos lasers infrarrojos con capacidad de detección en dos longitudes de onda: 700 y 800 nm. Para el análisis se registraron solamente bandas brillantes, claramente distinguibles entre 50 y 700 pares de bases.
Relaciones genéticas
A cada fragmento AFLP se le asignó una valor de 1 para presencia y 0 para su ausencia en cada individuo. De ésta manera se generó una matriz binaria, la cual sirvió como base para construir dendrogramas usando el coeficiente de Dice (Nei and Li, 1979), UPGMA (Unweighted Pair Group Method withArithmetic Mean, Sokal and Sneath, (1963), y el Método de Jackknifing para comprobación. El método de remuestreo bootstrap se ejecutó para determinar la robustez del dendrograma considerando 1000 iteraciones a partir de los datos originales, el límite de confianza para cada par se determinó con el algoritmo de Felsenstein et al. (1985). Los dendrogramas se graficaron con el programa TreeView 1.6.6 (Page, 2001).
Resultados y discusión
Colectas
Se identificaron y colectaron plantas de diferentes variedades de chile con síntomas de marchitamiento y amarillamiento; de 304 plantas colectadas en 128 se encontró Rhizoctonia solani, lo cual representa 42% de incidencia. A partir de ellas se obtuvieron 73 aislados de raíz y 115 de tallo. Los tipos de chile colectados fueron mirasol, serrano, güero, de árbol, puya, ancho, mirasol de tres venas, caribe, guajón, tornachile, pasilla, cristalino, páprika bola, cascabel, guajillo, piquín, jalapeño, anaheim y cayene; estos nombres provienen del conocimiento del agricultor del predio de donde proviene la colecta y de acuerdo a la apreciación de los autores. Los estados donde se obtuvieron aislados fueron Zacatecas, San Luis Potosí, Durango, Guanajuato, Chihuahua, Colima y Querétaro. Con base en los criterios de colecta establecidos se estimó que se tiene una muestra representativa de la diversidad genética existente en la región de muestreo, ya que se trató de una selección completamente al azar, por tanto es alta la probabilidad de tener los aislados más comunes presentes en los campos de colecta. Después de aplicar los criterios de selección para la obtención de aislados genéticamente puros se obtuvieron 60 aislados de tallo y 51 de raíz. El Cuadro 1 señala, además de los estados donde se colectó, el número de municipios considerados, el número de lotes y el número de aislados por municipio.

Las muestras procesadas originaron colonias fúngicas, no esporulantes, incoloras en etapa juvenil y café cuando maduran, constan de largas ramificaciones que crecen en ángulos rectos respecto a la hifa principal; presentan la formación de un septo de la ramificación cerca del punto de origen (Figura 1), lo cual concuerda con las características morfológicas de R. solani descritas por Parmeter y Whitney (1970). Por medio de tinción de núcleos con safranina se determinó que los aislados de Rhizoctonia solani de los diferentes estados donde se realizaron las colectas son polinucleados, con un promedio de 8 núcleos por célula llegando a tener hasta 23 (Figura 1).

Grupos de anastomosis
Con los resultados obtenidos mediante el uso de las cepas probadoras se construyó el Cuadro 2, donde se observa que el grupo GA4 fue el más común pues aislados de todos los estados bajo estudio pertenecen a él. Los grupos relativamente raros fueron GA2-1, GA2-2IIIB y GA11, presentes solamente en Guanajuato; el grupo GA2-2IV presente solamente en Zacatecas y el grupo GA12 presente solamente en Durango. El estado que tuvo mayor diversidad en cuanto a grupos de anastomosis fue Guanajuato con cuatro, seguido de Durango y Zacatecas con tres; en San Luis Potosí y Querétaro solamente se encontró un grupo.

Esta clasificación representa un avance importante en el conocimiento de las características de los aislados, ya que permitiran establecer criterios en relación al grado de diversidad genética entre ellos, lo cual además tendrá relevancia en el establecimiento de estrategias para su control. Otros grupos se han encontrado en México, aunque ninguno de ellos en chile donde la identificación de R. solani como integrante del complejo que ocasiona la marchitez es relativamente reciente; así Meza-Moller et al., (2007) documentaron la presencia del grupo GA 1 y GA2 en plantas de chile y uva, en Sayula, Jalisco y Horcasitas, Sonora, respectivamente. El grupo GA2-2IIIB se observó en frijol en el estado de Veracruz, México (López-Olmos et al, 2005). Mientras que el grupo GA3 se determinó en papa en Navidad, Nuevo León; Arteaga, Coahuila; Ayahualulco, Veracruz; Tapalpa, Jalisco, Huatabampo, Sonora y León Guanajuato (Virgen-Calleros et al, 2000). El grupo GA5, se encontró en el estado de Veracruz en cultivo de frijol (López-Olmos et al, 2005).
Los grupos GA6 y GA7, distribuidos en plantas de chile y papa en Sayula, Jalisco yToluca, Estado de México, respectivamente (Virgen-Calleros et al, 2000; López-Olmos et al, 2005). El grupo GA4, que tuvo la mayor frecuencia de aparición, es uno de los más reconocidos a nivel mundial y no ha sido asociado con un hospedero especifico, ya que se ha encontrado en varios cultivos como soya, remolacha, calabazas, cacahuate, entre otros; se considera como causante de la pudrición de la semilla y de la raíz en diferentes cultivos; además existen reportes de que este grupo junto con los grupos GA2. 1, GA2-2IV, también presentes en México según los resultados de este trabajo, ocasionan el "damping off" (ahogamiento) de la planta.
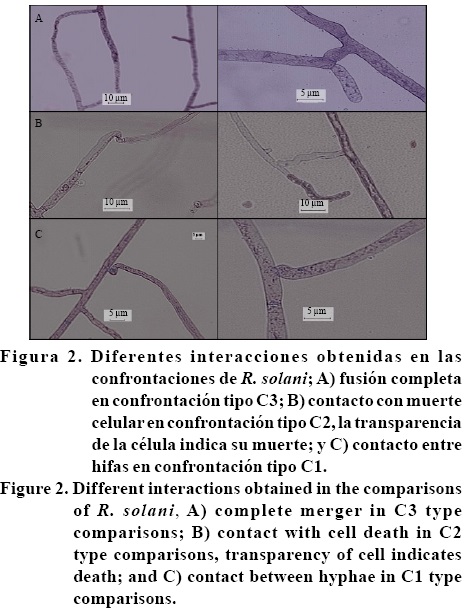
Además de las cepas probadoras también se establecieron confrontaciones pareadas entre aislados de los diversos estados, los cuales representan todos los grupos de anastomosis encontrados. Las confrontaciones probaron todas las combinaciones posibles; la gran mayoría de confrontaciones resultaron ser del tipo C1 (83.5%), las reacciones de compatibilidad fueron menores con sólo un 9.5% de confrontaciones, mientras que las confrontaciones de tipo C2 fueron únicamente 7% (Cuadro 3). Una vez más los resultados muestran que los aislados mexicanos son poco compatibles entre sí, lo cual sugiere amplia diversidad genética, lo cual puede explicarse por la gran diversidad de tipos de chiles que se siembran en la región estudiada, lo que probablemente indujo un proceso de coevolución ampliamente documentado en otros patosistemas. En este sentido se considera que el estado donde se presenta mayor variabilidad genética es Colima con 92% de interacciones C1 y el de menor variabilidad es Querétaro con 100% de interacciones C3. En general se puede apreciar en el resto de los estados un equilibrio entre las interacciones C1 y C3. La Figura 2 muestra los tipos de confrontaciones analizadas.
Análisis de diversidad genética
El análisis AFLP de R. solani dio como resultado la amplificación de 255 bandas de las cuales 232 fueron polimórficas (91%). Un ejemplo de los resultados obtenidos con AFLP se muestra en la Figura 3. En ella se observa que la variación observada a nivel genotípico es muy alta ya que todos los aislados fueron haplotipos AFLP únicos.

Con la matriz binaria de presencia/ausencia de bandas se calcularon las distancias genéticas entre aislados usando el algoritmo de Dice, con las cuales se construyó el dendrograma UPGMA con bootstrap de 1 000 iteraciones que se presenta en la Figura 4. La alta diversidad intrapoblacional fue tan alta como la diversidad interpoblacional lo cual determinó la construcción de un dendrograma con poca e structura ya que se observan dos grupo bien definidos, pero que contienen asilados de todos los estados; lo mismo se observa en la formación de los cuatro subgrupos: no existen subgrupos que contengan aislados preferentemente de alguna región.

Los reportes señalan a Phytophthora capsici como el causante de la marchitez del chile (Redondo, 1989; Mora, 1988), pero ya que se encuentra recurrentemente asociado a dos hongos reportados también como patógenos de plantas (Mora, 1988), es necesario identificar al agente causante de esta enfermedad y considerar la posibilidad de que sea un complejo fúngico el que se conjuga para causar el síndrome. Además la alta diversidad genética de estos patógenos, particularmente R. solani, dificulta su control debido a su gran capacidad adaptativa, lo cual se comprueba por la multitud de ambientes y hospederos donde puede prosperar. Esta puede ser la causa por la que no se han podido obtener variedades de chile resistentes a la marchitez.
El uso de los AFLP ha permitido aclarar las relaciones filogenéticas, de patogenicidad y la diversidad genética de aislados de éste patógeno provenientes de diversas especies. Así, Ceresini et al. (2002) encontraron que 32 aislados estudiados del grupo GA3 obtenidos de papa tenían distinto fenotipo AFLP, mientras que 28 fenotipos AFLP se determinaron en 36 aislados del grupo GA3 obtenidos de tabaco. Esta alta diversidad genética concuerda con los resultados obtenidos en el presente estudio. Por otra parte, mediante el uso de AFLP (López-Olmos et al., 2005) encontraron relación entre el genotipo y el grupo de anastomosis en aislados de R. solani obtenidos de plantas de frijol, aunque no se encontró asociación con la patogenicidad.
Taheri (2011) demostró por medio de análisis de varianza molecular (AMOVA) que la región geográfica fue el factor determinante de la estructura genética de las poblaciones del grupo GA4 HG-1; sin embargo en los resultados aquí presentados la región geográfica de origen no tuvo importancia, en el dendrograma los agrupamientos se dan independientemente de ésta, lo cual puede deberse a que en éste trabajo se analizaron simultáneamente aislados de diversos grupos de anastomosis.
Conclusiones
La diversidad genética de R. solani es alta, lo cual se comprobó por los perfiles AFLP que indican que todos los aislados estudiados pueden considerarse como haplotipos, ya que no se encontraron dos patrones similares. Otro indicativo de diversidad fue la gran mayoría de interacciones de tipo C1, que denotan incompatibilidad, en las confrontaciones entre aislados de los diferentes estados de México estudiados. Esta conclusión tiene implicaciones importantes tanto para el control de R. solani como para el control de la marchitez del chile, ya que una amplia diversidad genética frecuentemente conlleva una amplia capacidad adaptativa, lo que requiere el desarrollo continuo de estrategias integrales para reducir las poblaciones fúngicas.
Literatura citada
Bandoni, R. J. 1979. Safranin O as a rapid nuclear stain for fungi. Mycologia. 11(4):873-874. [ Links ]
Bensch, S. and Akesson, M. 2005. Ten years of AFLP in ecology and evolution: why so few animals? Mol Ecol. 14(10):2899-2914. [ Links ]
Boidin, J. 1998. Taxonomie moleculaire des Aphyllophorales. Mycotaxon. 66:445-491. [ Links ]
Çebi kiliçoglu, M. and Özkof, I. 2013. Phylogenetic analysis of Rhizoctonia solani AG-4 isolates from common beans in Black Sea coastal region, Turkey, based on ITS-5.8S rDNA. Turk. J. Biol. 37:18-24. [ Links ]
Ceresini, P. C.; Shew, H. D.; James, T. Y.; Vilgalys, R. J. and Cubeta, M. A. 2007. Phylogeography of the Solanaceae-infecting Basidiomycota fungus Rhizoctonia solani AG-3 based on sequence analysis of two nuclear DNA loci. BMC Evol. Biol. 7:163. [ Links ]
Cubeta, M. A. and Vilgalys, R. 1997. Population Biology of the Rhizoctonia solani Complex. Phytopathology. 87(4):480-484. [ Links ]
Felsenstein, J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution. 39(4):783-91. [ Links ]
Fiers, M.; Edel-Hermann, V.; Héraud, C.; Gautheron, N.; Chatot, C.; Le Hingrat, Y.; Bouchek-Mechiche, K. and Steinberg C. 2011. Genetic diversity of Rhizoctonia solani associated with potato tubers in France. Mycologia. 103(6):1230-44. [ Links ]
González-García, M. 2008. Aspectos de sistemática y biología del complejo Rhizoctonia. Fitosanidad. 12(3):147-159. [ Links ]
González-García, V.; Portal-Onco, M. A. and Rubio-Susan, V. 2006. Review. Biology and systematics of the form genus Rhizoctonia. Span. J. Agric. Res. 4(1):55-79. [ Links ]
González-Hernández, D. 2002. Estado actual de la taxonomía de Rhizoctonia solani Kühn. Rev. Mex. Fitopatol. 20(2):200-205. [ Links ]
Gour, R. 2012. Isolation and characterization of Actinomycetes against Macrophomina phaseolina and Rhizoctonia solani. Adv. J. Pharm. Sci. 1(2):31-30. [ Links ]
Guigón-López, C. y González-González, P. A. 2001. Estudio regional de las enfermedades de chile (Capsicum annum L.) y su comportamiento temporal en el Sur de Chihuahua, México. Rev. Mex. Fitopatol. 19:49-56. [ Links ]
Krechel,A.; Faupe,A.; Hallmann, J.; Ulrich, A. and Berg, G. 2002. Potato-associated bacteria and their antagonistic potential towards plant-pathogenic fungi and the plant-parasitic nematode Meloidogyne incognita (Kofoid and Whithe) Chitwood. Can. J. Microbiol. 48:772-786. [ Links ]
Liu, Z. L. and Sinclair, J. B. 1993. Differentiation of intraspecific groups within anastomosis group I of Rhizoctonia solani using ribosomal DNA internal transcribed spacer and isozyme analyisis. Can. J. Plant Pathol. 12:376-382. [ Links ]
López-Olmos, K.; Hernández-Delgado, S. and Mayek-Pérez, N. 2005. AFLP fingerprinting for identification of anastomosis groups of Rhizoctonia solani Kühn from common bean (Phaseolus vulgaris L.) en México. Rev. Mex. Fitopatol. 23(2): 147-151. [ Links ]
MacNish, G. C.; Carling, D. E. and Brainard, K.A. 1993. Characterization of Rhizoctonia solaniAG-8 from bare patches by pectic isozyme (Zymogram) and anastomosis techniques. Phytopathology. 83:922-927. [ Links ]
Meza-Moller, A.; Esqueda, M., Gardea, A.; Tiznado, M. y Virgen-Callero, G. 2007. Variabilidad morfológica, patogénica y susceptibilidad a fungicidas de Rhizoctonia solani aislado de rizósfera de Vitis vinifera var. perlette seedless. Rev. Mex. Micol. 24:1-7. [ Links ]
Meza-Moller, A.; Esqueda, M.; Sánchez-Teyer, F.; Vargas-Rosales, G.; Gardea, A. A. and Tiznado-Hernández, M. 2011. Genetic variability in Rhizoctonia solani isolated from Vitis vinifera based on Amplified Fragment Length Polymorphism. Am. J. Agr. Biol. Sci. 6(3):317-323. [ Links ]
Mirmajlessi, S. M.; Safaie, N.; Mostafavi, H.A.; Mansouripour, S. M. and Mahmoudy, S. B. 2012. Genetic diversity among crown and root rot isolates of Rhizoctonia solani isolated from cucurbits using PCR based techniques. Afr. J. Agr. Res. 7:583-590. [ Links ]
Mora, L. F, 1988. Guía de producción para chile picante, San José, Costa Rica. Laboratorios Griffith. 22p. [ Links ]
Nei, M. and Li, W. H. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc Natl Acad Sci USA. 76(10):5269-5273. [ Links ]
Page, R. D. M. 2001. TreeView version 1.6.6. TreeView online user's guide. Glasgow: University of Glasgow, http://taxonomy.zoology.gla.ac.uk/rod/rod.htm. [ Links ]
Parmeter,Jr.andWhitney,H.S.1970.Taxonomyandnomencal1ureoftheperfect stage. In Rhizoctonia solani, Biology and pathology. Parmeter, Jr. J. R. (Ed.). University of California Press, California. 7-19 pp. [ Links ]
Pérez-Moreno, L.; Durán-Ortiz, L. J.; Ramírez-Malagón, R.; Sánchez- Pale, J. R. y Olalde-Portugal, V. 2003. Compatibilidad fisiológica y sensibilidad a fungicidas de aislamientos de Phytophthora capsici Leo. Rev. Mex. Fitopatol. 21:19-25. [ Links ]
Raeder, U. and Broda, P. 1985. Rapid preparation of DNA from filamentous fungi. Lett Appl. Microbiol. 1(1):17-20. [ Links ]
Redondo, E.; Rodríguez, R.; Ortega, M.; Larqué, A. y Engleman, E. M. 1989. Mecanismo de infección y patología de las plantas de chile susceptibles y resistentes al hongo Phytophthora capsici Leo. Agrociencia. 77:123-137. [ Links ]
Servicio de Información Agroalimentaria y Pesquera (SIAP). 2012. Cierre de la producción agrícola. Chile verde. http://www.siap.gob.mx. (consultado mayo, 2013). [ Links ]
Silva-Rojas, H. V.; Fernández-Pavia, S. P.; Góngora-Canul, C.; Macías-López, B. C. y Ávila-Quezada, G. D. 2009. Distribución espacio temporal de la marchitez del chile (Capsicum annuum L.) en Chihuahua e identificación del agente causal Phytophthora capsici Leo. Rev. Mex. Fitopatol. 27(2):134-147. [ Links ]
Sneh, B.; Jabaji-Hare, S.; Neate, S. and Dijst, G. 1996. Rhizoctonia species: taxonomy, molecular biology, ecology, pathology and disease control. Kluwer Academic Publisher. 578 p. [ Links ]
Sneh, B.L. and Ogoshi, A. 1991. Identification of Rhizoctonia species. Am Phytopathol Soc Press. St. Paul, Minnesota. 133 pp. [ Links ]
Sokal, R. R. and Sneath, P. H. A. 1963. Principles of numerical taxonomy. San Francisco: H. Freeman & Company. 35 p. [ Links ]
Taheri, P. 2011. Classification and genetic diversity of Rhizoctonia solani populations causing tomato damping-Offin Iran. Proc. IIIrd IS on Tomato Diseases. Acta Hort. 914:131-134. [ Links ]
Velásquez, V R. y Victoriano, L. F. 2007. Presencia de patógenos en almácigos y semilla de chile (Capsicum annuum L.) en Aguascalientes y Zacatecas, México. Rev. Mex. Fitopatol. 25:75-79. [ Links ]
Virgen-Calleros, G.; Olalde-Portugal, V. and Carling, D. E. 2000. Anastomosis groups of Rhizoctonia solani on potato in central Mexico and potential for biological and chemical control.Amer. J. Potato Res. 77:219-224. [ Links ]
Vos, P.; Hogers, R.; Bleeker, M.; Reijans, M.; Vane Lee, T.; Hornes, M.; Frijters,A.; Pot, J.; Paleman, J.; Kuiper, M. and Zabeau, Y. 1995. AFLP:A new technique for DNA firgerprinting. Nucleics Acids Res. 23(21):4407-4414. [ Links ]














