Introducción
México es uno de los principales productores y exportadores de frutillas (o berries) en el mundo. En nuestro país existen más de 26 mil hectáreas dedicadas a la producción de berries, donde destaca la producción de zarazamora cuenta con un 47.79% de dicha superficie, la fresa con el 38.09%, la frambuesa (9.03%) y el arándano (5.09%), las cuales generaron entre 80 y 100 mil empleos durante el año 2014. Actualmente, esta tendencia productiva de frutillas sigue creciendo. Datos del Servicio de Información Agroalimentaria y Pesquera (SIAP) destacan al Estado de Michoacán como el productor más importante, seguido de Baja California, Jalisco y Guanajuato, ingresando al país cerca de 1,000 millones de dólares por exportación de frutillas (González et al., 2019; http://infosiap.siap.gob.mx [accesado 2019]). Lo anterior remarca la importancia económica de esta actividad agrícola en México.
Los hongos fitopatógenos causan graves pérdidas económicas en los cultivos agrícolas, incluyendo, por supuesto, la producción de berries, siendo una práctica común es el uso de fungicidas para controlarlos (Heydari y Pessarakli, 2010). Sin embargo, está ampliamente reportado que dichos compuestos químicos dañan la microbiota benéfica asociada a las plantas, contaminan el ambiente y pueden causar diversas enfermedades en humanos y animales. Además, de limitar la exportación de productos agrícolas debido a las restricciones arancelarias (Adesemoye y Kloepper, 2009). Por lo tanto, se han buscado nuevas estrategias que sean efectivas para controlar dichos patógenos de plantas, siendo el uso de agentes biológicos una de las más empleadas actualmente (Glick, 2014).
Las bacterias promotoras del crecimiento vegetal o PGPR (por sus siglas en inglés, Plant Growth-Promoting Rhizobacteria) son agentes que resaltan por sus diversos mecanismos para antagonizar y controlar el crecimiento de fitopatógenos, disminuyendo los efectos nocivos en plantas. También, se ha reportado que las PGPR pueden promover al mismo tiempo, el crecimiento y la salud de las plantas. Las actividades de promoción del crecimiento vegetal se clasifican en mecanismos directos e indirectos, y son diversos los estudios que apoyan el uso de PGPR como agentes biológicos para biocontrolar enfermedades en cultivos de interés agrícola (Coutinho et al., 2015; Luo et al., 2012; Pandya et al., 2015; Rashid et al., 2012; Saini et al., 2015; Santoyo et al., 2012). Dentro de los mecanismos directos se mencionan aquellos en los que las bacterias influyen directamente en el crecimiento de las plantas, por ejemplo: pueden estimular la adquisición de nutrientes tales como el nitrógeno, hierro o fósforo, sintetizan compuestos que potencian la inmunidad de las plantas (Resistencia Sistémica Inducida) y/o regulan los niveles de auxinas, giberelinas y citocininas (Ahmad et al., 2008; Santoyo et al., 2016). Por otra parte, los mecanismos indirectos incluyen aquellos en los que las bacterias producen compuestos con actividad antibiótica hacia los patógenos de las plantas, tales como lipopéptidos, fenazinas, sideróforos, el compuesto 2,4-diacetilfloroglucinol (DAPG) y volátiles como la dimetilhexadecilamina (DMHDA) y el ácido cianhídrico (HCN) (Hernández-León et al., 2015; Velázquez-Becerra et al., 2013). Estos últimos compuestos (DAPG y HCN) se han reportado en diversas especies de Pseudomonas, incluyendo trabajos con las cepas UM16, UM240, UM256 y UM270, las cuales producen ambos compuestos y mostraron ser agentes de biocontrol efectivos contra patógenos como Botrytis cinerea (Hernández-León et al., 2015). B. cinerea es un patógenos de más de 200 especies vegetales de importancia agrícola y causante de la enfermedad del moho gris, afectando las partes aéreas de la planta y pudrición de vegetales, frutas y flores después de la cosecha, incluyendo frutillas como la zarzamora (Williamson et al., 2007).
Los compuestos HCN y DAPG son ampliamente producidos por cepas PGPR, incluyendo diversas especies de Pseudomonas (Santoyo et al., 2012; Hernández-Salmerón et al., 2016). Para la producción del HCN, el gen hcnC es importante, ya que codifica la subunidad C de la sintasa de ácido cianhídrico (o HCN sintasa), la cual cataliza la reacción para la formación del HCN a partir del aminoácido glicina (Laville et al., 1998). Por otra parte, el gen phlD forma parte del operón phlACBD que participa en la biosíntesis del compuesto DAPG (Yang y Cao, 2012). En particular, el gen phlD codifica para una policétido sintasa, siendo la principal responsable de la producción de monoacetilfloroglucinol (MAPG), el cual es precursor directo de 2,4-DAPG mediante una acetilación llevada a cabo por los productos de los genes phlA, phlB y phlC (Bangera y Thomashow, 1999; Mavrodi et al., 2001). En un trabajo reciente, Hernández-Salmerón et al. (2018), detectaron que el patógeno B. cinerea modula la expresión in vitro de los genes phlD y hcnC de la cepa biofungicida Pseudomonas fluorescens UM270, destacando la comunicación e importancia de estos dos elementos genéticos en la interacción antagonista-fitopatógeno.
La producción de sideróforos (agentes quelantes de hierro) producidos por bacterias PGPR fue uno de los primeros mecanismos indirectos de promoción del crecimiento vegetal en ser descrito (Kloepper et al., 1980). Los autores propusieron que el hierro del suelo es captado por los sideróforos bacterianos, los cuales actúan como sistemas de transporte del elemento hacia las bacterias y lo hacen menos disponible para los patógenos. Diversos trabajos han demostrado que mediante este mecanismo, los sideróforos restringen el crecimiento de patógenos fúngicos que causan enfermedades en plantas (Weller , 2007). Cabe mencionar que la producción de sideróforos no está relacionada con la producción de compuestos como el HCN y DAPG, ya que su síntesis involucra rutas y elementos genéticos diferentes; pero que sin embargo, han sido encontrados en el mismo genoma de varias especies de pseudomonas fluorescentes (Almario et al., 2017; Ahmad et al., 2018; Hernández-Salmerón et al., 2016).
Debido a la importancia de generar nuevos métodos para la búsqueda de mecanismos indirectos de promoción del crecimiento vegetal (o biocontrol) en bacterias benéficas (Santoyo et al., 2019), en el presente trabajo se analizó si la producción de sideróforos en aislados bacterianos de la endósfera y rizósfera de plantas de zarzamora (Rubus fruticosus L.) como un marcador de selección de cepas de Pseudomonas, los cuales a su vez puedan encontrarse con otros elementos genéticos de biocontrol de patógenos, tales como los genes antifúngicos phlD y hcnC.
Método
Bacterias endófitas y rizosféricas asociadas a R. fruticosus
En el presente estudio se analizaron un total de 410 aislados bacterianos asociados a la endósfera y rizósfera de plantas de R. Fruticosus, obtenidos por Contreras et al. (2016). Brevemente, para aislar los endófitos los tejidos (raíz y tallo) de plantas sanas (o asintomáticas) se sumergieron en etanol al 70% durante 3 minutos, se lavaron con solución fresca de hipoclorito de sodio durante 5 minutos, se enjuagaron con etanol al 70% durante 30 segundos y finalmente se lavaron cinco veces con agua destilada estéril. Para confirmar que el proceso de esterilización fue exitoso, las partes alícuotas del agua destilada estéril utilizada en el enjuague final se colocaron en cajas Petri con medio agar nutritivo. Las placas se examinaron para determinar que no hubiera crecimiento bacteriano después de 5 días de crecimiento en incubación a 28°C. Los aislados rizosféricos se obtuvieron a partir del suelo que se encontraba fuertemente adherido a las raíces de las plantas, para ser colocado en medio mínimo (Contiene en g/L: 13g de KH2PO4; 2g de (NH4)2SO4; 5mg de FeSO4; 25mg de MgSO4; 4g de glucosa y 15g de agar; pH 7). Posteriormente, se realizaron diluciones seriadas para ser inoculadas en medio mínimo con agar e incubando durante 3 días a 28°C. Se realizaron también dos procesos de purificación con el fin de tener aislados puros. Los aislados que se obtuvieron fueron mantenidos en glicerol al 50% y resembrados de manera rutinaria en cajas Petri con agar nutritivo (el agar nutritivo se adquirió de BD Bioxon y contiene en g/L: 5 gramos de peptona, 3 g de extracto de carne y 15 g de agar, con un pH final 6.8) al 20%, las cuales se incubaron por 48 h a 30 °C, previo a cada análisis.
Identificación de cepas productoras de sideróforos
Para la determinación de la producción de sideróforos, se utilizó el medio sólido CAS (Contiene cromo azurol sulfonato) en el cual se sembraron las cepas, posteriormente se incubaron por 72 h a 30 °C. Al finalizar este periodo, se seleccionaron las bacterias productoras de sideróforos tomando en cuenta la formación de un halo (amarillo-naranja) alrededor de la colonia. La producción de sideróforos por cada cepa se realizó por triplicado. El cambio de color de azul a amarillo-naranja indica la producción de sideróforos, debido a que los sideróforos exhiben una mayor afinidad por el hierro que el cromógeno (Santoyo et al., 2019).
Amplificación de los genes phlD y hcnC
El ADN genómico se obtuvo de los aislados productores de sideróforos mediante el uso del Wizard® Genomic DNA Purification Kit de Promega®. A través de la PCR se amplificaron los genes phlD y hcnC. Para la detección de los genes phlD se emplearon los oligonucleótidos phl2a 5’-GAGGACGTCGAAGACCACCA-3’ y phl2b 5’-ACCGCAGCATCGTGTATGAG-3’ (Raaijmakers et al., 2001); mientras que para detectar los genes hcnC se usaron los oligonucleótidos FhcnC 5’-GCGGGGTGATTTTCTTTCGG-3’ y RhcnC 5’-GTACAGCGCGTTGGATTGC-3’, diseñados en este trabajo a través del alineamiento de múltiples secuencias hcnC de diversas cepas de Pseudomonas. Se empleó la cepa K12 de E. coli como control negativo para la amplificación de ambos genes; así como los respectivos controles sin ADN templado (no mostrados en el gel de la Fig. 2). Los productos de PCR se amplificaron por duplicado en cada cepa y se purificaron con un kit (Wizard® Genomic DNA Purification Kit de Promega®), se secuenciaron (Molecular Research LP, Texas, EE. UU.) y analizaron usando la herramienta BLASTn del GenBank. Las secuencias 16S de los aislados identificados se depositaron en la base de datos del GenBank con los números de accesos KP634997- KP635000 (Contreras-Pérez et al., 2016).
Bioensayos de antagonismo
Los bioensayos de antagonismo fúngico se realizaron por triplicado como lo descrito por Hernández-León et al. (2015). Para ello, se emplearon medios con agar nutritivo. Se incubaron 24 h a una temperatura de 28 ºC en obscuridad; transcurrido este tiempo se realizaron las mediciones del diámetro del crecimiento micelial de los patógenos, los cuales cumplieron 48 h en el medio, y se determinó el porcentaje de inhibición del crecimiento micelial de acuerdo a la siguiente fórmula: % de inhibición del crecimiento = [(A m-A t) / A m] × 100, donde A m es el área micelial de control y A t es el área micelial con tratamiento.
Resultados
En el presente trabajo se buscó la producción de sideróforos y la presencia de los genes antifúngicos hcnC y phlD en 410 aislados bacterianos asociados a plantas de R. fruticosus L.) de la región de Zamora, Michoacán, México. Los resultados muestran que 70 aislados produjeron sideróforos. La Fig. 1 (panel A) muestra un análisis general de los aislados que producen (o no) sideróforos en medio CAS, así como su confirmación al cultivarse en un medio con cuatro aislados (panel B).
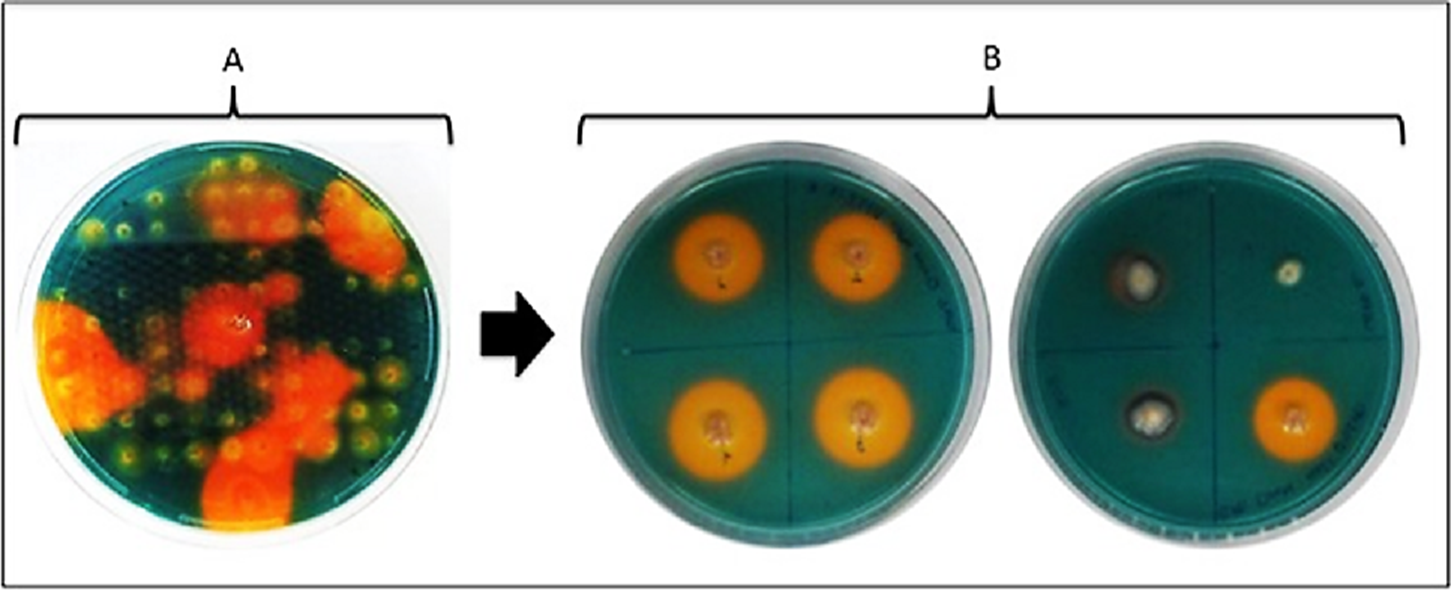
Fig. 1 Análisis de aislados asociados a R. fruticosus productores de sideróforos, observado como un halo de color amarillo-naranja alrededor del inóculo bacteriano. El panel A muestra una caja Petri con medio CAS (Cromo Azurol Sulfonato) con diferentes aislados. El panel B muestra la reinoculación y corroboración de aquellos aislados que producen (o no) sideróforos.
En un alto porcentaje (70%) de las cepas productoras de sideróforos, se detectaron ya sea uno o dos de los genes antifúngicos analizados (Cuadro 1); mientras que, el gen hcnC se detectó en el 51.4% del total de aislados productores de sideróforos analizados, siendo una mayor proporción que el gen phlD (19%). También, es relevante resaltar que se encontró una mayor detección de genes phlD y/o hcnC en cepas aisladas de la endósfera (43/70), comparado con aquellas cepas rizosféricas (6/70). Cabe mencionar que la producción de sideróforos fue un primer factor de selección de aquellos aislados con múltiples factores antagónicos; tales como presencia de genes phlD y/o hcnC, que podrían sintetizar HCN y DAPG, respectivamente, y que podrían ejercer un potencial papel de biocontrol hacia los patógenos probados en este trabajo.
Cuadro 1 Porcentajes y proporciones de detección de genes antifúngicos en aislados productores de sideróforos de origen endófito o rizosférico.
| Aislados/Genes | Porcentaje total | Aislados de origen endófito | Aislados de origen rizosférico |
|---|---|---|---|
| Aislados productores de | |||
| sideróforos | 17 % (70/410) | 65.7 % (46/70) | 34.2 % (24/70) |
| Detección de genes | |||
| antifúngicos | 70 % (49/70) | 61.4 % (43/70) | 8.5 % (6/70) |
| Detección del gen hcnC | 51.4 % (36/70) | 91.6% (33/36) | 8.3 % (3/36) |
| Detección del gen phlD | 19 % (13/70) | 27.7 % (10/36) | 8.3 % (3/36) |
*Números entre paréntesis muestra las proporciones de los aislados encontrados en el muestreo, ya sea totales (endófitos más rizosféricos) o analizados por origen endófito o rizosférico.
La Fig. 2 muestra un ejemplo de los geles de agarosa en donde se amplificaron los genes hcnC o phlD. Al detectarse dos o más bandas, los experimentos se repitieron, o bien las bandas se purificaron para ser secuenciadas, corroborando por búsquedas de homología la identidad con genes hcnC o phlD ya reportados en el GenBank. Cabe destacar que las identidades de las secuencias estuvieron entre un 99 y 100% con genes ya reportados de especies ampliamente estudiadas del género Pseudomonas.
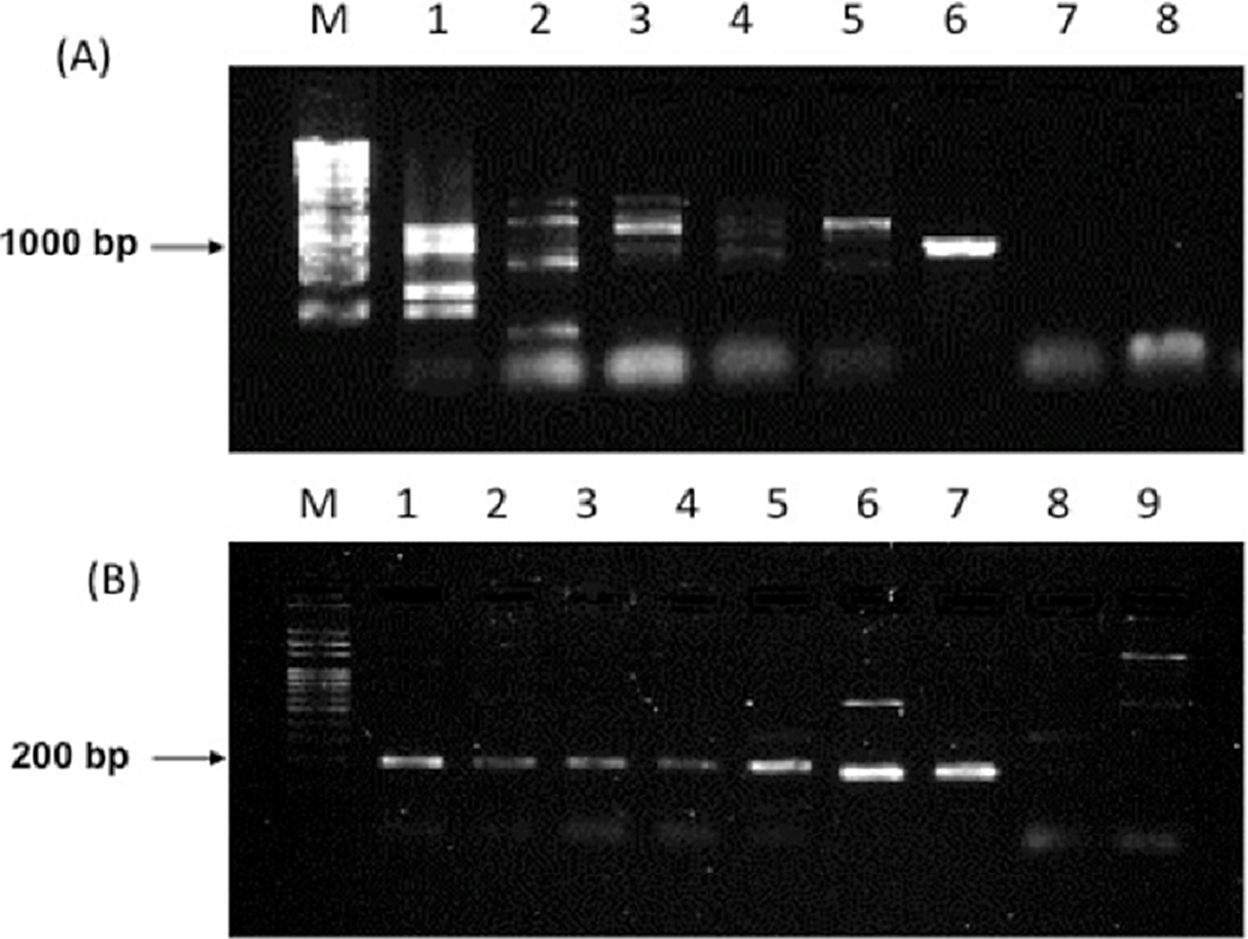
Fig. 2 Amplificación por PCR del gen phlD en aislados rizosféricos y endófitos (Panel A). La imagen muestra un gel agarosa al 1% representativo. El carril M muestra el marcador de peso molecular, el carril 1 el control positivo de P. fluorescens UM270, mientras que del carril 2 al 7 son diferentes aislados asociados a R. fruticosus. El carril 8 es un control negativo (E. coli). El Panel B muestra un gel agarosa al 2% representativo de los genes hcnC. El carril M muestra el marcador de peso molecular, el carril 1 el control positivo de P. fluorescens UM270, mientras que del carril 2 al 8 son diferentes aislados asociados a R. fruticosus. El carril 9 es un control negativo sin ADN templado.
Finalmente, se observó una alta relación entre la producción de sideróforos, la presencia de genes phlD y/o hcnC y la actividad antagónica hacia los fitopatógenos B. cinerea, Fusarium oxysporum, F. solani y Rhizoctonia solani (Cuadro 2). Como un control negativo se usó la cepa Escherichia coli K12, la cual no exhibe antagonismo hacia patógenos fúngicos de plantas. Por otra parte, como control positivo y comparativo del antagonismo presentado por las cepas endófitas o rizosféricas analizadas en este trabajo, se empleó la cepa P. fluorescens UM270, la cual ha sido anteriormente reportada por su producción de sideróforos, presencia de los operones hcnABC y phlACBD, entre otros mecanismos de biocontrol y promoción del crecimiento vegetal (Hernández-León et al., 2015; Hernández-Salmerón et al., 2016). Otras cepas que mostraron una tendencia a inhibir el crecimiento de los patógenos, pero que no fue significativa, no se incluyeron en el Cuadro 2.
Cuadro 2 Aislados productores de sideróforos de origen endófito y rizosférico que presentaron inhibición del crecimiento del micelio de los patógenos fúngicos B. cinerea, F. oxysporum, F. solani y R. solani.
| Patógenos fúngicos | ||||||
|---|---|---|---|---|---|---|
| Aislado o cepa | Características | Botrytis cinerea | Fusarium oxysporum | Fusarium solani | Rhizoctonia solani | |
| COPE63 | Sid+hcnC | 33.9 ± 8.7b | 50.9 ± 3.5c | 38.9 ± 14b | 44.0 ± 13b | |
| COPE17 | Sid+hcnC | 39.0 ± 8.9b | 27.1 ± 12b | 27.0 ± 6.9b | 41.0 ± 12b | |
| COPE19 | Sid+hcnC+phlD | 27.0 ± 7.9b | 77.1 ± 9.0c | 58.8 ± 11c | 29.0 ± 8.5b | |
| COPE39 | Sid+hcnC+phlD | 78.0 ± 21bc | 81.1 ± 8.0c | 85.2 ± 17c | 58.0 ± 17bc | |
| COPE40 | Sid+hcnC+phlD | 28.0 ± 4.5b | 31.0 ± 3.3b | 34.2 ± 3.4b | 25.8 ± 2.1b | |
| COPE40-1 | Sid+hcnC+phlD | 66.5 ± 13c | 70.4 ± 8.0c | 26.8 ± 3.8b | 33.0 ± 5.0b | |
| UM270 | Sid+hcnC+phlD | 76.2 ± 13c | 88.4 ± 9.2c | 53.0 ± 15c | 55.2 ± 8.8c | |
| E. coli K12 | Ninguna | 2.11 ± 5.9a | 3.08 ± 4.1a | 1.22 ± 4.2a | 3.31 ± 2.2a | |
Se muestran los porcentajes de inhibición y los valores de la desviación estándar (±) del crecimiento fúngico como la media de tres réplicas independientes medidas entre el día 5 y 7 (dependiendo del patógeno y su dinámica de crecimiento), en comparación con el experimento control (Sin bacteria). Se presentan en el cuadro únicamente los resultados de las cepas que mostraron una inhibición del crecimiento de los patógenos estadísticamente significativa entre el tratamiento y los experimentos de control. Los datos se analizaron estadísticamente con una prueba ANOVA y la prueba de separación de medias de Duncan para comparaciones múltiples. Las letras indican que las medias difieren significativamente (p ≤0.05). La cepa K12 se usó como control negativo. La cepa P. fluorescens UM270 se empleó como control positivo para la detección de actividad antifúngica, previamente reportada (Hernández-León et al., 2015; Hernández-Salmerón et al., 2016).
Discusión o Conclusiones
La producción de sideróforos por parte de bacterias promotoras del crecimiento vegetal es uno de los principales mecanismos de antagonismo hacia patógenos de plantas y uno de los primeros en describirse. Su acción implica la quelación de hierro en ambientes rizosféricos, haciendo este micronutriente menos disponible para los patógenos (Kloepper et al., 1980). En el presente trabajo se analizó la relación entre la producción de sideróforos en cepas bacterianas y su origen de aislamiento (rizósfera o endósfera). La producción de sideróforos se ha reportado en cepas rizosféricas de diversas plantas, así como en cepas de origen endófito (de los Santos-Villalobos et al., 2018). Interesantemente, las cepas que mostraron una producción clara de síntesis de sideróforos en medio CAS pertenecen al género Pseudomonas (Contreras-Pérez et al., 2016), la comparación de secuencias de los genes phlD y hcnC usando la herramienta BLASTn y mostrando una alta identidad con dicho género, aunque requieren un análisis adicional para asignar la especie a la que pertenecen los aislados. En cierto sentido podría esperarse esto, ya que las Pseudomonas son conocidas por su capacidad de producción de sideróforos (Kloepper et al., 1980), además de que son bacterias abundantes en ecosistemas como la rizósfera o la endósfera (Santoyo et al., 2012; Mendes et al., 2011). En contraste, Contreras y colegas (2016) reportaron que las especies del género Pseudomonas no fueron de las más abundantes en la endósfera de plantas de zarzamora, siendo otros géneros como Bacillus, Herbaspirillum, Burkholderia y Lysinibacillus los más abundantes. Sin embargo, a diferencia de la investigación de Contreras et al. (2016), en este trabajo se usó como marcador selectivo la producción de sideróforos, por lo que podría esperarse una mayor selección de especies de Pseudomonas. Sin embargo, no se descarta que un estudio más amplio pueda detectar cepas de otros géneros, como Burkholderia, que son también productores de sideróforos.
La producción y excreción de sideróforos bacterianos en ambientes como la rizósfera, donde pueden quelar el hierro disponible del medio y ser nuevamente adquirido por cepas de Pseudomonas, es uno de los primeros mecanismos indirectos de promoción del crecimiento vegetal (Kloepper et al., 1980). La clave está en hacer menos disponible dicho elemento (Fe) en la rizósfera para los potenciales patógenos fúngicos, restringiendo así su crecimiento (Santoyo et al., 2010). Cabe destacar que el papel de los sideróforos producidos por bacterias endófitas no ha sido estudiado a detalle, pues su papel ha sido endocado hacia estudios de fitoextracción de compuestos tóxicos para la planta (Rajkumar et al., 2010); sin embargo se sabe que dichos compuestos quelantes de hierro inducen el sistema de defensa de la planta contra potenciales patógenos, además de conferir al endófitos una ventaja para colonizar los tejidos internos de la planta y excluir otros microorganismos (Loaces et al., 2011; Ramos-Solano et al., 2010). Así mismo, se ha reportado que si no existe una limitante de Fe en el medio ambiente, el antagonismo se pierde (Kloepper et al., 1980). Por lo tanto, aquellas cepas bacterianas que contengan no sólo la producción de sideróforos, sino que presenten otros mecanismos de antagonismos, como la producción de volátiles (e.g. HCN) o compuestos difusibles (e.g. DAPG), podrían tener mejores actividades (y oportunidades) de biocontrol de fitopatógenos en abientes como la rizósfera y posiblemente la endósfera (Glick, 2014).
El gen hcnC forma parte del operón hcnABC, el cual es importante para la síntesis del compuesto volátil ácido cianhídrico o HCN (Laville et al., 1998). Dicho compuesto se ha reportado ampliamente como antifúngico (Hernández-León et al., 2015). Diversos trabajos reportaron que el compuesto HCN es responsable del antagonismo hacia patógenos fúngicos (Santoyo et al., 2019). Aunque en este trabajo se reporta únicamente la amplificación de un fragmento del gen hcnC, es muy probable que se encuentren presentes los demás genes del operón, así como también la posible producción del compuesto HCN; pero se requiere de trabajos adicionales para corroborar dicha hipótesis.
Como se había mencionado anteriormente, el gen phlD forma parte del operón phlACBD, que biosintetiza el compuesto 2,4-diacetilfloroglucinol (DAPG) (Yang y Cao, 2012). El gen phlD codifica para una policétido sintasa, siendo la principal responsable de la producción de monoacetilfloroglucinol (MAPG), precursor directo de 2,4-DAPG mediante una acetilación llevada a cabo por las demás enzimas PhlA, PhlB y phlC (Bangera y Thomashow, 1999). Al igual que el HCN, la síntesis de DAPG ha sido reportada como una de las principales caratcerísticas antifúngicas y de biocontrol en cepas de Pseudomonas (Hernández-León et al., 2015). En un trabajo realizado por Mavrodi et al. (2001) se reportó una amplia presencia del gen phlD en cepas de Pseudomonas de diferentes suelos. Un dato interesante que arrojó dicho trabajo fue la gran diversidad genética que existe en los genes phlD. Esto podría estar asociado a que en nuestro trabajo no detectamos este marcador en más cepas de Pseudomonas, por lo que el diseño y utilización de más oligonucleótidos, algunas con bases degeneradas, podría ayudar a detectarlo en más aislados. Otra investigación sugiere que las Pseudomonas productoras de DAPG no forman un sólo grupo filogenético, sino que la presencia del operón biosintético phlACBD podría estar distribuido en otros géneros bacterianos (Almario et al., 2017). Adicionalmente, en la base de datos Pseudomonas Genome Database (Winsor et al., 2010) se reportan cientos de genomas de especies del Pseudomonas, por lo que realizar una búsqueda preliminar para la detección de genes para la síntesis de sideróforos puede ser una herramienta rápida y fácil, que puede permitir la identificación de cepas con potencial para el biocontrol de fitopatógenos (aunque no todas presentan genes para la producción de sideróforos, HCN o DAPG.
En conclusión, los sideróforos son agentes quelantes de hierro y su producción por parte de cepas de Pseudomonas ha sido uno de los primeros mecanismos de promoción del crecimiento vegetal propuesto (Kloepper et al., 1980). Sin embargo, es deseable que un biofungicida bacteriano presente dos o más características antagónicas, como la producción del volátil HCN y el compuesto difusible DAPG (Santoyo et al., 2019). Así, en este trabajo se resalta la importancia de la detección inicial de actividad de producción de sideróforos en aislados asociados a plantas de zarzamora, lo cual puede ser útil para detectar múltiples mecanismos de antagonismo hacia patógenos fúngicos de plantas. Estudios adicionales con las cepas que mostraron mejores actividades de antagonismo (e.g. EZ8 y EZ16) se están llevando a cabo para detectar la efectividad fungicida en campo.











 nueva página del texto (beta)
nueva página del texto (beta)


