Introducción
El género Fragaria forma parte de la familia Rosaceae, una de las más importantes a nivel mundial, que agrupa aproximadamente a 3,000 especies de 107 géneros; presenta diferentes niveles de ploidia, desde diploides, tetraploides, hexaploides y octaploides. Fragaria × ananassa es una de las especies más ampliamente cultivadas, de naturaleza octaploide. Se originó a partir de la cruza de dos especies silvestres octaploides: F. virginiana y F. chiloensis. Todos los esfuerzos de mejoramiento genético en el cultivo de la fresa partieron de esta población inicial, por lo que la fresa tiene una base genética reducida (Hummer, 2008, 15; Hancock, 2008, 394; Ather et al., 2018, 221).
En México existen programas de mejoramiento genético tanto públicos (Colegio de Posgraduados e Instituto Nacional de Investigaciónes Forestales Agrícolas y Pecuarias) como privados (Driscoll´s, Berrimex, Splendor, Sunbelle, Agrana, Planamerica) que han intentado dar solución a la problemática de los productores y consumidores, generar variedades que se adapten a diferentes condiciones climáticas. En este contexto, surge la necesidad de desarrollar una variedad para clasificarla a nivel morfológico, fisiológico y molecular, con la finalidad de mantener su integridad genética en todas y cada una de las etapas de incremento del germoplasma; para este propósito una de las herramientas más efectivas son los marcadores moleculares (Whitaker, 2011, 116), que permiten identificar poblaciones con una diversidad genética reducida y más vulnerables a un posible cambio ambiental; así como, distinguir subpoblaciones genéticamente diferenciadas del resto, inferir genealogías y conocer el grado de parentesco entre individuos (Graham et al., 1996, 402; Powell et al., 1996, 226; Degani et al., 2001, 2; Sargent et al., 2006, 1350; Carbone et al., 2006, 128; Dangl et al., 2007, 118; Rousseau-Gueutin et al., 2008, 2046; Gil-Ariza et al., 2009, 338; Muhammad et al, 2018, 270). Adicionalmente los marcadores moleculares se utilizan para la identificación varietal, protección de la propiedad intelectual, evaluación de la pureza del material vegetal en viveros, la identificación de variación somaclonal en lotes de cultivo in vitro y para evitar la mezcla de material vegetal en bancos de germoplasma (Firas y Abdulkareem, 2015, 121).
Para la identificación varietal mediante marcadores moleculares en fresa se han usado diversas técnicas como los AFLP´s (del inglés Amplified Fragment Lenght Polymorphisms) y RAPD’s (del inglés Random Amplified Polymorphic DNA), (Powell et al., 1996, 225; Degani et al., 1998, 247; Congiu et al., 2000, 239; Degani et al., 2001, 1; Arnau et al., 2001, 302; Tyrka et al., 2002, 273; Garcia et al., 2002, 76; Melese et al., 2018, 2), además de los microsatélites SSR (del inglés Simple Sequence Repeat), que son arreglos de di-, tri- o tetra- nucleótidos, posicionados uno enseguida del otro, muy abundantes en todo el genoma eucariótico, codominantes, expresan varios alelos por locus, tienen un alto nivel de polimorfismo y su costo es menor en comparación con marcadores de generaciones anteriores (Arnau et al., 2002, 70; Shimomura y Hirashima, 2006, 399; González y Aguirre, 2007, 568; Debnath y Ricard, 2009, 83; Amom y Nongdam, 2017, 2). Los SSR se han convertido en los marcadores preferidos para la identificación varietal tanto en el sector público como en el privado (Debnath et al., 2008, 314; Nunes et al., 2013, 451) son robustos, confiables, reproducibles y consistentes como identificadores genotípicos; también se han usado en la caracterización de poliploides (Holton et al., 2002, 66; Beovides et al., 2006, 11; Han et al., 2006, 431; Zhang et al., 2010, 134; Novo et al., 2010,110; Hwang et al., 2011, 288; Nantoumé et al., 2013, 2131; Sánchez-Sevilla et al., 2015, 22), en la caracterización molecular del cultivo de fresa (Arnau et al., 2002, 69; Sargent et al., 2003, 550; Hadonou et al., 2004, 430; Carrasco et al., 2013, 1196) y se han utilizado para distinguir entre variedades de fresa altamente relacionadas (Shimomura y Hirashima, 2006, 399; Carrasco et al., 2007, 504; Dangl et al., 2007, 120; Debnath et al., 2008, 316; Hussein et al., 2008, 678; Debnath y Ricard, 2009, 85; Debnath, 2014, 338; Kim et al., 2019, 70). De esta manera, la caracterización molecular representa una herramienta complementaria a la caracterización morfológica, donde se puede evaluar la integridad genética del material vegetal de manera rápida y eficaz. Por lo anterior el objetivo de la presente investigación fue obtener la huella genética de un grupo de variedades de fresa desarrolladas en el Colegio de Postgraduados mediante el uso de SSR.
Método
Material vegetal
Se analizaron las variedades de fresa CP0201 (Zamorana), CP0204 (Jacona), provenientes de la cruza de la variedad Camarosa y una selección avanzada, ambas están registradas en el catálogo nacional de variedades vegetales del Servicio Nacional de Inspección y Verificación de Semillas (Rodríguez-Bautista et al., 2012, 115; SNICS, 2018, 9), CPLE7, CP0615, selecciones avanzadas desarrolladas en el Colegio de Postgraduados y la variedad comercial Festival desarrollada en el año 2000 por la Universidad de Florida en los Estados Unidos de América (Whitaker et al., 2011, 556).
Extracción de ADN
Se utilizaron hojas recientemente expandidas de 10 plantas de cada variedad cultivadas en condiciones de invernadero y se colocaron a -20 °C antes del proceso de extracción de ADN. Para el proceso de extracción se usó el kit comercial ChargeSwitch® (Invitrogene, 2006, 13), a partir de 100 mg de tejido foliar, 600 µL de lisis, 100 µL de reactivo A, el cual se modificó con PVP 40 000 en lugar de PVP 10 000, 2µL de ARNasa y 400 µL de buffer de precipitación (Nunes et al., 2011, 1384). La separación y elución del ADN se hizo con un robot de extracción KingFisher™ Flex Magnetic Particle Processor (Thermo Scientific, Waltham, MA, USA).
La concentración expresada en ng µL-1 y la calidad del ADN en términos de densidad óptica de absorbancia (DO260/280) se midió con un espectrofotómetro de ultra bajo volumen (NanoDrop® 2000, Thermo Scientific, Waltham, MA, USA), se verificó que la relación 260/280 se mantuviera entre 1.4 y 1.8; después se ajustó a una concentración de 20 ng µL-1 para su uso en la amplificación de los marcadores.
Amplificación por PCR
Para la amplificación de la reacción en cadena de la polimerasa (PCR, del inglés, Polymerase Chain Reaction) se usaron diez repeticiones de producto de DNA de cada variedad, por lo que se analizaron 100 muestras. Las reacciones se modificaron a partir de la metodología de Sargent et al. (2003, 552) y la de Hilmarsson et al., (2017, 5).
En la reacción de PCR se usó un volumen total de 25 µL con 20 ng µL-1 de ADN genómico, 1.5 µM de MgCl2, 10 pmol de cada iniciador, 1X PCR Buffer, 0.2 µM de DNTPs, una unidad de ADN polimerasa (kit GoTaq®, Promega). La configuración del programa de PCR con los tiempos modificados constó de un paso de desnaturalización inicial de 95 °C por 4 min; 30 ciclos de desnaturalización de 95 °C por 1 min, alineamiento por 2 min a las temperaturas de alineamiento (Ta) óptimas para cada primer determinadas por pruebas de PCR en gradiente, extensión de 72 °C por 2 min; por último extensión final a 72 °C por 60 min, lo anterior se realizó en un termociclador BIORAD Modelo C1000, Hercules, CA, USA.
Se utilizó un conjunto de nueve pares de iniciadores SSR (Cuadro 1), desarrollados para fresa diploide (Sargent et al., 2003, 551) y probados en fresa octaploide (Govan et al., 2008, 654; Brunings et al., 2010, 66; Sargent et al., 2012, 1231; Kim et al., 2019, 111). Los iniciadores se agruparon en reacciones multiplex usando el programa Multiplex Manager (Holleley y Geerts, 2009, 512); se tomó como base las temperaturas de alineamiento, determinadas de manera empírica para cada uno de los iniciadores.
Cuadro 1 Secuencia de los iniciadores y etiqueta fluorescente asignada
| Grupo | Marcador | Secuencia 5’→3’ | Etiqueta | |
|---|---|---|---|---|
| 1 | EMFn182 | F | GCAACAAAGGAGGTTAGAGTCG | [6-FAM] |
| R | TGGTGAGTGCTCATTGTTCC | |||
| EMFvi166 | F | ACCGACAGCTGAGTTAGAGGAG | [6-FAM] | |
| R | AGTCATAGGACCCCACTTCAAA | |||
| ARSFL11 | F | GCGAAGCATAACTGGCAGTATCTG | [HEX] | |
| R | GCGGGCCTAGGTGATCTTGGA | |||
| 2 | EMFv104 | F | TGGAAACATTCTTACATAGCCAAA | [6-FAM] |
| R | CAGACGAGTCCTTCATGTGC | |||
| EMFn181 | F | CCAAATTCAAATTCCTCTTTCC | [HEX] | |
| R | GCCGAAAAACTCAAACTACCC | |||
| EMFn121 | F | GGTCCCTAAGTCCATCATGC | [6-FAM] | |
| R | GAGTGGATGCAAACATGAGC | |||
| 3 | EMFvi136 | F | GAGCCTGCTACGCTTTTCTATG | [6-FAM] |
| R | CCTCTGATTCGATGATTTGCT | |||
| EMFn170 | F | CAGTTTGCCCAACAACAAGG | [HEX] | |
| R | TTGATGGCAACAAATCACG | |||
| EMFn111F | F | GAAGCTCCTCTCACAAAGTTAAGG | [6-FAM] | |
| R | TCAACAACAACATCAACAAAGG | |||
F: Forward; R: Reverse.
Análisis de fragmentos
Los productos de PCR se analizaron mediante electroforesis capilar en un secuenciador automático de ADN (ABI PRISM® 3100 Genetic Analyzer, Applied Biosystems, Foster City, CA, USA), se utilizó Liz-500 como marcador estándar interno de peso molecular; se usó un factor de dilución de cinco al momento de cargar los productos de PCR en el secuenciador. La lectura de los electroferogramas e identificación de alelos se realizó con el programa GeneMapper® versión 4.
Análisis de la información
Se construyeron patrones alélicos a partir de los cuales se identificaron las diferencias en número, tamaño e intervalo de amplificación para cada marcador. Posteriormente, con las frecuencias alélicas, calculadas a partir de la matriz binaria de presencia y ausencia elaborada con el paquete en Polysat bajo el ambiente R (Clark y Jasieniuk, 2011, 563), se compararon las similitudes o diferencias de los patrones alelicos expresados en cada una de las variedades; para ello se usó el programa de análisis de datos INFOGEN/E, v. 2014, a partir de ello se obtuvo la matriz de distancias con dos coeficientes distintos, el de Jaccard (Sj) (Sneath y Sokal, 1973, 121) y el de Dice (SD) (Dice, 1945, 297). Estos coeficientes expresan la probabilidad de que un alelo en un individuo se encuentre también en otro individuo (Lim et al., 2017, 230). A partir de estos coeficientes se construyeron fenogramas con el programa NTSYSpc v. 2.10.
Como estimadores poblacionales, se calcularon los siguientes índices:
Resultados
Amplificación de los marcadores SSR
Todos los marcadores usados se amplificaron vía PCR; además, el tamaño y número de las amplificaciones fueron consistentes con estudios previos que se pueden observar en el Cuadro 2, donde se presenta el número de picos, la temperatura de alineamiento para cada marcador y su tamaño expresado en pares de bases; donde se destaca que el peso molecular de los fragmentos amplificados se mantuvo dentro de los límites reportados por Govan et al. (2008, 653) y Brunings et al. (2010, 92).
Cuadro 2 Temperaturas de alineamiento, picos observados y tamaño de los fragmentos encontrados en el presente estudio en comparación con estudios previos
| Locus | -------Este estudio------ | Brunings et al . (2010) | Govan et al . (2008) | ||||
|---|---|---|---|---|---|---|---|
| Ta | PO | Rango | PO | Rango | PO | Rango | |
| (°C) | (Pb) | (Pb) | (Pb) | ||||
| ARSFL11 | 61.5 | 5 | 250 - 275 | 8 | 258-278 | 14 | 257-321 |
| EMFN111 | 61.5 | 3 | 230 - 250 | - | - | 24 | 208-269 |
| EMFn121 | 51.5 | 5 | 225 - 260 | 8 | 228-254 | 15 | 226-256 |
| EMFn170 | 58.0 | 9 | 185 - 230 | 9 | 188-233 | 15 | 188-238 |
| EMFn181 | 54.6 | 11 | 155 - 240 | 7 | 164-212 | 35 | 138-248 |
| EMFn182 | 59.8 | 5 | 170 - 210 | 8 | 179-202 | 15 | 191-219 |
| EMFv104 | 58.6 | 12 | 75 - 135 | 15 | 72-126 | 27 | 69-138 |
| EMFvi136 | 59.8 | 7 | 130 - 170 | 8 | 132-160 | 18 | 111-188 |
| EMFvi166 | 59.8 | 4 | 255 - 285 | 4 | 268-281 | 11 | 254-282 |
Ta: Temperatura de alineamiento; PO: Picos observados; Pb: Pares de bases
El número de alelos observados es diferente en comparación con lo reportado por otros autores, debido a la cantidad y naturaleza de las variedades evaluadas, ya que la riqueza alélica de los marcadores SSR está influenciada por el número de poblaciones que se analicen (Yoon et al. 2012, 5). Se determinaron 63 alelos diferentes en las cinco variedades estudiadas (Cuadro 3). Con los nueve marcadores, el número de alelos amplificados estuvo entre 3 y 12 por marcador. Los marcadores que más alelos amplificaron fueron el EMFn181 y el EMFv104, con 11 y 12, respectivamente. De acuerdo con Govan et al. (2008, 658) y Kim et al. (2019, 111) tan sólo uno de estos dos SSR puede discriminar entre variedades muy relacionadas. Todos los marcadores evaluados tuvieron un alto nivel de variabilidad y están distribuidos por todo el genoma del género Fragaria, lo que resultó en una huella única para cada variedad.
Cuadro 3 Número de alelos observados y peso molecular expresados en pares de bases en los marcadores probados considerando las cinco variedades de fresa estudiadas
| Marcadores | Número de alelos observados | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| †A1 | A2 | A3 | A4 | A5 | A6 | A7 | A8 | A9 | A10 | A11 | A12 | |
| ARSFL11 | ¶261 | 263 | 267 | 259 | 272 | |||||||
| EMFn182 | 176 | 185 | 188 | 198 | 190 | 191 | ||||||
| EMFvi166 | 278 | 271 | 278 | 269 | 260 | |||||||
| EMFn121 | 229 | 233 | 254 | 255 | 248 | |||||||
| EMFn181 | 166 | 172 | 175 | 185 | 188 | 211 | 218 | 231 | 159 | 209 | 236 | |
| EMFv104 | 78 | 88 | 91 | 104 | 108 | 112 | 123 | 125 | 128 | 95 | 110 | 117 |
| EMFN111 | 244 | 247 | 236 | |||||||||
| EMFN170 | 187 | 193 | 197 | 201 | 208 | 223 | 225 | 216 | 205 | |||
| EMFvi136 | 134 | 143 | 153 | 155 | 161 | 163 | 157 | |||||
†Alelos identificados; ¶Este valor corresponde al peso molecular de cada alelo expresado en pares de bases (Pb)
Huella genética
Con los pesos moleculares de cada alelo, expresados en pares de bases (Pb), se crearon geles virtuales de las variedades por cada SSR (Figuras 1 y 2). En los cuales se observan claramente las diferencias entre variedades en cuanto al tamaño y número de fragmentos amplificados; esta información sirve como base estándar para comparar los resultados obtenidos en ensayos de identificación varietal del germoplasma. Las variedades CP0201 y CP0204, comparten a uno de sus progenitores, ambas provienen de la cruza de Camarosa por una selección avanzada del programa del Colegio de Postgraduados (SNICS, 2008a, 14; SNICS, 2008b, 14), por lo que están altamente emparentadas, situación que queda de manifiesto en su patrón alélico; sin embargo, es posible detectar diferencias entre ellas en los marcadores: EMFvi166 y EMFn181, estas se confirman con el análisis de similitud y el fenograma generado a partir de los datos moleculares.

Figura 1 Gel virtual con los patrones alélicos derivados de nueve loci de microsatélites SSR de las variedades CP0201 y CP0204, identificados mediante electroforesis capilar

Figura 2 Gel virtual con los patrones alélicos derivados de nueve loci de microsatélites SSR de las variedades CPLE7 y CP0615, identificados mediante electroforesis capilar
Las variedades CPLE7 y CP0615, (Figura 2), presentaron diferencias en cuanto al tamaño de los fragmentos encontrados, en los marcadores EMFn 121, EMFn181 y EMFN170, la presencia de alelos con diferentes pesos moleculares en un mismo locus se debe a la naturaleza codominante de los marcadores moleculares SSR y a la naturaleza octaploide de la fresa cultivada (Whitaker, 2011, 117; Kim et al., 2019, 111).
Brunings et al. (2010, 68) reportaron la huella genética de la variedad Festival de Florida, Estados Unidos, donde se analizó el mismo juego de marcadores moleculares, las diferencias con la variedad Festival mantenida en el Colegio de Postgraduados fueron las siguientes: cuatro marcadores (EMFvi166, EMFn121, EMFn170) tuvieron el mismo número de alelos; sin embargo, tres SSR (ARSFL11, EMFn182, EMFvi136) mostraron alelos distintos en tamaño de los fragmentos. El SSR EMFn181 tuvo nueve alelos contra cuatro en el trabajo previo y el SSR EMFv104 tuvo ocho alelos en comparación con seis del perfil de Florida (Figura 3).

Figura 3 Comparativos entre el patrón alélico de la variedad Festival mantenida en el Colegio de Postgraduados y la mantenida en Florida, según Brunings et al. (2010, 68)
Brunings et al. (2010, 71) y Lim et al., (2017, 230), encontraron en otras variedades más alelos con tamaños similares a los detectados en el presente estudio, lo que sugiere que la variedad Festival procedente del Colegio de Posgraduados tuvo un patrón alélico diferente al de la variedad mantenida en Florida; probablemente debido a contaminación en alguna de las etapas de incremento del material vegetal, posiblemente en vivero, lo anterior indica que es de gran importancia revisar cuidadosamente la metodología empleada en la multiplicación asexual de las colecciones de germoplasma en el programa de mejoramiento de fresa institucional.
Análisis de similitud
El análisis de similitud mostró que las variedades se diferenciaron en función de su origen, de tal manera que tanto para el coefiente de Jaccard como para el coeficiente de Dice se obtuvieron resultados similares cuando se calcularon las matrices de distancias fenéticas (Cuadro 4), los resultados de ambos se pueden observar en la Figura 4, donde se presentan los fenogramas que resultaron de cada uno de los coeficientes.
Cuadro 4 Matriz de similitud generadas a partir del conteo de alelos amplificados con microsatélites, obtenidos mediante los coeficientes de Jaccard y Dice
| Variedad | Jaccard | ||||
| CP0201 | CP0204 | CP0615 | CPLE7 | Festival | |
| CP0201 | 1.00 | ⁃ | ⁃ | ⁃ | ⁃ |
| CP0204 | 0.88 | 1.00 | ⁃ | ⁃ | ⁃ |
| CP0615 | 0.00 | 0.00 | 1.00 | ⁃ | ⁃ |
| CPLE7 | 0.70 | 0.58 | 0.00 | 1.00 | ⁃ |
| Festival | 0.61 | 0.69 | 0.00 | 0.33 | 1.00 |
| Variedad | Dice | ||||
| CP0201 | CP0204 | CP0615 | CPLE7 | Festival | |
| CP0201 | 1.00 | ⁃ | ⁃ | ⁃ | ⁃ |
| CP0204 | 0.94 | 1.00 | ⁃ | ⁃ | ⁃ |
| CP0615 | 0.00 | 0.00 | 1.00 | ⁃ | ⁃ |
| CPLE7 | 0.83 | 0.74 | 0.00 | 1.00 | ⁃ |
| Festival | 0.76 | 0.82 | 0.00 | 0.49 | 1.00 |
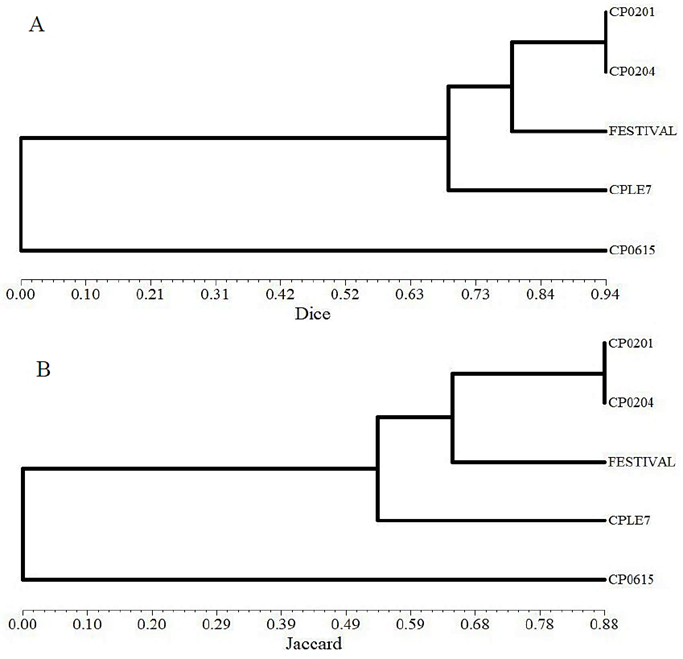
Figura 4 Fenogramas generados a partir de datos moleculares (SSR), resultado del cálculo de matrices de distancias fenéticas mediante el coeficiente de Dice (arriba) y de Jaccard (abajo)
El agrupamiento de las variedades fue la misma tanto al usar el coeficiente de Jaccard y el de Dice; CP0201 (Zamorana) y CP0204 (Jacona) tuvieron una huella genética similar y se situaron en la misma rama, así se confirmó su relación; los datos genealógicos de estas variedades indican que ambas comparten el mismo progenitor femenino (Rodríguez-Bautista et al., 2012, 115; SNICS, 2018, 9). Las variedades CP0615, CPLE7 y Festival poseen un patrón alélico distinto en comparación con las variedades CP0201 y CP0204. Lo anterior ratifica lo mencionado por Govan et al. (2008, 658) y Kim et al. (2019, 111) sobre el poder de discriminación de los microsatélites aquí empleados.
Estimadores poblacionales
En cuanto a los estimadores poblacionales calculados (Cuadro 5), el índice de uniformidad de una población (Uj), indica la distribución de cada uno de los alelos evaluados dentro de cada población, puede tomar valores de uniformidad máxima (Uj= 0 ó Uj= 1) o uniformidad mínima (0 < Uj < 1). El estimador Uj alimentado con los alelos derivados de los SSRs tuvo un nivel de uniformidad mínimo, debido a la naturaleza polimórfica de los microsatélites; el que más alelos presentó fue de nueve alelos de distintos tamaños; dado que el número de alelos amplificados por cada microsatélite cambia en función de las variedades, entonces la uniformidad entre poblaciones indicó que existieron diferencias entre los alelos observados.
Cuadro 5 Estimadores poblacionales de las variedades de fresa evaluadas
| Variedad | Uj | Hj | Hpop | Hsp |
|---|---|---|---|---|
| CP0615 | 0.54 | 3.96 | 4.89 | 24.45 |
| CP0201 | 0.46 | 5.10 | ||
| CP0204 | 0.50 | 4.32 | ||
| CPLE7 | 0.53 | 5.15 | ||
| Festival | 0.49 | 5.93 |
Uj : Índice de uniformidad de una población; Hj : Índice de diversidad de una población; Hpop: Polimorfismo de la población media; Hsp: Polimorfismo de la especie
En lo referente al índice de diversidad de una población (Hj), éste mide el grado de entropía o grado promedio de incertidumbre en identificar la categoría a que puede pertenecer un individuo elegido al azar de la colección. De esta manera, las variedades que tuvieron mayor diversidad alélica fueron Festival, CPLE7 y CP0201 (Zamorana), con respecto a las otras variedades consideradas en el estudio. En el Cuadro 5 se observa también el polimorfismo de la población media y el polimorfismo de la especie, dentro del espectro de las poblaciones analizadas.
Conclusión
Se obtuvo el patrón alélico o huella genética de cuatro variedades de fresa del Colegio de Postgraduados. Las diferencias entre variedades se expresaron en el número de alelos amplificados, así como en el tamaño de fragmentos. Se evidenciaron las similitudes entre las variedades CP0201 y CP0204, indicando su parentesco ya que comparten el progenitor femenino. Las variedades CP0615 y CPLE7 tienen un patrón alélico diferente a las demás variedades evaluadas.











 text new page (beta)
text new page (beta)


