Introducción
El protozoo llamado Trypanosoma cruzi (T. cruzi), es el agente etiológico que causa la enfermedad de Chagas. Esta enfermedad, es una de las causas más comunes de muerte relacionadas con insuficiencia cardiaca congestiva entre los adultos jóvenes en áreas endémicas de México, Sudamérica y Centroamérica (Cura and Viña, 2010). Actualmente la enfermedad de Chagas es considerada como una enfermedad infecciosa emergente de transmisión congénita, por transfusión sanguínea y/o por trasplante de órganos en los Estados Unidos y en países de Europa que reciben migrantes de países donde la enfermedad es endémica (Bern and Montgomery, 2009). La infección es transmitida de forma natural por insectos hematófagos de la subfamilia Triatominae, vectores que se distribuye fundamentalmente en áreas endémicas rurales de América, dónde las malas condiciones sociosanitarias favorecen su presencia (Hotez et al., 2014).
T. cruzi se caracteriza por mostrar una estructura poblacional clonal y a menudo multiclonal, es decir, que su población está compuesta por varias subpoblaciones de parásitos (Tibayrenc et al., 2018). Esta diversidad genética le confiere diferencias genotípicas y fenotípicas a cada una de las cepas del protozoario. Es posible que los humanos y otros hospederos, así como los triatominos se infecten con dos o más clones genéticamente diferentes (Andrade et al., 1999; Pinto et al., 2000). El comportamiento biológico de cada cepa puede estar relacionada con su estructura genética (Brenière et al., 2012). Esto puede tener influencia en las manifestaciones clínico-patológicas de la enfermedad, en el ciclo de transmisión y en la resistencia o sensibilidad a fármacos (Mejía-Jaramillo et al., 2012). Las variedades de T. cruzi estudiadas han sido clasificadas en seis linajes (TcI a TcVI) (Zingales et al., 2012). En pacientes chagásicos humanos, las variedades TcI, TcIV y TcV han sido asociadas a padecimientos cardiomiopáticos, mientras que los linajes TcII, TcV y TcVI a problemas digestivos (Zingales et al., 2018). En este contexto, resalta el TcI como el linaje más abundante en el continente Americano, el cual puede estar asociado con los ciclos silvestres y domésticos de la enfermedad (Zingales et al., 2018). Por otro lado, estudios contemporáneos que analizaron la resistencia o sensibilidad de diferentes cepas de T. cruzi a los fármacos más comunes, sugieren que una proporción importante de los parásitos de los diferentes linajes de T. cruzi presentan cierta resistencia a los fármacos comúnmente empleados para tratar la infección (Mendonça et al., 2018). Sin embargo, en aislados de T. cruzi de la región norte de Sudamérica, se han observado ciertas diferencias en la resistencia a los fármacos. Por ejemplo, se ha reportado que los parásitos del linaje TcI presentan resistencia al benznidazol más frecuentemente que los parásitos pertenecientes al biotipo II (Toledo et al., 2004). Los demás linajes (III, IV, V y VI) han sido poco estudiados, pero en ensayos in vitro ya se ha observado que algunas cepas correspondientes a los linajes III, V y VI presentan cierta resistencia a dicho fármaco (Hamuy et al., 2013). La resistencia a fármacos asociada a los diferentes linajes del parásito representa un problema clínico para el tratamiento efectivo de la enfermedad en humanos y es por eso importante determinar que linajes de T. cruzi circulan en cada región geográfica. En México, se ha reportado la presencia de los seis linajes de T. cruzi distribuidos de manera heterogénea en el país (revisado por López-Vivas et al., 2018). Sin embargo, en estos estudios pueden considerarse sólo como la fase inicial de la epidemiología molecular del parásito en México, y por eso se requiere seguir realizando más estudios que complementen el conocimiento de la situación que el país guarda en relación al los biotipos de parásito existentes. En el estado de Hidalgo, México, que es una región endémica del parásito, no se han hecho estudios moleculares para determinar los linajes de los parásitos. Por tal motivo resulta pertinente conocer la base genética de los aislados de T. cruzi que habitan en el estado. El objetivo fue determinar la diversidad genética del T. cruzi que infecta a los triatominos recolectados en las regiones endémicas del Estado de Hidalgo, a través de la amplificación por PCR- punto final del fragmento de la secuencia de la región intergénica del gen mini exón de T. cruzi, el cual es considerado un marcador molecular de tipificación.
Método
Área de estudio
Los insectos hematófagos de la subfamilia Triatominae fueron recolectados en 82 casas ubicadas en 14 municipios del Estado de Hidalgo (Atlapexco, Chapulhuacán, Huautla, Huejutla, Metztitlán, Orizatlán, Pacula, Pisaflores, San Agustín Metzquititlán, Tecozautla, Tenango, Tianguistengo, Xochiatipan y Zimapan). Se recolectaron 170 triatominos durante los meses de abril a septiembre de 2016, la metodología de búsqueda de triatominos fue realizada de acuerdo con la Norma Oficial Mexicana para la vigilancia epidemiológica, prevención y control de las enfermedades transmitidas por vector (NOM-032-SSA2-2002). Los insectos fueron recolectados por el personal de las brigadas antipalúdicas de las jurisdicciones sanitarias del Estado de Hidalgo, en un ejercicio de tres visitas por casa.
Identificación morfológica de los triatominos y examinación microscópica
Los triatomas se clasificaron por fechas y por localidades y se examinaron a través de microscopio estereoscópico. La identificación taxonómica de los triatominos se efectuó de acuerdo a las claves morfológicas de Lent and Wygodzinsky (1979). Se realizó el diagnóstico convencional (por la técnica de microscopia óptica), para determinar la presencia o ausencia de T. cruzi en los insectos. Por medio de la presión abdominal del triatomino se tomaron muestras de heces, se colocaron en un portaobjetos y se diluyeron con 50µl de solución PBS, pH 7.0, y se observaron a 40x a través del microscopio óptico (Souza, 2009).
Extracción del ADN
Para determinar la presencia o ausencia de T. cruzi en los triatominos colectados, además del diagnóstico microscópico, se realizó diagnóstico molecular por PCR. El ADN genómico del parásito se buscó en las heces de los triatominos. En triatominos vivos el ADN se extrajo de las heces y orina y en triatominos de los que no se podían extraer heces por alguna razón o porque estuvieran muertas, el ADN se obtuvo de los intestinos de cada insecto (la mezcla obtenida corresponde a 25 mg tejido, heces y orina). El ADN fue extraído y purificado utilizando el kit “Wizard® SV Genomic DNA Purification System” (Promega, USA), siguiendo las instrucciones del fabricante. Se confirmó la integridad del ADN de las muestras a través de la electroforesis horizontal en geles de agarosa al 0.8% y teñidos con bromuro de etidio (0.5 μg / ml) (Sambrook and Russell, 2001).
Detección de T. cruzi en triatominos mediante PCR múltiplex
Para la identificación y genotipificación de T. cruzi en los triatominos se realizó la técnica de reacción en cadena de la polimerasa múltiplex (PCR-mul) para la amplificación de los fragmentos del gen mini-exón de T. cruzi del parásito con los iniciadores descritos por Souto et al. (1996), que discrimina al grupo TcI de los grupos TcII-TcVI. Esta discriminación se da por el tamaño de banda amplificado: 350 pb para TcI y 300 pb para todos los demás linajes. Se usaron los siguientes oligonucleótidos: TcI (hacia adelante) 5'-GTGTCCGCCACCTCCTTCGGGCC-3' (amplifica fragmentos de 350 pb); TcII-TcVI (hacia adelante) 5'CCTGCAGGCACACGTGTGTGTG-3' (amplifica fragmentos de 300 pb) y TcI-TcVI (inverso) 5'-CCCCCTCCCAGGCCACACTG-3'. El volumen final para la reacción fue de 25 µl por tubo.
Se agregó 12.55 µl de H2O, buffer polimerasa 5µl, MgCl2 2µl, 1µl de dNTPs, 10 picomoles de cada primer, 0.25 de Taq polimerasa (GoTaq Flexi DNA Polymerase, Promega) y 45 ng de AND purificado. Las condiciones para la reacción fueron las siguientes: desnaturalización de 95 °C por 1 minuto, 30 ciclos con etapas de desnaturalización a 95 °C por 30 segundos, alineación a 61.6 °C durante 30 segundos y extensión a 72 °C por 30 segundos; por último se realizó una extensión final a 72 °C durante 6 minutos. Los productos finales de la PCR fueron separados mediante electroforesis en geles de agarosa al 1.5 % preparado con una solución amortiguadora de Tris base 40 mM, ácido acético 20 mM y EDTA 1 mM (TAE 1×). Se utilizó como amortiguador de corrida la misma solución mezclada con bromuro de etidio a una concentración final de 0.5 µg/ml para lograr la visualización de las bandas de ADN se utilizó el transiluminador de luz UV. Las fotos se obtuvieron con una cámara Canon power shot 5 MG pixeles.
Resultados y Discusión
El Cuadro 1 resume la información acerca de los sitios de búsqueda, las especies de insectos encontradas, las frecuencias de triatominos infectados con T. cruzi y el linaje de T. cruzi encontrado en su caso. La búsqueda de triatominos se realizó en un total de 82 casas distribuidas en 14 municipios del Estado de Hidalgo. Se recolectaron 170 ejemplares y se identificaron tres especies: La principal especie de insectos identificada fue T. dimidiata (87/170; 51.17%), seguida por T. mexicana (14/170; 8.23%) y finalmente T. gerstaeckeri (7/170; 4.11%). A un porcentaje importante de triatominos encontrados no se les pudo asignar una clasificación ni de especie, ni de sexo, dado que el método morfológico no es muy confiable para hacer estas clasificaciones en los estados ninfales. De ahí que se clasificaran como T. spp. al 36.47% (62/170) de los triatominos encontrados. Pero de acuerdo a las proporciones de adultos encontradas podría asumirse que las ninfas también en su mayoría deberían ser T. dimidiata. Pero para no dejar este tipo de información sin definir, en futuros estudios sería interesante considerar hacer la determinación molecular de la especie de las ninfas recolectadas. El 80.58% (137/170) de los triatominos recolectados fueron encontrados en el peridomicilio, en tanto que el restante 19.41% (33/170) de los insectos se recolectó del interior de los domicilios. De los ejemplares recolectados el 37.05% (63/170) fueron hembras adultas, 26.47% (45/170) machos adultos, y el resto eran ninfas no sexadas; 5.29% (9/170) del segundo estadio; 5.88% (10/170) del tercer estadio; 3.52% (6/170) del cuarto estadio y el 21.76% (37/170) del quinto estadio. Los ciclos de transmisión de T. cruzi se han clasificado en intradomiciliados (el de mayor riesgo para la salud pública), peridomiciliados y silvestres, principalmente de acuerdo al nicho ecológico que prefieren las distintas especies de triatominos (Salazar et al., 2010). Los hallazgos en el presente estudio concuerdan con lo reportado en otros autores (Salazar et al., 2010), en relación a que T. dimidiata se distribuye en el intradomicilio y el peridomicilio, en tanto que T. mexicana lo hace en el peridomicilio. T. gerstaeckeri, una especie que normalmente ha sido reportada en los tres ciclos de transmisión de T. cruzi (Martínez -Tovar et al., 2013), se encontró tanto en el peridomicilio como en el intradomicilio, en los municipios de Capulhuacan y Tianguistengo, lo que puede indicar que esta especie se está adaptando al nicho ecológico doméstico en las zonas mencionadas, lo cual podría representar un mayor riesgo de salud para la población humana. También es interesante señalar que en estas dos localidades (Capulhuacan y Tianguistengo), T. gerstaeckeri y T. dimidiata, parecen comportarse como especies simpátricas. La existencia de especies de triatominos simpátricas no es una novedad, ya que otros autores han reportado una asociación simpátrica de hasta tres especies diferentes en México (Rodríguez et al., 2011), pero hasta dónde sabemos, este es el primer reporte de la asociación simpátrica de T. gerstaeckeri y T. dimidiata.
Cuadro 1 Frecuencia de infección con T. cruzi e identificación entomológica de los triatominos capturados en los 14 municipios del Estados de Hidalgo
| Municipio | Número de localidades |
Triatominos positivos a T. cruzi | Biotipo | Ciclo Transmisión |
Especies de triatominos recolectadas |
||
|---|---|---|---|---|---|---|---|
| Microscopia óptica |
PCR-mul | Intra domicilio |
Peri domicilio |
||||
| Atlapexco (Casas=10) |
8 | 0 (0%) | 3(1.76%) | TcI | 0 | 33 |
T. dimidiata 14 (8.24%) y T.spp. 9 (11.18%) |
| Chapulhuacán (Casas=9) |
2 | 1 (0.59%) | 1 (0.59%) | TcI | 2 | 12 |
T. dimidiata 1 (0.59%), T. spp. 9 (5.29%) y T. gerstaeckeri 4 (2.35%) |
| Huautla (Casas=5) |
3 | 0 (0%) | 1 (0.59%) | TcI | 0 | 16 |
T. dimidiata 13 (7.65%) y T. spp. 3 (1.76%) |
| Huejutla (Casas=10) |
10 | 0 (0%) | 3 (1.76%) | TcI | 3 | 11 |
T. dimidiata 13 (7.65%) y T. spp. 1 (0.59%) |
| Metztitlán (Casas=5) |
3 | 1 (0.59%) | 1 (0.59%) | TcI | 0 | 5 | T. mexicana 5 (2.94%) |
| Orizatlán (casa=1) |
1 | 0 (0%) | 1 (0.59%) | TcI | 0 | 1 | T. dimidiata 1 (0.59%) |
| Pacula (Casas=1) |
1 | 0 (0%) | 0 (0%) | - | 0 | 1 | T. spp. 1 (0.59%) |
| Pisaflores (Casas=1) |
1 | 0 (0%) | 0 (0%) | - | 0 | 1 | T. gerstaeckeri 1 (0.59%) |
| San Agustín Metzquititlán (Casas=1) |
1 | 0 (0%) | 1 (0.59%) | TcI | 0 | 1 | T. mexicana 1 (0.59%) |
| Tecozautla (Casas=2) |
2 | 0 (0%) | 1 (0.59%) | TcI | 0 | 2 | T. mexicana 2 (1.18%) |
| Tenango (Casas=1) |
1 | 0 (0%) | 0 (0%) | - | 0 | 1 | T. gerstaeckeri 1 (0.59%) |
| Tianguistengo (Casas=13) |
3 | 0 (0%) | 0 (0%) | - | 14 | 5 |
T. dimidiata 14 (8.24%), T. spp. 4 (2.35%) y T. gerstaeckeri 1 (0.59%) |
| Xochiatipan (Casas=18) |
4 | 0 (0%) | 6 (3.53%) | TcI | 14 | 41 |
T. dimidiata 31 (18.24%) y T. spp. 24 (14.12%) |
| Zimapán (Casas=5) |
3 | 1 (0.59%) | 1 (0.59%) | TcI | 0 | 7 |
T. mexicana 6 (3.53%) y T. spp. 1 (0.59%) |
| Porcentajes | 43 | 1.76% | 11.17% (p=0.0004) | TcI | 19.41% | 80.58% |
|
En este estudio observamos que el PCR tiene mayor sensibilidad que la microscopía óptica en la detección de parásitos en heces de los triatominos (p= 0.0004) (Cuadro 1). La frecuencia de infección por T. cruzi encontrada entre las muestras de triatominos analizadas fue de 1.76% (3/170) por la técnica de microscopía óptica y el 11.17% (19/170) por PCR. Se observa una diferencia de la sensibilidad entre las dos técnicas de diagnóstico utilizadas era de esperarse, ya que previamente ha sido demostrado que la técnica de PCR es más sensible que la microscópica para la detección de T. cruzi a partir de sangre (Kirchhoff et al., 1996) y de heces de chinches (Almeida et al., 2008; Lardeux et al., 2016).
Aunque el diagnóstico por PCR es más sensible que el microscópico, es posible que aún por PCR la prevalencia de infección por T. cruzi en triatominos encontrada en el presente trabajo esté siendo subestimada, ya que ha sido reportado que dependiendo del tipo de marcador molecular utilizado en la prueba de PCR, la capacidad de detección puede cambiar (Lardeux et al., 2016; Almeida et al., 2008). De ahí que futuros estudios deberían determinar que marcadores son los más adecuados para hacer más sensible y confiable la técnica de PCR para la detección de infecciones por T. cruzi en insectos vectores. El promedio de chinches infectadas encontrado por el método microscópico en este estudio en el municipio de Metztitlán (20%) es aparentemente mucho mayor que lo reportado en un estudio previo para el mismo municipio (7.7%) (Becerril et al., 2010; Castro-Tenorio et al., 2017). Sin embargo, en el presente reporte sólo se analizaron 5 chinches en Metztitlán, y la diferencia en la prevalencia de infección por T. cruzi en las chinches estudiadas en ese municipio, podría tener más que ver con un problema del tamaño de muestra que con una diferencia real en prevalencia. La prevalencia general de triatominos infectados encontrada en el presente estudio, aunque no es alta en comparación con otras regiones de México, como la que se ha reportado en la región sur del Estado de México (aproximadamente 30%; Estrada-Franco et al., 2006, Medina-Torres et al., 2016, López Vivas et al., 2018), debe ser tomada en cuenta por las autoridades sanitarias del estado de Hidalgo en su esfuerzo por prevenir y controlar el riesgo de infecciones por T. cruzi en humanos.
En cuanto a la genotipificación de T. cruzi por la amplificación de la región intergénica del gen mini exón, se encontró únicamente el biotipo TcI en las tres especies de triatominos colectados (Figura 1). Hasta donde sabemos, éste es el primer reporte de tipificación molecular de T. cruzi en el Estado de Hidalgo. Estudios previos en esta área geográfica, sólo se han reportado los porcentajes de prevalencia que varían de entre 1.5% al 8.21% en humanos seropositivos a T. cruzi y la prevalencia de infección en cuatro especies de triatominos endémicos de la región: T. barberi (Usinger), T. dimidiata (Latreille), T. mexicana (Herrich-Schaeffer) y T. gerstaeckeri (Stål) (Velasco et al., 1992; Gómez et al., 2006). En estudios previos se ha reportado la presencia de los seis linajes (TcI-TcVI) en México, mediante el análisis de la secuencia del gen mini-exón del T. cruzi. Pero los estudios de epidemiología molecular de T. cruzi son incipientes en nuestro país. El linaje más reportado y por lo tanto mejor conocido es el TcI, que se ha encontrado en humanos, en diversos mamíferos y en varias especies de triatominos infectados. En el caso de los linajes TcII-TcVI existen pocos estudios en México, y en estos se ha reportado la presencia de los diferentes linajes de T. cruzi en heces de varias especies vectoras (Triatoma dimidiata, Triatoma longipennis y Meccus pallidipennis) distribuidas en varias áreas geográficas de la República Mexicana (Ramos-Ligonio et al., 2012; Ibáñez-Cervantes et al., 2013; López-Vivas et al., 2018). Sin embargo, hace falta hacer estudios más minuciosos acerca de la epidemiología molecular de T. cruzi en humanos ya que estos linajes no se han reportado en pacientes chagásicos en México (Salazar-Schettino et al., 2016). Por lo que queda como una asignatura pendiente, el determinar la frecuencia con la que los diferentes linajes de T. cruzi encontrados en México están produciendo infecciones en humanos. Los datos del presente reporte sugieren que las infecciones por T. cruzi en humanos en el estado de Hidalgo, deben estar siendo causadas principalmente por parásitos del linaje TcI. Esto es relevante dada la resistencia que ha sido encontrada en parásitos de este biotipo a los fármacos utilizados terapéuticamente para combatir la infección. De ahí que se recomienda seguir buscando alternativas terapéuticas y métodos de prevención y control de la enfermedad más efectivos, si se pretenden reducir los riesgos a la salud pública ocasionados por T. cruzi tanto a nivel regional como global.
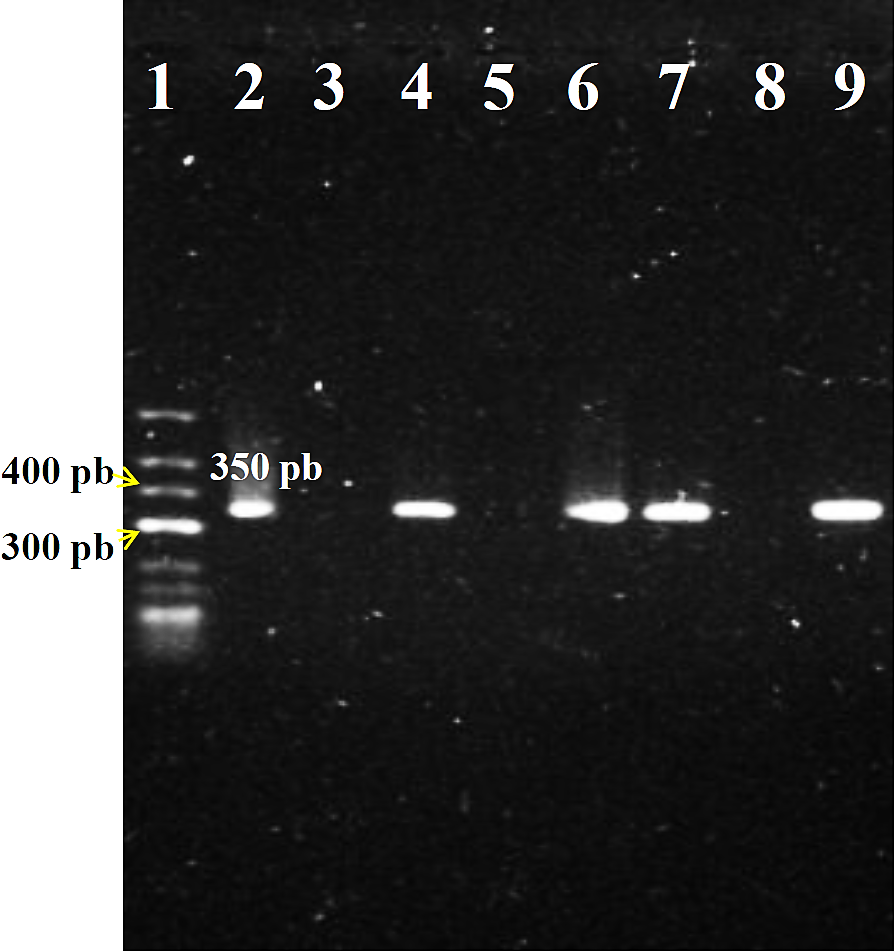
Figura 1 Amplificación de los fragmentos específicos del gen mini-exón (PCR-mul) con iniciadores reportados por Souto et al. 1996. En la figura la línea 1 corresponde al: MPM (Low DNA Mass ladder, Invitrogen™); línea 2: Control positivo T. cruzi Sylvio X10/4 (TcI); línea 3: Control negativo (H20); líneas 4 a la 9, corresponde a las muestras 1 - 6
Conclusiones
Existe una frecuencia del 11.17 % (19/ 170 muestras) de triatominos infectados por T. cruzi a través de la técnica molecular de PCR multiplex que amplifica al gene mini-exón. Se encontró el biotipo TcI de T. cruzi en tres especies de insectos hematófagos; T. dimidiata, T. mexicana y T. gerstaeckeri. Los datos sugieren que existe una circulación activa del linaje TcI en el ecotopo peridoméstico en 10 municipios del Estado de Hidalgo y abre un riesgo importante de infección en humanos, que habitan en estas zonas endémicas.











 nova página do texto(beta)
nova página do texto(beta)


