Introducción
El fósforo (P) es uno de los principales nutrientes que necesitan las plantas para su crecimiento. Generalmente, los suelos contienen altas cantidades de P inorgánico y orgánico; el primero corresponde a las hidroxiapatitas y fluoroapatitas, mientras que el segundo se encuentra como ácido fítico o fitato en los residuos animales y vegetales. Sin embargo, el P presente en los compuestos orgánicos no es aprovechado por las plantas (Mengel et al., 2001a). Los microorganismos juegan un papel importante en la solubilización del P. Algunas bacterias y hongos tienen la capacidad de producir enzimas, las cuales son las encargadas de liberar el ion ortofosfato del ácido fítico, compuesto soluble para la planta (Sharma et al., 2013). En el caso del fósforo inorgánico, algunos microorganismos producen y liberan ácidos orgánicos al suelo capaces de disolver el P mineral, como resultado del intercambio aniónico de fosfato por el anión ácido o pueden quelar iones de Fe3+, Al3+ y Ca2+ asociados con P (Omar, 1997). Actualmente, se ha logrado estudiar a fondo diversos mecanismos de solubilización de P. Se sabe que dichos mecanismos, en los cuales actúan los microorganismos, dependen de las limitaciones de nutrimentos que podrían llegar a presentarse y del estrés ambiental al que se sometan (Liu et al., 1992; Antelman et al., 2000).
Con las nuevas tecnologías como la proteómica, se puede identificar, caracterizar y determinar la función de las proteínas dentro de la célula y obtener un amplio panorama del funcionamiento de un microorganismo (Cahill et al., 2001).
En el presente trabajo se hace uso de la proteómica para identificar las proteínas implicadas en la solubilización de fosfatos inorgánicos por bacterias nativas de La Comarca Lagunera.
Método
Evaluación de fuentes de C y N para la solubilización de Pi en medio sólido PVK
Se trabajó con las cepas SP1, SP2, SP3, SP4, SP6, SP7, SP8 y SP9, aisladas previamente de cultivos forrajeros de La Comarca Lagunera (datos no publicados) y conservadas a -20 °C en glicerol al 25 %. Se preparó un inóculo con las cepas en caldo nutritivo a 30 °C, 150 rpm por 24 horas. Se usó el medio sólido Pikovskaya (PVK) con la siguiente composición en gramos por litro: glucosa, 10; Ca3(PO4)2, 5; (NH4)2SO4, 0.5; NaCl, 0.2; MgSO4·7H2O, 0.1; KCl, 0.2; extracto de levadura, 0.5; MnSO4·H2O, 0.002; FeSO4·7H2O, 0.002, agar bacteriológico, 15. Se utilizaron diferentes fuentes de C, N y Pi en este medio y se realizó un cálculo para tener la misma cantidad de estos elementos en los medios de cultivo, independientemente de su fuente (Tabla 1). El inóculo se ajustó a una densidad óptica de 0.5 a 600 nm y se inocularon 5 μL de éste en las cajas conteniendo el medio sólido PVK. Las cajas se incubaron a 30 °C por 72 horas, los ensayos se realizaron por triplicado. A los tres días se determinó el Indicé de Solubilización (IS) relacionando el diámetro total (colonia + el halo)/el diámetro de la colonia.
Evaluación de fuentes de C y N para la solubilización de Pi en medio líquido PVK
Los inóculos se prepararon bajo las condiciones descritas anteriormente, se utilizaron 5 mL para inocular matraces de 125 mL conteniendo 25 mL del medio líquido PVK con diferentes fuentes de C, N y P. Los cultivos se incubaron a 30 °C, 150 rpm por 72 horas, el ensayo se realizó por triplicado. Se determinó el contenido de fosfato soluble en medio líquido mediante la metodología reportada en la Norma NMX-AA-029-SCFI-2001.
Identificación molecular
Se realizó la amplificación de un fragmento de 400 pares de bases del gen 16S rDNA de una muestra de DNA extraído. Los primers utilizados fueron 16S f 5’ -AGGAGGTGATCCAACCGCA - 3’ y 16S r 5’ AACTGGAGGAAGGTGGGGAT - 3’. Se utilizaron 14.5 µL de agua destilada estéril, 3.5 µL de Buffer 10X/MgCl2 50 mM, 0.5 µL dNTP’s 10 mM, 2 µL primers f y r 10 mM, 0.5 µL de Taq polimerasa paq5000 (Agilent Technologies) y 2 µL de DNA 100 ng µL-1 para tener un volumen total de reacción de 25 µL. La reacción se llevó a cabo en termociclador Axigen Maxigen® bajo el siguiente programa: 1 ciclo de desnaturalización a 95 °C por 5 minutos, 35 ciclos de desnaturalización a 94 °C por 1 minuto, alineamiento a 54 °C por 1 minuto y elongación a 72 °C por 5 minutos y una temperatura final de 4 °C.
La secuenciación de los productos amplificados se realizó en un secuenciador automático (Perkin Elmer modelo 3730 Applied Biosystem®). Las secuencias obtenidas fueron alineadas y analizadas con la herramienta BLAST (Basic Logical Aligment Search Tool) de la base de datos National Center of Biotechnology Information (NCBI) del GenBank con secuencias altamente similares.
Estudios de expresión de proteínas en la cepa seleccionada
Cultivos bacterianos
Se realizaron cultivos de la cepa SP3 para estudiar la expresión de proteínas, esta cepa presentó mayor capacidad de solubilización de P inorgánico. Se preparó medio líquido PVK usando como fuente de C la glucosa, para el N urea y para P se utilizaron fosfato de calcio y fosfato de aluminio. El inóculo se preparó bajo las condiciones descritas anteriormente y se utilizaron 5 mL para inocular matraces de 250 mL conteniendo 70 mL del medio PVK. Los cultivos se realizaron por duplicado y se incubaron a 30 °C, 150 rpm por 72 horas. Se realizó la medición de pH al inicio y al final de la fermentación y se determinó la concentración de azúcares reductores por DNS.
Precipitación y cuantificación de proteína
La precipitación de proteínas se llevó a cabo con TCA al 100 %. A una muestra de 30 mL del cultivo bacteriano se le agregó el mismo volumen de TCA, la mezcla se homogenizó y se dejó precipitar toda la noche a -20 °C y después fue centrifugada a 10 000 rpm durante 15 min a 4 °C. El sobrenadante fue retirado para lavar la pastilla obtenida con 500µL de acetona fría (cuatro veces), se dejó secar la pastilla por 3 horas a 7°C y se resuspendió en 1 mL de buffer Tris-HCL pH 7 con 2% SDS.
Para la cuantificación de proteína se utilizó el BCATM Protein assay kit (Pierce®), con Albumina Bovina como estándar (SIGMA®).
Geles de poliacrilamida
Se cargaron 50 μg de proteína de cada una de las muestras en un gel de poliacrilamida al 12% ((Bolt 12% Bis-Tris Plus, Thermo Fisher Scientific®) y se realizó la electroforesis a 95 V. Los geles se tiñeron con Azul de Comassie y se lavaron con agua destilada.
Cada banda del gel se cortó en tres secciones iguales, cada sección se cortó en piezas de aproximadamente 1 mm y se pasaron a tubos Eppendorf, se destiñó el gel con una solución de metanol 50 %/ácido acético 5 %, se hicieron lavados cada 30 min hasta que los geles se destiñeron por completo.
In-gel Digestión de proteínas
Día 1. Lavados
Las muestras se lavaron con H2O destilada durante 5 minutos y se decantó el agua, se adicionaron 125 µL de solución 50/50 acetonitrilo (ACN) 60%/H2O para realizar un lavado por 5 minutos y retirar el reactivo. Este procedimiento se repitió una vez más. Se agregaron 125 µL de Ambic (bicarbonato de amonio al 100 mM) y se lavó por 5 min, después se adicionaron 125 µL de ACN (100%) y se lavó durante 5 min más, se retiraron ambas soluciones, se repitió el lavado con ACN, se retiró y se dejaron secando las muestras en una centrifuga de vacío.
Día 2. Alquilación y reducción
Se adicionaron 125 µL de DTT (1,4 Dithiothreitol 10 mM preparado en AMBIC) a las muestras y se incubaron a 65 °C por 1 hora. Las muestras se dejaron enfriar y se descartó la solución. Se agregaron 125 µL de IDA (iodoacetamida 55 mM preparado en AMBIC) y se incubó en la oscuridad por 1 hora y se decantó la solución.
Se realizó un lavado con 125 µL de Ambic por 5 min, y se adicionaron 125 µL de ACN (100%) por 5 min, se descartó la parte líquida y se dejaron secar las muestras en la centrifuga de vació toda la noche.
Digestión por enzima tripsina
Se preparó una solución de tripsina 20 µg/mL Ambic. Se agregó esta solución en una proporción de 50:1 (w/w proteína tripsina). Se adicionaron 50 µL y 100 µL de AMBIC para cubrir completamente los geles, se dejó incubar a 37 °C por 18 horas.
Día 3. Extracción de péptidos
Después de la incubación de los péptidos, se agregaron 150 µL de buffer de extracción {50% ACN, 50 % H2O, 0.1% ácido fólico (AF)}, se mezcló en el vórtex y se incubó por 10 min, se obtuvo el sobrenadante y se pasó a un tubo Eppendorf nuevo, este paso se realizó dos veces más y se dejaron secar las muestras toda la noche en la centrifuga de vacío. Las muestras se resuspendieron en 100 µL de buffer de extracción, se filtraron y se dejaron secar toda la noche en la centrifuga de vacío. Las muestras se almacenaron a -20 °C hasta su análisis.
Análisis por cromatografía líquida acoplada a espectrometría de masas
Los péptidos se resuspendieron en 19 µL del buffer A (ácido fórmico al 0.1 %) y 1 µL del buffer B (80 % de acetonitrilo y 0.1 % de ácido fórmico). Las muestras se pasaron a través de un filtro de 0.2 μm (Nanosep, Pall Corp®) antes de cargarse en un tubo de muestreador automático y se insertaron en un sistema Ultimate 3000 LC (Thermo Scientific - Dionex®). El análisis LC-MS/MS se realizó en un Orbitrap Fusion Tribrid (Thermo Scientific®) utilizando una fuente de ionización de nanopulverización. Para cada muestra, se inyectaron 6 µl y los péptidos se separaron mediante un gradiente de 90 minutos de buffer B creciente, a un caudal de aproximadamente 200 µL por minuto. Se recolectó el espectro de masa completo cada tres segundos, se fragmentaron continuamente los iones más intensos con 38 % de disociación inducida por colisión (CID) y se registraron los espectros MS/MS resultantes. La exclusión dinámica se utilizó para excluir iones precursores del proceso de selección durante 60 segundos después de una segunda selección dentro de una ventana de 10 segundos.
Búsqueda de bases de datos e identificación de proteína
Los espectros de masas tándem se convirtieron en archivos genéticos de Mascot, utilizando el procesador MSConvert (http://proteowizard.sourceforge.net/tools.shtml). Se realizaron búsquedas en los archivos MGF utilizando el programa Mascot (Matrix Scientific, Boston, MA). Para la identificación de proteínas, se utilizó una base de datos, en noviembre del 2017, del National Center for Biotechnology Information (NCBI) específica para K. aerogenes.
La base de datos reversa se generó invirtiendo todas las secuencias de las proteínas de la base de datos de destino, con la verificación de que todas las secuencias invertidas creadas no coincidieran con las secuencias presentes en la base de datos de destino. Los parámetros del Mascot fueron los siguientes: segmentaciones enzimáticas trípticas, que permiten hasta dos divisiones omitidas, tolerancia a péptidos de 20 ppm, tolerancia iónica a fragmentos de 0.8 Da, modificación fija debida a carboxiamidometilación de cisteína (+57 Da), modificaciones variables de oxidación de metionina (+16 Da) y desamidación de asparagina o glutamina (+0.98 Da). Se determinaron proteínas estadísticamente significativas para todas las muestras.
Anotación funcional de las proteínas
La búsqueda de la función de las proteínas se realizó en los sitios web http://pfam.xfam.org/ y http://www.uniprot.org/ utilizando los archivos fasta de la base de datos para K. aerogenes desarrollada en noviembre del 2017.
Análisis estadístico
Se realizó un ANOVA simple para comparar los resultados de la cuantificación del fosfato inorgánico solubilizado y el índice de solubilización de las cepas y en los casos donde hubo diferencia significativa se realizó una prueba de LSD de Fisher con un α=0.05. Se utilizó el programa Statgraphics centurión.
Resultados
Evaluación de fuentes de C y N para la solubilización de Pi en medio sólido PVK
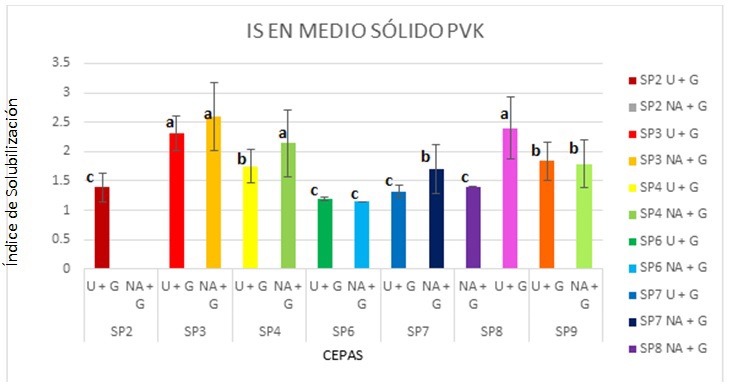
Cuando la glucosa, la urea, el nitrato de amonio y el fosfato de calcio estaban presente en el medio de crecimiento como fuente C, N y P, se observó la presencia de halos de solubilización (Tabla 2). La cepa SP1 fue la única que no presentó halo de solubilización por lo que fue descartada, la cepa SP2 solo presentó halo en el medio de crecimiento enriquecido con urea y glucosa (Figura 1). El ANOVA donde se compararon cepas y medios de crecimiento, arrojó que para ambos factores hubo diferencia significativa (Figura 2).
Tabla 2 Índice de solubilización (IS) en medio sólido PVK. La expresión NA aplica donde no hubo formación de halos de solubilización. ANOVA y prueba LSD de Fisher con un α=0.05.
| Cepa | Índice de solubilización | |
|---|---|---|
| Urea y glucosa | Nitrato de amonio y glucosa |
|
| SP1 | NA | NA |
| SP2 | 1.39c | NA |
| SP3 | 2.31a | 2.59a |
| SP4 | 1.75b | 2.13a |
| SP6 | 1.19c | 1.14c |
| SP7 | 1.32c | 1.7b |
| SP8 | 1.7c | 2.13a |
| SP9 | 1.83b | 1.79b |
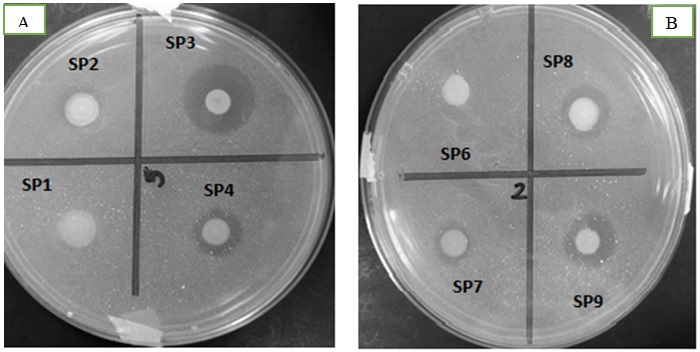
Figura 1 Halos de solubilización de fosfato de calcio en medio sólido. 1A. Medio con fuente de C-N glucosa y urea respectivamente. 1B. Medio con Fuente de C-N glucosa y NA respectivamente
Evaluación de fuentes de C y N para la solubilización de Pi en medio líquido PVK
Para esta metodología solo se analizaron las cepas que presentaron mayor IS en medio sólido PVK, la cuales fueron la SP3 (2.31 y 2.59), SP4 (1.75 y 2.13), SP8 (1.7 y 2.13) y SP9 (1.83 y 1.79) en medios de crecimiento urea-glucosa-fosfato de calcio y nitrato de amonio-glucosa-fosfato de calcio, respectivamente.
Comparando los valores de solubilización arrojados en ambos tipos de fosfato, la cepa SP3 fue la que presentó hasta 1.97 ppm, siendo la mayor cantidad de fosfato de calcio solubilizada en el medio U+G, seguida de la SP9 con un valor de 1.88 ppm en dicho medio. En el caso del medio NA+G, la máxima solubilización de P se obtuvo con la cepa SP9, alcanzando un valor de 1.85 ppm, seguida de la cepa SP3 con un valor de 1.74 ppm. Para el FA nuevamente la cepa SP3 alcanzó el valor más alto con 1.36 ppm, seguida de la SP8 con 0.79 ppm en el medio U+G. Para el medio NA+G SP9 y SP8 obtuvieron valores de 0.94 ppm y 0.55 ppm respectivamente, siendo los valores más altos (Tabla 3).
Tabla 3 Cuantificación del Pi solubilizado en medio líquido PVK. Glucosa (G) como fuente de C, urea (U) y nitrato de amonio (NA) como fuentes de N. ANOVA y prueba LSD de Fisher con un α=0.05.
| Cepa | Fosfato de calcio (ppm) |
Fosfato de aluminio (ppm) |
||
|---|---|---|---|---|
| U+G | NA+G | U+G | NA+G | |
| SP3 | 1.97a | 1.74a | 1.36b | 0.53c |
| SP4 | 0.61c | 0.23d | 0.77c | 0.20d |
| SP8 | 1.28b | 1.00b | 0.79c | 0.55c |
| SP9 | 1.88a | 1.85a | 0.66c | 0.94b |
El ANOVA donde se compararon cepas y medios (Glucosa-Urea y Nitrato de amonio-Glucosa con fosfato de calcio y fosfato de aluminio arrojó que para ambos factores hubo diferencia significativa (Figura 3)
Identificación molecular
Debido a sus altos valores de solubilización, la cepa SP3 fue seleccionada para su identificación molecular y posteriores estudios de proteómica. La comparación del fragmento de ADN, obtenido por PCR y secuenciado, con la base de datos del NCBI arrojo un valor positivo para la bacteria Klebsiella aerogenes (Figura 4)
Estudios de expresión de proteínas en la cepa seleccionada
Medición de pH y azúcares reductores en cultivos bacterianos
Se monitoreó el pH al principio y al final de los cultivos bacterianos (Tabla 4), se observa en ésta que en los medios que contenían FC no hubo un decremento significativo en el pH, caso contrario al medio que contenía FA como fuente de P, lo anterior se puede atribuir a una mayor presencia de ácidos orgánicos en el medio. Además se midieron azúcares reductores, se alcanzaron valores de hasta 96 y 95.5 % de consumo de glucosa para los cultivos en FA y FC respectivamente.
Identificación y anotación funcional de proteínas
Se estudió la influencia de la fuente de P inorgánico en la expresión de proteínas en K. aerogenes, considerada como Bacteria Solubilizadora de Fosfato (BSF). El fosfato de calcio y fosfato de aluminio fueron las únicas fuentes en los cultivos bacterianos. Se lograron identificar un total de doscientos dieciséis proteínas. Bajo las condiciones de experimentación, no se detectaron proteínas únicas para los cultivos en FC o FA. La sobreexpresión de proteínas sólo se consideró cuando se obtuvieron valores mayores a 2. La tabla 5 muestra las proteínas identificadas involucradas en el aprovechamiento del P, como las ABC transportadores. Para este caso, no se observó un cambio de diferencia en la expresión mayor a 2. Además, se observó la presencia de la proteína histidina quinasa Pho y proteína del sistema de transporte específico de fosfato Pho U con un cambio de diferencia (fold) de 1.10 el cual no se considera significativo.
Tabla 5 Proteínas ABC transportadoras transmembrana registradas y su abundancia en el cultivo celular. El incremento corresponde a la cantidad de veces que se encuentra la proteína en cada condición, se considera significativo si el cambio de diferencia es ›2 (2 fold). NA (no aplica) cuando se encuentra en la misma cantidad en ambas condiciones. Fosfato de calcio (FA) y Fosfato de Calcio (FC).
| Nombre de la proteína | Incremento | Función | Péptidos identificados |
|
|---|---|---|---|---|
| FA | FC | |||
| Proteína de unión al sustrato del transportador ABC de galactosa | 1.04 | Transportar la galactosa a través de una bicapa lipídica | 37 | |
| Proteína de unión al sustrato del transportador de oligopéptidos ABC OppA | 1.16 | Transporte transmembrana | 35 | |
| Glutamato / aspartato ABC transportador sustrato proteína de unión | NA | NA | Permite el movimiento dirigido de sustancias hacia dentro y/o fuera de la célula | 28 |
| Proteína de unión al sustrato del transportador ABC del aminoácido de cadena ramificada | 1.70 | Proteínas de unión periplasmáticas | 20 | |
| Proteína de unión al sustrato transportadora de cistina ABC | NA | NA | Catálisis de la transferencia transmembrana de un ion | 26 |
| Proteína transportadora de sustrato del transportador ABC | 1.19 | Proteínas de unión a soluto extracelulares | 14 | |
| Spermidine / putrescine transportador de ABC proteína de unión al sustrato PotF | 1.31 | Proteínas de unión a soluto extracelulares | 14 | |
| Proteína transportadora de sustrato del transportador ABC | NA | NA | Transporte | 21 |
| Transportador de ABC de azúcar proteína de unión a ATP | 1.2307 | Dominio soluble en agua de transportadores ABC transmembrana | 25 | |
| Proteína de unión al sustrato del transportador de histidina ABC HisJ | 1.91 | Transporte | 10 | |
| D-ribosa ABC transportador sustrato-proteína de unión | 1.58 | Catálisis de la transferencia de un grupo fosfato | 12 | |
| Transportador de Xylose ABC Proteína de unión a ATP | NA | NA | Exportación o importación de sustratos que van desde iones pequeños hasta macromoléculas | 14 |
| Proteína de unión al sustrato del transportador de metionina ABC MetQ | 1.11 | Pueden estar involucrados en la virulencia bacteriana | 9 | |
Además, se lograron identificar proteínas que participan en rutas metabólicas de las cuales se obtiene la mayor producción de ácidos orgánicos, como se comentó con anterioridad estos ácidos orgánicos son uno de los principales mecanismos de solubilización de Pi que emplean las bacterias (Tabla 6).
Tabla 6 Proteínas identificadas en los cultivos bacterianos que participan en rutas metabólicas.
| Nombre de la proteína | Función | Incremento | Péptidos identificados |
|
|---|---|---|---|---|
| F. A. | F.C. | |||
| aldehído deshidrogenasa | Glicólisis | 1.07 | 33 | |
| subunidad grande de carbamoil fosfato sintasa | Ciclo de la urea | 1.06 | 45 | |
| malato deshidrogenasa | Ciclo del ácido cítrico | 1.13 | 13 | |
| complejo piruvato deshidrogenasa dihydrolipoyllysine-residuo acetiltransferasa | Oxidación anaerobia del piruvato | 1.17 | 24 | |
| piruvato oxidasa | 1.04 | 31 | ||
| fosfoglicerato quinasa | Glucólisis | 1.42 | 21 | |
| 6-fosfofructoquinasa dependiente de ATP | 1.21 | 21 | ||
| succinato - subunidad beta CoA ligasa | Ciclo de krebs | 1.16 | 17 | |
| succinato - subunidad alfa de la CoA alfa | 1.28 | 10 | ||
| NAD (P) oxidorreductasa dependiente | Ciclo de beta-oxidación. | 1.05 | 9 | |
| beta-cetoacil-ACP reductasa | ácidos grasos | 1.3 | 7 | |
| clase II fructosa-bisfosfato aldolasa | Glucólisis, gluconeogénesis y el ciclo de Calvin | 1.2 | 13 | |
Discusión y Conclusiones
La glucosa como única fuente de carbono en el medio de crecimiento, propició una mayor solubilización. Rubio et al., (2016) y Relwani, et al., (2008), estudiaron la influencia de las fuentes de C y N en la solubilización de fosfato de calcio en los hongos Talaromyces flavus (BAFC 3125) y Aspergillus tubingensis, respectivamente. Los primeros autores concluyen que la presencia de glucosa aumentó la producción de ácidos orgánicos, especialmente ácido glucónico, el cual es considerado uno de los principales ácidos solubilizadores de P. Relwani et al., (2008), reportaron que A. tubingensis fue capaz de degradar todas las fuentes de C, sin embargo, la glucosa y sacarosa promovieron significativamente la solubilización de P. Con respecto a la fuente de N, la urea arrojó valores altos de solubilización en el medio de crecimiento, caso contrario a lo observado por Rubio et al. (2016), quienes determinaron que los tratamientos que contenían NA y SA presentaban una mayor solubilización, así mismo lo reportaron Relwani et al., (2008), cuando el KNO3 se encontraba en el medio de crecimiento.
En el presente estudio se encontraron las proteínas ABC transportadoras transmembrana (Tabla 5) responsables de adquirir y transportar el fosfato. Pegos et al., (2014) reportaron la presencia de estas proteínas cuando la cepa Xanthomonas citri se encontraba bajo condiciones de estrés (falta de P). Se concluye que las proteínas ABC transportadoras pertenecen al sistema de privación del fosfato {phosphate starvation system (Pst)}, el cual es uno de los mecanismos que activan las bacterias para mantener el control de la homeostasis del fosfato, se activa cuando la bacteria se encuentra bajo estrés ambiental (falta de nutrimentos) o si el suelo contiene diversos tipos de fosfato (Rouached et al., 2010).
Además, se encontró la proteína histidina quinasa PhoR, la cual es uno de los dos componentes del sistema regulador del regulon Pho y desempeña un papel clave en la homeostasis del P. La función de dicha enzima es interactuar con el sistema de transporte específico de fosfato (proteínas ABC transportadoras) y una proteína llamada PhoU para detectar los niveles de P inorgánico ambiental (Lamarche, et al., 2008).
Se concluye que las fuentes de Carbono y Nitrógeno influyen en la capacidad de solubilización de fosfato inorgánico en bacterias nativas de La Comarca Lagunera. Los mejores resultados de solubilización en fosfato inorgánico se presentaron cuando la glucosa se encontraba en el medio como única fuente de carbono y la urea como fuente de nitrógeno. Se clasifica a las bacterias nativas de La Comarca Lagunera como bacterias solubilizadoras de fosfato porque mostraron tener la capacidad de solubilizar fosfato de calcio y aluminio. K. aerogenes resultó ser la cepa bacteriana que presentó una mayor capacidad para solubilizar fosfato de calcio (2.31 cm) en medio sólido PVK y fosfato de calcio (1.97 ppm) y fosfato de aluminio (1.36 ppm) en medio líquido PVK. Bajo las condiciones experimentales de este estudio, no se identificaron proteínas únicas de K. aerogenes para solubilizar el fosfato de calcio y fosfato de aluminio, la bacteria solo varió su nivel de expresión de proteínas en cada una de las condiciones estudiadas.











 nueva página del texto (beta)
nueva página del texto (beta)