Introducción
La Comarca Lagunera, ubicada en los estados de Coahuila y Durango es la cuenca lechera más importante de México, concentra la mayor parte del inventario bovino lechero del país, con un promedio de 464,086 cabezas de ganado que representan alrededor de 20% del hato nacional (SAGARPA, 2015). Dada la importancia que los bovinos de leche representan para la región, la selección temprana de vaquillas con alto potencial lechero representaría un ahorro por conceptos de alimentación y cuidados sanitarios.
En la producción de leche influyen factores como la raza, edad, estado sanitario, condición fisiológica y del ambiente, y en todos los casos están influenciadas por los genes, que en un alto porcentaje determinan el comportamiento productivo y reproductivo del animal (Vallone et al., 2014,41; Vásquez et al., 2007, 60; Thomas et al., 2002,757). La producción de leche es una variable continua y está determinada en parte por las enzimas que participan en la ruta metabólica. Cada una de estas enzimas es codificada por un gen específico cuya expresión y variante (alelo) es determinante en la producción y calidad de la leche. En los programas de mejoramiento genético tradicional se busca identificar aquellos animales que portan genes (alelos) que mejoran esta característica, aunque no necesariamente se conozca cuál es ese gen o su forma (alelo).
Los estudios realizados en ganado bovino a nivel molecular han permitido estudiar características productivas de gran importancia para el mejoramiento animal, y han llevado a la búsqueda de marcadores genéticos para la identificación de genes asociados a caracteres de interés (Martínez et al., 2012, 294; Piñeira et al., 2012, 45; White et al., 2005, 2001). Entre los genes relacionados con la producción y calidad de leche, se encuentra el gen Ob también llamado gen leptina. El producto de este gen es la hormona proteica leptina de 16-KDa, compuesta por 146 aminoácidos, secretada por los adipocitos a la sangre y desempeña funciones importantes en el control del peso corporal, la deposición de grasa, consumo de alimento, la función inmune, y la reproducción en ganado. (Javanmard et al., 2010, 10; Quintero y Ruíz, 2008, 98; Nkrumah et al., 2005, 26; Huang et al., 2014, 11997).
Se han descrito varios polimorfismos (SNP, Single Nucleotide Polymorphism) en el exón dos del gen Ob (Yoo et al., 2005, 1551). Uno de estos polimorfismos es la sustitución de citosina(C) por timina (T), que a su vez conduce al remplazo del aminoácido arginina por cisteína en la proteína leptina, originando una modificación en la estructura de la proteína que bloquea la capacidad para ser identificada por los receptores en el hipotálamo, suprimiendo la sensación de saciedad del animal, por lo que consumen más alimento, reflejándose en un aumento de producción de leche entre otras características (Cerón-Muñoz et al., 2009, 372).
Este polimorfismo genera dos alelos, el T y el C, siendo el alelo T el mutado y el C el alelo normal, asociados respectivamente a la leptina modificada y leptina común. El alelo T también se ha asociado con el mayor contenido de grasa en la canal (Dias et al., 2007, 4; Souza et al., 2010, 440; Cerón-Muñoz et al., 2009, 379).
Como el animal recibe sólo un alelo por par de genes de cada progenitor, en relación con el gen Ob el genotipo de las vacas puede ser: CC (ambos alelos codifican la leptina común); TT (ambos alelos codifican la leptina modificada) y CT (cada alelo codifica un tipo de leptina). En su comportamiento, los animales con genotipo CC son más lentos para engordar, comen menos durante el pico de lactancia y producen menos leche. Los animales con genotipo TT producen más leche, de mejor calidad y canales con mayor marmoleo (Montoya et al., 2009, 43). Los tres genotipos para la producción de leptina han sido encontrados en todas las razas bovinas, pero en diferentes proporciones (Corva et al., 2007, 243).
Debido a que el gen de Ob y sus polimorfismos representan una función muy importante en los sistemas de producción y calidad de leche, el uso de este gen como marcador genético podría ser una opción eficiente para selección de becerras en edad temprana, y se asume que conociendo la condición genética del animal, se puede conocer el tipo de leptina que produce y por lo tanto ser un indicador de cuál será su potencial productivo y de calidad de la leche desde su nacimiento.
En este contexto, el objetivo de esta investigación fue determinar la relación existente entre los genotipos generados (CC, CT, TT) por el polimorfismo del exón 2 del gene Ob y las características de producción y calidad de la leche en vacas Holstein en lactancia.
Método
Selección de animales
Para el estudio se seleccionaron 100 vacas de raza Holstein en servicio activo con 2 a 7 lactancias y con 60 a 70 días posparto, de tres grupos de producción de leche: 34 altas productoras (µ = 43.42 litros/día ± 4.3 L), 31 medias productoras (µ = 33.47 litros/día ± 1.4 L), 35 bajas productoras (µ = 24.68 litros/día ± 2.8 L). Todas las vacas pertenecían al establo “Lácteos Florida” ubicado en el municipio Francisco I. Madero Coahuila, México.
Obtención de muestras de sangre
De las vacas seleccionadas se recolectaron muestras de sangre de la arteria coccígea en tubos vacutainer con anticoagulante EDTA y las muestras se transportaron inmediatamente al laboratorio en un congelador y almacenadas a -20 °C para la posterior extracción de ADN en el laboratorio de Biotecnología de la Facultad de Agricultura y Zootecnia, ubicada en el ejido Venecia municipio de Gómez Palacio, México.
Extracción de DNA
La extracción de ADN a partir de la sangre de las vacas se realizó mediante la metodología Saltin-Out (Rivera et al., 2010, 5), con algunas modificaciones. Se tomaron 300 μL de sangre y se colocaron en tubos eppendorf de 1.5 ml; enseguida se agregaron 540 μL de solución lisis de glóbulos rojos (Tris-HCl 10 mM pH 8, TritonX-100 1% y Sacarosa 11%), luego se centrifugó a 1200 rpm durante 3 min y se descartó el sobrenadante, este paso se realizó 3 veces hasta que se lavó perfectamente de las células anucleadas. Posteriormente se añadieron 180 μL de la solución lisis de glóbulos blancos (Tris-HCl 10 mM pH 8, NaCl 400 mM; EDTA 2 mM), más 30 μL de la solución de proteinasa K (Proteinasa K 20 mg/ml, SDS 1%, EDTA 2mM), más 6 μL SDS 20%, y la mezcla se agitó durante 5 minutos con vórtex hasta obtener la disolución completa de la pastilla. Enseguida se incubó a 65°C por 1 h agitándolo cada 10 min. Posteriormente se añadieron 30 μL de acetato de potasio 3M, se centrifugó a 1200 rpm por 10 min, y se recuperó el sobrenadante a un tubo eppendorf de 1.5 ml. Para conseguir la precipitación del ADN se agregaron 200 μL de isopropanol frío (-20°C) y se dejó reposar durante 30 min, transcurrido el tiempo se centrifugó a 1200 rpm por 10 min, y se descartó el sobrenadante para obtener el pellet; con el objetivo de conseguir mayor pureza de ADN se añadió 200 μL de etanol 70% frío (-20°C) y se centrifugó a 200 rpm por 5 min, se eliminó el sobrenadante, y el pellet se dejó secar a 37°c por 12 h en incubadora (Boekel Scientific; BOEKEL). Finalmente el pellet se resuspendió en 30 μL de agua libre ARNasas y se almacenó a -20°C para su uso posterior. El ADN obtenido se visualizó en gel de agarosa al 0.8%, y se evaluó la concentración y pureza del ADN por el método de espectrofotometría (NANODRP 1000, Thermo Scientific).
PCR y Digestión enzimática
Las reacciones de PCR se llevaron a cabo en un termociclador TC-512 (Tecchne). Los primers utilizados fueron: P2F: 5´-TAACGGAGCACGTGGGTG-3´ y P2R: 5´-GCCACGGTTCTACCTCGTCT-3´ (accesion GenBank: HE605298). Las reacciones de amplificación fueron las siguientes: se utilizó un volumen de 20 μL que contenía aproximadamente 50 ng de ADN, buffer de reacción 1X, 1.5 mM MgCl2, 0.02 mM dNTPs, 0.125 μM de cada uno de los primer, 1U de Taq DNA Polimerasa (BIOLINE®) y agua Milli-Q para completar el volumen final de 20 μL. El perfil térmico de amplificación de los productos de PCR consistió en lo siguiente: 95°C durante 1 minuto para desnaturalización inicial. Enseguida 35 ciclos con las siguientes temperaturas y tiempos: 95°C durante 30 segundos, 62°C durante 30 segundos para pegado de los primers, 72°C por 30 segundos para la generación del producto de PCR, y una extensión final a 72°C por 10 minutos. Los productos de PCR se corrieron en gel de agarosa 1.0 % durante 60 minutos a 86 voltios y se tiñeron con bromuro de etidio. Asimismo, los productos de PCR se sometieron a digestión con la enzima Aci I (Arthrobacter citreus) que reconoce el sitio de corte CCGC, a 37 °C durante 3 horas con la siguiente mezcla reactiva: 10 μl del producto de PCR, 2 μl del amortiguador de digestión, 1 μl enzima de restricción y 18 μl de agua desionizada. Los productos de digestión se observaron por electroforesis en gel de agarosa al 1.5 %, teñido con bromuro de etidio. Los genotipos se determinaron basados en la diferencia de tamaño de los fragmentos en el gel. Adicionalmente, se realizó una digestión “In Silico” con el software GENtle (V 2.0) para corroborar el número y tamaño de los fragmentos esperados y que éstos coincidieran con los productos esperados a partir de la digestión “In Vitro” del producto de PCR.
Identificación del polimorfismo
El producto de PCR obtenido a partir del gen Ob de las vacas de cada grupo de producción de leche se secuenció en el IPICYT (Instituto Potosino de Investigación Científica y Tecnológica), San Luis Potosí, S.L.P, México, con el método de didesoxinucleótidos marcados en el secuenciador automático 3130 Genetic Analyzer (Applied Biosystems). Las secuencias obtenidas se sometieron al análisis BLAST servidor NCBI (http://blast.ncbi.nlm.nih.gov/Blast.cgi) para detectar homología de las secuencias de interés con secuencias reportadas del gen Ob en el GenBank.
Muestras de leche
Se colectaron 50 ml en tubos falcon (Falcon®) de plástico de muestra de leche individual de cada una de las 100 vacas ya identificadas durante las ordeñas de la mañana y de la tarde. Esta actividad se realizó durante ocho semanas. Los tubos conteniendo las muestras de leche se identificaron con el número de la vaca, fecha de colecta y ordeña, e inmediatamente después de obtenidas, etiquetadas y selladas, las muestras se transportaron en hielo al laboratorio para su análisis dentro de las siguientes cuatro horas.
Análisis físico-químico de muestras de leche
Las muestras de leche se analizaron en el Laboratorio de lácteos de la Facultad de Agricultura y Zootecnia de la Universidad Juárez del Estado de Durango. Las muestras se calentaron a baño María hasta alcanzar 35ºC y mezcladas suavemente antes de ser analizadas en el equipo LactiCheck LC-02 (PAGE & PEDERSEN) el cual determina de forma automática los porcentajes de grasa, proteína y sólidos totales de la muestra.
Variables de producción y calidad evaluadas
En el estudio se realizaron registros diarios de producción de leche (PL) en litros, consumo de materia seca (CMS) en kilogramos, peso vivo del animal (PVA) en kilogramos. Las variables de calidad de leche porcentaje de grasa (PG), porcentaje de proteína (PP), contenido de solidos totales (ST), fueron determinadas a partir del análisis fisicoquímico realizados con el equipo Lacticheck LC-02 (PAGE & PEDERSEN).
Análisis estadístico
Se realizó un análisis de correlación de Spearman para determinar asociaciones entre las variables de producción y calidad de leche, además se llevó a cabo un análisis de regresión lineal para probar la dependencia de los componentes de producción y calidad de la leche con los genotipos de leptina. Se utilizó el siguiente modelo estadístico:
Dónde: Yijk valor observado para la variable de interés: producción de leche (PL), consumo de materia seca (CMS), peso vivo del animal (PVA), porcentaje de grasa (PG), porcentaje de proteína (PP), sólidos totales (ST); b0 es la media general de la población para las diferentes características; b1iNL es el coeficiente de regresión lineal de las variables dependientes sobre el número de lactancias; b2jDL es el coeficiente de regresión lineal de las variables dependientes sobre los días lactando; b3Gk es el efecto medio que se espera ocurra sobre b0 en función del genotipo leptina (k = TT, CT, CC), el cual se analizó como una variable Dummy; eijk es el efecto residual aleatorio. Los datos se analizaron en SAS (V 9.0), utilizando las opciónes GLM, correlación de Spearman y comparación de medias con la prueba diferencia mínima significativa (DMS).
Resultados
La extracción de ADN de las muestras de sangre se obtuvo en cantidad y calidad adecuada para poder realizar PCR. Los productos de PCR fueron visualizados por medio de electroforesis en gel de agarosa al 1.0 % mostrando que los primers diseñados fueron eficientes para generar a partir del gen Ob el producto de PCR esperado de 272 bp (Figura 1).
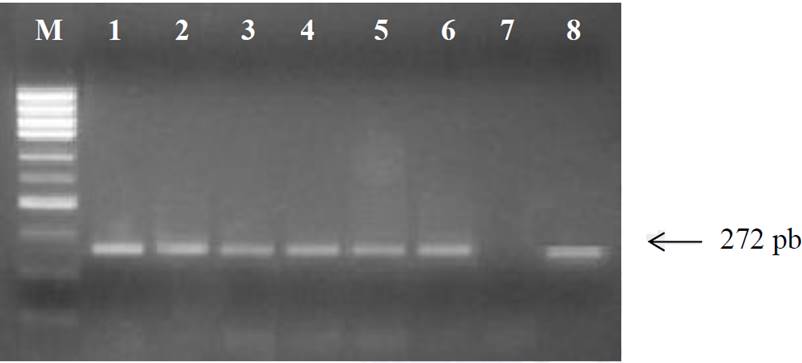
Figura 1 Productos de PCR a partir gen Ob con un fragmento de 272 pb de longitud. M: Marcador molecular de 1000 pb. Carriles 1-6 y 8 productos de PCR, carril 7: control negativo.
La digestión de los productos de PCR de 272 bp de longitud con la enzima de restricción Aci I generó tres patrones de restricción. El primer patrón de restricción consistió en el fragmento intacto de PCR de 272 bp, el cual no fue cortado por la enzima debido a que no presenta el sitio de corte ya que posee el polimorfismo o sustitución de citosina por timina C/T, y el cual está asociado con el genotipo homocigoto TT (Figura 2).
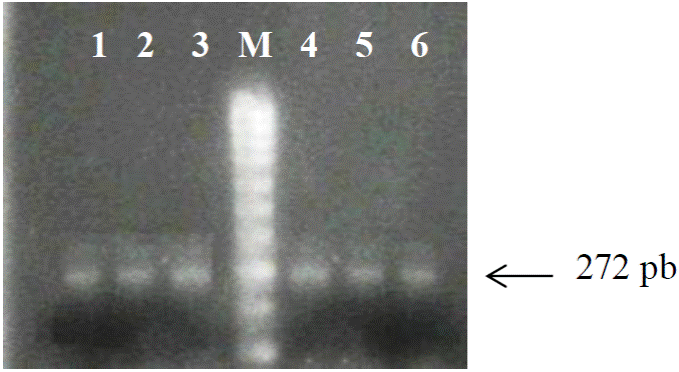
Figura 2 Patrón de restricción para el genotipo TT. Carriles 1-3, productos de PCR de 272 pb sin digerir. Carriles 4-6, mismos productos de PCR después de la digestión enzimática (enzima Aci I) mostrando un único fragmento de 272 pb., sin sitio de corte, asociado con el genotipo TT. M: marcador molecular de 1000 pb.
El segundo de los patrones de restricción presentó dos fragmentos, uno de 106 y otro de 166 pb de longitud asociados con el genotipo homocigoto CC. El alelo C codifica para la leptina normal y no presenta la mutación, y al no estar modificado el sitio de corte CCGC la enzima lo reconoce y genera estos dos fragmentos de diferente longitud (Figura 3).

Figura 3 Patrón de restricción para el genotipo CC. Carriles 1-3, productos de PCR con un tamaño de 272 pb de longitud. M: marcador molecular de 1000 pb. Carriles 4-6, productos de la digestión enzimática (enzima Aci I) mostrando dos fragmentos, uno de 106 pb y otro de 166 pb asociados con el genotipo CC.
El tercero de los patrones de restricción y asociado con el genotipo heterocigoto CT, el cual genera los dos tipos de leptina modificada y común, origina tres fragmentos de diferente peso molecular, un fragmento de 106 y otro de 166 pb originados por el alelo de la leptina normal, y un fragmento de 272 pb correspondiente al alelo de la leptina modificada (Figura 4).
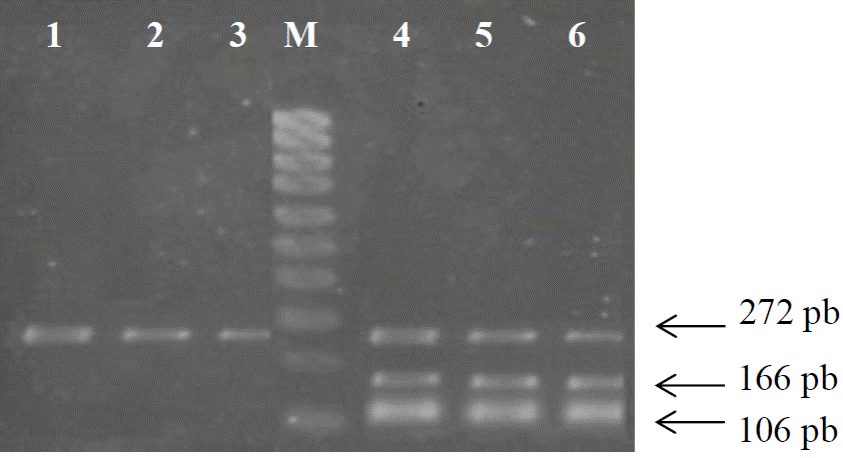
Figura 4 Patrón de restricción para el genotipo CT. Carriles 1-3, productos de PCR con un tamaño específico de 272 pb de longitud, M: Marcador molecular de 1000 pb. Carriles 4-6, productos de la digestión enzimática (enzima Aci I) mostrando tres fragmentos de 272, 106 y 166 pb.
En la Figura 5 se presentan las secuencias del gen leptina mutada y normal obtenidas del Gen Bank (accesion HE605298), en donde se puede observar la transición de citosina (C) por timina (T) ubicada en el cromosoma 4 del exón 2 posición 1115, también se muestran las secuencias genéticas de los productos de PCR del gen leptina de vacas de los tres grupos según su estatus de producción: altas, medias y bajas productoras. La secuencia del gen Ob para la leptina de las vacas de alta producción fue similar a la secuencia del gen Ob de la leptina mutada del GeneBank, ya que en el sitio específico de la mutación se encontró el nucleótido timina, contrario a la secuencia del gen Ob para la leptina de las vacas de baja producción el cual fue igual al alelo normal con la presencia del nucleótido citosina.
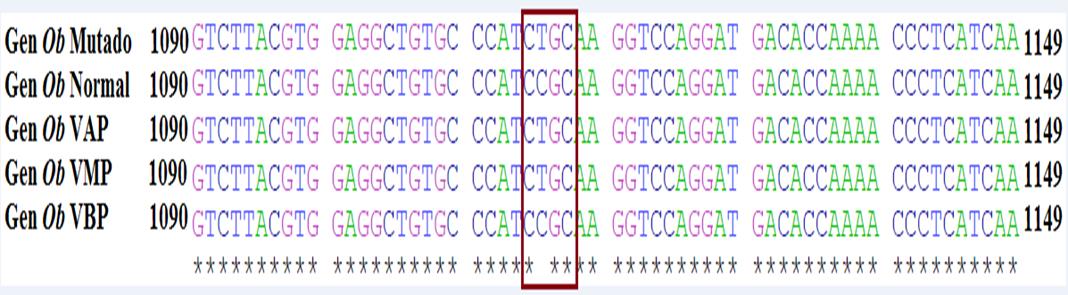
Figura 5 Secuencia del gen Ob para leptina mutada y normal obtenidas del GenBank (Gen Ob Mutado y Gen Ob Normal), y secuencia de los productos de PCR del gen Ob de vacas altas productoras (VAP) medias productoras (VMP) y bajas productoras (VBP). *= nucleótidos comunes en las secuencias.
El anális de varianza (Tabla 1) muestra que hubo significancia (p<0.01) en la fuente de variación para el modelo en las variables PL, CMS y PV. Asimismo se observó significancia (p<0.01) para los genotipos TT y CT en las mismas variables.
Tabla 1 Análisis de varianza.
| FV | GL | PL | CMS | PV | PG | PP | ST |
|---|---|---|---|---|---|---|---|
| Modelo | 4 | 1533.6** | 141.1** | 137839.4** | 14.72 | 2.63 | 2.39 |
| Error | 95 | 9.5 | 1.3 | 1273.9 | 2.09 | 0.507 | 0.41 |
| NL | 1 | 0.08 | 3.3 | 3228.3 | 13.698 | 0.023 | 0.13 |
| DL | 1 | 49.3 | 4.8 | 4719.6 | 10.041 | 0.076 | 0.05 |
| TT | 1 | 4709.2** | 422.1** | 412384.2** | 14.404 | 7.633 | 4.27 |
| CT | 1 | 664.1** | 79.0** | 77211.1** | 1.160 | 0.080 | 0.35 |
| R2 | 0.87 | 0.81 | 0.82 | 0.22 | 0.17 | 0.19 | |
| CV | 9.0 | 4.9 | 4.9 | 18.17 | 20.5 | 6.7 |
CMS=Consumo de Materia Seca, PV=Peso Vivo, PL= Producción de leche, PG= Porcentaje de grasa, PP= Porcentaje de proteína, ST= Sólidos totales. ** = (P<0.01). Nota: Los valores que se muestras corresponden a los cuadros medios de las variables estudiadas.
En la comparación de medias entre genotipos (Tabla 2) se observa que las diferencias entre los valores medios del genotipo TT y los de los genotipos CT y CC, son significativamente diferentes en todas las variables estudiadas, y entre los genotipos CT y CC sólo hubo diferencias significativas en las variables PL, CMS y PV.
Tabla 2 Comparación entre genotipos y diferencia entre medias.
| Genotipos | PL | CMS | PV | PG | PP | ST |
| TT vs CT | 10.11** | 2.64** | 82.55** | 1.08** | 0.65** | 0.55** |
| TT vs CC | 18.77** | 5.67** | 177.43** | 1.41** | 0.70** | 0.71** |
| CT vs CC | 8.65** | 3.03** | 94.88** | 0.33 | 0.05 | -0.15 |
CMS=Consumo de Materia Seca, PV=Peso Vivo, PL= Producción de leche, PG= Porcentaje de grasa, PP= Porcentaje de proteína, ST= Sólidos totales; TT, CT y CC son los genotipos posibles asociados a las vacas según el SNP del gen Ob determinado en cada vaca. **= (P<0.01).
En la Tabla 3 se muestran los modelos de predicción para la producción de leche (PL), consumo de materia seca (CMS) y peso vivo del animal (PV) en función del número de lactancias, días lactando y genotipo del animal. El modelo para producción de leche tuvo un coeficiente de determinación del 0.87. Al hacer la comparación respectiva del modelo para producción de leche (PL) de cada genotipo se obtuvo que cuando las vacas son de genotipo TT, tienen un aumento en la producción de leche diaria de 18.06 L más que el genotipo CC y 10.47 L más que el genotipo CT (ES 0.8122, p<0.0001); mientras que las vacas con el genotipo CT tienen una producción mayor de 7.59 L sobre el genotipo CC (ES 0.9095, p<0.0001). Para el caso del consumo de materia seca (CMS), las vacas con el genotipo TT consumieron 5.40 kg más de materia seca que las vacas con genotipo CC, y 2.78 kg más que las vacas con genotipo CT (ES 0.3138, p<0.0001), asimismo, estás ultimas tuvieron un consumo mayor de 2.62 kg de materia seca que las vacas con genotipo CC (ES 0.3424, p<0.0001). Respecto al peso vivo del animal (PV), las vacas con genotipo TT tuvieron en promedio 169.02 kg más peso que las vacas con genotipo CC y 88.02 kg más que las vacas con genotipo CT (ES 9.8066, p<0.0001). Por su parte, las vacas con el genotipo CT fueron 81 kg más pesadas que las vacas con genotipo CC (ES 10.7012, p<0.0001). En general, las vacas con el genotipo TT tuvieron valores más altos en las tres variables dependientes que las vacas con cualquiera de los genotipos CT y CC. Los parámetros estimados no fueron significativos para las variables porcentaje de grasa (PG), porcentaje de proteína (PP), y contenido de solidos totales (ST).
Tabla 3 Modelos de predicción por genotipo para las variables producción de leche, consumo de materia seca y peso vivo.
| Variable | Modelo | R 2 |
|---|---|---|
| PL | PL = 23.91 + 0.023NL + 0.006DL + 18.06TT | 0.87 |
| PL = 23.91 + 0.023NL + 0.006DL + 7.59CT | 0.87 | |
| PL = 23.91 + 0.023NL + 0.006DL + 0.0CC | 0.87 | |
| CMS | CMS = 19.46 + 0.14NL + 0.002DL + 5.40TT | 0.82 |
| CMS = 19.46 + 0.14NL + 0.002DL +2.62CT | 0.82 | |
| CMS = 19.46 + 0.14NL + 0.002DL + 0.0CC | 0.82 | |
| PV | PV = 608.24 + 4.63NL + 0.06DL + 169.02TT | 0.82 |
| PV = 608.24 + 4.63NL + 0.06DL + 81CT | 0.82 | |
| PV = 608.24 + 4.63NL + 0.06DL + 0.0CC | 0.82 |
PL= Producción de leche, CMS=Consumo de Materia Seca, PV=Peso Vivo, NL= No. De lactancias; DL = Días lactando; TT, CT y CC son los genotipos posibles asociados a las vacas según el SNP del gen Ob determinado en cada vaca.
Se observaron correlaciones (Tabla 4) positivas y altamente significativas (P<0.01) de la producción de leche (PL) con días de lactancia (DL), consumo de materia seca (CMS), peso vivo de la vaca (PV), porcentaje de grasa (PG), porcentaje de proteína (PP) y con el genotipo TT, y correlación significativa (P<0.05) con sólidos totales (ST). También se observó correlación negativa y altamente significativa (P<0.01) con el genotipo CC. La correlación entre producción de leche y el genotipo heterocigoto (CT) no fue significativa.
Tabla 4 Matriz de coeficientes de correlación de Spearman.
| NL | DL | CMS | PV | PL | PG | PP | ST | |
|---|---|---|---|---|---|---|---|---|
| DL | 0.011 | |||||||
| CMS | 0.175 | 0.407** | ||||||
| PV | 0.165 | 0.397** | 1.000** | |||||
| PL | 0.091 | 0.384** | 0.945** | 0.935** | ||||
| PG | 0.230 | 0.235* | 0.369** | 0.364** | 0.349** | |||
| PP | 0.037 | 0.031 | 1.000** | 0.337** | 0.935** | 0.369** | ||
| ST | -0.063 | 0.038 | 0.248* | 0.249* | 0.253* | 0.051 | 0.260** | |
| TT | 0.020 | 0.120 | 0.830** | 0.767** | 0.830** | 0.371** | 0.420** | 0.427** |
| CT | 0.158 | 0.389 | -0.032 | 0.042 | -0.032 | -0.104 | -0.179* | 0.285** |
| CC | 0.174 | -0.497** | -0.792** | -0.803** | -0.792** | -0.267** | -0.243* | -0.147 |
NL= Número de Lactancias, DL= Días de lactancia, CMS= Consumo de Materia seca, PV= Peso Vivo, PL= Producción de Leche, PG= Porcentaje de grasa, PP= Porcentaje de proteína, ST= Sólidos totales, TT, CT, y CC= Son los genotipos posibles para el SNP del gen Ob. *=P<0.05, **=P<0.01.
El consumo de materia seca (CMS) mostró correlación positiva y altamente significativa (P<0.01) con peso vivo de la vaca (PV), porcentaje de grasa (PG), porcentaje de proteína (PP) y con el genotipo TT, y correlación significativa (P<0.05) con sólidos totales (ST). Presentó correlación negativa y altamente significativa (P<0.01) con el genotipo CC, y no hubo correlación con el genotipo CT.
El peso vivo de la vaca (PV) mostró correlación en el mismo sentido y magnitud que el CMS con las de más variables.
El porcentaje de grasa (PG) tuvo correlación positiva y altamente significativa (P<0.01) con el porcentaje de proteína (PP) y con el genotipo TT, y correlación negativa y altamente significativa (P<0.01) con el genotipo CC. La correlación con el genotipo heterocigoto (CT) no fue significativa.
La correlación del porcentaje de proteína (PP) con los sólidos totales y el genotipo TT fue positiva y altamente significativa (P<0.01), pero con los genotipos CT y CC fueron negativas y significativas (P<0.05).
Los sólidos totales (ST) también mostraron correlación positiva y altamente significativa (P<0.01) con los genotipos TT y CT, y correlación no significativa con el genotipo CC.
El genotipo TT mostró correlación positiva y altamente significativa (P<0.01) con el CMS, PV, PL, PG, PP y ST; el genotipo CT tuvo correlación positiva y altamente significativa (P<0.01) con ST y correlación negativa y significativa (P<0.05) con PP; el genotipo CC presentó correlación negativa y altamente significativa (P<0.01) con DL, CMS, PV, PL, PG, y correlación negativa y significativa (P<0.05) con PP y correlación no significativa con ST.
Las correlaciones entre el número de lactancias con las demás variables no fueron significativas (P>0.05).
Discusión
Existe una relación entre los genotipos estudiados y las características de producción y calidad de la leche, éstos resultados coinciden con los reportados por otros autores (Ngu et al., 2015, 305; Schenkle et al., 2005, 2018). El presente estudio demostró que la digestión del producto de PCR del gen Ob para la leptina con la enzima Aci I así como el número y tamaño de los fragmentos observados, se puede usar como herramienta para determinar el genotipo del ganado Holstein respecto al gen Ob. Estos genotipos generados se presentan en la mayoría de razas bovinas, pero en diferentes proporciones (Ayres et al., 2010, 2056) y concuerdan con los del ganado Bos taurus según estudios realizados por Choudhary et al., (2005, 741).
Los resultados de secuenciación mostraron la sustitución de citosina por timina en el alelo mutado proveniente de las vacas altas productoras (VAP), el cual coincide con la mutación de la secuencia del gen Ob mutado obtenida del GenBank. Mientras que en la secuencia del alelo de las vacas bajas productoras (VBP) se observó la presencia de citosina al igual que en la secuencia del alelo normal obtenida del GenBank. Lo anterior coincide con Raoof et al., (2008, 348).
En relación con el genotipo y su potencial productivo, las vacas que se identificaron con el genotipo TT mostraron ser superiores a las vacas con cualquiera de los otros dos genotipos (CT y CC) en todas las variables estudiadas, ya que las vacas con dicho genotipo tuvieron una capacidad de mayor consumo de materia seca lo que a su vez conllevó a un aumento de peso vivo, esto también lo menciona Vallone et al., (2014,43), al igual que Liefers et al., (2002, 1637), quienes señalan que la correlación entre el tamaño de la vaca y el consumo de alimento es alta y positiva, de manera tal que las vacas de mayor tamaño tienen mayor consumo, además se asocia el incremento del consumo de alimento con una mayor producción de leche (Banos et al., 2007, 3196,) ya que la vacas con el genotipo TT alcanzaron mayor producción en el pico de lactancia. Mojtaba et al., (2015, 21) informa de los efectos positivos del genotipo TT con mayor producción diaria de leche. Así mismo para las vacas con genotipo TT se observó un mayor contenido en el porcentaje de grasa y proteína en comparación a las vacas con cualquiera de los otros dos genotipos, esto mismo lo menciona Liefers et al., (2002, 1636). En el caso de homocigoto CC se caracterizaron por tener un menor consumo y por consiguiente tuvieron un menor peso (Woronuk 2011, 2), además de tener un decremento importante en la producción de leche coincidiendo con lo reportado por Casas et al., (2007, 2810). Por otro lado el genotipo heterocigoto CT tuvo un comportamiento intermedio definido por su estructura en la cual tiene la característica de generar los tipos de leptina común y mutada (Corva et al., 2007, 244).
El principal sitio de acción de la leptina es en el núcleo arcuato del hipotálamo, el cual comprende dos poblaciones de neuronas: la primera libera el neuroléptico y representa la vía orexigénica (inductora del apetito); la segunda comprende neuronas que secretan propiomelancortina y representa la vía anorexigénica (inductora de saciedad), la leptina inhibe la vía orexigénica y estimula la anorexigénica. La función más importante de la leptina es de reguladora del peso corporal (Dias et al., 2007, 3; Frühbeck 2006, 7). Al producirse la sustitución de citosina por timina en el alelo normal, se altera la estructura de la proteína y bloquea la capacidad para ser identificada por los receptores en el hipotálamo (Buchanan et al., 2002, 113), originando características relacionadas con ganancia de peso.
La correlación entre los días de lactancia, consumo de materia seca, peso vivo y producción de leche fue positiva y significativa (P<0.01) lo cual significa que entre más días de lactancia haya transcurrido, el animal tiende a consumir más y por consiguiente su producción es mayor, es importante mencionar que los muestreos de leche se realizaron durante el pico de lactancia de 60 a 70 días posparto, ya que el objetivo era conocer si el polimorfismo del gen Ob afectaba de forma directa el consumo de materia seca y la producción de leche, en donde se pudo observar la existencia de correlaciones significativas entre peso vivo de la vaca y la producción de leche lo cual coincide con Vallone et al., (2014, 43).
Por otro lado se puede observar los efectos positivos del genotipo TT versus el genotipo CC con respecto al aumento de los componentes de la leche como en el caso del porcentaje de grasa y proteína. Además de tener un impacto significativo (P<0.01) sobre la producción de leche, las vacas con genotipo TT tienen un mayor consumo de materia seca lo que a su vez conduce a una ganancia de peso. El genotipo CT no estuvo correlacionado con la ganancia de peso y con las variables de calidad de la leche, tuvo una producción media, asumiendo que su comportamiento intermedio se debe a que generan dos tipo de leptina; normal y mutada; por el contrario el genotipo CC tuvo una alta correlación negativa con la producción y con los componentes de la leche de manera específica el porcentaje de grasa y proteína, así como un menor consumo de materia seca y menor peso vivo.
Estos resultados demuestran la importancia de sustitución C/T la cual trae consigo un cambio en uno de los aminoácidos alterando de forma radical la estructura la proteína leptina generando que el polimorfismo se relacione con los parámetros como producción y los componentes de calidad de la leche (Liefers et al., 2002,1636)
Se puede concluir que los resultados sugieren que el genotipo TT del gen leptina tiene un efecto positivo sobre el consumo de materia seca, el volumen de producción de leche, y está asociado con el incremento en el contenido porcentual de los distintos componentes de la leche, representando así este polimorfismo un indicador para evaluaciones futuras, y su aplicación práctica en el mejoramiento genético asistido teniendo ventajas económicas para los productores lecheros. Es necesario seguir analizando información sobre otros polimorfismos en el gen leptina y la interacción que pueda existir con polimorfismos en otros genes para poder hacer una mejor selección de animales con características sobresalientes.




![[ARTÍCULO RETRACTADO]Teleconexiones atmosféricas-oceánicas (océanos Pacífico y Atlántico) moduladoras de veranos húmedos y secos en el Núcleo del Monzón de Norte América](/img/es/prev.gif)






 nueva página del texto (beta)
nueva página del texto (beta)


