Introducción
La contaminación con Hidrocarburos Aromáticos Policíclicos (HAP) causa la inhibición de microorganismos del suelo (Eom et al. 2007). Sin embargo, algunos grupos de microorganismos pueden adaptarse y crecer en presencia de estos compuestos orgánicos (Peixoto et al. 2011). Por ejemplo, bacterias aeróbicas pueden tolerar los HAP (Si-Zhong et al. 2009), y son comunes en sitios contaminados por plaguicidas y metales pesados. Las bacterias resistentes pueden utilizar los HAPs como fuente de carbono y energía para su crecimiento y reproducción; además, liberan enzimas como dioxygenasas, peroxidasas y catalasas, para que oxiden a los HAP y consecuentemente lo transformen o degraden a compuestos menos tóxicos (Harvey et al. 2002). La biodegradación de HAP se ha estudiado principalmente en bacterias vivas libres; aunque hay pocos casos de biodegradación estudiados en bacterias simbióticas. En este sentido, las bacterias simbióticas pertenecientes al género Rhizobium son bien conocidas por establecer una simbiosis mutualista con las leguminosas y por su contribución a la fijación biológica del nitrógeno atmosférico (Oldroyd y Downie, 2008). Barret y Parker (2005), identificaron bacterias del género Burkholderia como simbiontes de nódulos en Mimosa pigra, Mimosa casta, Mimosa pudica y Abarema macradenia en la isla de Barro Colorado, Panamá. También estudios de plantas de Mimosa spp. en América Central y del Sur, reportaron que forman simbiosis con Burkholderia, Cupriavidus y Rhizobium en sus ambientes nativos (Barrett y Parker, 2006), pero la mayoría de las especies en Brasil, que es el centro más grande de la diversidad Mimosa, tienen una preferencia abrumadora por Burkholderia (Bontemps et al. 2010, dos Reis et al. 2010; Simon et al. 2011). La ventaja de utilizar bacterias simbióticas en los procesos de biorremediación de HAP está relacionada con su capacidad para fijar nitrógeno que es limitante durante la biorremediación, ya que la deposición de hidrocarburos de petróleo aumenta significativamente la acumulación de carbono orgánico en el suelo y genera una relación C/N muy alta (López-Martínez et al. 2005; Radwan et al. 2007). Rhizobium tropici CIAT 899 ha sido reportado como tolerante a los metales pesados y antibióticos como cloranfenicol, espectiomicina, carbenicilina y estreptomicina (Martínez-Romero et al. 1991). Otros miembros del género Rhizobium o Mesorhizobium son capaces de tolerar y degradar sustancias tóxicas tales como dibenzotiofeno, 2,4-dinitrotolueno (Dutta et al. 2003), y algunos HAP como fenantreno y acenaftileno (Poonthrigpun et al. 2006). Rhizobium tropici CIAT 899 mostró tolerancia en crecimiento a 40 g/mL de fenantreno degradando el 50%, y benzopireno a 60 g/mL, degradando 45% a las 120 h (González et al. 2013). El objetivo de este estudio fue aislar bacterias de nódulos de mimosas y evaluar su capacidad degradadora de fenantreno en medios de cultivo líquidos.
Métodos
Aislamiento
Se colectaron plantas de mimosas en Municipios del estado de Chiapas, México; posteriormente se les extrajeron los nódulos de la raíz (Tabla 1). Los nódulos colectados fueron lavados y sumergidos en etanol al 70 % durante 5 minutos, posteriormente desinfectados con hipoclorito de sodio al 25 % durante 15 minutos, el exceso de hipoclorito fue eliminado con lavados de agua destilada estéril. Finalmente los nódulos desinfectados fueron macerados y resuspendiendo en una solución de MgSO4·7H2O al 10 mM (Wong y Caballero, 2010). Se inocularon 100 µL de la suspensión en el medio de cultivo de Extracto de Levadura- Manitol (YMA) las cajas inoculadas fueron incubadas a 29 °C durante 2 días, posteriormente fueron seleccionadas colonias que presentaron diferente morfología.
Las cepas bacterianas aisladas fueron seleccionadas con base al crecimiento en los medios de cultivo diferenciales de agar MacConkey y agar hierro de Kliger para descartar enterobacterias.
Secuenciación del gen 16S rARN
Para realizar la identificación de las cepas, se realizó la extracción del ADN de los aislados seleccionados con el kit ZR Fungal/Bacterial DNA Miniprep™. Posteriormente se amplificó por PCR el gen 16S rARN, utilizando los oligonucleótidos fD1 y rD1 (Weisburg et al. 1991), el producto de amplificación obtenido de 1.4 Kb fue purificado de gel con el kit Thermo Scientific GeneJET Gel Extraction de acuerdo con las instrucciones del proveedor. Los productos de PCR purificados fueron enviados a secuenciar a la unidad de síntesis del Instituto de Biotecnología de la UNAM. Las secuencias del gen 16S rARN fueron alineadas con el programa CLUSTALW (Thompson et al. 1994). Los árboles filogenéticos fueron construidos con el método neirghbor-joining (Saitou y Nei, 1987), empleando 1400 nucleótidos del gen 16S ribosomal de acuerdo con la matriz de distancia de Jukes y Cantor (1969), utilizando el programa MEGA versión 6 (Tamura et al. 2013).
Cinética de crecimiento microbiano
Las cepas BB24, BB26 y BY8 fueron crecidas en medio de cultivo PY (peptona extracto de levadura) durante 24 h a 29°C con agitación a 200 rpm. Posteriormente, los cultivos bacterianos fueron ajustados a una densidad óptica de 0.2 a 600 nm, e inoculados en matraces que contenían 200 mL del medio de cultivo Bushnell Haas Broth (BHB), composición (g/L): 0.2 MgSO4· 7H2O, 0.02 CaCl2 2H2O, 1 KH2PO4, 1 K2HPO4, 1 NH4NO3, 0.05 FeCl3, y Tween-80, como fuente de carbono fenantreno 500 ppm y succinato de sodio (5g/L) como control (sigma-aldrich).
Los cultivos fueron crecidos durante 168 h a 29°C con agitación a 200 rpm, el crecimiento bacteriano durante el proceso de degradación del compuesto aromático se determinó mediante diluciones seriadas de los cultivos para cuantificar el crecimiento microbiano por cuenta viable, 100 (L de las diluciones fueron inoculadas en cajas Petri con medio de cultivo PY durante 48 h e incubados a 29°C. Las mediciones se realizaron a las 0, 24, 48, 72, 96 y 168 h del periodo de incubación. Los resultados fueron analizados por comparación de medias con la prueba de Tukey, con un nivel de significancia de P≤0.05, utilizando el programa “Statistical Analysis System” (SAS versión 9.3).
Extracción de HAPs de la fase líquida
Se empleó la técnica de extracción en fase sólida (SPE) en su modalidad de cartuchos con C18. Las columnas de extracción se activaron previamente con 5 mL de metanol, 5 mL de hexano y 5 mL de agua mili-Q. Se pasaron 50 mL del medio de cultivo a través de los tubos de SPE a una presión de 25 psi y con un flujo de 1-2 mL/min. Después, se adicionaron 5 ml de agua mili-Q. La elución de los HAPs se realizó con 10 mL de hexano. Se concentró el extracto hasta sequedad bajo un flujo de argón de alta pureza 99.997% (INFRA) y se volvió a suspender el extracto con 1 mL de acetonitrilo. El extracto concentrado se almacenó en un vial de borosilicato ámbar de 2 mL con tapón de rosca y septa de PTFE, se le adicionó 50 µg/L de p-Terfenilo (sigma-aldrich) como estándar interno y fue refrigerado a 4ºC hasta su análisis cromatográfico.
Cuantificación por Cromatografía de gases acoplada a espectrometría de masas (GC-MS)
Para la determinación de fenantreno se utilizó un cromatógrafo de gases (Thermo Trace GC Ultra) acoplado a un espectrómetro de masas con una trampa de iones (ITQ 900) (Thermo Fisher Scientific Inc, Austin, TX USA). Las condiciones de operación del CG-MS fueron las siguientes: Se utilizó 2 μl de muestra por análisis. La inyección fue en modo splitless con un automuestreador (TriPlus Autosampler). La temperatura del inyector fue de 250 ºC y de la línea de transferencia fue de 270 °C. La temperatura de la fuente de iones fue de 200 °C. Los componentes fueron separados en una columna capilar modelo TR-5MS 5% fenilmetilsiloxano (30 m longitud x 0.25 mm diámetro interno) con 0.25 μm de espesor de la fase estacionaria. La rampa de temperatura del horno fue una temperatura inicial de 70°C mantenida durante 1 min, seguido de un gradiente de 15ºC/min hasta alcanzar 180ºC mantenidos durante 1 min, posteriormente incrementos de temperatura de 5°C/min hasta 270 ºC mantenidos 15 min (tiempo de corrida: 42 min). El gas portador fue helio de ultra alta pureza (INFRA) (99.9999%). El detector de masas operó en modo de monitoreo de ion selectivo (SIM).
Resultados
Aislamiento
De los nódulos de mimosas colectadas en el estado de Chiapas, se aislaron 122 cepas bacterianas en el medio de cultivo YMA, posteriormente fueron crecidas en el medio de cultivo MacConkey; se identificaron 80 cepas que pertenecen a enterobacterias. Estos resultados fueron confirmados con el medio de cultivo agar hierro de Kliger, descartándolas del grupo de cepas aisladas. Las 42 que no crecieron en el medio de cultivo MacConkey fueron crecidas en medio de cultivo Bushnell Haas Broth (BHB) que contenía como única fuente de carbono fenantreno a 500 ppm, solamente las cepas BB24, BB26 y BY8 tuvieron la capacidad de crecer en presencia de este compuesto.
Identificación
Las secuencias obtenidas de las tres cepas fueron comparadas con la base de datos de nucleótidos del GenBank. Dos cepas presentaron relación genética con especies del género Burkholderia. La secuencia de la cepa BB24 presentó el 99% de identidad con Burkholderia phymatum, la cepa BB26 presentó 99 % identidad con Burkholderia phenoliruptrix y la secuencia de la cepa BY8 99 % identidad con Rhizobium paknamense. La relación genética de las cepas aisladas con las especies de Burkholderia y Rhizobium reportadas se puede observar en el árbol filogenético construido con el método neirghbor-joining (Saitou y Nei, 1987), basado en 1400 nucleótidos de las secuencias del gen 16S ribosomal de acuerdo a la matriz de distancia Jukes y Cantor 1969 (Figura 1).
Cinética de crecimiento microbiano
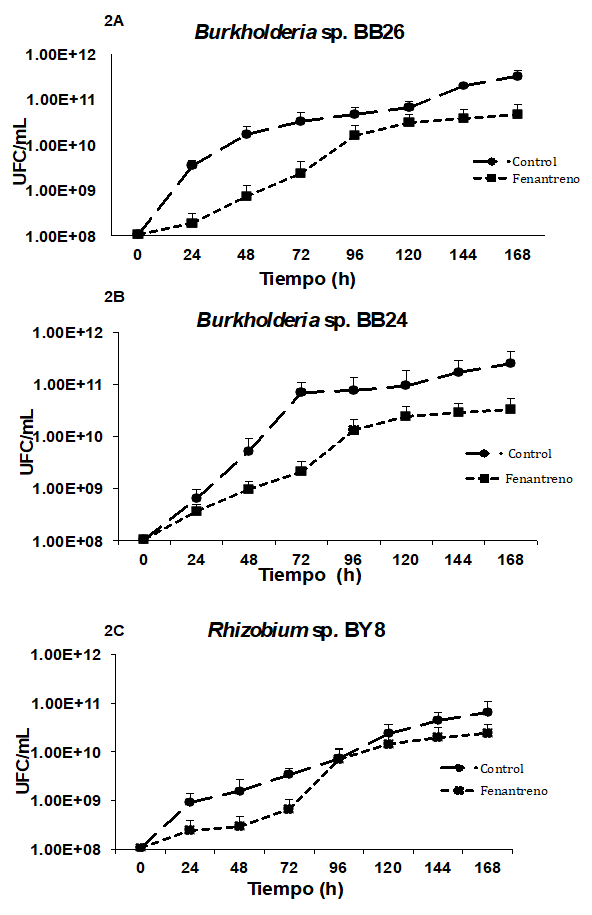
Las cepas de Burkholderia sp. BB26, Burkholderia sp. BB24 y Rhizobium sp. BY8 en presencia de fenantreno 500 ppm como fuente de carbono, tuvieron un comportamiento de crecimiento similar al tratamiento control, el cual utilizó como fuente de carbono succinato de sodio. Los resultados obtenidos fueron analizados por comparación de medias, y no se encontró diferencia significativa en los tratamientos (Figura 2, A, B, C).
Cuantificación de compuestos aromáticos por Cromatografía de gases
Las cepas de Burkholderia sp. BB26, Burkholderia sp. BB24 y Rhizobium sp. BY8, crecieron en presencia de fenantreno (500 ppm). En la Figura 3 se reporta el porcentaje de degradación de fenantreno realizado por las tres cepas bacterianas, se observa que durante 168 horas de la cinética, el contaminante va siendo removido del medio de cultivo. A las 168 h de incubación, la cepa Rhizobium sp. BY8 fue capaz de degradarlo en 99% (Figura 3C), Burkholderia sp. BB26 78.5 % (Figura 3B) y Burkholderia sp. BB24 68.5% de fenantreno a una concentración de 500 ppm (Figura 3A).
Discusión o Conclusiones
Las tres cepas aisladas de los nódulos de mimosas que tuvieron la capacidad de crecer en presencia de fenantreno están relacionadas genéticamente por análisis de secuencia del gen 16S ribosomal con Burkholderia phenoliruptrix, Burkholderia phymatum y Rhizobium paknamense. La cepa de Burkholderia sp. BB26 tuvo el mayor crecimiento a las 120 h en presencia de fenantreno. El comportamiento del crecimiento microbiano en presencia de este hidrocarburo fue muy similar al control, el cual contenía como fuente de carbono succinato, que es una fuente de carbono que liberan las plantas en los exudados radicales y que es fácilmente metabolizable por las bacterias en la rizosfera. El mismo comportamiento de crecimiento lo presentaron las cepas de Burkholderia sp. BB24 y Rhizobium sp. BY8 en presencia de fenantreno. Durante el crecimiento bacteriano se evaluó la capacidad de degradar fenantreno de las cepas; Burkholderia sp. BB26 degradó el 78.5% de fenantreno a las 120 h de crecimiento. Sin embargo es el primer reporte de una cepa relacionada genéticamente a Burkholderia phenoliruptrix como degradadora de fenantreno, debido a que esta especie ha sido reportada como degradadora de xenobióticos recalcitrantes (Coenye et al. 2004). Mientras que Burkholderia phymatum ha sido reportada como osmotolerante, pero no con la capacidad de degradar fenantreno, en este trabajo la cepa Burkholderia sp. BB24 relacionada genéticamente a esta especie degradó 65% a las 120 h de crecimiento. Algunas especies del género Burkholderia como Burkholderia cepacia G4, han sido reportadas como degradadoras de tricloroetileno (Kim et al. 2014), Burkholderia unamae, Burkholderia kururiensis y Burkholderia xenovorans son especies reportadas con capacidad de crecer en presencia de benceno, fenol, tolueno y bifenilos (Caballero et al. 2007). Especies del género Rhizobium han sido reportadas como degradadoras de compuestos xenobióticos, entre ellas Rhizobium tropici CIAD 899, que tiene la capacidad de degradar fenantreno el 50% y 45% de benzopireno a las 120 h; Rhizobium leguminosarum fue reportada como degradadora de tolueno, mientras que Rhizobium paknamense no ha sido reportado como degradadora de xenobióticos, es el primer reporte de una cepa relacionada genéticamente con esta especie como degradadora de fenantreno. Durante el crecimiento de esta cepa, se encontró que degradada el 99% de fenantreno 500 ppm a las 168 h de crecimiento, esta capacidad de Rhizobium sp. BY8 para degradar fenantreno es comparable a bacterias del género Pseudomonas que degradan el 98% después de diez días de incubación, mientras que Stenotrophomonas y Bacillus thuringiensis FQ1 degradan el 95% en suelo después de tres meses de incubación, Alcaligenes faecalis y Brevundimonas degradan del 90-95% a las 72 h, estas bacterias utilizan este compuesto como fuente de carbono y energia (Xia et al. 2010; Gónzalez et al. 2013; Galazka y Galazka, 2015; Jiang et al. 2015). Los resultados de degradación de fenantreno por las cepas de Burkholderia sp. BB26 Burkholderia sp. BB24 y Rhizobium sp. BY8 sugieren que las tres cepas tienen potencial para utilizarse en procesos de biorremediación de suelos contaminados con fenantreno.











 nueva página del texto (beta)
nueva página del texto (beta)