Introducción
El queso de hebra en la región de la cuenca del Papaloapan (Oaxaca, México) se elabora en áreas antisépticas de casas y traspatios con leche sin pasteurizar. El producto puede estar contaminado con Listeria monocytogenes, Salmonella typhi, Mycobacterium bovis (causante de la tuberculosis bovina) y representar un riesgo para la salud. La estandarización del proceso con cultivos iniciadores endémicos a partir del queso o sus subproductos podría contribuir a la obtención de queso que cumpla con lo indicado en la norma oficial mexicana NOM-243-SSA1-2010 (Diario Oficial de la Federación 2010, 125). En la industria quesera, el suero fermentado con cultivos indefinidos se utiliza en Francia, Suiza, Italia y otros países para la producción artesanal de quesos Gruyere, Emmental, Grana (Beresford 2001, 259) y Parmesano (Coppola 1997, 305).
La microbiota de diferentes alimentos fermentados son caracterizados con base en su morfología, fisiología, estructura de sus componentes celulares y, recientemente, mediante marcadores moleculares, como el gen ADNr 16S. Dentro de los consorcios microbianos que se desarrollan en este tipo de alimentos fermentados se encuentran las bacterias ácido lácticas (BAL), como Lactobacillus acidophilus, L. plantarum, L. casei, L. casei sbp rhamnosus, L. delbrueckii sbp bulgaricus, L. fermentum, L. reuteri, Lactococcus lactis sbp lactis, L. Lactis sbp. Cremoris, Bifidobacterium bifidum, B. infantis, B. adolecentis, B. longum, B. breve, Enterococcus faecalis, E. faecium, y otros (Farnworth 2001, 335). Las BAL proporcionan sabor, textura e incrementan el valor nutricional de los alimentos, se utilizan en la industria alimentaria como bioconservadores debido a la producción de bacteriocinas y otras sustancias que ejercen acción antibacteriana, además, las cepas mencionadas han mostrado características nutracéuticas (Palou y Serra 2000, 76; Vasquez 2009, 64; Shiphrah 2013, 118). Estudios microbiológicos recientes en quesos frescos de México: queso de aro (Teotitlan de las Flores, Oaxaca) y queso Oaxaca (de hebra), han revelado que estos quesos contienen una elevada concentración microbiana: levaduras (9 Log10 UFC/g), bacterias de interés industrial (BAL) (35.5 Log10 UFC/g), y coliformes (28 Log10 UFC/g) (Castro-Castillo et al., 2013, 107; Gonzalez-Montiel y Franco-Fernandez, 2015, 254). Sin embargo, los estudios se han efocado principalmente al queso o bien a la cuajada, sin considerar el suero de leche, por lo que resulta interesante explorar esta matriz para el aislamiento de BAL con la posibilidad de que estas cepas se utilicen como iniciadores en la elaboración de quesos frescos a partir de leche pasteurizada. Considerando lo anterior, el objetivo de este estudio fue evaluar la dinámica poblacional de BAL y aislarlas a partir de SFQH, con base en sus características morfológicas y moleculares.
Método
El suero se recolectó al finalizar la producción de queso de hebra elaborado con leche cruda, se distribuyó en dos matraces de 250 mL estériles con tapa de rosca que fueron incubados a 37°C por 120 h, se colectaron muestras a las 0, 24, 48 y 120 h que fueron utilizadas para determinar el pH; además con éstas se realizaron diluciones decimales en amortiguador de fosfato salino, inoculando 100 µL de cada dilución decimal, por duplicado, en placas de agar APT (lactobacilos heterofermentativos y otros microorganismos exigentes), Elliker (Streptococcus, Lactococcus, Leuconostoc, lactobacilos homo y heterofermentativos), MRS (lactobacilos) y M17 (estreptococos lácticos) (Fluka, Alemania). Las placas de agar se sellaron con parafilm e incubaron a 37ºC durante 48 h. El conteo en placa se realizó en cajas que contenían entre 30-300 colonias y se reportaron las UFC mL-1 y el número de colonias. Todas las colonias obtenidas se sometieron a pruebas microbiológicas y fueron seleccionadas aquellas con características propias de las BAL: tinción de Gram (positivo) (Hycel, México), prueba de la catalasa (negativa) (JT Baker, USA), morfología colonial (Ramírez-Baca 2009, 14) y celular (cocos y bacilos); y se purificaron en el medio de aislamiento correspondiente. Las BAL presuntivas se analizaron con pruebas bioquímicas API 20A (bioMerieux, Francia) y moleculares. Las galerías API 20A constan de 20 sustratos deshidratados que son L-triptófano (Ind), urea (Ure), D-glucosa (Glu), D-manitol (Man), D-lactosa (Lac), D-sacarosa (Sac), D-maltosa (Mal), salicina (Sal), D-xilosa (Xyl), L-arabinosa (Ara), gelatina (Gel: actividad proteasa), esculina (Esc: actividad β-glucosidasa), glicerol (Gly), D-celobiosa (Cel), D-manosa (Mne), D-melezitosa (Mlz), D-rafinosa (Raf), D-sorbitol (Sor), L-ramnosa (Rha) y D-trehalosa (Tre). Para la caracterización molecular, el ADN genómico se extrajo con el kit Ultra Clean microbial DNA isolation (Mo BIO, USA). El gen ADNr 16S se amplificó con los oligonucleótidos (UNAM, México) y bajo las condiciones de reacción reportadas (Weisburg 1991, 697). Los amplicones se purificaron, secuenciaron (Macrogen Inc. Korea) y analizaron bajo el método de máxima verosimilitud, con el modelo Tamura Nei, y 1500 réplicas de bootstrap (Tamura 2013, 2725). Las secuencias del gen ADNr 16S de las cepas identificadas como BAL fueron depositadas en la base de datos del NCBI (National Center of Biotechnological Information) y sus números de registro se reportan entre paréntesis después de cada cepa.
Resultados
Durante el proceso de fermentación del suero, en todos los medios de cultivo el mayor recuento microbiano se observó a las 48 h (Figura 1). El mayor recuento celular se obtuvo en el medio Elliker (Streptococcus, Lactococcus, Leuconostoc, lactobacilos homo y heterofermentativos), seguido en orden descendente en los medios MRS (lactobacilos), APT (bacilos heterofermentativos) y M17 (estreptococos lácticos). Una disminución del crecimiento se observó a las 120 h en los medios restantes, con excepción del medio APT.

Figura 1 Cinética de crecimiento en diferentes medios de cultivo selectivos durante la fermentación del suero de queso. APT (bacilos heterofermentativos), Elliker (Streptococcus, Lactococcus, Leuconostoc, lactobacilos homo y heterofermentativos), MRS (lactobacilos) y M17 (estreptococos lácticos).
La selección de 22 aislados de BAL presuntivas se realizó en función de la morfología colonial específicas para cada grupo microbiano en función del medio de cultivo utilizado. El 100% de los aislados fueron Gram positivo, 86% catalasa negativo y 25% fueron identificados como BAL, de acuerdo al análisis filogenético del marcador ADNr 16S. La cepa VK (KJ175072) se relacionó filogenéticamente con el grupo de Lactobacillus (Figura 2, Cuadro 1), ya que se localizó en una rama aparte.
Cuadro 1. Información general de los alineamientos locales de las secuencias obtenidas de los aislados con la base de datos del GenBank y su número de acceso.
| Aislado | Descripción Best-hit | Cobertura | Identidad | Acceso |
|---|---|---|---|---|
| VK | Lactobacillus sp. VK | 100% | 100% | KJ175072.1 |
| Lactobacillus oris strain J-1 | 100% | 99% | CP014787.1 | |
| VD | Enterococcus sp. VD | 100% | 100% | KJ175073.1 |
| Enterococcus faecium isolate EFE10021 | 100% | 99% | LN999844.1 | |
| VL | Enterococcus sp. VL | 100% | 100% | KJ175074.1 |
| Enterococcus faecium isolate EFE10021 | 100% | 99% | LN999844.1 | |
| VH | Enterococcus sp. VH | 100% | 100% | KJ175075.1 |
| Enterococcus faecium isolate EFE10021 | 100% | 99% | LN999844.1 | |
| VB | Enterococcus sp. VB | 100% | 100% | KJ175076.1 |
| Enterococcus faecium strain TS4E3 | 98% | 99% | KJ571216.1 |
Las cepas VD (KJ175073), VH (KJ175075), VB (KJ175076) y VL (KJ175074) se relacionaron filogenéticamente con Enterococcus(Figura 3, Cuadro 1).
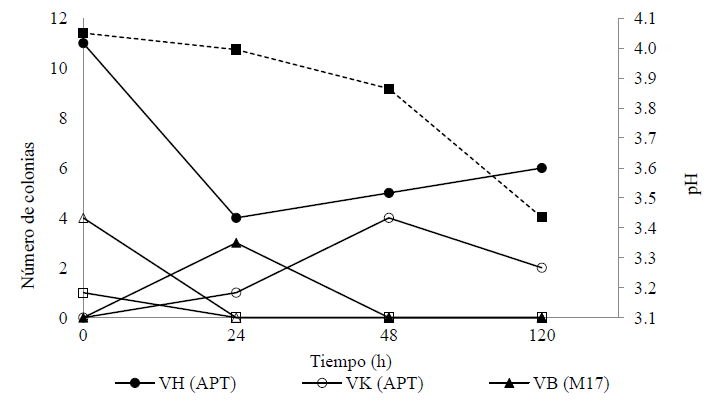
La dinámica poblacional mostró cuatro comportamientos durante la fermentación (Figura 4). Las cepas VD y VL estuvieron presentes desde el inicio de la fermentación y no se detectaron después de las 24 h. La cepa VH presentó un comportamiento similar a VD y VL hasta las 24 h, posteriormente su población se incrementó (6 x 105 UFC mL-1) hasta el final del bioproceso. La cepa VB mostró su máximo crecimiento a las 24 h, posteriormente disminuyó, sin detectarse después de las 48 h. La población de la cepa VK se incrementó desde el inicio de la fermentación, mostrando el máximo nivel a las 48 h (4 x 105 UFC mL-1), disminuyendo en 50 % hacia el final del bioproceso. Finalmente, la población de la cepa VH fue más abundante en relación a las poblaciones de las otras cepas y junto con la cepa VK estuvieron presentes hasta el final de la fermentación. Por otro lado, el pH descendió desde 4.05 hasta 3.4 (cambio de pendiente a las 24 h), debido posiblemente al incremento de la población del aislado VB. Un descenso más pronunciado del pH se observó a partir de las 24 h, éste puede asociarse con metabolitos producidos por los aislados VH y VK. Desde el punto de vista poblacional, el crecimiento de la cepa VB y la acidificación del medio de cultivo en las primeras 24 h de fermentación posiblemente tuvieron un efecto antagónico sobre el crecimiento de las cepas VD, VL y VH pertenecientes al género Enterococcus. Posterior a ese tiempo, un segundo incremento de acidez en el medio (cambio más pronunciado de pH), afectó positivamente la presencia de las cepas VH y VK.
Desde el punto de vista metabólico, las cinco cepas fueron capaces de utilizar glucosa, lactosa y sacarosa. Las cepas VD, VH y VL presentaron el mismo perfil metabólico (Cuadro 1), indicando que pertenecen a la misma especie, lo que coincide con el análisis filogenético (Figura 3). Además, las cepas VD, VH, VK y VL fueron capaces de metabolizar mono, di y trisacáridos (enlace α o β) con radicales y polioles como el glicerol y sorbitol. La cepa VB, a diferencia del resto, fue capaz de hidrolizar esculina, actividad inusual en bacterias de este género (Giraffa 2002, 163).
Cuadro 2. Perfil bioquímico de las cepas aisladas de SQHF.
| Cepa | Sustratos | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Ind | Ure | Glu | Man | Lac | Sac | Mal | Sal | Xyl | Ara | Gel | Esc | Gly | Cel | Mne | Mlz | Raf | Sor | Rha | Tre | |
| VB | - | - | + | - | + | + | - | - | - | - | - | + | - | - | + | - | - | - | - | - |
| VD | - | - | + | + | + | + | + | + | + | + | - | - | + | + | + | + | + | + | + | + |
| VH | - | - | + | + | + | + | + | + | + | + | - | - | + | + | + | + | + | + | + | + |
| VK | - | - | + | + | + | + | + | + | + | + | - | - | + | + | + | + | + | + | + | + |
| VL | - | - | + | + | + | + | + | + | + | + | - | - | + | + | + | + | + | + | + | + |
Discusión o Conclusiones
La presencia de Enterococcus en el SFQHF puede indicar contaminación alimentaria, sin embargo, su presencia se ha reportado en cultivos iniciadores derivados de suero para la producción a pequeña escala de quesos Gruyere, Emmental y Grana (Beresford 2001, 259), también en productos lácteos artesanales (Dolci 2008, 302), en quesos madurados y salchichas fermentadas (Giraffa 2002, 163; Giraffa 2004, 251). E. faecium y E. faecalis microencapsuladas han sido utilizadas recientemente como probióticos en alimentación humana y animal (Weis 2010, 187; Coeuret 2003, 269). Los lactobacilos como L. casei ssp. Casei, L. paracasei ssp. Paracasei, L. rhamnosus, L. plantarum, L. fermentum, L. brevis, L. buchneri, L. curvatus, L. acidophilus y L. pentosus han sido reportados como BAL no iniciadoras (NSLAB por sus siglas en inglés) debido a su baja población al inicio de la fermentación (102 - 103 UFC g-1), como se observó con la cepa VK (Figura 4) (Coeuret 2003, 269).
La disminución del pH en el SFQH, posiblemente se debió a la producción de ácido láctico proveniente del metabolismo de los aislados mencionados (Shiphrah 2013, 118); siendo ésta la causa principal de la modificación de la población de BAL durante el bioproceso como se mostró para L. casei(García 2013, 136). Otras variables que pudieron tener un efecto sobre la dinámica poblacional, son la competencia nutricional o la posible producción de enterocinas por la cepa VH (Vasquez 2009, 64) o bacteriocinas por la VK.
La dinámica poblacional de BAL en el SFQH se evaluó mediante técnicas microbiológicas; además se aislaron e identificaron cinco cepas BAL relacionadas con los géneros Enterococcus (VB, VD, VH y VL) y Lactobacillus (VK) por medio de técnicas moleculares. Las cinco cepas fueron capaces de utilizar glucosa, lactosa y sacarosa como fuente de carbono. Particularmente las cepas VH y VK, debido a su resistencia en cultivos con pH ácido, podrían utilizarse como iniciadores para la elaboración de quesos regionales (fresco y Oaxaca) inocuos que cumplan con las especificaciones de la NOM-243-SSA1-2010; es decir utilizando leche pasteurizada, pero sin comprometer la calidad sensorial de estos productos.











 text new page (beta)
text new page (beta)