Introducción
Las comunidades microbianas del suelo, formadas principalmente por bacterias y hongos son un componente clave de los ecosistemas naturales y agrícolas, debido a que participan de manera activa en numerosos procesos ecológicos fundamentales para el mantenimiento de la vida sobre nuestro planeta (Maestre, 2016). Las demandas de nutrientes para la regeneración de los bosques tropicales se satisfacen en parte con árboles de leguminosas en simbiosis con bacterias fijadoras de nitrógeno, llamadas comúnmente rizobios. Nuestra comprensión de la abundancia de esas especies está sesgada hacia las regiones tropicales húmedas mientras que las comunidades microbianas del suelo en los bosques tropicales estacionalmente secos son poco conocidos (Gei et al., 2018).
Los bosques secos tropicales (BST) son uno de los ecosistemas más amenazados del mundo, por su acelerada pérdida de cobertura vegetal (Espinosa et al., 2012). Tienen una gran diversidad y abundancia de leguminosas con aporte a la fijación biológica de nitrógeno (FBN). Sin embargo, hay pocas estimaciones de cantidades de nitrógeno fijadas, lo que dificulta la comprensión de los factores que controlan la FBN en esos ecosistemas (Ferreira-Da Silva et al., 2017). Las regiones de BST de Tumbes, Piura y Puyango, localizadas en el noroeste de Perú y suroeste de Ecuador, son de las zonas con gran endemismo y riqueza de especies más importantes del mundo (Aguirre, 2012; Espinosa et al., 2012; Linares-Palomino et al., 2010).
En los valles secos de la costa y callejón interandino de Ecuador y Perú, se encuentra la especie Clitoria brachystegia Benth., una leguminosa representante de la familia Fabaceae, reportada como endémica y en peligro de extinción (FAO, 2015). Esta planta está clasificada en el libro rojo de la Unión Internacional para la Conservación de la Naturaleza (UICN), como amenazada bajo criterio A4c (UICN, 2012).
Las leguminosas han impulsado la evolución de los rizobios para adaptarse a la necesidad de nodular su planta hospedera bajo condiciones locales, principalmente de pH y disponibilidad de nutrientes en el suelo (Cao et al., 2014; Saeki et al., 2013; Suzuki et al., 2014). La asociación rizobio-leguminosa se considera un proceso de alta eficiencia en fijación biológica de nitrógeno (FBN) y puede ser capaz de abastecer hasta un 90% de las necesidades de nitrógeno en dichas plantas (López-Alcocer et al., 2017). El grupo de los rizobios está formado por alfaproteobacterias o betaproteobacterias del suelo que forman simbiosis con sus hospederas leguminosas, compitiendo con otros miembros de la microbiota, formando los bacteroides en el interior de los nódulos fijadores de nitrógeno (Poole et al., 2018).
Los rizobios de leguminosas del género Clitoria han sido poco estudiados y hasta la fecha no existen reportes sobre los simbiontes asociados a C. brachystegia. La mayor parte de la información disponible se refiere a las bacterias de Clitoria ternatea (Duangkhet et al., 2018; López-López et al., 2012; de Oliveira-Nunes et al., 2018), y en menor medida a los simbiontes de Clitoria fairchildiana (Coelho et al., 2018; Menna et al., 2006), Clitoria javitensis (Parker, 2008) y Clitoria mariana (Parker y Rousteau, 2014).
Este estudio pretende establecer la diversidad de los rizobios asociados con la leguminosa endémica y amenazada C. brachystegia, en 5 remanentes del BST que comparten Ecuador y Perú; así como el potencial de contribución a la fijación de nitrógeno a un ecosistema amenazado por la actividad antrópica y el cambio climático.
Materiales y métodos
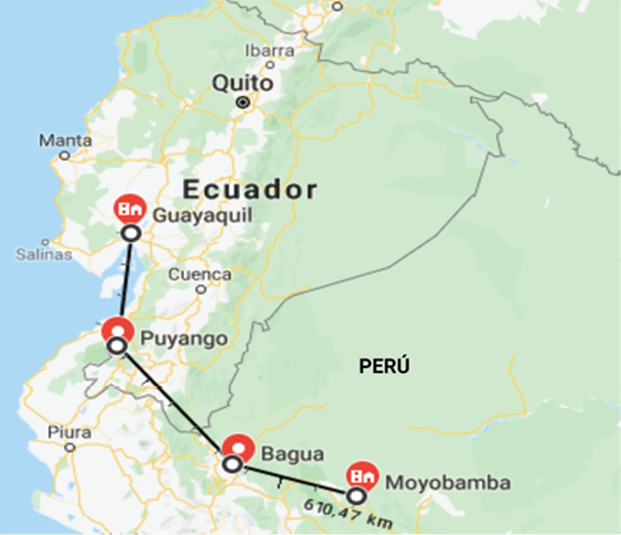
De acuerdo con la información encontrada en los herbarios de la Universidad de Guayaquil en Ecuador; Universidad Nacional Pedro Ruiz Gallo y Universidad Nacional Agraria La Molina (UNALM) en Perú, se ubicaron y monitorearon los sitios de recolecta de la leguminosa endémica C. brachystegia Benth., registrada en sus fichas de identificación. Se recorrieron varios sitios mencionados por las colecciones de los herbarios consultados; sin embargo, solo se logró encontrarla en 5 de 10 localidades de BST reportadas en los herbarios; 3 ubicadas en Ecuador (2 en Guayaquil, provincia del Guayas y 1 en Puyango, provincia de Loja) y 2 en Perú (Moyobamba y Bagua, Departamentos de Lambayeque y Amazonas, respectivamente) (fig. 1). Cabe mencionar que sólo en las localidades de Puyango y Bagua, se encontraron árboles silvestres, en las otras localidades las plantas fueron sembradas varios años atrás. Se recolectaron muestras vegetales (flores y vainas) que fueron identificadas en el Herbario de la Universidad de Guayaquil (Ecuador). Así como también muestras de suelo que fueron analizadas en el Laboratorio del Departamento de Recursos Hídricos de la UNALM.
Los aislamientos, pruebas de identificación y tipificación molecular, nodulación y fijación de nitrógeno de las bacterias se realizaron en el Laboratorio de Ecología Microbiana y Biotecnología (LEMyB) del Departamento de Biología de la UNALM. La tabla 1 muestra las localidades de muestreo, promedios de temperatura y humedad relativa anuales, la ubicación geográfica, altitud, el número de plantas y el número de nódulos efectivos que originaron los aislados de rizobios para este estudio. Los resultados del análisis de suelo se presentan en la tabla 2.
Tabla 1 Ubicación y condiciones ambientales de las localidades donde se obtuvieron muestras de C. brachystegia.
| Localidad, país (código) | Temperatura | Humedad relativa | Ubicación | M snm | Número de plantas muestreadas / número de nódulos obtenidos |
|---|---|---|---|---|---|
| Jardín Botánico de Guayaquil, Ecuador (BG) | 27 °C | 88% | 2°4’47.14” S, 79°54’36.94” O | 60 | 6/4 |
| Universidad de Guayaquil, Ecuador (UG) | 27 °C | 88% | 2°8’55.83” S, 79°54’56.75” O | 7 | 2/10 |
| Puyango*, Ecuador (PY) | 22.5 °C | 79% | 3°52’27.0” S, 80°02’56.3” O | 366 | 3/4 |
| Moyobamba, Perú (MY) | 19 °C | 79% | 6°02’00” S, 76°58’00” O | 858 | 2/14 |
| Bagua*, Perú (PB) | 23 °C | 83% | 5°45’26” S, 78°26’43” O | 576 | 2/14 |
* Localidades donde se encontraron plantas silvestres.
Tabla 2 Análisis físicoquímicos de los suelos obtenidos en las 5 localidades donde se tomaron las muestras de C. brachystegia.
| Características del suelo | Ecuador | Perú | |||
|---|---|---|---|---|---|
| BG | UG | PY | MY | PB | |
| Textura | Franco arenoso | Franco arenoso | Arcilloso | Arena franca | Franco arenoso |
| pH | 6.98 | 6.27 | 4.28 | 6.98 | 7.32 |
| Fósforo (ppm) | 12.59 m | 33.18 a | 11.28 m | 26.22 a | 13.91 ma |
| Potasio (ppm) | 62.4 b | 490 a | 176 m | 84 b | 91.2 b |
| Materia orgánica (%) | 3.62 m | 8.69 a | 0.33 b | 2.15 b | 2.66 m |
| CaCO3 (%) | 0.23 b | 0 | 0 | 0.21 b | 5.46 ma |
a = alto, m = medio; b = bajo, ma = medianamente alto (Depto. Recursos Hídricos UNALM).
Con una pala de jardín se extrajeron las raíces y se recolectaron con mucho cuidado los nódulos, que fueron guardados en microtubos de 1.5 mL con sílica gel y algodón para su conservación hasta llegar al laboratorio, donde se hidrataron en agua destilada por 30 min en sobres de papel filtro. Luego se empleó un proceso de desinfección, que consistió en sumergirlo en alcohol al 70% y luego en solución de lejía al 3% por 3 min, respectivamente (Zúñiga-Dávila, 2012). Cumplido el proceso de desinfección se enjuagaron 5 veces con agua destilada hasta eliminar residuos de cloro. Con pinzas estériles se abrieron los sobres con los nódulos y se colocaron en placas de Petri, agregando una gota de agua destilada estéril a cada uno de los nódulos, luego se procedió a macerar cada nódulo sobre una caja de Petri estéril (Zúñiga-Dávila, 2012). Cada macerado se sembró por estrías paralelas en placas de Petri con medio extracto de levadura manitol agar con rojo Congo (LMA-RC) (Zúñiga-Dávila, 2012). El rojo Congo se añadió a razón de 10 mL L-1 de medio (pH 7). Las placas se incubaron a 28 °C por 24 a 72 h para Rhizobium sp. y de 5 a 7 días para Bradyrhizobium sp.
Para la identificación y caracterización molecular, las bacterias fueron cultivadas en tubos con 3 mL de caldo peptona extracto de levadura (PY) (Noel et al., 1984) o caldo extracto de levadura manitol (LMC) (Zúñiga-Dávila, 2012) a 28 °C y 200 rpm de agitación. Una vez obtenida la concentración bacteriana mínima (1x106 UFC mL-1) se extrajo el ADN de las células siguiendo el protocolo del Gene Jet Genomic DNA Purification kit (Thermo Scientific, USA).
Se generaron perfiles genómicos mediante la técnica rep-PCR (Versalovic et al., 1994), empleando el primer BOX A1R (5’-CTACGGCAAGGCGACGCTGACG-3`). Se consideró un volumen de reacción de 50 µL. La concentración final y volúmenes de reacción de los reactivos fueron: 2 μL primer BOX A1R (50 pmoles), 3.125 μL dNTPs (1.25 mM), 2.5 μL buffer KCl - MgCl2 (1×), 7.5 μL MgCl2 (7.5 mM), 0.4 μL Taq polimerasa (2 U), 0.4 μL DMSO (10%), 5 μL ADN diluido (10 ng µL-1) (50 ng) y 2.24 μL agua ultra pura (Milli-Q). El programa para la amplificación empezó por la desnaturalización inicial a 95 ºC por 3 min; seguido de 25 ciclos de desnaturalización a 93 ºC por 45 seg, alineamiento a 53.4 ºC por un min y extensión a 65 ºC por 8 min; finalizando con una extensión final a 65 ºC por 16 min. Se comprobó la presencia de bandas amplificadas mediante revelado en gel de agarosa al 1% en buffer TBE a 80 V por 180 min. Se utilizó como referencia el marcador de peso molecular Gene Ruler 1kb DNA Ladder Plus (Thermo Scientific, USA). Las imágenes de los geles electroforéticos revelados con los amplicones se alinearon junto al marcador de peso molecular. Se compararon los perfiles generados de cada cepa obtenida, con bandas en común que mostraron el mismo peso molecular. Se consideraron perfiles diferentes, a los que mostraron al menos una banda diferente.
Se amplificaron fragmentos del gen ribosomal 16S casi completos, mediante PCR (Weisburg et al., 1991). La concentración final y volúmenes de reacción de los reactivos fueron: 2 μL de primer fD1y primer rD1 (0.4 µM, c/u), 1 μL dNTPs (200 µM, c/u), 5 μL Taq Buffer KCl (1×), 3 μL MgCl2 (1.5mM), 0.2 μL Taq ADN polimerasa (1U), 5 μL ADN diluido (10 ng µL-1) (50 ng) y 31.8 μL agua ultra pura (Milli-Q). El programa para la amplificación se inició con la desnaturalización inicial a 94 ºC por 3 min; seguido de 30 ciclos de desnaturalización a 94 ºC por 45 seg, alineamiento a 55 ºC por 45 seg y extensión de 72 ºC por 2 min; y finalizando con una extensión final a 72 ºC por 5 min. Esta fue verificada mediante la corrida en gel de electroforesis a 60 V por 150 min. Se utilizó como referencia el marcador de peso molecular Gene Ruler 1kb DNA Ladder Plus (Thermo Scientific, USA).
Una vez amplificados los fragmentos del gen 16S, se enviaron a Macrogen Inc. (Seúl, Corea del Sur), para su secuenciación. Los cromatogramas resultantes fueron editados con el programa Chromas Lite, versión v2.01. Las secuencias fueron comparadas con las de la base de datos GenBank (Benson et al., 2017) y las del servidor EzTaxon (Kim et al., 2012). Las relaciones filogenéticas se infirieron usando el método de máxima verosimilitud escogiendo el mejor modelo de substitución para cada alineamiento de acuerdo al criterio de información Bayesiano según se sugiere en el software MEGA 7.0 (Kumar et al., 2016). El soporte de los nodos se determinó mediante un análisis de bootstrap inferido con 500 réplicas (Felsenstein, 1985). Las secuencias generadas fueron depositadas en la base de datos GenBank recibiendo los números de acceso MT422153 a MT422185. La relación entre la diversidad de los rizobios de C. brachystegia con las condiciones edáficas de cada localidad fue evaluada mediante análisis de correlación de Pearson con la riqueza de especies y con los índices de Shannon-Weaver y Simpson (Magurran, 1988), calculados con el software PAST (Hammer et al., 2001).
Debido a la poca cantidad, adaptación y capacidad de germinación de las semillas de C. brachystegia, verificada en ensayos previos, para el test de nodulación se emplearon variedades de leguminosas que normalmente son noduladas por Rhizobium y Bradyrhizobium (CIAT, 1988), como el frijol Red Kidney (Phaseolus vulgaris L.) y caupí (Vigna unguiculata), respectivamente. Las semillas fueron hidratadas en vasos de precipitado con 400 mL de agua destilada estéril por 2 h. Luego se desinfectaron con alcohol al 70% e hipoclorito de sodio al 3% por 3 min en ambos casos (Zúñiga-Dávila, 2012), luego se enjuagaron con agua destilada estéril hasta 5 veces hasta llegar a nivel cloro 0 (cero), comprobado con kit de cloro Hanna Instruments®. Las semillas de cada variedad se colocaron en cámara de germinación a 24 °C por 72 h en oscuridad, luego 2 semillas germinadas fueron transferidas a macetas esterilizadas de 1 kg con sustrato arena-vermiculita fina (1:1 v/v) esterilizada, a los 15 días se dejó solo la planta que presentaba mayor vigor. Las cepas de rizobios fueron reactivadas en medio LMA-RC y luego de 72 h se inoculó una asada en tubos con 4 mL de caldo LMC, los cuales fueron incubados a 28 °C por 2 a 5 días hasta obtener una población de 108 UFC mL-1. La inoculación de las plántulas se realizó en 2 tiempos, al momento de sembrar en maceta y a los 7 días posteriores a la siembra. En cada tiempo, se colocó 1 mL del inóculo bacteriano respectivo. Las plantas se mantuvieron en condiciones de vivero en el LEMyB. El ensayo incluyó 24 tratamientos: 9 cepas de Rhizobium (1J, 2J, 14J, 27J, 30J, 37J, 83J, 88J, 151J) y 9 cepas de Bradyrhizobium (99J, 102J, 124J, 125J, 134J, 136J, 139J, 142J, 143J) elegidas al azar, R. tropici CIAT 899 (cepa Fix+ para frijol), Bradyrhizobium yuanmingense LMTR 28 (cepa Fix+ para caupí), 2 controles sin nitrógeno y sin inocular (para frijol y caupí), y 2 controles con nitrógeno y sin inocular (para frijol y caupí). Cada tratamiento tuvo 5 réplicas. Las plántulas se regaron con solución nutritiva estéril de Broghton y Dillworth diluida 1:4 sin nitrógeno, cada 3 o 5 días, acorde a requerimiento (Somasegaran y Hoben, 1985). Los controles con nitrógeno se regaron con la misma solución adicionada de 70 ppm de N en forma de nitrato de amonio (Somasegaran y Hoben, 1985).
La fijación biológica de nitrógeno se evaluó mediante la prueba de reducción de acetileno descrito por Hardy et al. (1968) según CIAT (1988). Las evaluaciones se realizaron entre 25 a 30 días después del trasplante de las semillas germinadas a macetas. Se tomó una muestra de nódulos de 1 gramo de peso de cada repetición de los tratamientos inoculados o 1 g de raíces de los tratamientos no inoculados y se introdujeron en frascos con tapa de goma apropiados de 28 mL que fueron sellados adicionalmente con parafilm. Se sustituyó 10% del volumen de la atmósfera de cada frasco con acetileno y se incubó durante 1 hora a 30 °C. Con el fin de medir la concentración de etileno generada, 1 mL de la atmósfera de cada frasco fue inyectado en el cromatógrafo de gases (Agilent Technologies 7890A, USA). El cromatógrafo estuvo acoplado a un detector de ionización de llama y una columna HP-PLOT/Q (30; 0.530; 40), detector FID-GC. Las condiciones del cromatógrafo para realizar este experimento fueron temperatura del horno a 60 °C, temperatura FID a 250 °C y temperatura del puerto inyector a 250 °C. Se realizaron 2 mediciones por cada tratamiento y los resultados se expresaron como μmol etileno h-1 g-1.
Resultados
La mayoría de los nódulos de C. brachystegia (43 de 46) presentaron coloración rosada interna, que indica presencia de leghemoglobina y fijación de N (Ott et al., 2005), y tuvieron tamaño promedio de 3 mm aproximadamente. Los nódulos macerados y luego estriados generaron alrededor de 55 colonias con crecimiento entre las 48 a 96 horas. De las cuales, se escogieron 46 colonias que presentaron consistencia mucosas, correosas o acuosas con coloración rosada a blanquecina en el medio LMA-RC.
Mediante la caracterización molecular por BOX-PCR se identificaron 33 perfiles genómicos entre los 46 aislados obtenidos de los nódulos de la planta hospedadora C. brachystegia (tabla 3). La mayoría de aislados que presentaron el mismo perfil se recuperaron de la misma localidad, sin embargo; se observaron algunos casos de aislados con idéntico perfil que fueron hallados en localidades diferentes pero cercanas (perfiles D y α en la tabla 3). Estos resultados indican una limitada capacidad de dispersión de los rizobios entre diferentes localidades.
Tabla 3 Perfiles BOX-PCR y origen de cepas identificadas por la secuenciación del gen 16S rRNA de C. brachystegia. *La cepa en negritas se usó para la secuenciación.
| Código de cepas* | Total aislados | Perfil genómico (BOX-PCR) | Primera coincidencia en EzTaxon | Identidad (%) | Origen cepa |
|---|---|---|---|---|---|
| 1J | 1 | A | R. multihospitium | 99.52 | UG |
| 26J-27J-30J | 3 | C | R. miluonense | 99.24 | BG |
| 2J-29J-35J | 3 | D | R. radiobacter | 99.62 | UG (2J,29J), BG (35J) |
| 54J | 1 | G | R. radiobacter | 99.62 | PY |
| 17J | 1 | J | R. miluonense | 99.24 | UG |
| 21J | 1 | M | R. viscosum | 99.81 | UG |
| 83J-84J | 2 | K | R. larrymoorei | 99.9 | PY |
| 14J | 1 | L | R. miluonense | 99.24 | UG |
| 37J-38J-48J-49J | 4 | S | R. multihospitium | 99.52 | UG |
| 88J | 1 | l | R. radiobacter | 99.62 | PY |
| 90J | 1 | m | B. tropiciagri | 100 | MY |
| 91J | 1 | n | B. liaoningense | 100 | MY |
| 96J | 1 | o | B. tropiciagri | 100 | MY |
| 97J | 1 | p | B. tropiciagri | 100 | MY |
| 98J | 1 | q | B. tropiciagri | 100 | MY |
| 99J-102J-108J | 3 | r | B. japonicum | 99.8 | PB |
| 106J | 1 | t | B. liaoningense | 100 | PB |
| 117J | 1 | w | B. liaoningense | 100 | PB |
| 118J | 1 | x | B. japonicum | 99.8 | PB |
| 119J | 1 | y | R. multihospitium | 98.44 | PB |
| 124J-125J | 2 | α | B. daqingense | 100 | PB (124J), MY (125J) |
| 127J | 1 | β | B. liaoningense | 100 | MY |
| 132J | 1 | γ | B. tropiciagri | 100 | MY |
| 134J-139J-144J | 3 | δ | B. tropiciagri | 100 | MY |
| 136J | 1 | ε | B. tropiciagri | 100 | MY |
| 146J | 1 | ζ | B. japonicum | 99.8 | PB |
| 142J | 1 | η | B. liaoningense | 100 | MY |
| 143J | 1 | θ | B. tropiciagri | 100 | MY |
| 148J | 1 | ι | R. endophyticum | 98.44 | PB |
| 150J | 1 | κ | R. endophyticum | 98.44 | PB |
| 151J | 1 | λ | R. endophyticum | 98.44 | PB |
| 153J | 1 | µ | R. endophyticum | 98.44 | MY |
| 159J | 1 | ρ | R. endophyticum | 98.34 | PB |
Los perfiles genéticos BOX-PCR se agruparon de acuerdo a su similitud, escogiendo una cepa representante por cada distribución de bandas a las cuales se les secuenció el gen ribosomal 16S. Las secuencias editadas, se analizaron en la base de datos EzTaxon para determinar las cepas tipo más relacionadas (Kim et al., 2012). Se encontraron 24 cepas de Rhizobium, género que se recuperó en las 5 localidades muestreadas (tablas 3, 4). Mientras que el género Bradyrhizobium aportó 22 cepas, encontradas solamente en las localidades de Perú.
Tabla 4 Abundancia de cada grupo filogenético de rizobios asociados con C. brachystegia y diversidad en las 5 localidades del bosque seco tropical de Ecuador y Perú.
| Ecuador | Perú | ||||
|---|---|---|---|---|---|
| BG | UG | PY | MY | PB | |
| Grupo | Número de aislados | ||||
| R1 | 3 | 2 | |||
| R6 | 1 | 2 | 2 | ||
| R5 | 2 | ||||
| R4 | 1 | ||||
| R3 | 1 | 5 | |||
| R2 | 3 | 2 | |||
| B4 | 10 | ||||
| B1 | 3 | 2 | |||
| B3 | 5 | ||||
| B2 | 1 | 1 | |||
| Índice de diversidad | Valor | ||||
| Riqueza de especies | 2 | 4 | 2 | 4 | 4 |
| Shannon-Weaver | 0.56 | 1.22 | 0.69 | 0.95 | 1.22 |
| Simpson | 0.5 | 0.27 | 0.33 | 0.46 | 0.27 |
Se evaluaron las relaciones filogenéticas de las cepas secuenciadas construyendo filogramas en base a las secuencias del gen ribosomal 16S. Las cepas de Rhizobium se distribuyeron en 6 grupos marcados como R1 a R6 en la figura 2. Los 2 primeros estuvieron relacionados con especies del grupo tropici (Ribeiro et al., 2012). El grupo 1 con Rhizobium multihospitium y Rhizobium tropici pero presentó la mayor identidad a nivel de secuencia con la primera especie (99.52%), mientras que el grupo 2 se posicionó en forma independiente siendo Rhizobium miluonense la especie con la que mostró la mayor identidad (99.24%). El tercer grupo estuvo formado por 5 cepas, se ubicó en forma independiente en la filogenia y mostró la mayor identidad con Rhizobium endophyticum (98.44%). La cepas 21J y 84J se agruparon con Rhizobium viscosum y Rhizobium larrymoorei compartiendo identidades de 99.81% y 99.9%, respectivamente, con dichas especies.

Figura 2 Filograma obtenido por el método de máxima verosimilitud en base a secuencias del gen ribosomal 16S de cepas de Rhizobium. Los números de las ramas representan valores de bootstrap. Las cepas obtenidas de C. brachystegia se muestran en negrita y los grupos en los que se distribuyeron se indican con letras R. Los números de acceso de las secuencias en la base de datos GenBank se muestran entre paréntesis.
El último grupo, formado por 3 cepas, se relacionó con la especie Rhizobium radiobacter con un nivel de identidad de 99.62%. La mayoría de aislados de Rhizobium (18 de 24) se obtuvieron de Ecuador, la localidad donde se encontró mayor cantidad de Rhizobium fue Universidad de Guayaquil (UG) con 41.7% del total de aislados de ese género. Las únicas cepas de Rhizobium encontradas en Perú fueron las del grupo 3 en Moyobamba y Bagua.
Las cepas de Bradyrhizobium se distribuyeron en 4 grupos señalados como B1 a B4 en la figura. 3. Las cepas de los 3 primeros grupos se ubicaron dentro del supergrupo japonicum que incluye a Bradyrhizobium japonicum (Beukes et al., 2016) mientras que las del cuarto grupo se aliaron con el supergrupo elkanii que contiene a las relacionadas con Bradyrhizobium elkanii (Beukes et al., 2016). Los grupos B1, B2 y B3 se ubicaron en un clado medianamente soportado y poco resuelto donde las secuencias del grupo B1 fueron idénticas a las de Bradyrhizobium liaoningense, la del grupo B2 idéntica a la de Bradyrhizobium daqingense y las del grupo B3 compartieron una identidad de 99.8% con B. japonicum. Las cepas del grupo B4 estuvieron igual de relacionadas a varias especies incluyendo B. tropiciagri (fig. 3). Este último grupo contribuyó con 10 de las 22 cepas de bradyrizobios (45.5%), y todas fueron encontradas en Moyobamba (Perú).
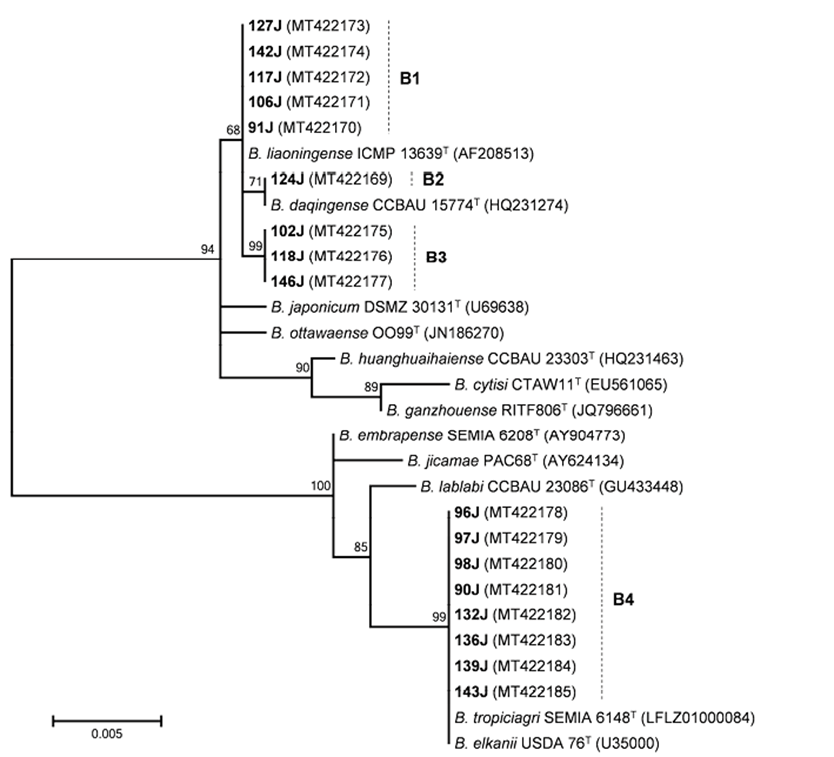
Figura 3 Filograma obtenido por el método de máxima verosimilitud en base a secuencias del gen ribosomal 16S de cepas de Bradyrhizobium. Los números de las ramas representan valores de bootstrap. Las cepas obtenidas de C. brachystegia se muestran en negrita y los grupos en los que se distribuyeron se indican con letras B. Los números de acceso de las secuencias en la base de datos GenBank se muestran entre paréntesis.
Las localidades que presentaron los mayores valores de diversidad según el índice de Shannon-Weaver y de menor dominancia según el índice de Simpson fueron Universidad de Guayaquil (UG, en Ecuador) y Bagua (PB, en Perú) (tabla 4). En ambas localidades se encontraron rizobios relacionados a 4 grupos diferentes. La localidad UG presentó textura del suelo franco arenosa, pH ligeramente ácido, valores considerados altos de fósforo (P), potasio (K) y materia orgánica (MO) y ausencia de carbonato (CaCO3) (tabla 2). Para el suelo franco arenoso de la localidad PB se registraron valor neutro en pH, alto a mediano en P, y bajos a medianos en K, MO y CaCO3 (tabla 2).
Las localidades de Jardín Botánico (BG) y Puyango (PY) evidenciaron los valores más bajos de diversidad en el índice de Shannon-Weaver (tabla 4). Así mismo el valor del índice de Simpson de 0.5 para BG puede interpretarse como el más dominante donde un solo grupo representó 75% de los aislados. La baja diversidad relativa se evidencia también considerando los pocos grupos de rizobios encontrados en esos sitios (tabla 4). La localidad PY presentó suelo arcilloso, con pH extremadamente ácido, valores bajos de MO, medio en P y K, y ausencia de CaCO3; mientras que el suelo de la localidad BG fue franco arenoso, de pH neutro, medio en MO y P, y bajo en K y CaCO3.
Se encontraron correlaciones entre algunos parámetros del suelo y los valores de diversidad. Así el índice de Shannon-Weaver presentó correlaciones positivas (> 0.51) con el fósforo, MO y carbonato, mientras que el potasio mostró correlación negativa (-0.6) con el valor de dominancia de Simpson. Así mismo, la riqueza de grupos mostró correlación positiva con el fósforo (0.7) y el pH (0.54). Sin embargo, estas correlacionen no fueron significativas (p > 0.05). Los valores climáticos de temperatura, humedad relativa y altitud de las 5 localidades muestreadas no presentan relación alguna con los índices de diversidad considerados en este trabajo.
En la determinación del potencial de fijación de N mediante el test de reducción de acetileno, se obtuvieron valores que variaron de 0.5 a 54.27 μmol etileno h-1 g-1 con las cepas de rizobios probadas. En el caso de las 9 cepas de Rhizobium, la media fue de 11.69 μmol etileno h-1 g-1 (s = 11.3; rango = 0.5 a 33.82). Los valores más altos de ARA con las cepas de Rhizobium fueron de 33.82 μmol etileno h-1 g-1 con la cepa 30J relacionada a R. miluonense, y de 21.29 y 21.2 μmol etileno h-1 g-1 correspondientes a las cepas 1J y 30J, respectivamente, ambas aliadas a R. multihospitium. En comparación, el valor para la cepa de Rhizobium utilizada como control Fix+ (CIAT 899) fue de 22.2 μmol etileno h-1 g-1. En el caso de las 9 cepas de Bradyrhizobium se observó una media de 16.38 μmol etileno h-1 g-1 (s = 16.38; rango = 4.86-54.27). Para Bradyrhizobium las cepas 125J (aliada a B. daqingense), 134J (aliada a B. tropiciagri) y102J (relacionada a B. japonicum) obtuvieron los valores más altos siendo 54.27, 38.69 y 34.7 μmol etileno h-1 g-1, respectivamente. La cepa control Fix+ de Bradyrhizobium (LMTR 28) mostró un valor de 15.55 μmol etileno h-1 g-1.
Discusión
Los 46 aislados presentaron el tiempo de crecimiento, y colonias con consistencia y coloración típica de cepas descritas de rizobios (CIAT, 1988; Ferrera et al., 1993; Somasegaran y Hoben, 1985; Zúñiga-Dávila, 2012). Los análisis moleculares de genotipado mediante BOX-PCR y la comparación del gen ribosomal 16S, complementaron la identificación morfológica de los aislados de rizobios de la planta hospedadora C. brachystegia obtenidos bajo condiciones locales y disponibilidad de nutrientes únicos de cada localidad (Cao et al., 2014). Los aislados caracterizados correspondieron a los géneros Rhizobium y Bradyrhizobium (α-Proteobacterias), los cuales son reportados como fijadores de nitrógeno y colonizadores típicos de los nódulos de las leguminosas (Allen y Allen, 1981; Ferrera et al., 1993; Lloret y Martínez-Romero, 2005; Poole et al., 2018; Somasegaran y Hoben, 1985).
Se encontraron números similares de aislados de Rhizobium y Bradyrhizobium, sin embargo; la distribución geográfica de estos géneros fue desigual encontrándose la mayoría de Rhizobium (75%) en las localidades de Ecuador mientras que todos los Bradyrhizobium se aislaron de las localidades de Perú. Esto podría explicarse considerando que la diversidad y composición de las poblaciones de leguminosas difieren de sus rizobios asociados, en relación directa con las condiciones locales (Li et al., 2012; Ribeiro et al., 2013; Wu et al., 2011; Yang et al., 2013).
En trabajos similares -en Brasil, México y Tailandia- como los de Menna et al. (2006), López-López et al. (2012), de Oliveira et al. (2018) y Duangkhet et al. (2018), respectivamente, también se han caracterizado cepas de rizobios de Clitoria pero de la especie C. ternatea. La gran mayoría de aislados obtenidos de dicha planta pertenecen al género Bradyrhizobium, solamente en los 3 primeros estudios mencionados se reportó una cepa de Rhizobium. En forma similar, Coelho et al. (2018) aisló solamente cepas de Bradyrhizobium al estudiar nódulos de C. fairchildiana en Brasil. Así mismo, Parker (2008) y Parker y Rousteau (2014) reportaron solamente Bradyrhizobium a partir de nódulos de C. javitensis y C. mariana en Panamá y Guadalupe, respectivamente. En conjunto estos reportes indican que los simbiontes preferidos de Clitoria spp. son del género Bradyrhizobium, por lo que sorprende haber encontrado un número relativamente grande de Rhizobium en nuestro estudio lo que podría estar en relación a las características ambientales de las zonas de muestreo (Alexander, 2010).
Las características de suelo pueden influir en las poblaciones microbianas, por ejemplo Pahuara-Hernández y Zúñiga-Dávila (2001) mencionan que la disponibilidad del fósforo tuvo efectos positivos en la riqueza de varios grupos bacterianos incluyendo rizobios. Las concentraciones de fósforo y potasio también influyeron en la composición de una comunidad de rizobios según Li et al. (2012). Por otro lado, Yang et al. (2013) encontraron que el pH puede influir en la diversidad de rizobios aumentando ésta con mayores valores de pH. En este estudio, observamos una tendencia a mayor riqueza de especies en suelos con mayor contenido de fósforo y mayor pH, y también mayores índices de diversidad en relación a suelos ricos en fósforo, potasio y materia orgánica; sin embargo, estas relaciones no fueron significativas quizás en relación a un tamaño de muestra pequeño. Aun así, es de resaltar que la localidad con los menores índices de diversidad de rizobios fue la que tuvo un suelo extremadamente ácido lo que concuerda con lo expresado por Fierer y Jackson (2006) quienes indican que el pH es un factor determinante en la diversidad bacteriana del suelo.
La determinación de la reducción de acetileno de las 18 cepas escogidas, permitieron establecer diferencias entre Bradyrhizobium respecto Rhizobium, donde las cepas de este último género presentaron menor potencial de fijación de N. Los valores determinados para las cepas de C. brachystegia en caupí fueron similares a los declarados por Sarr et al. (2009) en la misma planta (0.8-35.9 µmol etileno h-1 g-1). Duangkhet et al. (2018), reportaron valores de 15 a 35 etileno µmol h-1 g-1 con cepas de Bradyrhizobium inoculadas en su planta hospedera C. ternatea, valores que son similares a los obtenidos en este estudio.
En conclusión, reportamos por primera vez la diversidad de rizobios asociados a la planta leguminosa C. brachystegia endémica del ecosistema BST de Ecuador y Perú. A diferencia de reportes sobre simbiontes de otras especies de Clitoria, encontramos que rizobios de los géneros Rhizobium y Bradyrhizobium son igualmente abundantes en los nódulos de C. brachystegia. Entre los rizobios analizados, encontramos cepas con buen potencial de fijación de nitrógeno. Estas cepas podrían usarse como biofertilizantes agrícolas para frijol o caupí, pero también, y de mayor relevancia, como inoculantes para plantas de C. brachystegia que sean usadas en esfuerzos de conservación o restauración de esta leguminosa en peligro de extinción, como se ha hecho con otras leguminosas amenazadas (Navarro et al., 2014).











 nova página do texto(beta)
nova página do texto(beta)